基于线粒体12S rRNA基因序列鉴别牛肉的种源
王兰萍,耿荣庆,王 伟,邓乔文,徐永新,陈 钥
(盐城师范学院 生命科学与技术学院,江苏 盐城 224051)
牛肉是广受欢迎的肉类食品,也是我国人民消费的重要肉类食品之一。由于牛肉含有丰富的蛋白质,脂肪含量低,氨基酸组成比猪肉更接近人体需要,且能提高机体抗病能力,许多消费者逐渐倾向于选择牛肉,牛肉在肉类消费中所占的比重逐渐增加[1]。近年来,受牛肉消费需求急剧上升、饲养成本提高等多种因素影响,牛肉价格持续上涨[2-3]。牛肉消费还受到消费习惯、地域条件等方面的影响,导致来源于不同牛种的牛肉及肉制品价格差异明显[4-5]。受经济利益的驱使,市场上经常出现用价格相对较低的黄牛肉假冒价格较高的水牛肉和牦牛肉及其肉制品的现象。
为了保护消费者的利益和监督牛肉产业链,寻求快捷、准确的方法对牛肉制品中牛源性成份进行鉴别十分必要。本研究拟运用PCR扩增,结合DNA测序技术,建立鉴别不同牛种来源牛肉的有效分子生物学方法。
1 材料与方法
1.1 牛肉样本来源及处理
分别购买市售的生鲜黄牛肉、牦牛肉和水牛肉各20份。每份样品分两部分,一部分直接保存于-20 ℃,另一部分在沸水中将牛肉煮熟后保存于-20 ℃。
1.2 牛肉样本总DNA提取
将冷冻的生牛肉和熟牛肉取出,每个样本约取30 mg,采用离心柱式组织基因组DNA小量抽提试剂盒(北京天根生化科技有限公司)提取总DNA。提取的总DNA样品溶于100 μL洗脱液TE中,于-20 ℃保存备用。
1.3 引物设计、PCR扩增与检测
根据Kocher等[6]设计的通用引物,进行优化设计后用于扩增12SrRNA基因片段,一对上、下游引物序列分别为5′-AAACTGGGATTAGATACCCCACTAT-3′和5′-GAGGGTGACGGGCGGTGTGT-3′。
PCR扩增体系为25 μL,其中10× buffer 2.5 μL,25 mmol/L MgCl22 μL, 10 mmol/L dNTP 2 μL,10 pmol/L引物各1 μL,5 U/μL Taq酶0.3 μL,DNA模板100 ng。PCR扩增条件为:94℃ 3 min;94℃ 1 min,60 ℃ 1 min, 72 ℃ 1 min,共35个循环;72℃延伸10min。PCR扩增产物在1%的琼脂糖凝胶电泳上检测。
1.4 PCR扩增产物的测序及数据分析
PCR扩增产物采用凝胶回收纯化试剂盒(北京天根生化科技有限公司)纯化后直接送南京金斯瑞生物科技有限公司进行正反双向测序,结合观察测序峰图人工核对测序结果。
引用GenBank数据库中已有的普通牛(GenBank序列号JN817322)、瘤牛(GenBank序列号DQ867006、AF492350)、牦牛(GenBank序列号AY684273、AF091686、EF494179、GQ464260、JQ846022)和水牛(GenBank序列号GU119956、HM623876、AF547270)的12SrRNA基因序列作为参照。采用Clustalx1.83[7]软件对所有测序获得的12SrRNA基因序列和引物序列进行多序列比对。在序列比对的基础上,基于Kimura双参数模型,运用MEGA5.1[8]软件以邻接法(Neighbor joining,NJ)构建基因进化树,进化树各分支的置信度都采用1000次自展分析进行重复检验。
2 结果与分析
2.1 12S rRNA基因片段的PCR扩增结果
一对引物在所有生牛肉DNA样品和熟牛肉DNA样品中均能扩增出一条明亮的特异性产物条带(图1),而且所扩增的12S rDNA片段大小与预期相符,约为440 bp。该结果不仅验证了所提取DNA模板质量的有效性,也表明引物具有通用性,且无非特异性扩增,可用于后续测序分析。
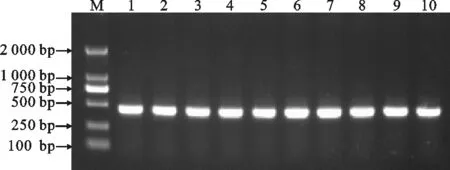
图1 12S rDNA基因扩增片段的电泳图Fig. 1 Electrophoresis picture of amplification fragment of 12S rDNA gene
2.2 基于12S rDNA基因序列特征鉴别牛肉样品的种源
分别将生黄牛肉、牦牛肉、水牛肉及相应熟牛肉样品的PCR扩增产物纯化后测序,每个生牛肉与对应的熟牛肉样本的12S rDNA基因测序结果均相同。根据测序结果可知,扩增的牦牛、水牛12S rDNA基因片段大小都为440 bp,而黄牛12S rDNA基因片段大小都为439 bp;在黄牛、牦牛、水牛的样本中,分别定义了1种(HN)、3种(MN-1、MN-2、MN-3)和2种(SN-1、SN-2)单倍型序列(图2)。多序列比对结果发现,黄牛、牦牛、水牛12S rDNA基因不仅在片段长度上存在一定差异,而且在碱基序列上存在更为显著的差异,显示出明显的种间特异性。牛种间12S rDNA基因序列差异百分比显示,牦牛与水牛之间最大(约7.5%),黄牛与水牛之间居中(约6.8%),黄牛与牦牛之间最小(约3.2%)。

图2 牛种间12S rDNA基因的多序列比对Fig. 2 Mutiple sequence alignment of 12S rDNA gene among bovine species
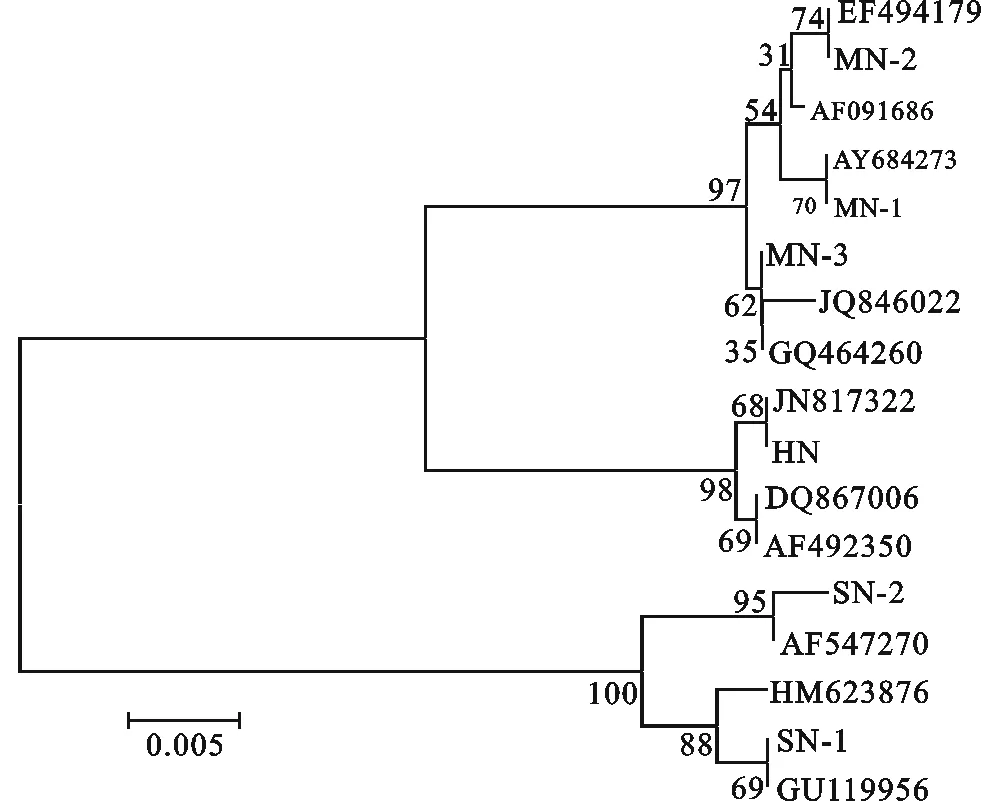
图3 基于NJ 法构建的12S rDNA基因进化树Fig. 3 Gene evolutionary tree of the 12S rDNA gene constructed based on the NJ method
基于试验获得的3个牛种12S rDNA基因序列和引用的牛12S rDNA基因序列,构建的系统进化关系如图3所示。系统进化树显示,所有的黄牛、牦牛、水牛12S rDNA基因序列各自独自聚为一类,本试验测序获得的黄牛、牦牛、水牛12S rDNA基因序列分别与引用的黄牛、牦牛、水牛12S rDNA基因序列聚类。由此可见,参照已知牛种12S rDNA基因序列,与测序获得的牛种序列共同构建系统进化树,能够清晰地鉴别测序样品的牛种来源。
3 讨 论
鉴别肉类的种源特征在打击掺假、保护消费者权益等方面具有十分重要的意义。用于肉类物种的鉴定主要有显微观察法、蛋白质分析法和DNA检测法等[9-10]。以DNA为基础的检测是一类简便、准确、灵敏的方法,在实际应用中最为广泛,可分为核酸探针杂交、PCR-RFLP分析、DNA指纹分析、PCR特异扩增和荧光定量PCR等技术[11-12]。
线粒体DNA既具有一定的种间变异性,又具有高度的种内保守性,是物种鉴定的理想遗传标记。线粒体12SrRNA基因还同时具有抗腐蚀和耐高温的特点,已经成为肉类鉴别中的重要标记基因之一[13-15]。运用12SrRNA基因作为标记基因,基于PCR扩增方法进行肉类种源鉴别的常用方法主要有PCR-RFLP、PCR特异性片段扩增、实时荧光定量以及PCR产物直接测序法。PCR产物直接测序法可以使用通用引物扩增,不仅能够获得丰富的基因序列信息,而且无需其它复杂的后续分析,甚至能够鉴定出亲缘关系非常近的物种。本研究中,通过使用哺乳动物通用引物,首先分别扩增黄牛、牦牛和水牛的12SrRNA基因片段,然后引用已知物种相应基因的序列作为参照,将直接测序获得的各牛种基因序列进行聚类分析,就能够很好地鉴定不同物种来源的牛肉,鉴定结果的准确率为100%。因此,结合运用PCR扩增和DNA测序技术是一种精确可靠的方法,能够有效地运用于牛肉的种源鉴别。
参考文献:
[1] 佚 名. 欧洲牛肉消费观念对我国肉牛业的启示[J]. 农村使用技术,2009(5):21-22.
[2] 曹建民,田 露,张越杰. 我国牛肉消费及其对猪肉价格变化的反应研究[J]. 中国畜牧杂志,2012,48 (12):12-15.
[3] 潘 力,田 露,曹建民,等. 世界牛肉消费趋势及影响因素分析[J]. 中国畜牧杂志,2012,48 (16):20-23.
[4] 司智陟. 我国牛肉消费现状与预测[J]. 中国食物与营养,2012,18(9):54-57.
[5] 吕 品. 我国城乡居民牛肉消费特征及影响因素分析[J]. 中国食物与营养,2012,18(9):45-49.
[6] Kocher T D, Thomas W K, Meyer A,et al. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers[J]. Pro Natl Acad Sci U S A,1989, 86(16):6 196-6 200.
[7] Thompson J D, Gibson T J, Plewniak F,et al. The CLUSTAL-X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4 876-4 882.
[8] Tamura K, Peterson D, Peterson N,et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods[J]. Mol Biol and Evol,2011, 28:2 731-2 739.
[9] Lees M, Popping B.Food Authenticity and Traceability[M]. Cambridge England: Woodhead Publishing Ltd,2003.
[10] Mahajan M V, Gadekar Y P, Dighe V D,et al. Molecular detection of meat animal species targeting MT 12SrRNAgene[J]. Meat Sci,2011, 88(1): 23-27.
[11] Mafra I, Ferreira I M P L V O, Oliveira M B P P. Food authentication by PCR-based methods[J]. Eur Food Res and Technol,2008,227(3):649-665.
[12] Violeta Fajardo, Isabel González, María Rojas,et al. A review of current PCR-based methodologies for the authentication of meats from game animal species[J]. Trends Food Sci & Technol,2010, 21(8):408-421.
[13] Girish P S, Anjaneyulu A S R, Viswas K N,et al. Sequence analysis of mitochondrial 12SrRNAgene can identify meat species[J]. Meat Sci, 2004, 66(3): 551-556.
[14] Zhang J B, Hui H, Ze P C,et al. Species identification in salted products of red snappers by semi-nested PCR-RFLP based on the mitochondria 12SrRNAgene sequence[J]. Food Contr, 2006, 17(7): 557-563.
[15] Wang Q, Zhang X, Zhang H Y,et al. Identification of 12 animal species meat by T-RFLP on the 12SrRNAgene[J]. Meat Science, 2010, 85: 265-269.

