基于网络药理学的个体化治疗与药物组合设计
熊江辉,聂舒媛,李莹辉
(1.中国航天员科研训练中心航天医学基础与应用国家重点实验室,北京 100094;2.武汉大学药学院,湖北武汉 430072;3.香港中文大学深圳研究院,广东深圳 518057)
药物发现的中心任务是确定人体基因/分子组成与疾病表型之间的依赖关系。而疾病的表型可由个体的基因决定,干扰这些基因的表达可能导致表型的改变,如从疾病到正常的转变。复杂疾病一般依赖于多种基因而非单一基因的改变。因此,研究与同一表型相关的多个基因之间的相互作用十分重要。网络药理学通过解析分子网络中多种组分之间的相互关系来研究疾病条件下的特征性网络改变,并研究药物作用下该网络受到扰动的模式,从而实现网络层面上的药物作用机制研究[1]。
协同致死(synthetic lethality)是肿瘤药物发现及网络药理学研究中的一个重要概念。协同致死是指两个基因间一种特定的相互关系:两基因中任一基因发生突变,细胞仍可存活,但若同时突变便会使细胞死亡[2]。利用协同致死原理有利于发现更为有效的癌症治疗方案。例如,ADP-核糖聚合酶〔poly(ADP-ribose)phosphatase,PARP〕与乳腺癌Ⅰ型敏感性蛋白(breast cancer typeⅠ susceptibility protein,BRCA)是某癌症中协同致死的两个基因,通过抑制PARP可开发出针对BRCA突变型肿瘤的靶向药物[3]。一项最近的综述表明,基于协同致死原理的处于临床开发阶段的药物已超过21种,而基于PARP和BRCA协同致死原理进行的临床试验已超过63个[4]。通过基因测试检测BRCA突变情况,据此选择是否采用PARP抑制剂给药治疗是一种典型的个体化治疗策略[5]。多项实验证明,药物组合联用可明显提高单一肿瘤药物的敏感性[6-8]。将此方法扩展到全基因组网络层面上,即通过筛选基因之间的协同致死关系,建立协同致死的基因网络,识别多个节点的扰动,可能成为一种药物组合理性设计的方法。
1 基于基因表达模式的药物组合理性设计方法
Lamb等[9]设计的 CMAP项目(Connectivity Map)是最早利用基因表达谱来建立小分子、基因和疾病之间相互关系的成功案例。通过CMAP可指导设计药物组合及个体化治疗。以地塞米松为例,地塞米松为治疗急性淋巴细胞白血病药物,作者首先通过基因芯片实验,得到地塞米松敏感细胞系和耐受性细胞系之间的差异基因表达谱,然后通过查询各种小分子化合物处理时细胞基因表达谱的变化,成功地识别出可逆转地塞米松耐药性(提高地塞米松敏感性)的药物西罗莫司。
该方法的基本过程(图1)如下:
步骤1:首先选择对某疾病有治疗效果的药物1,通过敏感细胞系和耐受细胞系的比较,或者药敏患者与耐受患者的比较研究,得到药物敏感性相关的基因表达谱。如图1所示,图中基因1为与药物1敏感性呈正相关的基因(图1A),基因2为负相关基因(图1B)。
步骤2:CMAP数据库中包含有多种小分子化合物处理引起的细胞基因表达谱变化模式。因此,在数据库中寻找筛选与药物1敏感性正相关的药物2。如图1C所示,若药物2能上调基因1,并同时下调基因2,则药物2即为提高药物1药敏性的候选药。
作者同时给出证据证实,药物1和药物2联用比单独药物1处理条件下对肿瘤细胞的疗效更显著(图1D)。
值得注意的是,该方法使用全基因组表达谱寻找最佳药物组合,而不是显示地使用基因网络进行药物组合理性设计,该种方法实际上是“基于基因表达标签(signature)的方法”而非“基于网络的方法”。

图1 基于基因表达谱的药物组合理性设计[10].A:基因1是与药物1敏感性呈正相关的基因;B:基因2是与药物1敏感性呈负相关的基因;C:以药物1药敏性为查询条件,寻找能上调基因1同时下调基因2的药物作为候选组合药物;D:药物1(地塞米松)与药物2(西罗莫司)的组合确实可以提高药物敏感性.
2 基于协同致死进行药物组合理性设计
通过遗传筛选(genetic screening)寻找与已知化合物有协同致死效应的基因,可以发现与已知药物敏感性显著相关的新基因,从而通过靶向这些新基因的药物,可以发现有效的化合物组合,从而增强单个药物的疗效。Whitehurst等[8]将高通量和高内涵的细胞筛选平台与全基因组RNA干抗(RNA interference,RNAi)文库技术相结合,建立了一个与紫杉醇相关的协同致死基因筛选实验体系,发现并鉴定了一系列紫杉醇处理条件下能选择性降低细胞存活率的基因靶标。这些靶标基因的功能分类主要涉及蛋白酶体、微管相关和细胞黏附等生物学过程。值得注意的是,对某些新识别出来的靶标的干扰可以使肺癌细胞对紫杉醇的敏感性提高1000倍。由本例可以看出,协同致死策略能给药物组合设计提供一个有效的途径:将靶向紫杉醇协同致死基因的药物与紫杉醇联用,从而使肿瘤细胞致死(图2)。据此可设计蛋白酶体抑制剂和紫杉醇的药物组合。事实上,硼替佐米(蛋白酶体抑制剂)与紫杉醇联用的药物组合已应用于临床研究中[11]。
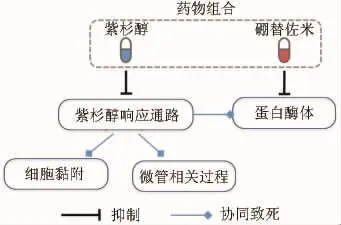
图2 基于协同致死的药物组合设计原理[10].
协同致死也可用于通过药物组合,提高靶向药物的疗效,抑制其耐药性。Astsaturov等[12]通过集成多种数据集建立了以表皮生长因子受体(epidermal growth factor receptor,EGFR)为中心的信号网络,进而通过RNAi文库筛选,得到了对EGFR抑制剂具有增敏作用的基因集合。以该基因集合中的信号转导及转录激活因子3(signal transducerand activator of transcription 3,STAT3)基因为例,作者将针对STAT3的抑制剂stattic与厄洛替尼(一种针对EGFR的酪氨酸激酶抑制剂)组合,证实药物组合可协同降低肿瘤细胞的存活率,抑制肿瘤生长。
3 基于协同致死概念设计个性化治疗
Luo等[5]设计了基于协同致死筛选设计个体化癌症治疗的方法。通过全基因组RNAi筛选,作者鉴定了一系列与癌基因KRAS(一种常见的突变型人类癌症基因)具有协同致死效应的基因。这些基因主要涉及与有丝分裂激酶PLK1(Polo-like kinase 1)和蛋白酶体相关的信号转导通路。抑制这些通路可导致Ras突变型细胞死亡。从上述角度出发可设计个体化治疗策略,以细胞分裂周期蛋白16(cell division cycle protein 16,CDC16)为例,其设计过程如下(图3):
第一步:分析Ras正常型癌症患者群体中CDC16基因的表达值与预后的相关性。如图3A所示,CDC16高表达(红线)与CDC16低表达(蓝线)患者的生存曲线无明显的差别,Log-rank分析的P值为0.67,表明在Ras正常型患者中,CDC16基因表达与患者的预后无关。因此,在这组患者中进行针对CDC16的靶向治疗可能无效。
第二步:分析Ras突变型癌症患者群体中CDC16基因表达值与预后的关系。如图3B所示,CDC16高表达(红线)与CDC16低表达(蓝线)患者的生存曲线具有显著差别,Log-rank分析的P值为0.02,表明在Ras突变型患者中,CDC16基因表达与预后显著相关,推测对这组患者进行针对CDC16的靶向治疗可能有效。
第三步:综合以上分析结果,可设计如下个体化治疗策略,在进行CDC16靶向治疗前,对患者Ras基因突变情况进行检测。如果结果为阳性即Ras突变型,可推荐CDC16靶向治疗;若为阴性,则不考虑采用CDC16靶向治疗。
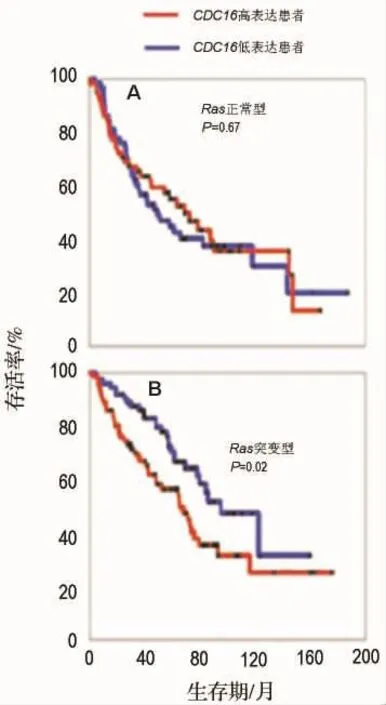
图3 基于协同致死设计个体化治疗的原则[8]
类似地,Scholl等[13]使用高通量RNAi筛选确定了癌症细胞中与KRAS协同致死的基因,并发现丝氨酸/苏氨酸激酶33(serine/threonine kinase 33,STK33)可以作为KRAS突变型癌症患者的治疗靶标。然而,在癌症细胞系或者初期癌症患者样本中STK33并没有结构异常或表达异常,这说明STK33并不是一个传统意义上的致癌基因。最近有研究表明,癌症的发生不仅仅依赖于驱动癌变的传统意义上的“癌基因”的突变,还取决于一些“正常”基因,该现象被称为“非致癌基因成瘾”(non-oncogene addiction)[14-15]。实际上,上述协同致死基因的筛选即为鉴定“非致癌基因成瘾”现象提供了一种有效的方式。
4 协同决定预后(synergistic outcome determination,SOD)——一种与协同致死类似的遗传相互作用
最近,本课题组提出了一种新的遗传相互作用,称为“SOD”。SOD与协同致死类似,是指由一对基因协同影响癌症患者的预后表型,而单个基因的表达与患者预后无显著的相关性[16]。如图4所示,基因(基因A和基因B)的表达及与表型(预后)的关系如下:①基因A和基因B都有低表达和高表达两种状态;② 红色三角形代表“预后差”的患者(生存时间较短或转移),绿色矩形代表“预后好”的患者(存活时间更长或非转移);③单个基因的表达与患者的预后无关,基因A为低表达时,所有基因A低表达的患者都分布在两个簇,50%分布在“预后差”(图4中的“左下”象限),50%分布在“预后好”(图4中的“左上”象限);④ 集成两个基因的表达信息可确定患者的预后情况,若基因A为低表达、同时基因B为高表达时(图4中的“左上”象限),所有的患者都为“预后好”。
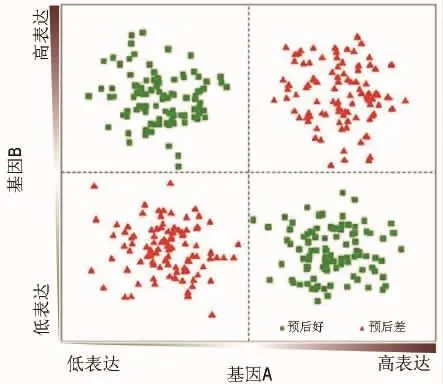
图4 协同决定预后(SOD)的概念[16].
在上述方法中,协同决定患者预后的基因-基因之间的配对可用一种基于信息学的算法来确定[16]。SOD与协同致死有几个不同的特征(表1):①“协同致死”涉及的表型在细胞水平(如细胞死亡),而SOD涉及的表型在生理层面(如患者的生存期等表型)。因此,SOD可以建立“基因层”事件和临床表型之间的直接联系。②由于伦理受限,无法在人体内通过实验筛选协同致死的基因。目前,高通量协同致死筛选仅仅局限于体外培养人体细胞系[8]。而SOD可通过患者预后信息及其肿瘤组织的基因表达数据计算得出。③协同致死筛选使用的是体外细胞系的基因表达数据,而SOD使用的是源自体内的肿瘤组织的基因表达数据。理论上,SOD能够捕捉“组织层面”的事件,而不仅是“细胞层次”事件。这对于肿瘤学研究十分重要,因为从肿瘤组织中得到的基因表达谱实际上包括上皮细胞及微环境中其他细胞混合物的表达信息。

表1 SOD与协同致死的概念比较
5 基于SOD进行药物组合理性设计
基于SOD理论,可以建立与预后等临床表型相关的基因-基因间协同作用网络。基于此基因-基因间相互依赖关系,可将药物敏感性基因映射到SOD网络中,从而建立一种药物组合设计的方法,其原理与计算过程如下:
第一步:计算某癌症特异SOD网络中每个基因节点受到某特定药物扰动的扰动值,从而将药物对网络的扰动信息通过药物敏感性基因投射到基因网络中[16]。如图5A,主药物有4个敏感性基因(基因1,2,3和4),标记“1”,表示主药物的作用模式。
第二步:计算某特定药物对网络中的“边”的扰动情况。SOD网络中的边表示2个基因之间存在协同关系。只有当两节点同时标记为“1”,此扰动边才能被标记为“1”,反映药物对2个基因的共同扰动。如图5A中主药物同时扰动基因1和基因4,因此,基因1和基因4之间的边可标记为“1”。
第三步:通过以下公式计算药物对全局网络的扰动系数(perturbation index,PI):

其中,N为网络中总的边数;M为网络中总的基因节点数;Di为基因节点i的扰动值;Dj为边j的扰动值。举例如下:① 如图5A所示,主药物扰动1个边(基因1和基因4间的连接)和4个节点,因此,主药物的PI为1/4=0.25;②考虑辅药物1与主药物的组合,其作用模式如图5B所示。辅药物1增加了一个对基因5的扰动,从而改变了基因5相关的三条边(基因1和基因5之间的连接,基因2和基因5之间的连接,基因3和基因5之间的连接),因此,该药物组合的PI为4/5=0.8;③ 考虑另一候选辅药物2,主药物与辅药物2组合的作用模式如图5C所示,辅药物2增加了对基因6的扰动,但是只改变了一条边(基因3和基因6之间的连接),该药物组合的PI为2/5=0.4;④ 药物组合(主药物+辅药物1)的PI高于组合(主药物+辅药物2),因此,主药物+辅药物1的组合要优于主药物+辅药物2。
上述例子中,候选辅药物1和辅药物2都扰动了一个基因,但是结果却显著不同。原因在于,辅药物1扰动的基因5与其他基因有较多的协同作用。
6 结论和展望
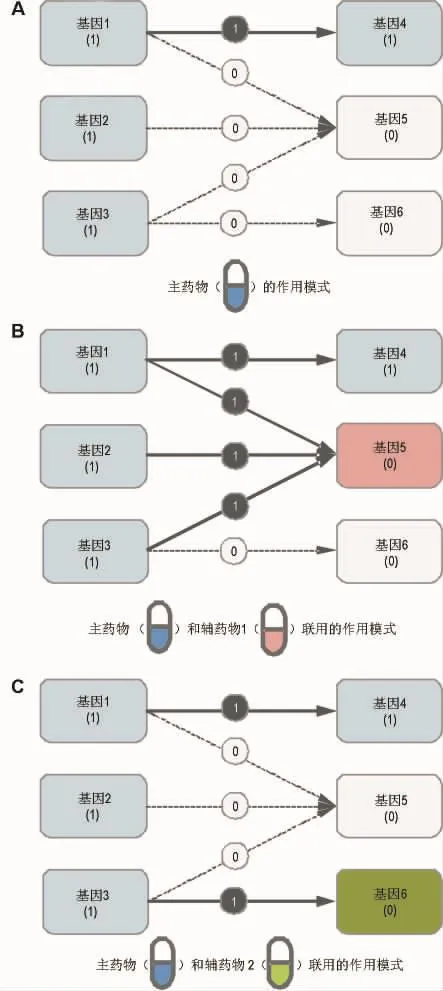
图5 基于SOD进行药物组合理性设计[10].
综上所述,在用药物对抗疾病的“战役”中,以描述基因和疾病表型之间依赖关系的网络可以成为药物组合理性设计与个体化治疗设计的“作战地图”。除了通过RNAi等功能筛选实验得到的协同致死网络,通过生物信息学方法计算得到的遗传相互关系网络亦可用于测试药物组合的协同效果。在有关网络药理学的早期文献中讨论了许多基于蛋白质相互作用网络的研究,而本文中讨论的通过计算得到的遗传相互作用网络则极大地丰富了“网络药理学作战地图”的内涵,理论上,可以通过各种层面定义这种遗传相互作用网络。①一种表型确定一种遗传相互作用。协同致死关系是通过体外实验中细胞存活率来决定,该关系中涉及的细胞表型可以是模式生物(如酵母菌)或体外培养的人体细胞的生。长情况也可以通过药物与基因之间的相互作用来定义新的遗传相互作用。例如,某肿瘤药的药敏性依赖于某个基因,该基因可通过该药物处理下的RNAi实验而筛选获得[8]。该种遗传相互作用中涉及的表型是细胞在药物和RNAi双重干扰下的细胞存活率。②生物系统中不同系统层面事件可以定义不同的遗传相互作用。由于复杂的生物系统可分成多种层面,因而不同层面上各事件(如基因水平、基因模块水平和细胞水平等)之间的相互作用研究也很重要。例如,在基因模块水平,可通过某协同算法推算出决定疾病表型的异常基因模块之间的相互作用关系[8],进而可应用于药物组合设计[16]。在组织水平,不同细胞之间的依赖性与表型有关,以组织微环境内细胞间的相互关系(如癌症细胞和基质细胞[16-17],肿瘤干细胞及其生态位[18])为靶标的药物组合将有可能逆转体内癌症药物的耐药性。
以蛋白质-蛋白质相互作用为代表的物理相互作用(physical interaction)本身不包含表型方面的信息,而遗传相互作用(genetic interaction)是“基因-基因-表型”(或“基因-药物-表型”)三元组之间的一个特定关系。在这种关系中,由于特定表型信息的引入,使得遗传相互作用更适于药理学研究。生物体不同系统层面中可以定义不同的表型,表型的多样性给定义新型的遗传相互作用提供了极大的创新空间。可以预见,遗传相互作用网络将在基于网络的药理学研究中发挥更大、更广泛的作用。
[1]Hopkins AL.Network pharmacology:the next paradigm in drug discovery[J].Nat Chem Biol,2008,4(11):682-690.
[2]Kaelin WG Jr.The concept of synthetic lethality in the context of anticancer therapy[J].Nat Rev Cancer,2005,5(9):689-698.
[3]Fong PC,Boss DS,Yap TA,Tutt A,Wu P,Mergui-Roelvink M,et al.Inhibition of poly(ADP-ribose)polymerase in tumors from BRCA mutation carriers[J].N Engl J Med,2009,361(2):123-134.
[4]Shaheen M,Allen C,Nickoloff JA,Hromas R.Synthetic lethality:exploiting the addiction of cancer to DNA repair[J].Blood,2011,117(23):6074-6082.
[5]Luo J,Emanuele MJ,Li D,Creighton CJ,Schlabach MR,Westbrook TF,et al.A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene[J].Cell,2009,137(5):835-848.
[6]Kim YW,Liu TJ,Koul D,Tiao N,Feroze AH,Wang J,et al.Identification of novel synergistic targets for rational drug combinations with PI3 kinase inhibitors using siRNA synthetic lethality screening against GBM[J].Neuro Oncol,2011,13(4):367-375.
[7]Toledo LI,Murga M,Zur R,Soria R,Rodriguez A,Martinez S,et al.A cell-based screen identifies ATR inhibitors with synthetic lethal properties for cancer-associated mutations[J].Nat Struct Mol Biol,2011,18(6):721-727.
[8]Whitehurst AW,Bodemann BO,Cardenas J,Ferguson D,Girard L,Peyton M,et al.Synthetic lethal screen identification of chemosensitizer loci in cancer cells[J].Nature,2007,446(7137):815-819.
[9]Lamb J,Crawford ED,Peck D,Modell JW,Blat IC,Wrobel MJ,et al.The Connectivity Map:using gene-expression signatures to connect small molecules,genes,and disease[J].Science,2006,313(5795):1929-1935.
[10]Xiong JH,Liang FJ,Li YH.The principle of rational design of drug combination and personalized therapy based on network pharmacology[M]∥Azmi AS.Systems Biology in Cancer Research and Drug Discovery.Springer Science+Business Media Dordrecht:Springer,2012:325-337.
[11]Davies AM,Lara PN Jr,Mack PC,Gumerlock PH,Bold RJ,Gandara DR.Bortezomib-based combinations in the treatment of nonsmall-cell lung cancer[J].Clin Lung Cancer,2005,7(Suppl 2):S59-S63.
[12]Astsaturov I,Ratushny V,Sukhanova A,Einarson MB,Bagnyukova T,Zhou Y,et al.Synthetic lethal screen of an EGFR-centered network to improve targeted therapies[J].Sci Signal,2010,3(140):ra67.
[13]Scholl C,Fröhling S,Dunn IF,Schinzel AC,Barbie DA,Kim SY,et al.Synthetic lethal interaction between oncogenic KRAS dependency and STK33 suppression in human cancer cells[J].Cell,2009,137(5):821-834.
[14]Solimini NL,Luo J,Elledge SJ.Non-oncogene addiction and the stress phenotype of cancer cells[J].Cell,2007,130(6):986-988.
[15]Luo J,Solimini NL,Elledge SJ.Principles of cancer therapy:oncogene and non-oncogene addiction[J].Cell,2009,136(5):823-837.
[16]Xiong J,Liu J,Rayner S,Tian Z,Li Y,Chen S.Pre-clinical drug prioritization via prognosis-guided genetic interaction networks[J].PLoS One,2010,5(11):e13937.
[17]Aharinejad S,Sioud M,Lucas T,Abraham D.Targeting stromalcancer cell interactions with siRNAs[J].Methods Mol Biol,2009,487:243-266.
[18]Malanchi I,Santamaria-Martínez A,Susanto E,Peng H,Lehr HA,Delaloye JF,et al.Interactions between cancer stem cells and their niche govern metastatic colonization[J].Nature,2011,481(7379):85-89.

