过敏性鼻炎的遗传学研究进展
赵延明 张 罗,2*
(1.首都医科大学附属北京同仁医院耳鼻咽喉头颈外科,北京100730;2.教育部耳鼻咽喉头颈外科重点实验室,北京市耳鼻咽喉科研究所,北京100005)
过敏性鼻炎(allergic rhinitis,AR)是IgE介导的鼻黏膜炎症反应,近年来其发病率呈上升趋势,在全世界范围内的发病率约为23%,中国中心城市的自报患病率为8.7% ~24.1%[1]。2002年美国在AR 上的医药花销为600万美元[2],AR给世界卫生支出造成的负担已不容忽视。从1916年Cooke和Vander Veer提出过敏性疾病为“单基因遗传疾病”至今,过敏性疾病的遗传学研究经历了近一个世纪的时间,如今已公认过敏性疾病为多基因遗传疾病,并且基因与环境之间,基因与基因之间的相互作用共同决定疾病的发生发展。AR作为过敏性疾病中的一种,其遗传学研究也经历了一个世纪的曲折发展。
1 过敏性疾病的遗传学研究方法
回首前人在过敏性疾病遗传学研究上取得的成就是令人振奋的,大量的致病基因被确定,过敏性疾病的发病机制趋于明朗,而随着生物实验技术的发展,统计分析方法的改进,在未来的研究中注定会取得更多的成果。但同时也无法忽略这些研究中存在的问题,包括如何明确所有参与疾病发生发展的易感基因及致病基因以及过敏性疾病发生的确切机制、如何解释实验研究的可重复性低以及怎样明确发病过程中环境和基因间的相互作用等。要了解并解决这些问题就要先从有关过敏性疾病遗传学研究的方法上着手。在探究过敏性疾病的致病基因的过程中基因组连锁研究和候选基因相关性研究是最早应用的主要研究手段。
1.1 基因组连锁研究
基因组连锁研究的研究群体是家系成员,检测到基因组中某一区域与疾病相关后,再通过进一步提高分辨率,最终确定哪些基因与过敏性疾病相关。应用于家系研究群体的基因组连锁研究的主要优点是:①检测的单核苷酸多态性(single nucleotide polymorphisms,SNP)位点少(可以少于500个)。由于家族成员间的基因来自共同的祖先,使得个体之间存在相同的较大区域的基因序列,因此对于需检测SNP位点数目可以少于500就足够达到实验要求;②不需要候选基因假设。相对于候选基因相关性研究,基因组连锁研究不需要挑选与过敏性疾病相关的候选基因,而是直接对实验对象的整个基因组进行检测;③ 可以发现新的疾病相关基因。由于候选基因研究无法探测新的致病基因,而基因组连锁研究弥补了这一缺点,在发现新的致病基因的研究中发挥了重要作用,早期的研究中发现的基因位点为后续的研究提供了依据及线索;④在多表型混杂因素的影响下基因组连锁研究是检测目的等位基因的理想手段。
1.2 候选基因研究
候选基因研究关注的是在过敏性疾病发生中参与炎症反应的各个因子的编码基因及相关基因,例如IgE候选基因和细胞因子候选基因,这些基因编码的产物在过敏性疾病中的作用已经得到公认,候选基因的变异与疾病的相关性是通过比较病例组和对照组人群的等位基因频率或者是基因型频率得出的。候选基因研究的主要缺憾来自人口结构的差异,由于病例组和对照组人群可能来于不同人口结构,其等位基因的分布频率必然存在差异,干扰了候选基因研究结果的可靠性,但在校正了统计学分析方法后,候选基因研究已经可以用于人口结构差异的样本研究。虽然与基因组连锁研究相比,候选基因相关性研究不能用于发现新的疾病易感基因位点,但其主要优点为:①基因检测功效高。相对于基因组连锁研究,相同样本量的研究中,候选基因研究对危险度低的致病基因的检测功效较高,这反映了对于危险度低的致病基因,其等位基因的分布频率在家系人群中与非相关人群相比差异并不明显;② 样本获取容易。候选基因研究不要求大样本量的家系人群,非家系的样本人群即可满足要求,相对于基因组连锁研究而言更容易获取研究样本,而且对同样本量大小的研究候选基因相关性研究比基因组连锁研究具有更高的统计学效力[3];③对标记物密度要求高。研究人群为非相关个体使得基因组中相同序列较少,所以候选基因研究要求更高密度的标记物。
在AR的研究过程中,无论是基因组连锁研究还是候选基因研究,都面临同样的问题,即研究结果的可重复性低。在不同人群中独立的可重复的基因型-表型相关性是确定AR相关基因最有力的证据。何为独立的重复研究?先前的研究中把一个等位基因变异当作重复实验的基本单位,但随着SNP检测序技术的发展,更多的学者认为应当把某个基因区域作为重复实验的基本单位,因为已经有先例证明,同一基因的功能变异与过敏性疾病易感性相关联[4]。ADAM33基因就很好的说明了这一问题,基因中SNP及单体型在所有被检测人群中都无相关性,而在不同人群中存在着不同的疾病相关风险SNP和单体型,这提示了疾病易感性的复杂性,可能在同一基因上存在不只一个风险SNP,他们共同决定了疾病的易感性。就重复性实验而言,相对于表型,重复的实验标准是与原始实验完全相同的表型下得出的阳性结果,而对于基因型,严格的标准是与原始实验完全相同的SNP、单体型及风险等位基因下得出阳性结果,对于一个疾病易感基因最有力的支持就是在严格标准的基因型及表型下得到的阳性重复结果。
2 过敏性鼻炎的遗传学研究
在过去的10年中,基因组连锁研究最早用于探索过敏性鼻炎的风险等位基因。确定了一系列AR易感基因位点区域,表1中列出了近年在过敏性鼻炎家族人群进行的遗传学研究成果。
基因组连锁研究中收集样本量足够的过敏性鼻炎家族人群是研究面临的主要困难。丹麦学者在收集大规模家族性过敏性鼻炎患者方面优势突出,并利用基因组连锁研究的方法在AR的早期遗传学研究中做出贡献,发现了多个风险等位基因区域。但是即使在同一国家,来自丹麦的4个过敏性家族研究中确定的基因区域也没能完全重叠。这可能与AR临床表型的选择及患者人群差异有关,但也从另一个方面说明了过敏性鼻炎作为复杂疾病在遗传学研究中的困难性。利用基因组连锁研究家族人群的成果,为后来的进行定位克隆确定具体的易感基因提供了宝贵的线索。
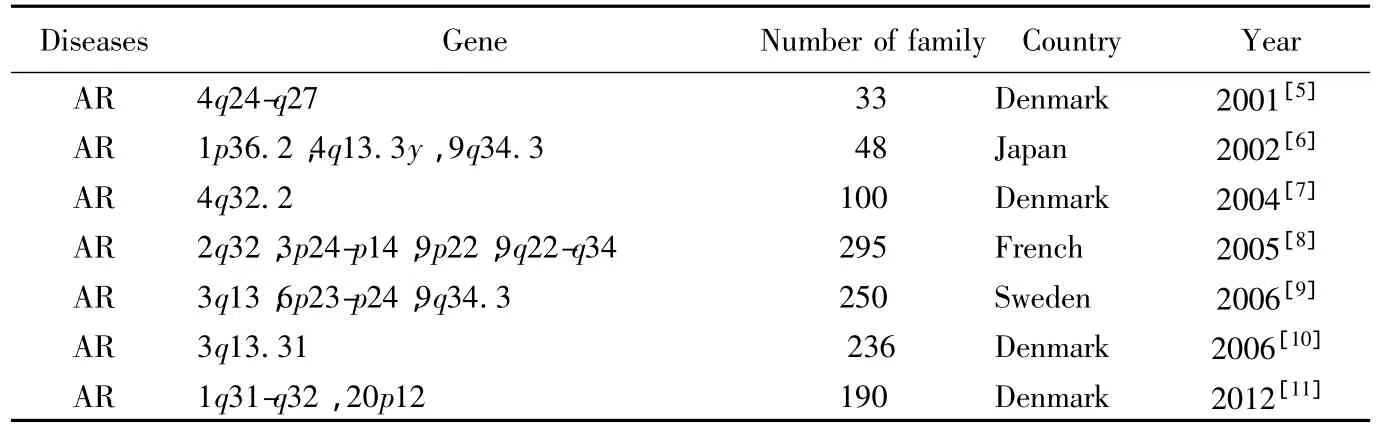
表1 过敏性鼻炎基因组连锁研究Tab.1 Genome-wide linkage study of AR
而对于候选基因相关性研究,也取得很多成果,根据参与AR发病机制的因子不同,候选基因研究可以分成几个主要的集中研究方向。
2.1 趋化性细胞因子及其受体的候选基因研究
趋化性细胞因子是一个蛋白质家族,主要作用是招募血液中的单核细胞、中性粒细胞及淋巴细胞等进入炎症反应发生部位,在AR的发病过程中发挥重要作用。Nakamura等[12]分析了趋化性细胞因子受体CCR1、CCR2、CCR3、CCR5、CCXCR1 的编码基因,发现了8个关于日本雪松特异性花粉症的SNP,又通过遗传不均衡测试及与非相关人群的比较,得出了CCR2的SNP 64Ile及CCR3的SNP 51C与日本雪松特异性花粉症相关,还发现了在病例组中单体型64Ile/780C/51C的频率高于对照组。Zhang等[13]选取了48个日本家庭检查44SNP,在患有AR的病例组及健康人群的对照组中发现在 SDAD1、CXCL9、CSCL10及CXCL11基因附近的单体型在后代的遗传频率明显增高。对于嗜酸细胞活化趋化因子,Chae等[14]研究后发现嗜酸细胞活化趋化因子3基因+2497T>G多态性与韩国人群中AR的易感性相关。
2.2 白细胞介素的候选基因研究
AR的发病机制理论中,有关人类CD4+T细胞分化为辅助性T细胞(help T cell,Th)Th1及Th2的失衡理论已经得到认可,而相关的Th2细胞因子IL-4、IL-5、IL-6、IL-9、IL-10 及IL-13 的基因研究也得到了广泛的关注。Movahedi等[15]对IL-4及IL-4R基因多态性进行分析后,发现IL-4基因的位点rs2243250变异的人群AR的发生率是正常人群的7.17倍,IL-4R基因区域内虽然未发现风险等位基因但位点rs1801275的变异与AR患者的某些临床特征具有相关性(嗜酸性粒细胞百分比,血清IgE水平等)。然而,Yadav[16]报道 IL-4 T589C,IL-4RA I50V,IL-4RA Q576 3个基因位点在AR患者和正常人群中未发现统计学差异,而IL-13R130Q多态性则与AR的发病风险相关。中国学者报告了尘螨过敏的AR患者中IL4 C-590基因多态性与疾病具有相关性,而IL-13基因和IL-4R基因的变异则未发现与尘螨过敏AR具有明显相关性[17]。IL-10基因多态性研究结果表明了IL-10RA基因上的S138G位点的变异可升高人群过敏性疾病发病风险[18]。
除了经典的Th1、Th2细胞因子对过敏性鼻炎的炎性反应机制发挥调节作用,有研究[19-20]表明Th17细胞也在炎性反应的调节过程中发挥了重要作用。Wang等[21]在对过敏性鼻炎伴哮喘的患者的研究中,发现位于IL-17A基因上的rs3819024位点,及IL-17A基因附近的2个位点rs1892280、rs10807439与AR的发病具有显著相关性。
2.3 血管紧张素转换酶候选基因的研究
血管紧张素转换酶(angiotensin converting enzyme,ACE)通过钝化炎性反应前肽类分子如P物质及缓激肽等发挥抗炎作用。Kim等[22]分析了137个AR患者中ACE基因插入和缺失型的基因型和等位基因频率,及186个AR患者(两项研究的健康对照者均为219名)中的血管紧张素原基因M235T多态性,最后未得出阳性结果。Kuet等[22]在韩国人群中做相似的研究同样未得出阳性结果。Lue等[23]在选取了106例AR不伴哮喘的儿童,及105例AR同时患有哮喘的儿童,并有102例健康儿童作为对照,研究发现ACE基因DD型在AR伴哮喘的儿童中的比例大于单纯的AR患儿,提出ACE基因DD型多态性与哮喘并AR的患者有相关性。最近,国内学者[24]对ACE的基因多态性与AR的相关性进行了荟萃分析,对6项研究结果共1 410个体进行了分析,发现ACE基因多态性与AR发病风险具有相关性,但是在亚型的分析中发现ACE仅提高了成人AR患者的发病风险而在儿童中未得到具有统计学意义的结果。
2.4 其他相关候选基因研究
Nakamura等[25]报道了嗜酸性粒细胞过氧化酶的编码基因Pro358Leu多态性与日本雪松过敏性鼻炎发生的相关性。白三烯是AR的重要调节因子,近年抗白三烯药物已经广泛应用于AR及哮喘的治疗。LTC4S作为白三烯合成调节的关键酶其基因A-444C多态性也在哮喘研究中得到证实[26],Eskandari等[27]报道了A-444C多态性可增加AR的发病风险。另外,在汉族人群中发现CD14基因多态性与AR相关但与血清IgE水平无相关性[28],FOXp3基因及EBI3基因多态性也在汉族人群中发现了与AR相关性[29]。这些候选基因的研究成果虽然重复性未得到证实,但也为后来的研究提供的线索。
相对于基因组连锁研究和候选基因研究,近年来日益成熟的全基因组相关研究(genome wide association scans,GWAS)在多基因遗传疾病的体现出了其自身的优势性。GWAS得到飞速发展主要得益于:①人类基因组SNP分型;② 高密度基因测序芯片技术的发展。由于同时可以对受试个体基因组上大于500 000的SNP进行检测,促使GWAS中要求大容量的SNP检测得以实施;③ 常见疾病的大样本量且分型完善的临床研究样本的确定。这些都极大的推动了全基因组相关性研究发展,多基因复杂疾病的遗传学研究提供了更广阔的空间。
GWAS的主要优势在于其对新的疾病易感基因的发现并由此揭示疾病的发病机制,并且能弥补候选基因研究及基因连锁研究中对中等风险的疾病易感基因检出率低的缺陷。GWAS存在的不足是实验需要测序的SNP位点多,一些经济实力较差的研究组织无力承担。为解决这一问题,研究者发明了基因混合测序的方法(pooling-based genome-wide association scans,pGWAS),即将病例组和对照组待测基因分别混合为两个基因库,这样只需检测两个样本的SNP,大大节省了实验费用。而pGWAS结果的可靠性也得到了证实[30],但是无论是GWAS或是最早应用的基因组连锁及侯选基因研究,最终致病的真正易感基因只能通过进一步建立基因及其变异的功能分型或是通过动物的基因功能实验来确定。
AR的遗传学研究最终的价值需要依靠临床实践来实现,对于在研究中取得的成果主要有以下几个方向的临床应用。①AR发病机制的发展性认识。早期的基因组连锁研究作为检测AR新的易感基因主要手段发挥了重要作用,GWAS在未来的研究中必定会发现更多的新的易感基因,最终完善AR发病机制网络。同时,新的易感基因的确定也将为AR治疗提供了新的线索,确定环境因素对AR发生发展的作用,明确环境中哪些因子与疾病的发生存在因果联系,最终可以通过调节环境因子来干预AR的发生发展。②确定疾病易感人群。AR的遗传学研究提示了其相应的易感人群,在发育早期对AR易感性进行评估,通过早期干预来降低人群中AR的发病率。③AR的导向意义。AR的遗传学研究提供了在基因水平上对疾病进行分型的可能,根据基因水平的疾病分型来指导临床实施治疗。确定哪些个体有可发展成重度AR的风险,并对这些人去实施一级预防干预。同时,通过药物遗传学的研究尚可明确个体对药物的敏感度,实施个体化治疗方案。
3 展望
AR的遗传学研究已经取得了很多成果,但由于其多基因遗传机制的复杂性,基因与环境间的相互作用,很多遗传学的研究可重复性比较低,甚至得出相反的结果,但从AR“单基因”遗传理论到Tips等研究提出“多基因理论”,经历了近40年的时间,而从1966年阐明IgE的生物学性质到今天AR众多候选基因的报告,又大约历时40年的时间,但我们可以看到后40年AR的研究进展速度是前一个40年无法比拟的,这其中的最大的原因就是分子生物学飞速发展。遗传学的实验技术随着科技发展取得的进步,更多更新的实验技术为研究AR提供有效的手段,同时各个国家的多中心协作也为过敏性鼻炎的研究提供有力的支持,早日建立起一个完整统一的研究体系,明确不同种族及环境对于AR的影响,并在此基础上构建一个有机的研究体系是最终解决AR遗传学研究各种困难的有效途径。相信随着研究的深入以及DNA相关技术的发展,AR遗传学的神秘面纱最终会被揭开。
[1]Zhang L,Han D,Huang D,et al.Prevalence of self-reported allergic rhinitis in eleven major cities in China[J].Int Arch Allergy Immunol,2009,149(1):47-57.
[2]Law A W,Reed S D,Sundy J S,et al.Direct costs of allergic rhinitis in the United States:estimates from the 1996 Medical Expenditure Panel Survey[J].J Allergy Clin Immunol,2003,111(2):296-300.
[3]Risch N,Merikangas K.The future of genetic studies of complex human diseases[J].Science,1996,273(5281):1516-1517.
[4]Ober C,Hoffjan S.Asthma genetics 2006:the long and winding road to gene discovery[J].Genes Immun,2006,7(2):95-100.
[5]Haagerup A,Bjerke T,Schoitz P O,et al.Allergic rhinitis——a total genome-scan for susceptibility genes suggests a locus on chromosome 4q24-q27[J].EJHG,2001,9(12):945-952.
[6]Yokouchi Y,Shibasaki M,Noguchi E,et al.A genomewide linkage analysis of orchard grass-sensitive childhood seasonal allergic rhinitis in Japanese families[J].Genes Immun,2002,3(1):9-13.
[7]Haagerup A,Borglum A D,Binderup H G,et al.Finescale mapping of type I allergy candidate loci suggests central susceptibility genes on chromosomes 3q,4q and Xp[J].Allergy,2004,59(1):88-94.
[8]Dizier M H,Bouzigon E,Guilloud-Bataille M,et al.Genome screen in the French EGEA study:detection of linked regions shared or not shared by allergic rhinitis and asthma[J].Genes Immun,2005,6(2):95-102.
[9]Bu L M,Bradley M,Soderhall C,et al.Genome-wide linkage analysis of allergic rhinoconjunctivitis in a Swedish population[J].Clin Exp Allergy,2006,36(2):204-210.
[10]Brasch-Andersen C,Haagerup A,Borglum A D,et al.Highly significant linkage to chromosome 3q13.31 for rhinitis and related allergic diseases[J].J Med Genetics,2006,43(3):e10.
[11]Kruse L V,Nyegaard M,Christensen U,et al.A genomewide search for linkage to allergic rhinitis in Danish sib-pair families[J].EJHG,2012,20(9):965-972.
[12]Nakamura H,Higashikawa F,Nobukuni Y,et al.Genotypes and haplotypes of CCR2 and CCR3 genes in Japanese cedar pollinosis[J].Int Arch Allergy Immunol,2007,142(4):329-334.
[13]Zhang J,Noguchi E,Migita O,et al.Association of a haplotype block spanning SDAD1 gene and CXC chemokine genes with allergic rhinitis[J].J Allergy Clin Immunol,2005,115(3):548-554.
[14]Chae S C,Park Y R,Oh G J,et al.The suggestive association of eotaxin-2 and eotaxin-3 gene polymorphisms in Korean population with allergic rhinitis[J].Immunogenetics,2005,56(10):760-764.
[15]Movahedi M,Amirzargar A A,Nasiri R,et al.Gene polymorphisms of Interleukin-4 in allergic rhinitis and its association with clinical phenotypes[J].Am J Otolaryngol,2013,34(6):676-681.
[16]Yadav A,Govindasamy G K,Naidu R.Polymorphic variants of interleukin-13 R130Q,interleukin-4 T589C,interleukin-4RA I50V,and interleukin-4RA Q576R in allergic rhinitis:A pilot study[J].Allergy Rhinol,2012,3(1):e35-e40.
[17]Lu M P,Chen R X,Wang M L,et al.Association study on IL4,IL13 and IL4RA polymorphisms in mite-sensitized persistent allergic rhinitis in a Chinese population[J].PloS One,2011,6(11):e27363.
[18]Hussein P Y,Zahran F,Ashour Wahba A,et al.Interleukin 10 receptor alpha subunit(IL-10RA)gene polymorphism and IL-10 serum levels in Egyptian atopic patients[J].J Invest Allergol Clin Immunol,2010,20(1):20-26.
[19]Crome S Q,Wang A Y,Levings M K.Translational minireview series on Th17 cells:function and regulation of human T helper 17 cells in health and disease[J].Clin Exp Immunol,2010,159(2):109-119.
[20]Afzali B,Mitchell P,Lechler R I,et al.Translational mini-review series on Th17 cells:induction of interleukin-17 production by regulatory T cells[J].Clin Exp Immunol,2010,159(2):120-130.
[21]Wang M,Zhang Y,Han D,et al.Association between polymorphisms in cytokine genes IL-17A and IL-17F and development of allergic rhinitis and comorbid asthma in Chinese subjects[J].Hum Immunol,2012,73(6):647-653.
[22]Kim J J,Kim H J,Lee I K,et al.Association between polymorphisms of the angiotensin-converting enzyme and angiotensinogen genes and allergic rhinitis in a Korean population[J].Ann Otol,Rhinol Laryngol,2004,113(4):297-302.
[23]Lue K H,Ku M S,Li C,et al.ACE gene polymorphism might disclose why some Taiwanese children with allergic rhinitis develop asthma symptoms but others do not[J].Pediatr Allergy Immunol,2006,17(7):508-513.
[24]Lin H,Lin D,Zheng C Q.Angiotensin-converting enzyme insertion/deletion polymorphism associated with allergic rhinitis susceptibility:Evidence from 1410 subjects[J].JRAAS,2013,
[25]Nakamura H,Miyagawa K,Ogino K,et al.High contribution contrast between the genes of eosinophil peroxidase and IL-4 receptor alpha-chain in Japanese cedar pollinosis[J].J Allergy Clin Immunol,2003,112(6):1127-1131.
[26]Sanak M,Simon H U,Szczeklik A.Leukotriene C4 synthase promoter polymorphism and risk of aspirin-induced asthma[J].Lancet,1997,350(9091):1599-1600.
[27]Eskandari H G,Unal M,Ozturk O G,et al.Leukotriene C4 synthase A-444C gene polymorphism in patients with allergic rhinitis[J].Otolaryn Head Neck,2006,134(6):997-1000.
[28]Han D,She W,Zhang L.Association of the CD14 gene polymorphism C-159T with allergic rhinitis[J].Am J Rhinol Allergy,2010,24(1):e1-e3.
[29]Zhang Y,Duan S,Wei X,et al.Association between polymorphisms in FOXP3 and EBI3 genes and the risk for development of allergic rhinitis in Chinese subjects[J].Hum Immunol,2012,73(9):939-945.
[30]Distefano J K,Taverna D M.Technological issues and experimental design of gene association studies[J].Meth Mol Biol,2011,700:3-16.

