细菌对噬菌体的抵抗机制研究进展*
陈灿煌,赵 霞 综述,谭银玲 审校
(第三军医大学微生物学教研室,重庆 400038)
噬菌体是感染细菌、真菌、放线菌或螺旋体等微生物的病毒,专性细胞内寄生,具有严格的宿主特异性。噬菌体可以感染宿主菌并将其裂解;而宿主菌为了对抗噬菌体的感染,也发展出相应的防御机制,即噬菌体抵抗(bacteriophage resistance)。噬菌体是迄今地球上数量最多的微生物,其数目是细菌的10倍[1],也可能是最多样化的微生物。噬菌体通过捕获细菌而维持着自然界中微生态的平衡,因此噬菌体和噬菌体的对抗机制其实在调节微生物种群中起着重要作用。
近来,细菌对抗菌药物耐药性的广泛传播引起全世界关注,人们开始重新考虑利用噬菌体来治疗细菌感染,即噬菌体治疗。而噬菌体治疗最显著的一个缺点是不能治疗抗噬菌体的细菌性感染疾病,并且可能会导致耐受噬菌体的菌株出现。不管是自然条件还是人为条件,细菌对噬菌体的抗性问题都很难解决,因此迫切需要弄清细菌对噬菌体的抵抗机制,为今后噬菌体治疗的靶点研究及耐受噬菌体细菌性感染的治疗提供可靠的依据。本文就此对细菌的抗噬菌体机制以及噬菌体的反防御策略进行综述。
1 吸附抑制
噬菌体感染的第一步是识别位于细菌表面的特异性受体。噬菌体面对的不仅是宿主菌数量庞大类型繁多的细胞膜和细胞壁成分,还有细菌通过突变产生的各种抗吸附机制,如噬菌体受体的突变、细胞外基质分泌的调节和竞争性抑制物的合成等。
细菌可以通过改变细胞表面受体的结构或构象来限制噬菌体的吸附。如金黄色葡萄球菌产生的一种细胞壁锚定毒性因子——IgG结合蛋白A,可以掩盖细菌表面的噬菌体受体,从而减少噬菌体的吸附数量;大肠埃希菌的外膜蛋白A(outer membrane protein A,OmpA)是T-偶数样噬菌体的受体,能被一种F质粒编码的外膜蛋白TraT包裹和修饰,使噬菌体无法识别。还有一些细菌通过相变(phase variation)改变细胞的表面结构,如博德特菌(Bordetella spp.)[2]。百日咳杆菌黏附素自主转运体(pertactin autotransporter,Prn)为博德特菌噬菌体受体,只能在博德特Bvg+相菌中表达,不能在Bvg-相菌中表达,因此博得特菌噬菌体BPP-1感染Bvg+相菌的效率比感染Bvg-相菌高出百万倍。尽管Bvg-相博德特菌中不存在噬菌体受体Prn,但BPP-1噬菌体仍然能较小程度地感染Bvg-相博德特菌,提示噬菌体产生了一种不结合主要受体也能感染细菌的变体。博得特菌噬菌体噬菌体基因mtd编码主要趋向性决定蛋白质,具有宿主识别的作用;而一些博得特菌噬菌体利用差异性反转录因子(diversity-generating retroelements,DGRs)介导的模板依赖逆转录作用在基因mtd的可变区引入核苷酸替换,引起主要趋向性决定蛋白的末端变异,使其可与不同的细菌表面结构相互作用,从而导致噬菌体吸附,最终逆转因细菌表面受体变异而引起的吸附抑制作用。最近的比较基因组学研究表明,包括双歧杆菌在内的19种噬菌体均含有DGRs系统,该系统可能在不同环境中噬菌体与其宿主菌的共进化过程中发挥重要作用。
细胞外的聚合物不仅有利于细菌在恶劣的环境中存活,还能为细菌在噬菌体和受体间提供一个物理屏障。但是在快速进化过程中,一些噬菌体可通过多糖水解酶或裂合酶识别甚至破坏细胞外多糖聚合物。一些假单胞菌,固氮菌可产生藻酸盐增加对噬菌体的抵抗;而以假单胞菌为宿主的F116噬菌体能合成一种藻酸盐裂解酶,能减低细胞外基质的黏滞性,更有利于噬菌体在细胞外基质中扩散。大肠埃希菌(E.coli)细胞表面具有脂多糖O抗原和荚膜多糖K抗原;噬菌体随着细菌的多样性进化而进化,一些噬菌体能与这些抗原特异性结合,如荚膜缺陷的菌株对K抗原特异性的噬菌体不敏感。在沙门氏菌噬中也能观察到同样的现象:P22噬菌体能识别O抗原。P22噬菌体的尾部具有内切糖苷酶活性,能使噬菌体穿过100 nm的O抗原层。E.coliO157:H7的O抗原特异性噬菌体ΦV10具有乙酰转移酶活性,能对O157抗原进行修饰,阻止噬菌体ΦV10和其他相似噬菌体的吸附。流感嗜血杆菌的噬菌体HP1c1同样面临宿主菌细胞表面结构的相变,Lic2A基因编码的蛋白参与表面脂低聚糖的合成,对Lic2A进行修饰能使流感嗜血杆菌发生相变,阻止HP1c1的吸附。
细菌可以利用环境中的小分子特异性结合到噬菌体的受体上,从而使噬菌体不能与这些受体结合。如大肠杆菌合成的铁转运蛋白FhuA[3],同时也是大肠杆菌噬菌体T1、T5和Φ80进入细菌的通道;抗菌小分子蛋白质J25也能利用FhuA作为受体,可与T5噬菌体竞争性结合FhuA受体。J25是细菌在养分耗尽时合成的,通过抑制近源菌株的生长来避免噬菌体的感染,在与其他微生物的竞争中发挥着重要作用。
2 DNA穿入阻滞
超感染排除(Superinfection exclusion,Sie)系统是细菌阻滞噬菌体DNA穿入细胞的主要机制。Sie系统主要涉及细胞膜蛋白质,编码这些蛋白质的基因常常也存在于噬菌体中。因此,除了在噬菌体和宿主菌相互作用中发挥效应外,Sie系统在噬菌体与噬菌体相互作用中也扮演着重要的角色。
目前已经鉴别出多种Sie系统,但只有少数Sie系统的机制被研究清楚,以革兰阴性菌中的Sie系统为例。T4噬菌体是众所周知的裂性噬菌体,其宿主大肠杆菌具有两种Sie系统,分别由imm基因和sp基因编码(Imm系统和Sp系统)。这两种系统能快速地抑制DNA进入细胞,阻止其他与T4噬菌体类似噬菌体的次级感染。尽管Imm系统和Sp系统的功能相似,但各自独立并具有不同的作用机制。Imm系统具有两个仅存在于细胞膜的特殊跨膜区域,通过改变DNA注入位点的构象来阻止噬菌体DNA进入细菌;Imm系统不能独自完成对噬菌体的免疫,只有在其他膜蛋白的协助下才能正常发挥作用。T4噬菌体的溶菌酶存在于尾部,能在细胞壁上产生空洞,利于噬菌体DNA进入细胞[4];Sp系统通过抑制T4噬菌体溶菌酶对肽聚糖的降解活性来防止噬菌体DNA入侵。
目前只有少数革兰阳性菌抑制噬菌体DNA注入的机制为人所知,它们大多数是用于工业牛奶发酵生产的乳酸杆菌。其中最具特征性的DNA注入机制是在乳酸杆菌噬菌体Tuc2009基因组中发现的Sie2009系统,之后又在乳酸杆菌其他菌种的噬菌体基因组中发现了该系统。包括Tuc2009在内的大多数乳酸杆菌噬菌体都属于P335乳酸杆菌噬菌体,来自这些噬菌体的Sie2009系统能够抵抗遗传特异性的936乳酸杆菌噬菌体。936噬菌体在乳酸杆菌特异性噬菌体中处于优势地位。有学者推测这个Sie系统位于细胞膜,通过抑制噬菌体DNA进入宿主细胞而发挥噬菌体抑制作用。随后,一种类似于Sie系统的机制在另一种用于乳品发酵工业的细菌——嗜热链球菌的前噬菌体中被发现。前噬菌体TP-J34编码一种耐受142-氨基酸脂蛋白的单信号肽,能阻止噬菌体DNA进入细胞。但是,这一机制能对进入乳酸菌的乳酸噬菌体发挥抑制作用。
3 降解噬菌体DNA
3.1 限制-修饰(restriction-modification,R-M)系统 几乎所有细菌都具有R-M系统,其活性依赖于几种不同的蛋白质,这些蛋白质至少可以分Ⅰ~Ⅳ型。R-M系统最主要的功能是防止外来DNA及病毒入侵细胞[5-6]。细菌对噬菌体的R-M系统由能降解侵入的噬菌体DNA的限制性内切酶和保护宿主DNA免遭消化的甲基化成分组成。其“经典”机制就是当进入细菌体内的噬菌体DNA在某些特定位点上未被甲基化(即修饰作用)时,那么它就被宿主的限制性内切酶裂解(即限制作用)[7],这样噬菌体就不能在细菌体内复制。
但噬菌体也发展出多种抗R-M系统的策略,与细菌进行着限制与反限制的多重较量。策略之一即是获得宿主菌的甲基化酶基因或利用宿主菌的甲基化系统来修饰噬菌体基因组DNA,如λ及其衍生噬菌体编码的Ral及Lar蛋白可促进噬菌体DNA的甲基化修饰;策略之二是通过基因组点突变的积累而造成细菌核酸内切酶识别位点的缺失,如多价葡萄球菌噬菌体的双链DNA上缺失Sau3位点,该位点具5′-GATC-3′识别序列,或阻断核酸内切酶的活性,如T3噬菌体编码的Ocr蛋白可通过水解S-腺苷蛋氨酸而抑制核酸内切酶的作用。其中最令人称奇的例子是在T4噬菌体中发现的抗R-M系统。该裂解性噬菌体的基因组中含有稀有碱基羟甲基胞嘧啶(hydroxymethyl cytosine,HMC),而宿主 DNA中的则是胞嘧啶。R-M系统只能识别具有胞嘧啶的特异序列,T4噬菌体DNA的这一特殊修饰使R-M系统无法识别。而在细菌与噬菌体的共进化过程中,一些细菌已经发展出相对应的机制来破坏这些经过修饰的噬菌体DNA,如与R-M系统不同的修饰依赖性系统(modified dependent systems,MDSs),可特异性地降解甲基化或羟甲基化的DNA。目前,仅少数MDSs酶被认知,如肺炎链球菌中的DpnI和大肠杆菌中的McrA、McrBC及Mrr。由于HMC残基的糖基化,T4噬菌体对MDS酶也具有抵抗力。但T4噬菌体仍然能逃避MDSs的作用,因其基因组HMC残基已被糖基化。而大肠杆菌CT596则发展出由葡萄糖修饰抑制剂(glucose modified inhibitor,Gmr)GmrS和GmrD两种前噬菌体蛋白构成的二元系统,能识别和裂解DNA中糖基化的HMC,但对未糖基化的DNA无效。而多种T4样噬菌体能够编码一种特异性针对GmrS-GmrD系统的内部蛋白(internal protein,IPI),感染时,成熟的IPI伴随噬菌体基因组进入宿主细胞,与GmrS-GmrD复合物相互作用并使复合物失活。一些细菌能利用单一的融合多肽来对付噬菌体的IPI,以维持其对噬菌体的抵抗作用。
3.2 CRISPR-Cas系统 CRISPR-Cas系统是细菌对付噬菌体的又一武器,研究者已经对规则性集簇间隔短重复序列(CRISPRs)及其伴侣因(cas)进行了详细的研究[8]。自1987年首次对CRISPR-Cas系统进行描述以来[9],比较性分析表明,该基因座存在于日益增多的细菌和古细菌的全基因组中。这些基因座一般含有21~48bp的正向重复序列,而这些重复序列由长度相似的26~72bp的非重复性间隔子(non-repetitive spacers)隔开,整个基因座的一侧则排列着大量的cas基因,数量从4个到20个不等。近来研究认为,CRISPR-Cas系统是细菌所特有的获得性免疫系统,可特异性抵抗外来核酸包括噬菌体基因组和质粒的入侵[10]。另有研究表明,CRISPRCas系统可能与铜绿假单胞菌的溶原性依赖的生物膜合成抑制及群集运动性有关。
当嗜热链球菌的噬菌体敏感株被烈性噬菌体感染后,就会产生抗噬菌体的突变菌株。这些突变株在CRISPR基因座的重复序列-间隔子区域的5′端获得了至少一个新的重复序列-间隔子单位。这个新产生的间隔子(通常在嗜热链球菌中为30bp)与位于噬菌体基因组的所谓前间隔子(proto-spacer)序列完全一致,其分子机制目前尚不清楚。不同的宿主菌能从相同的噬菌体基因组中获得各自特异的间隔子序列,惟一的共同特征是在该噬菌体基因组中前间隔子的两侧分布着一些短的保守核苷酸序列(如NNAGAAW),被称为前间隔子相关性基序(proto-spacer-associated motif,PAM)。值得注意的是,不论PAM是位于前间隔子的上游或下游,PAM的序列及位置在CRISPR-Cas系统中是变化的。
关于CRISPR-Cas系统的作用机制已有多个假说提出。其中一个假说认为,CRISPR-Cas系统能通过细菌的RNA干扰机制以RNA为目标发挥作用。另有学者认为,CRISPR-Cas系统也可以针对DNA发挥作用。几组研究人员纯化了Cas蛋白,发现这些蛋白质可在重复区域将CRISPR mRNA切成小RNAs片段,且这些小RNA片段可靶向降解刚进入细胞的噬菌体或质粒的RNA或DNA。
噬菌体在与细菌的相互作用中其实也已进化出相关途径对抗CRISPR-Cas系统的作用,如嗜热链球菌噬菌体可通过前间隔子区的点突变、缺失或在保守的PAM区的突变来抵抗CRISPR-Cas系统。细菌仍然有可能获得源自噬菌体突变株的新的间隔子序列,从而建立新的噬菌体抵抗状态。因此事实上,CRISPR-Cas系统与噬菌体的应答正好说明了不同微生物种类间动态的共进化关系,并且被公认的是,CRISPR-Cas系统在微生物群落结构的塑造中扮演了重要角色。
4 顿挫感染
细菌能够合成各种同源蛋白使噬菌体的发生顿挫感染。
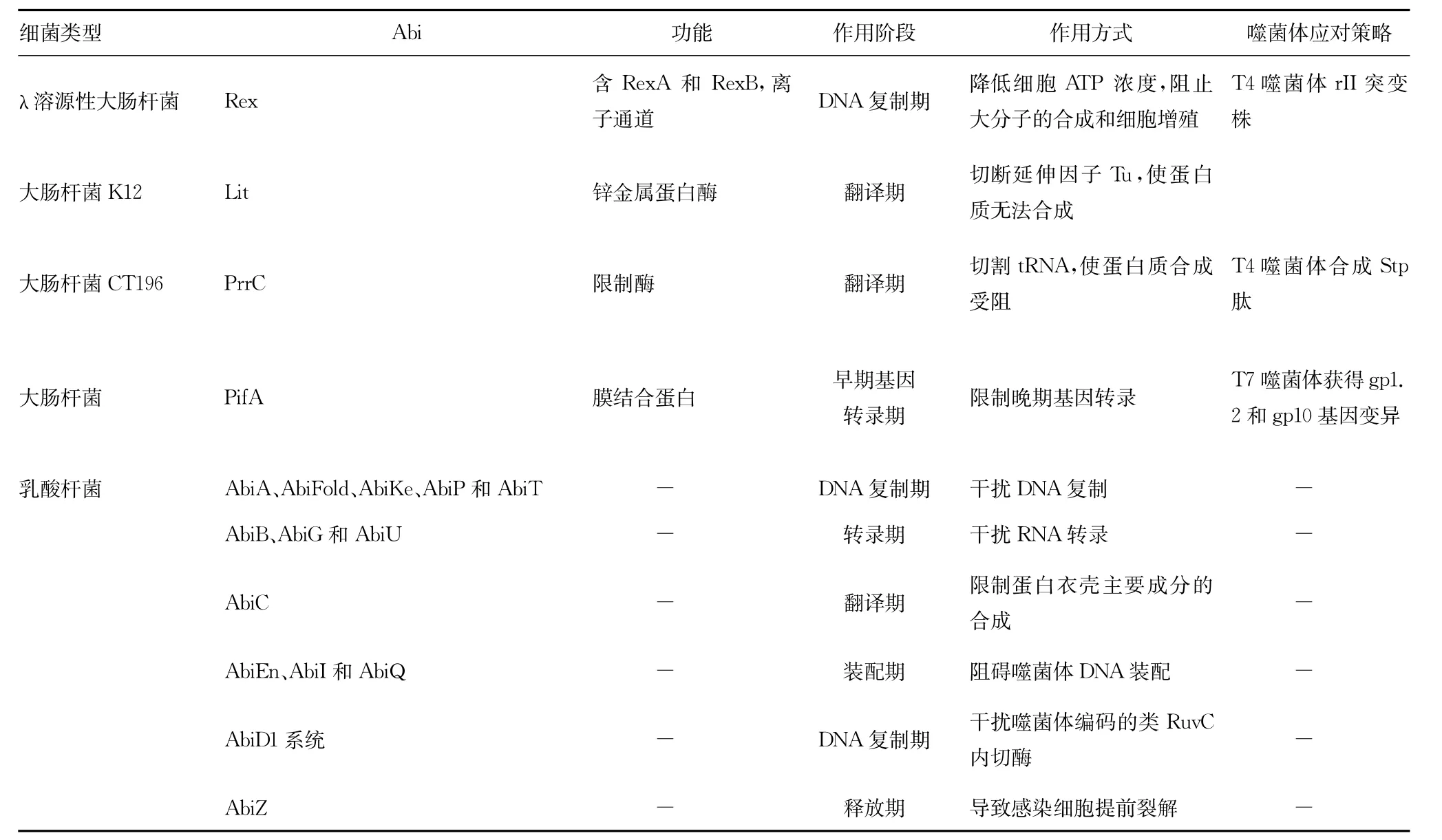
表1 细菌中的Abi
与前几种抗噬菌体系统有所不同,顿挫感染系统(abortive infection,Abi)在噬菌体感染的复制、转录或翻译等关键步骤发挥作用,会导致感染细胞的死亡。对Abi的研究开始于50年前,但是直到现在仅部分了解其在少数几种细菌中的作用方式,大多数细菌中的Abi还有待进一步鉴定[11-26],见表1。
除此以外,在细菌中广泛存在的毒素-抗毒素系统(toxinantitoxin systems,ToxIN)也与噬菌体的顿挫感染有关,如黑腐果胶杆菌属中的ToxIN系统、大肠杆菌中的hok-shok系统及mazeF系统。
5 展 望
细菌对噬菌体的抗性机制通常都是在实验条件下进行一次性单一的噬菌体-宿主的研究,而且细菌的菌株之间往往存在复杂多重的抗噬菌体屏障。同一个细菌中还有许多无法估计的各种系统之间的相互协作,噬菌体群体的共同作用和进化效应也常常被人们忽略,不同噬菌体之间的相互抵抗也同样未被人重视。由于自然条件下的抗病毒系统反映出细菌性病毒显著的多样性,也反映出抗病毒系统在维护自然和人类环境中噬菌体-宿主平衡的作用,更多的噬菌体屏障还有待发现。现在,迫切需要超越以往对噬菌体的认识,尽力去了解这些抗病毒效应背后的分子机制,有了对噬菌体的进一步认识,就能更深入的了解整个生命系统。
[1]Brüssow H,Hendrix RW.Phage genomics:small is beautiful[J].Cell,2002,108(1):13-16.
[2]Uhl MA,Miller JF.Integration of multiple domains in a two-component sensor protein:the Bordetella pertussis BvgAS phosphorelay[J].EMBO J,1996,15(5):1028-1036.
[3]Destoumieux-Garzon D,Duquesne S,Peduzzi J,et al.The iron-siderophore transporter FhuA is the receptor for the antimicrobial peptide microcin J25:role of the microcin Val11-Pro16β-hairpin region in the recognition mechanism[J].Biochem J,2005,389(Pt 3):869-876.
[4]Lu MJ,Stierhof YD,Henning U.Location and unusual membrane topology of the immunity protein of the Escherichia coli phage T4[J].J Virol,1993,67(8):4905-4913.
[5]Pingoud A,Fuxreiter M,Pingoud V,et al.Type II restriction endonucleases:structure and mechanism[J].Cell Mol Life Sci,2005,62(6):685-707.
[6]Pingoud A.Restriction endonucleases[J].Springer,2004,14(7):63-90.
[7]Hill C,Miller LA,Klaenhammer TR.In vivo genetic exchange of a functional domain from a typeⅡA methylase between lactococcal plasmid pTR2030and a virulent bacteriophage[J].J Bacteriol,1991,173(14):4363-4370.
[8]Sorek R,Kunin V,Hugenholtz P.CRISPR-a widespread system that provides acquired resistance against phages in bacteria and archaea[J].Nature Rev Microbiol,2008,6(3):181-186.
[9]Ishino Y,Shinagawa H,Makino K,et al.Nucleotide-sequence of the iap gene,responsible for alkaline-phosphatase isozyme conversion in Escherichia coli,and identification of the gene-product[J].J Bacteriol,1987,169(12):5429-5433.
[10]Marraffini LA,Sontheimer EJ.CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA[J].Science,2008,322(5909):1843-1845.
[11]Molineux IJ.Host-parasite interactions:recent developments in the genetics of abortive phage infections[J].New Biol,1991,3(3):230-236.
[12]Bingham R,Ekunwe SIN,Falk S,et al.The major head protein of bacteriophage T4binds specifically to elongation factor Tu[J].J Biol,2000,275(30):23219-23226.
[13]Snyder L.Phage-exclusion enzymes:a bonanza of biochemical and cell biology reagents[J].Mol Microbiol,1995,15(3):415-420.
[14]Cheng X,Wang W,Molineux IJ.F exclusion of bacteriophage T7occurs at the cell membrane[J].Virology,2004,326(2):340-352.
[15]Hill C,Miller LA,Klaenhammer TR.Nucleotide sequence and distribution of the pTR2030resistance determinant(hsp)which aborts bacteriophage infection in lactococci[J].Appl Environ Microbiol,1990,56(7):2255-2258.
[16]Garvey P,Fitzgerald GF,Hill C.Cloning and DNA sequence analysis of two abortive infection phage resistance determinants from the lactococcal plasmid pNP40[J].Appl Environ Microbiol,1995,61(12):4321-4328.
[17]Domingues S,Chopin A,Ehrlich SD,et al.The lactococcal abortive phage infection system AbiP prevents both phage DNA replication and temporal transcription switch[J].J Bacteriol,2004,186(3):713-721.
[18]Bouchard J,Dion E,Bissonnette F,et al.Characterization of the two-component abortive phage infection mechanism AbiT from Lactococcus lactis[J].J Bacteriol,2002,184(22):6325-6332.
[19]Dai G,Allison GE,Geller BL,et al.Molecular characterization of a new abortive infection system(AbiU)from Lactococcus lactis LL51-1[J].Appl Environ Microbiol,2001,67(11):5225-5232.
[20]Cluzel PJ,Chopin A,Ehrlich SD,et al.Phage abortive infection mechanism from Lactococcus lactis subsp.lactis,expression of which is mediated by an Iso-ISS1element[J].Appl Environ Microbiol,1991,57(12):3547-3551.
[21]O′Connor L,Tangney M,Fitzgerald GF.Expression,regulation,and mode of action of the AbiG abortive infection system of Lactococcus lactis subsp.cremoris UC653[J].Appl Environ Microbiol,1999,65(1):330-335.
[22]Durmaz E,Higgins DL,Klaenhammer TR.Molecular characterization of a second abortive phage resistance gene present in Lactococcus lactis subsp.lactis ME2[J].J Bacteriol,1992,174(2):7463-7469.
[23]Emond E,Holler BJ,Boucher I,et al.An abortive infection mechanism from Lactococcus lactis[J].Appl Environ Microbiol,1998,64(12):4748-4756.
[24]Bidnenko E,Ehrlich SD,Chopin MC.Lactococcus lactis phage operon coding for an endonuclease homologous to RuvC[J].Mol Microbiol,1998,28(4):823-834.
[25]Durmaz E,Klaenhammer TR.Abortive phage resistance mechanism AbiZ speeds the lysis clock to cause premature lysis of phage-infected Lactococcus lactis[J].J Bacteriol,2007,189(4):1417-1425.
[26]Paterson S,Vowill T,Buck A,et al.Antagonistic coevolution accelerates molecular evolution[J].Nature,2010,464(7286):275-278.

