羊驼皮肤基因转录因子及其结合位点分析
张俊珍,范瑞文,白俊明,董常生
(山西农业大学动物科技学院,山西 太谷030801)
皮肤包括表皮、真皮和皮下组织,是机体的保护性器官。皮肤内组织决定毛纤维生长、毛色形成、皮肤性质等功能。羊驼毛纤维柔软纤细且具有多种颜色,是纺织原料的“软黄金”,其遗传资源丰富[1],是对毛纤维进行生物学特性研究的资源库或活标本。羊驼皮肤韧性强,也是优质的皮革原料。这些特点都是由皮肤中表达的基因决定的,本课题组已构建了羊驼皮肤的cDNA文库,并进行了大规模测序(以库名为 ASCD提交到 NCBI)[2],为研究羊驼皮肤基因的表达提供了基础。基因的表达受到转录因子的调节,而其结合位点决定着转录的效率。
真核生物转录起始十分复杂,往往需要多种蛋白因子的协助,转录因子与RNA聚合酶Ⅱ形成转录起始复合物,共同参与转录起始的过程。转录因子的 结 合 位 点 (transcription factor binding site,TFBS)是转录因子调节基因表达时,与转录因子mRNA结合的区域,TFBS长度一般为6~12bp范围内,位于基因非编码区,大部分位于5′侧翼区,但有时也位于3′侧翼区或内含子区。转录因子通过特异性结合调控区域的DNA序列来调控基因转录过程。一个转录因子往往同时调控若干个基因,而它在不同基因上的结合位点具有一定的保守性,又不完全相同[3]。本文通过对获得的表达序列标签(expressed sequence tags,EST)进行分析,以发现可能的转录因子结合位点,并用实时定量PCR法对表达丰度高的转录因子及其结合位点进行不同毛色皮肤中的相对定量检测,为研究调控羊驼皮肤发育和毛色生物学的分子机制提供基础。
1 材料与方法
1.1 材料 本文所用材料为本室构建的白色青年羊驼皮肤cDNA文库,该文库经大规模测序,并提交到 NCBI的EST序列(库名为 ASCD)(http://www.ncbi.nlm.nih.gov/nucest?term=alpaca &dong)。
1.2 方法
1.2.1 序列信息处理 用CAP3软件(华大基因公司)将EST序列进行装配,再用BLASTN软件将装配好的序列与NCBI nr数据进行比对,并用GO软件(华大基因公司)对发现的已知序列进行注释和聚类。
1.2.2 对皮肤基因的转录因子结合位点进行比较基因组分析 应用oPOSSUM分析软件(http://www.cisreg.ca/cgibin/oPOSSUM/opossum),参照人皮肤中表达的同类已知基因,分析羊驼皮肤可能的转录因子结合位点 。脊椎动物转录因子结合位点的Fisher score<0.1时认为是有效的。
1.2.3 高表达的转录因子及其结合位点在不同毛色羊驼皮肤中的转录表达分析 取液氮保存的羊驼皮肤组织用 Trizol Reagent提取总 RNA,并用Nanodrop测定其浓度。分别取1μg总RNA用SYBR Prime ScriptTMRTPCR KIT(Perfect Real Time)进行反转录:10μL反应体系中含5×Prime ScriptTM Buffer 2μL,Prime ScriptTMRTEnzyme Mix10.5μL,Oligo dT Primer25pmol,随机引物50 pmol,Total RNA 1μL,DEPC水加至10μL。上述成分混匀后置于PCR仪器中,反应条件为37℃15 min,85℃5s。RT产物保存于-20℃备用。
取RT产物,进行实时荧光定量PCR反应:20 μL反应体系中含SYBR Premix ExTaqTM1×,上下游引物各0.2μmol/L(MITF F:5′-AAGTTGCAACGAGAACAGCA-3′,R:5′-CAGTTCTCAAGAGCGGGTTC-3′;CREB1F:5′-GTGTTACGTGGGGGAGAGAA-3′,R:5′-GGGCTAATGTGGCAATCTGT-3′),引物由北京六合华大基因科技股份有限公司合成),ROX Reference DyeⅡ(50×)1×,cDNA模板2μL(约500ng),ddH2O加至20 μL。设置95℃预变性30s后,95℃5s,55℃30s,72℃10s,43个循环。温度反应结束,由熔点曲线判定PCR反应的特异性,根据荧光曲线的CT值计算定量结果。内参基因18SrRNA(引物 F:5′-GAAGGGCACCACCAGGAGT-3′,R:5′-GAGACAAATCACTCCACCAA-3′)与目的基因在同一条件下不同管内扩增,每个样本设3次重复。
2 结果
2.1 羊驼皮肤中发现的转录因子 为了找出调节皮肤基因表达的可能的转录机制,本文选择530个基因进行分析,结果发现在羊驼皮肤中有8个转录因子,而且大部分为低丰度表达。但MITF的表达量稍高于其他7个转录因子(表1)。

表1 羊驼皮肤中表达的转录因子
2.2 羊驼皮肤中发现的转录因子结合位点 针对羊驼皮肤中表达的转录因子,应用oPOSSUM分析软件发现6个不同类型的转录因子结合位点。其中,CREB1属于BZIP类型,与靶基因结合的预测分值最高(表2)。
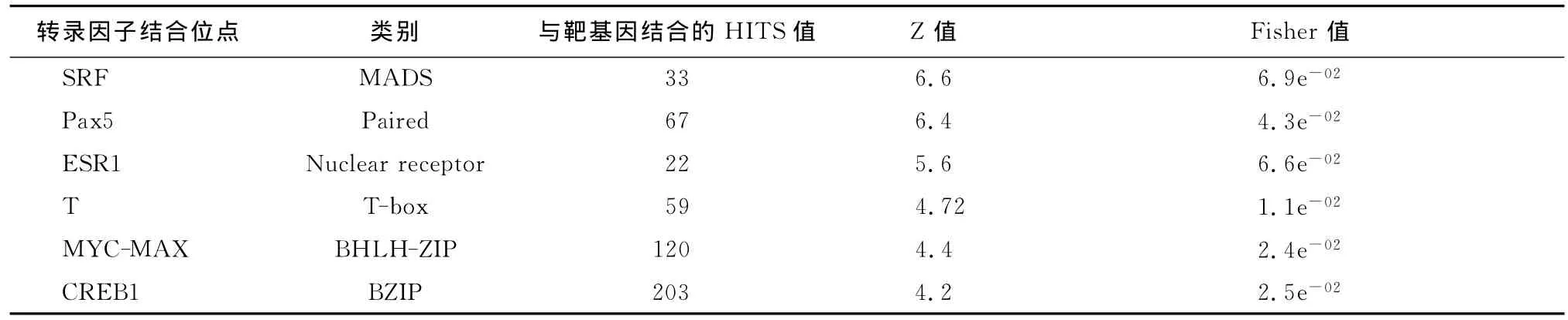
表2 羊驼皮肤中转录因子可能的结合位点
2.3 MITF与CREB1在不同毛色羊驼皮肤中的表达分析 实时定量PCR结果显示,MITF在棕黑色羊驼皮肤中的表达量显著高于其在白色羊驼皮肤中的表达量,呈5.938倍数关系;同时,CREB1在棕黑色羊驼皮肤中的表达量显著高于其在白色羊驼皮肤中的表达量,呈2.997倍数关系(见图1)。
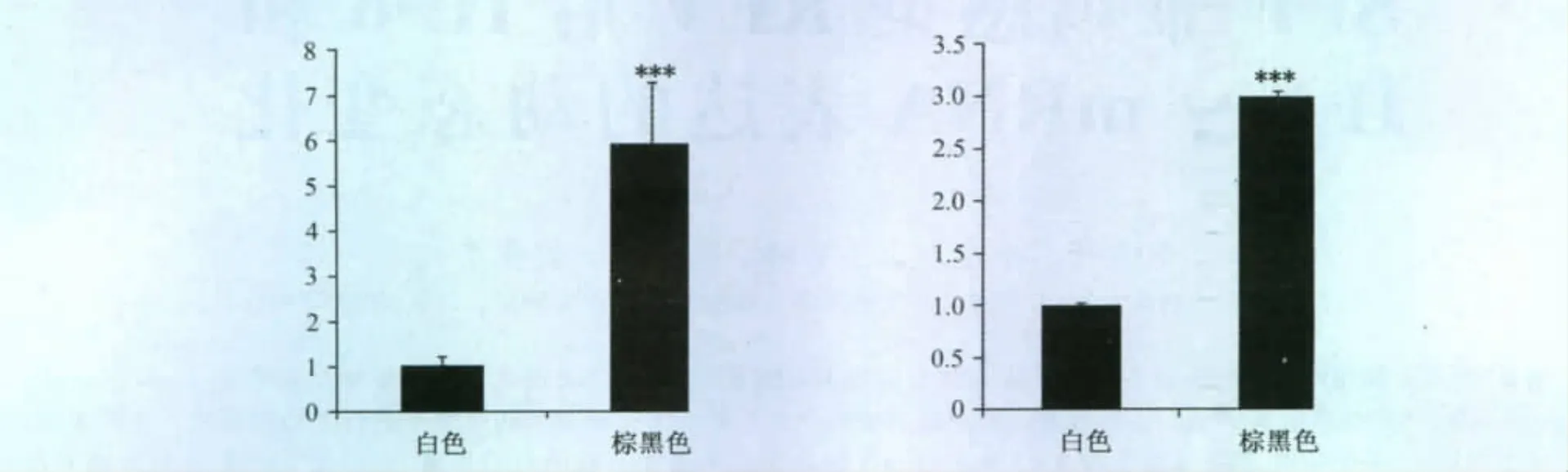
图1 MITF和CREB1基因在不同毛色羊驼皮肤中的实时荧光定量PCR结果
3 讨论和结论
根据转录因子的作用特点可分为两类:第一类为普遍转录因子它们与RNA聚合酶Ⅱ共同组成转录起始复合物,转录才能在正确的位置开始。第二类转录因子(Transcription Factor,TF)为组织细胞特异性转录因子,这类TF是在特异的组织细胞或是受到一些类固醇激素、生长因子或其他刺激后,开始表达某些特异蛋白质分子时需要的一类转录因子。从表1可以看出,羊驼皮肤中存在与皮肤发育和毛色调控的转录调节因子,关于这些因子有过相关的报道。如热激基因转录因子(heat shock transcription factor family member 5)是调控热激基因活性的信号传导链中的最终成员[4]。而热激蛋白是机体处在热胁迫和大量化学胁迫的环境中时产生一系列的应激反应,而致体内相关基因的活性迅速增加,并以分子伴侣的形式帮助相关蛋白折叠、稳定、组装、胞内运输和降解,使机体免受环境胁迫的影响,在热境中存活[5]。
Verdoni发现将角化细胞中的SRF失活可对鼠表皮的发育起转录调节作用[6]。虽然众所周知Pax 5可防止淋巴癌,但它在免疫系统外所起的作用也有了相关报道,与其相关的含有成对box的转录因子Pax 3在黑色素细胞的发育过程中起着重要的作用[7]。在羊驼皮肤中,尤其是发现了组织细胞特异的转录因子MITF,MITF是黑色素细胞内黑色素生成相关酶基因的特异的转录因子[8-9],属bHLH-LZ(basic helix-loop-helix-leucine-zipper)的成员[10],通过识别靶基因启动子区的E-box(CAYRTG)、M-box(TCAYRTG or CAYRTGA)[11]和T-box[12]的序列而使靶基因转录。黑色素细胞内调节毛色形成的转录因子MITF的结合位点之一,即CREB-1,CREB-1的靶基因预测分值最高,该位点在羊驼黑色素细胞中是一个重要的转录因子结合位点[[Mypaper]13]。而 且,相对定量PCR 结果显示,MITF和CREB1在不同毛色羊驼皮肤中的表达量存在显著差异。现已发现CREB通过结合cAMP反应元件在黑色素细胞中刺激转录,从而在cAMP信号路径中起调节作用[14]。在羊驼黑色素细胞中已发现了 MITF的靶基因 Melanocortin receptor-1(MC1R)、endothelin receptor B (EDNRB)、tyrosinase(TYR)、TYR-related protein 1 (TYRP1)和TYR-related protein 2(DCT)等毛色基因(图2),这对掌握羊驼皮肤中决定毛色以及其他生理功能的分子机理提供重要的基础,为后续研究其他哺乳动物皮肤生物学以及色素形成的调控提供了资源。
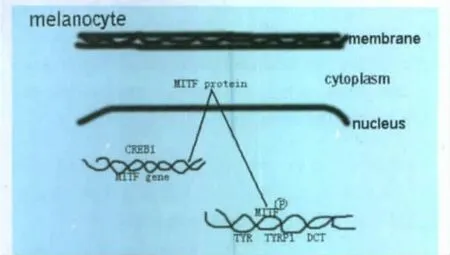
图2 MITF转录因子与转录结合位点和靶基因间的关系
[1] Cecchi T,Cozzali C,Passamonti P,etal.Melanins and Melanosomes From Llama(Lama glama L)[J].Pigment Cell Research,2004,17(3):307-311.
[2] 范瑞文,董常生,游蓉丽,等.白色青年羊驼皮肤的基因表达分析[J].中国农业科学,2009,42(1):290-298.
[3] Zhang Z L,Gerstein M.Of mice and men:phylogenetic footprinting aids the discovery of regulatory elements[J].BMC Biology,2003,2:11.
[4] Morimoio R I.Regulation of the heat shock transcriptional response:cross talk between a family of heat shock factors,molecular chaperones,and negative regulators[J].Genes Dev,1998,12:3788-3796.
[5] Hartl F U,Hayer-Hartl M.Molecular chaperones in the cytosol:from nascent chain to folded protein[J].Science,2002,295:1852-1858.
[6] Verdoni A,Ikeda S and Ikeda A.Serum response factor is essential for the proper development of skin epithelium[J].Mammalian Genome,2010,21:64-76.
[7] Wang Q,Fang W,Krupinski J,etal.Pax genes in embryogenesis and oncogenesis[J].Journal of Cellular and Molecular Medicine,2008,12:2281-2294.
[8] Tachibana M.MITF:A stream flowing for pigment cells[J].Pigment cell Research,2000,13(4):230-240.
[9] Yasumoto K,Yokoyama K,Takahashi K,etal.Functional analysis of microphthalmia-associated transcription factor in pigment cell-specific transcription of the human tyrosinase family genes[J].Journal of Biological Chemistry,1997;272(1):503-509.
[10]Hirofumi Aoki,Osamu Moro.Involvement of microphthalmiaassociated transcription factor(MITF)in expression of human melanocortin-1receptor(MC1R)[J].Life Sciences,2002,71:2171-2179.
[11]Levy C,Khaled M,Fisher D E.MITF:master regulator of melanocyte development and melanoma oncogene[J].Trends Mol Med,2006,12(9):406-414.
[12]Suzanne Carreira,Baoguo Liu and Colin R.Goding.The Gene Encoding the T-box Factor Tbx2Is a Target for the Microphthalmia-associated Transcription Factor in Melanocytes[J].The Journal of Biological Chemistry,2000,275:21920-21927.
[13]Ping Wan,Yongqing Hu,Li He.Regulation of melanocyte pivotal transcription factor MITF by some other transcription factors[J].Mol Cell Biochem,2011,354(1-2):241-246.
[14]Kim J,Jia L,Stallcup M R,etal.The role of protein kinase A pathway and cAMP responsive element-binding protein in androgen receptor-mediated transcription at the prostate-specific antigen locus[J].J Mol Endocrinol(England),2005,34(1):107-108.

