优雅蝈螽卵巢Piwi蛋白亚家族成员Giwi cDNA序列全长的克隆与生物信息学分析
刘静 周志军 常岩林
生殖干细胞(germline stem cells,GSC)是精巢和卵巢中配子发生的中枢,GSC的自我更新与分化在动物体内保持平衡,从而使GSC和成熟配子的数量保持稳定[1]。关于GSC的研究在模式生物果蝇中最为深入。Piwi基因广泛存在于各种多细胞生物类群的干细胞之中。迄今为止,尚没有关于其在半变态昆虫中研究的报道。Lin和Spradling[2]首先在果蝇卵巢中发现piwi基因对GSC的分裂有调控作用。随后,Cox等[3,4]证明piwi基因在果蝇雌性和雄性GSC中以自主方式表达并促进GSC分裂。Piwi蛋白通过与piRNA(Piwi-interacting RNA)结合形成Piwi-piRNA复合物引起基因沉默,进而对生殖细胞进行调控[5-7]。目前在多种生物(如人、小鼠、斑马鱼、线虫)中都有果蝇piwi基因的同源基因发现,piwi基因的突变可导致生殖细胞发育缺陷[4,8-12]。
虽然近年来已对多种昆虫的piwi同源基因进行了研究[13,14],但昆虫种类繁多,生殖方式多样,生殖生理差异较大。已研究的果蝇、蜜蜂、家蚕均为完全变态类昆虫,其卵巢管为滋养式(meroistic type),piwi基因在生殖干细胞龛(stem cell niche)的端丝和冠细胞中均有表达[4],piwi基因突变导致GSCs过早丢失[15]。直翅目为不完全变态类昆虫,其卵巢管是无滋式(panoistic type),卵子发生过程中没有滋养细胞提供营养和信号[16]。无滋式的直翅目昆虫卵巢是否存在调控GSC发育的因子,是否存在Piwi蛋白同源物,GSC发育的调控模式等尚无报道。
优雅蝈螽(Gampsocleis gratiosa)隶属于昆虫纲(Insecta)直翅目(Orthopter)螽斯总科(Tettigonoidea),是直翅目重要的模式昆虫。本研究利用piwi基因主要表达于生殖细胞的特点,通过转录组测序(RNASeq)、RACE-PCR及相关生物信息学技术,对优雅蝈螽雌性成虫卵巢中piwi同源基因及预测的蛋白序列进行分析,为厘清无滋式卵巢管的原卵区(germarium)内部细胞的组成及GSC龛模式在不完全变态昆虫是否适用等问题提供理论依据。
1 材料与方法
1.1 材料
供试用的优雅蝈螽采自河北省顺平县(38 83' N,115 13' E),将捕获的末龄若虫带回实验室内饲养至成虫。
1.2 方法
1.2.1 总RNA提取 选取发育良好的优雅蝈螽雌性成体,快速解剖取出卵巢置于含有RNAiso Plus(TaKaRa)的匀浆器中,充分研磨,进行总RNA提取。具体操作依照RNAiso Plus试剂盒说明书进行,提取获得的总RNA溶解于RNase-free(TIANGEN)无菌水中。
1.2.2 引物设计 通过对本实验室前期测得的优雅蝈螽转录组数据进行搜索,筛选出与昆虫piwi基因同源的序列片段。由于piwi基因序列全长接近3 kb,因此将中间段序列分为A、B、C 3个互相重叠的片段进行引物设计。利用Primer Premier 5.0[17]软件,共设计了3对中间段序列扩增引物(GAF/GAR,GBF/GBR,GCF/GCR)和4条基因两端RACE引物(G3F1,G3F2,G5F1,G5F2)(表 1),并交由上海铂尚生物技术有限公司合成。
1.2.3 cDNA第一条链合成 用Oligo dT引物对总RNA(约1μg)进行反转录,具体操作依照Prime-Script®RT-PCR Kit(TaKaRa)试剂盒说明书进行。
1.2.4 piwi基因中间段序列RT-PCR 以反转录得到的cDNA为模板,分别用GAF/GAR,GBF/GBR,GCF/GCR引物对进行扩增。扩增程序:94℃预变性3min;94℃变性30s,55℃退火30s,72℃延伸90s,30个循环;72℃延伸10min。

表1 RACE-PCR引物
1.2.5 piwi基因3'/5'端RACE-PCR反应 3'RACE依照3'-Full RACE Core Set Ver.2.0试剂盒说明书对总RNA(约1μg)进行反转录。反应程序:42℃ 60min,70℃ 15min。以反转录得到的cDNA为模板,用G3F1和3' Outer Primer及G3F2和3' Inner Primer分别进行Outer PCR扩增和Inner PCR扩增。反应程序:94℃预变性3min;94℃变性30s,55℃退火30s,72℃延伸1min(Outer PCR 20个循环/Inner PCR 30个循环);72℃延伸10min。5' RACE依照5'-Full RACE Kit试剂盒说明对总RNA(约2μg)进行去磷酸化、去帽子、5' RACE Adaptor连接及反转录反应。反应程序:30℃ 10min,42℃ 1h,70℃15min。以反转录得到的cDNA为模板,用5' Outer Primer和G5R1及5' Inner Primer和G5R2分别进行Outer PCR和Inner PCR扩增(以上Outer Primer和Inner Primer均由TaKaRa公司试剂盒提供)。反应程序同3' RACE。
1.2.6 克隆及测序 RT-PCR和3'/5'端RACE-PCR产物经1%琼脂糖凝胶电泳检测,切取目的条带经DNA片段凝胶回收试剂盒(北京三博远志)纯化、回收后,连接到pMD19-T载体(TaKaRa)上,并转化到大肠杆菌DH5α中。转化后生成的菌落,通过蓝白斑筛选,随机选取3个阳性单克隆,交由上海铂尚生物技术有限公司进行双向测序。
1.2.7 生物信息学及系统进化分析 测序结果经Lasergene[18]软件校对、拼接获得cDNA序列全长。使用NCBI中的ORFfinder程序进行开放阅读框(ORF)及基因两端非编码区预测并推导出相应的氨基酸序列。运用BioEdit[19]进行多重序列比对,寻找PIWI结构域活性催化模体。蛋白质的理论分子量、等电点及其它基本性质的分析采用Prot Param[20]进行,二、三级结构分别通过PROSITE[21]和Phyre2[22]软件进行预测。采用 MEGA5[23]软件基于邻接法(Neighbor-Joining,NJ)构建系统进化树。
2 结果
2.1 cDNA全长序列分析
将测序所得序列片段进行剪接,得到全长3 462bp的cDNA序列,GenBank序列登录号为JX998175。该序列包含2 742bp的开放阅读框,共编码913个氨基酸残基,5'非编码区(5' UTR)111bp和3'非编码区(3' UTR)609bp。预测的理论蛋白分子量为102.7kD,等电点为9.55。Giwi蛋白具备Piwi蛋白亚家族全部特征,C端的保守PIWI结构域,中部的保守PAZ结构域和N端可变结构域(图 1)。
2.2 序列比对及系统进化分析
从NCBI数据库中选取具有代表性的脊椎动物和截止2012年10月10日登录的全部昆虫Piwi蛋白亚家族序列,包括:斑马鱼Danio rerio(NP_89918-1.1、ABM46842.2)、 人 Homo sapiens(AAC97371.2、AAK92281.1、BAC81341.1、BAC81343.1、BAC813-42.1)、 小 鼠 Musmusculus(BAA93706.1、ABM691-81.1、AAN75583.1)、大鼠 Rattus norvegicus(NP_00-1102323.1)、埃及伊蚊 Aedes aegypti(XP_0016576-26.1、XP_001652831.1、XP_001652945.1、XP_0016-53082.1、XP_001663408.1、XP_001663409.1、XP_-001663870.1)、切叶蚁 Acromyrmex echinatior(EGI6-4222.1)、 意 大 利 蜜 蜂 Apismellifera(ACV84372.1、NP_001159378.1)、家蚕 Bombyxmori(NP_0010980-67.2、NP_001098066.2、BAF73718.2)、弓背蚁Camponotus floridanus(EFN67778.1)、致倦库蚊 Culex quinquefasciatus(XP_001867947.1、XP_001844024.1、XP_001844067.1、XP_001844068.1、XP_0018470-30.1、XP_001860347.1、XP_001862491.1、XP_001-867946.1)、黑腹果蝇 Drosophilamelanogaster(ABO-26294.1、ABO27430.1、AAD38655.1、AAD08705.1、NP_476875.1)、大红斑蝶 Danaus plexippus(EHJ69-790.1、EHJ75824.1)、印度跳蚁Harpegnathos saltator(EFN77932.1、EFN83189.1)、褐飞虱Nilaparvata lugens(AEI25513.1)、人虱 Pediculushumanus corporis(XP_002431988.1)、 赤 拟 谷 盗 Tribolium castaneum(EFA-02921.1、EFA07425.1)和优雅蝈螽Gampsocleis gratiosa共47条。运用BioEdit进行多重序列比对分析,得出PIWI结构域亦具有类似RNA酶H活性中心的DDH(Asp/Asp/His)三联催化模体(图1),3D模型中可见PAZ结构域围成的袋状结构,DDH催化位点深埋袋中(图2)。

图1 优雅蝈螽giwi基因 cDNA核苷酸序列和氨基酸序列
用黑腹果蝇Drosophilamelanogaster Ago1(NP_725341.1)蛋白序列作为外群,采用NJ法构建分子进化树。结果(图3)显示,Piwi蛋白亚家族聚类形成两个大的分枝A和B。分枝A,由28条昆虫Piwi和Aubergine蛋白组成,虽然又可进一步划分为两个较小的分枝I和Ⅱ,但是这两个分枝与Piwi和Aubergine蛋白并不对应。例如:果蝇的Piwi和Aubergine蛋白都位于分枝I,而家蚕的Piwi和Aubergine蛋白,及本研究所获得Giwi蛋白均位于分枝Ⅱ。分枝B,由7条昆虫的Ago3、2条昆虫的Piwi蛋白及11条脊椎动物的Piwi蛋白组成,可进一步划分为3个较小的分枝III、IV和V。分枝III和IV分别由8条和3条脊椎动物的Piwi蛋白构成;分枝V则由昆虫的Ago3和2条昆虫Piwi蛋白构成,且与分枝IV形成姊妹群。
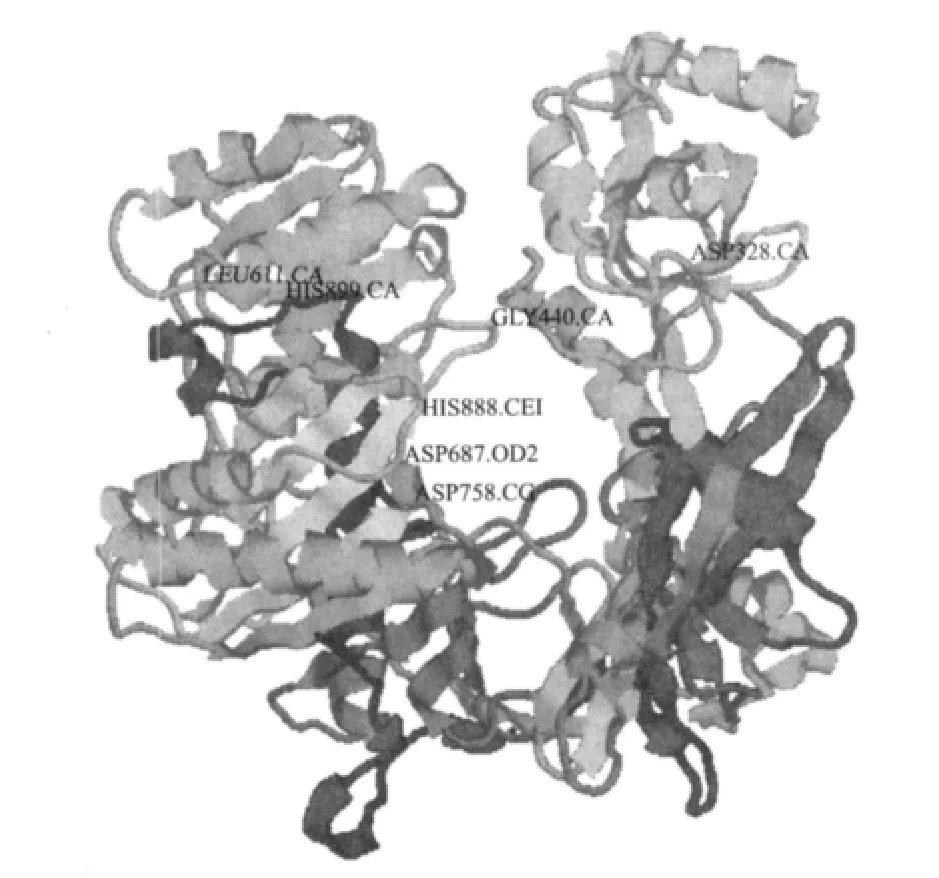
图2 优雅蝈螽Giwi预测蛋白的3D结构
3 讨论
本研究预测所得Giwi蛋白为Piwi蛋白亚家族成员。该家族蛋白进化保守,均含有C端的PIWI结构域,中部的PAZ结构域和N端的可变结构域[24]。PAZ结构域是小RNA结合区域,PIWI结构域具有类似RNA酶H的DDH三联催化模体,是行使切割功能的活性中心,与piRNAs的生成有密切关系[24,25]。通常认为含有活性DDH三联催化模体的Piwi蛋白具有剪切活性或类剪切活性[24]。Giwi蛋白亦含有DDH三联结构,暗示该蛋白具有切割活性。
Piwi蛋白亚家族偏好表达于生殖系细胞,对生殖干细胞的自我更新和生殖系发育起着重要的调控作用[2,26]。该亚家族根据果蝇Piwi蛋白成员被分为三类 :Piwi、Aubergine和 Ago3[27]。然而,本研究系统发育分析发现,昆虫的Piwi和Aubergine在进化树上位于相同的枝上,果蝇和家蚕的Piwi和Aubergine蛋白都没有按照物种差异区分开来。有报道推测piwi和aubergine基因可能来源于远古基因的重复事件[24]。脊椎动物的Piwi蛋白和昆虫的Ago3蛋白在进化树上亦没有明显的分开,Ⅴ号进化枝上的2个昆虫Piwi蛋白指出该蛋白亚家族在命名过程中可能存在问题,以上问题有待进一步的研究来加以阐明。
随着分子生物学技术的发展,关于未知新基因的cDNA全长的获得方法日新月异,包括基于构建cDNA文库法、基于通用简并引物的RT-PCR法和计算机基因克隆法,又称硅片克隆、电子克隆[28]。构建cDNA文库工作量较大,获得的序列片段长度有限,且文库覆盖范围有限[29]。通用简并引物PCR法是根据数据库中已有物种序列设计简并引物进行扩增,方便简单,但是由于个体内通常存在同一基因家族的多个成员,而这些简并引物在基因上的位置又经常位于该基因家族的结构域,因此,所获得的测序结果往往是该基因家族的其他成员[30]。电子克隆依靠电脑和网络资源,速度快、成本低是其最大的优点,但电子克隆可依赖的EST序列有限,而且这些序列由于测序丰度的限制,有些序列甚至仅仅是单次测序结果,其精确性有待验证[31]。本研究则在基于454高通量测序获得的转录组序列的基础上,筛选感兴趣的目的功能基因,通过RT-PCR和RACE末端扩增技术克隆获得该基因的cDNA序列全长。随着高通量测序平台的市场化,转录组测序成本的进一步降低,转录组测序的研究方法在分子生物学相关研究领域的应用会更加广泛。
4 结论
本研究以半变态昆虫优雅蝈螽为材料,通过对二代测序获得的转录组数据进行搜索,成功挑选出piwi基因的同源体并对其进行研究,得到giwi cDNA序列全长。序列分析预测所得Giwi蛋白具备Piwi蛋白亚家族全部特征。同源比对分析得到PIWI结构域具有类似RNA酶H活性中心的DDH三联催化模体。系统发育分析指出昆虫的Piwi和Aubergine可能源于远古基因的重复事件,Piwi蛋白亚家族在命名过程中可能存在问题,以上问题有待进一步研究。

图3 优雅蝈螽及其他Piwi亚家族蛋白的系统发育树
[1] 冯振月, 潘敏慧, 鲁成.果蝇生殖腺干细胞和它们的微环境[J].细胞生物学杂志, 2006, 28:169-172.
[2] Lin H, Spradling AC.A novel group of pumiliomutations affects the asymmetric division of germline stem cells in the Drosophila ovary[J].Development, 1997, 124(12):2463-2476.
[3] Cox DN, Chao A, Baker J, et al.A novel class of evolutionarily conserved genes defined by piwi are essential for stem cell selfrenewal [J].Genes Dev, 1998, 12(23):3715-3727.
[4] Cox DN, Chao A, Lin H.piwi encodes a nucleoplasmic factor whose activitymodulates the number and division rate of germline stem cells [J].Development, 2000, 127(3):503-514.
[5] Lu J, Clark AG.Population dynamics of PIWI-interacting RNAs(piRNAs)and their targets in Drosophila [J].Genome Res, 2010,20(2):212-227.
[6] Frost RJ, Hamra FK, Richardson JA, et al.MOV10L1 is necessary for protection of spermatocytes against retrotransposons by Piwiinteracting RNAs [J].Proc Natl Acad Sci USA, 2010, 107(26):11847-11852.
[7] Siomi MC, Mannen T, Siomi H.How does the Royal Family of Tudor rule the Piwi-interacting RNA pathway? [J].Genes Dev, 2010, 24(7):636-646.
[8] Kuramochi-Miyagawa S, Kimura T, Yomogida K, et al.Twomouse piwi-related genes:miwi andmili [J].Mech Dev, 2001, 108(1-2):121-133.
[9] Lau NC, Lim LP, Weinstein EG, et al.An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans [J].Science, 2001, 294(5543):858-862.
[10] Qiao D, Zeeman AM, Deng W, et al.Molecular characterization ofhiwi, ahumanmember of the piwi gene family whose overexpression is correlated to seminomas [J].Oncogene, 2002, 21(25):3988-3999.
[11] Tan CH, Lee TC, Weeraratne SD, et al.Ziwi, the zebrafishhomologue of the Drosophila piwi:Co-localization with vasa at the embryonic genital ridge and gonad-specific expression in the adults[J].Mech Dev, 2002, 119(Suppl 1):221-224.
[12] Houwing S, Kamminga LM, Berezikov E, et al.A role for Piwi and piRNAs in germ cellmaintenance and transposon silencing in zebrafish [J].Cell, 2007, 129(1):69-82.
[13] Kawaoka S, Minami K, Katsuma S, et al.Developmentally synchronized expression of two Bombyxmori Piwi subfamily genes,SIWI and BmAGO3 in germ-line cells [J].Biochem Biophys Res Commun, 2008, 367(4):755-760.
[14] Liao Z, Jia Q, Li F, et al.Identification of two PIWI genes and their expression profile inhoneybee, Apismellifera [J].Arch Insect Biochem Physiol, 2010, 74(2):91-102.
[15] Li L, Xie T.Stem cell niche:structure and function [J].Annu Rev Cell Dev Biol, 2005, 21:605-631.
[16] Nation JL.Insect physiology and biochemistry [M].Boca Raton:CRC Press, 2002:1-544.
[17] Singh VK, Mangalam AK, Dwivedi S, et al.Primer premier:program for design of degenerate primers from a protein sequence[J].BioTechniques, 1998, 24(2):318-319.
[18] Burland TG.DNASTAR's Lasergene sequence analysis software[J].Methods Mol Biol, 2000, 132:71-91.
[19] Thomas AH.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT [J].Nucleic Acids Symposoum Series, 1999(41):95-98.
[20] Gasteiger E, Hoogland C, Gattiker A, et al.Protein identification and analysis tools on the ExPASy server[M]//Walker JM.The Proteomics Protocols Handbook.Totowa:Humana Press, 2005:571-607.
[21] Castro Ed, Sigrist CJA, Gattiker A, et al.ScanProsite:Detection of PROSITE signaturematches and ProRule-associated functional and structural residues in proteins [J].Nucleic Acids Research, 2006,34:362-365.
[22] Kelley LA, Sternberg MJE.Protein structure prediction on the Web:a case study using the Phyre server [J].Nature Protocols 2009, 4(3):363-371.
[23] Tamura K, Peterson D, Peterson N, et al.MEGA5:molecular evolutionary genetics analysis usingmaximum likelihood,evolutionary distance, andmaximum parsimonymethods [J].Mol Biol Evol, 2011, 28(10):2731-2739.
[24] Zhou X, Liao Z, Jia Q, et al.Identification and characterization of Piwi subfamily in insects [J].Biochem Biophys Res Commun,2007, 362(1):126-131.
[25] 李超, 杜志游, 陈集双.解读 AGO蛋白结构及其功能[J].中国生物化学与分子生物学报, 2009, 25(11):969-976.
[26] Thomson T, Lin H.The biogenesis and function of PIWI proteins and piRNAs:progress and prospect [J].Annu Rev Cell Dev Biol, 2009, 25:355-376.
[27] Samji T.PIWI, piRNAs, and Germline Stem Cells:What’s the link? [J].Yale Journal of Biology And Medicine, 2009, 82(2009):121-124.
[28] 樊红, 李钰.克隆新基因cDNA全长的策略和方法[J].国外医学遗传学分册, 2002, 25(1):11-13.
[29] 晏慧君, 黄兴奇, 程在全.cDNA文库构建策略及其分析研究进展[J].云南农业大学学报, 2006, 21(1):1-6.
[30] 曹存巍, 刘伟, 李若瑜.简并PCR结合RACE技术克隆马尔尼菲青霉素未知基因[J].中国皮肤性病学杂志, 2007, 21(11):660-662.
[31] 胡皝, 萧浪涛.生物信息学在新基因全长cDNA电子克隆中的应用[J].生物技术通报, 2007(4):93-96.

