决明查尔酮合成酶的同源模建和分子模拟对接
李云双,周 虹,钟德馨,方袁梦梦,王万军,廖 海,周嘉裕
西南交通大学生命科学与工程学院,成都 610031
类黄酮物质是一类重要的植物次级代谢产物,在植物花色形成、植物育种、抵抗紫外线辐射、防止病原微生物侵染中都发挥了重要作用。并且,类黄酮物质还具有预防心血管疾病、抗癌、调节免疫、抗衰老、抗菌、抗炎等多种生物活性,是一些药用植物的主要活性成分[1]。查尔酮合成酶(Chalcone synthase,CHS,EC 2.3.1.74)是植物中类黄酮物质合成途径的关键酶,是调控植物类黄酮物质合成的关键基因之一。CHS催化该途径的第一步,即丙二酰单酰CoA和香豆酰CoA缩合形成具有C15骨架的黄酮类化合物-查尔酮。此中间物的异构化和功能基团的进一步取代都能导致黄酮、异黄酮和花色素苷的合成。目前,已从多种植物中克隆了CHS基因,包括兰花、矮牵牛、拟南芥、金鱼草和欧洲赤松等[2]。但是,迄今为止仅有苜蓿(Medicago sativa)中的CHS有X-ray晶体结构报道,并用于CHS催化机制的研究[3]。
决明(Senna tora)为豆科草本植物,其干燥成熟种子被称为决明子。决明子是一种常用的中药,有效成分主要是蒽醌类及黄酮类化合物[4]。鉴于CHS在植物生理生化活动中的重要性,有关CHS结构、催化机理及分子工程的研究已成为植物生理生化及分子生物学研究的热点之一。本课题组在前期研究中已获得决明CHS基因的全长cDNA序列[5]。在此基础上,本文利用已有苜蓿CHS晶体结构为模板进行决明CHS三维结构的同源建模,并对获得的三维结构模型进行动力学模拟和合理性验证,再将该模型与香豆酰CoA、丙二酸单酰CoA进行分子对接,为利用此类CHS三维模型研究其催化机理和分子工程改造奠定基础。
1 材料和方法
1.1 序列比对和同源模建
决明CHS的一级序列(编号:ACB78187.1)来源于NCBI数据库。在美国国立生物技术信息中心(NCBI)的PDB数据库(http://www.rcsb.org/pdb)中用protein blast(http://blast.ncbi.nlm.nih.gov/Blast)方法搜寻同源性和一致性较高且已知晶体结构的蛋白为模板。利用Discovery StudioTM(DS)2.5(Accelrys,San Diego,USA;http://accelrys.com/products/discovery-studio/)的 Align Multiple Sequence模块对决明CHS进行二级结构预测并与苜蓿CHS进行序列比对。然后将目标与模板序列提交到自动建模服务器SWISS-MODEL(http://swissmodel.expasy.org),得到初始模型。利用DS的Proteins Superimposing模块叠合决明CHS与苜蓿CHS的三维结构。
1.2 初始模型的能量优化与修正
采用分子动力学计算能量最小化的方法来优化不利的非共价接触,保持正确的肽键构型及能量最低状态。本文选用Discovery StudioTM(DS)2.5的能量最小化(EM)和分子动力学模拟(DM)模块对初始模型进行能量优化,力场为CHARMm力场。能量优化的步骤为:(1)Minimization_1:考虑到溶剂化效应,首先将初始模型放入正立方体的TIP3P水模型中溶解,对整个体系采用1000步最陡下降法(Steepest Descent)优化;(2)Minimization_2:应用5000步共轭梯度法(Conjugate Gradient)进一步优化;(3)Heating:从50 K到300 K梯度进行5 000步的动态模拟;(4)Equilibration:在热力学温度(T=300 K)进行了1000ps的分子动力学计算,模拟步长为1 ps,每隔500fs收集1个分子构象保存到结果文件中,选定能量最低的构象为最终的建模结果。
1.3 模型验证
利用在线软件PROCHECK验证立体化学性质的合理性;利用ERRAT(http://nihserver.mbi.ucla.edu/SAVES/)评估模型的构建精炼程度;利用Profile_3D对模建结构中氨基酸序列的相容性进行初步评价打分。
1.4 分子对接
丙二酸单酰辅酶A(Compound ID:10663;MF:C24H38N7O19P3S)和对-香豆酰辅酶A(Compound ID:5280329;MF:C30H42N7O18P3S)的结构来源于NCBI Pubchem(http://Pubchem.ncbi.nlm.nih.gov/)数据库,并进行三维结构转化备用。
利用Molegro Virtual Docker(MVD)(http//:www.molegro.com/products.php)软件的Detect Cavities模块寻找配体结合口袋。在相同的对接条件下,利用MVD的Docking Wizard将配体(对-香豆酰辅酶A和丙二酸单酰辅酶A)与决明CHS进行对接。根据构象聚类分析的结果,选取最低能量构象作为最终结合构象,生成决明CHS与配体的复合物结构,并进行能量最小化(EM)和动力学模拟(MD),用于底物结合位点和催化机制的分析。
2 结果和讨论
2.1 序列比对和同源模建
PDB数据库搜索结果表明,苜蓿CHS的氨基酸序列与决明CHS的氨基酸序列同源性最高,一致性(Identity)为88%,相似性(Positives)为95%,因此选择苜蓿CHS晶体(PDB编号:1CHW_B)作为同源建模的模板。对决明CHS进行二级结构预测并与苜蓿CHS进行序列比对(图1),预测结果表明决明CHS中有13个α螺旋,占32.82%,15个β折叠,占19.23%,无规则卷曲占47.95%。将决明CHS与苜蓿CHS序列提交到自动建模服务器SWISS-MODEL,获得了决明CHS的初始模型。初始模型与模板叠合后的root-mean-square deviation(RMSD)值为0.105Å,表明决明CHS的三维结构与苜蓿CHS的三维结构十分相似(图2)。

图1 决明CHS与苜蓿CHS的序列比对Fig.1 Sequence alignment of Chalcone synthase from S.tora and M.sativa using proteins aligning module of DS,the secondary structure was demonstrated with“α”for α-helices,“β”for β-sheets and“c”for random coils

图2 模型决明CHS与模板苜蓿CHS的三维结构叠合Fig.2 3D superimposition structure of Chalcone synthase from S.tora and M.sativa,the yellow ribbon represented the model and the red one represented the template
2.2 同源模型的验证
利用PROCHECK分析优化后模型的立体化学合理性,模型的主链和侧链立体化学构型均较为合理,并且从Ramachandran图(图3)中可见该模型的主链结构合理:蛋白的φ、ψ二面角90.6%在核心区域,9.1%在允许区域,绝大多数φ-ψ角均在正常范围内,没有落在不允许区域内的残基。用ERRAT对模型进行评估,得到其综合质量因子为93.684,表面模型的精炼程度符合分析要求。用Profile-3D对模建结构中氨基酸序列的相容性进行评价(图4),结果显示大约95.64%的残基的得分均大于0.2,表面该模型的侧链环境是合理的。以上分析结果表明,决明CHS的三维结构模型能够用于对接研究。

图3 决明CHS建模结构的Ramachandran plotFig.3 Ramachandran plot of the modeled structure of Chalcone synthase from S.tora
2.3 对接研究与活性位点
为了进一步确定模型的稳定性,分别将决明CHS,决明CHS-丙二酰辅酶A复合物和决明CHS-(对-香豆酰辅酶A)复合物进行动力学模拟,计算得到模拟过程中所有构象与初始构象的RMSD值。如图5所示,在100 ps的平衡模拟的轨迹中,决明CHS-丙二酰辅酶A复合物和CHS-(对-香豆酰辅酶A)复合物的RMSD值(0.442Å与0.472Å)均低于决明CHS的RMSD值(0.852Å)。该结果表明当CHS-配体复合物形成后,由于配体与酶分子间存在大量的氢键和范德华力,使得CHS的三维结构更趋于稳定。

图4 决明CHS建模结构的Profile-3D图Fig.4 Profile-3D score of the modeled structure of Chalcone synthase from S.tora
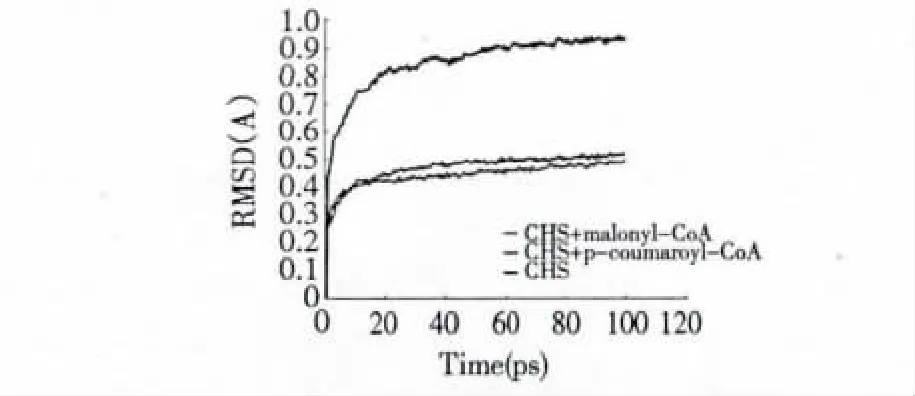
图5 动力学模拟过程中所有构象与初始构象的均方根偏差值(RMSD)Fig.5 The root-mean-square deviations(RMSD)calculated for backbone atoms of ligands-bound complex and for backbone atoms of SMT without ligands during MD simulations
与苜蓿CHS类似,决明CHS的三维结构中含有两个重要的结构域:对-香豆酰辅酶A结合域与丙二酰辅酶A结合域[3]。对-香豆酰辅酶A结合域主要由 133Ser、164Cys、193Ile、194Thr、197Thr、210Val、254Ile、263Leu、265Phe、269Lys、305Gly、338 Ser 和374Gly等残基构成。对-香豆酰辅酶A与决明CHS对接后,其 MolDockScore为-188.745,结合自由能为-242.575KJ/mol。对-香豆酰辅酶 A结合域中133Ser、164 Cys、194Thr、197Thr、269Lys、305Gly、338 Ser与374Gly等残基与对-香豆酰辅酶A形成氢键,剩余的残基(193Ile、210Val、254Ile、263Leu、265Phe)与对-香豆酰辅酶A通过范德华力相互作用,从而稳固对-香豆酰辅酶A在复合物的空间位置(图6)。对-香豆酰辅酶A分子中的泛硫醇部分伸入到CHS酶中,从而定位了最后的已连接硫酯的底物结合于残基Cys 164(催化残基)上。Cys 164的巯基硫原子与对-香豆酰辅酶A分子中香豆酰基氧原子形成氢键,距离为 3.412 Å。

图6 对-香豆酰辅酶A与决明查尔酮合酶的结合位点Fig.6 Binding sites of p-coumaroyl-CoA with Chalcone synthase from S.tora
丙二酰辅酶 A结合域主要由 Lys55、Met59、Lys62、Ser133、Cys164、Thr197、Leu206、Asp207、Val210、Phe215、Ile254、Phe265、Val271、Gly305、Gly306和Ala308等残基构成。丙二酰辅酶A与决明CHS对接后,其 MolDockScore为-180.226,结合自由能为-220.396 KJ/mol。丙二酰辅酶A结合域中Met59、Ser133、Asp207与Gly305等残基与丙二酰辅酶 A形成氢键,剩余的残基(Lys55、Lys62、Cys164、Thr197、Leu206、Val210、Phe215、Ile254、Phe265、Val271、Gly306、Ala308)与丙二酰辅酶 A 通过范德华力相互作用,从而稳固丙二酰辅酶A在复合物的空间位置(图7)。

图7 丙二酰辅酶A与决明查尔酮合酶的结合位点Fig.7 Binding sites of malonyl-CoA with Chalcone synthase from S.tora
2.5 催化机制的推测
如上所示,底物与查尔酮合酶特异性结合的立体化学特征阐明了 Cys164、Phe215、Phe265、His303、Asn336在反应机制中的作用[5]。Phe215和Phe265主要负责底物丙二酸单酰辅酶A结合的特定方向,从而控制聚酮中间物的延伸。Cys164的巯基氢原子与 His303咪唑环上的 N原子相距3.493 Å,Cys164、His303与活性中心的H2O能够形成电子传递体系。首先,水分子转移1个电子至His303的咪唑环上,然后该电子再转移至Cys164的巯基上使后者为负电子基团。随后该负电子基团进攻对-香豆酰辅酶A的香豆酰基,形成1分子CHS-对-香豆酰基的中间产物,而辅酶A从CHS中分离出来[3](图8)。
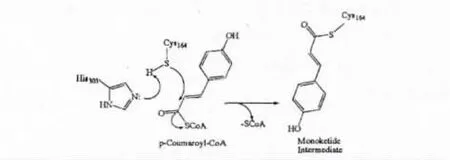
图8 对-香豆酰辅酶A与决明CHS结合产生单烯酮中间产物的假设机制Fig.8 Hypothetical mechanism for p-coumaroyl-CoA binding with CHS from S.tora with the generation of a monoketide intermediate
3 结论
查尔酮合成酶(CHS)是植物中类黄酮生物合成途径的关键酶,是调控植物类黄酮物质合成的关键基因之一,对植物具有非常重要的意义。迄今为止未有CHS与对-香豆酰CoA复合物的结晶结构报道,使得人们无法进一步认识CHS与对-香豆酰辅酶A的结合与催化机制。因此,本研究利用同源建模的方法构建决明CHS的三维模型,PROCHECK与Profile-3D检验发现模型中几乎所有氨基酸残基的二面角与侧链环境均处于合理区,表明所建模型可信度高,能够用于后续的对接研究。通过分子对接,筛选到若干与底物(对-香豆酰辅酶A和丙二酸单酰辅酶A)结合的关键氨基酸残基,它们主要通过氢键、范德华力与底物结合,从而稳定底物在酶分子中的空间位置。根据CHS的酶学特性与对接结果,推测决明CHS中Cys164、His303与活性中心的H2O能够形成电子传递体系,参与酶促反应过程。以上研究成果能够为利用此类CHS三维模型研究其催化机理和分子工程改造奠定基础。
1 Ma LQ(马兰青),Shi GL(师光禄),Ye HC(叶和春),et al.Plant-specific type III polyketide synthase superfamily:gene structure,function and metabolistes.Chin J Biotechnol(生物工程学报),2010,26:1482-1492.
2 Dao TTH,Linthorst HJM,Verpoorte R.Chalcone synthase and its functions in plant resistance.Phytochem Rev,2011,10:397-412.
3 Ferrer JL,Jez JM,Bowman ME,et al.Structure of chalcone synthase and the molecular basis of plant polyketide biosynthesis.Nat Struc Biol,1999,6:775-784.
4 Hao YJ(郝延军),Shang YL(桑育黎),Zhao YQ(赵余庆),et al.The progress of Cassia tora.Chin Tradit Herb Drugs(中草药),2001,32:858-859.
5 Liao H(廖海),Zhou JY(周嘉裕).Molecular cloning and analysis of a Chalone synthase gene of Cassia tora.Acta Botan Boreali-Occidentalia Sin(西北植物学报),2008,28:1728-1733.

