鸭瘟病毒LH2011强毒株部分基因序列的测定与分析
王继春, 张传健, 许梦微, 王志胜, 侯继波
(江苏省农业科学院国家兽用生物制品工程技术研究中心,江苏 南京 210014)
鸭瘟病毒(Duck enteritis virus,DEV)属于疱疹病毒目疱疹病毒科疱疹病毒甲亚科,是鸭病毒性肠炎(俗称鸭瘟)的病原,能感染48种以上的雁形目水禽,如鸭、鹅和天鹅等[1-2]。鸭瘟呈急性发病,有高度的接触传染性,发病率和死亡率可达100%,常表现为消化道黏膜糜烂、血管损伤引起的实质性脏器出血及退性行变化、淋巴器官病变等[1,3-4]。
鸭瘟灭活疫苗和弱毒活疫苗均能对鸭瘟产生保护,其中弱毒活疫苗的免疫效果更佳,鸭瘟弱毒疫苗分为人工鸡胚适应毒和天然弱毒,免疫接种后1 d即可产生保护,免疫持续期可达1年以上[1,5-6]。疫苗株接种鸭只后不能排出足够量的病毒,难于对同群鸭只产生有效感染,不能诱发接触感染免疫。
鸭瘟病毒共有78个开放阅读框。在建立感染性克隆的基础上[7],对鸭瘟病毒 2 085株进行全基因测序,与GenBank中发表的强毒株和弱毒株序列对比发现 UL47、UL41、UL12、UL2、US10 和 LORF11中的碱基缺失、移码或非同义替代仅在弱毒株中出现,提示这6个开放阅读框的变异可能与鸭瘟病毒的致病力变化高度相关[8],而且这些变异不损伤鸭瘟病毒在鸡胚上的增殖能力,但会有损于鸭瘟病毒在鸭只间的水平传播能力。
2011年国家兽用生物制品工程技术研究中心从南京六合某鸭场的病死鸭中分离获得一株鸭瘟病毒强毒株(LH2011株),本研究对 LH2011株LORF11、UL47、UL41、UL12、UL2 和 US10 等位基因序列进行测定,并结合GenBank中发表的其他鸭瘟强、弱毒株的序列,对其进行进一步对比分析,为深入研究鸭瘟病毒的机理提供依据。
1 材料与方法
1.1 材料
鸭瘟病毒LH2011株由国家兽用生物制品工程技术研究中心实验室分离、鉴定和保存,对鸭的最小致死剂量为1 ml 1×10-5~1×10-7。SPF鸡胚,购自中牧集团南京药械厂。Taq酶(Ex Tap酶)和PCR反应试剂,购自大连宝生物工程有限公司。
1.2 方法
1.2.1 PCR引物 根据已发表的鸭瘟病毒的LORF11、UL47、UL41、UL12、UL2 和 US10(JF999965;EU082088.2;JQ647509.1)开放阅读框的上、下游序列中的保守序列分别设计1对引物(表1),由南京金斯瑞生物科技有限公司合成。

表1 PCR引物序列Table 1 Primers for PCR
1.2.2 模板DNA提取 病毒制备:取9~10日龄SPF鸡胚,按常规方法制备鸡胚成纤维细胞,将DEV LH2011株按0.01的感染复数(MOI)接种鸡胚成纤维细胞,当细胞病变(CPE)达60%~70%时,将上清液与细胞一起反复冻融3次,8 000 r/min离心15 min,取上清液备用。
病毒DNA的提取:①取制备的上清液445 μl,加入10%十二烷基磺酸钠50 μl,20 mg/ml蛋白酶K 2.5 μl,混匀;置56 ℃ 水浴1 h,99 ℃ 水浴10 min,冷却至室温;②加入三羟甲基氨基甲烷饱和酚200.0 μl,混匀,4 ℃ 12 000 r/min,5 min,取上清液。重复2次;③加入三羟甲基氨基甲烷饱和酚100.0 μl和氯仿 100.0 μl,混匀,4 ℃ 12 000 r/min,5 min,取上清液;④加入1/10体积的3 mol/L醋酸钠和1~2倍体积无水乙醇,上下颠倒混匀,置-20℃ 40 min至3~5 d;⑤4 ℃ 12 000 r/min,20 min,弃上清液,沉淀用70%乙醇洗涤,4℃ 12 000 r/min,1 min,弃上清液,洗涤 2次;⑥置超净台中风干,加入 20.0~100.0 μl经高压灭菌的超纯水溶解。
1.2.3 PCR扩增与序列测定 PCR反应体系为:DNA 模板1.0 μl,Taq酶 (LORF11用Ex Taq酶)0.5 μl,上游引物 (10 pmol/μl)2.0 μl,下游引物 (10 pmol/μl)2.0 μl,dNTP 混合物 (dATP、dCTP、dGTP和 dTTP 浓度均为 2.5 mmol/L)4.0 μl,10 × Buffer 5.0 μl,Mg2+(25 mmol/L)4.0 μl,ddH2O 31.5 μl。样品经2 000 r/min离心瞬间混匀进行PCR反应。
PCR反应程序为:94℃ 3 min预变性;94℃变性30 s,52℃退火40 s,72℃延伸(LORF11是3 min 40 s,UL47 是 2 min 30 s,UL41 是 1 min 30 s,UL12是2 min,UL2 是1 min,US10 是1 min),循环40次,最后72℃延伸10 min,于4℃保存待检。取5.0 μl PCR产物与Marker一起分别点入1%琼脂糖凝胶上,电泳40 min,观察扩增片段的长度。将PCR反应获得的特异性片段的条带切胶回收后由华大生物科技有限公司进行序列测定。
1.2.4 序列的对比分析 对测定的LORF11、UL47、UL41、UL12、UL2 和 US10 等位基因序列应用VectorNTI(Invitrogen)进行开放阅读框(ORF)分析,并将其与GenBank上已发表的鸭瘟病毒的等位同源序列进行对比分析,其中包括2085株(JF999965)、VAC株(EU082088.2)和CHv株(JQ647509.1)的所有这6个ORF序列,Holland株(JQ595417)、Jansen株(JQ430740.1)、D11-JW-016株(JQ430739.1)和clone-03株 (EU294364.1)的 LORF11,clone-03(EF492886.1)的 UL47,clone-03(EF524094.1)的UL12,Attenuated strain 2株(JQ347518.1)、Attenuated strain 1株 (JQ347517.1)、N3株(JQ248597.1)、LS株(JQ248598.1)、N2 株(JQ248596.1)、N1 株(JQ043216.1)和 clone-03株(EF449516.1)的 UL2,clone-03株(EF524095.1)的US10。
2 结果
2.1 LORF11同源子的典型特征
序列测定结果(图 1A)显示,LH2011的LORF11同源子长4 341 bp(JX885588),与另一株分离自中国的强毒株CHv株的同源子阅读框结构一致,而与其他毒株的LORF11则表现出高度的变异性。与欧洲毒株(2085株、Jansen株、Holland株和D11-JW-016株)相比,一个1 071 bp的插入序列将 LH2011株的同源子分为了 2个 ORF,即LORF11A和 LORF11B,LORF11A的 ORF有235个氨基酸,其中氨基端164个与欧洲毒株同源,而LORF11B的ORF有923个氨基酸,其中羧基端892个氨基酸与欧洲毒株的羧基端氨基酸同源。与Clone-03株对比发现,后者LORF11中缺失了一个1 930 bp的碱基序列,LORF11A氨基端有211个氨基酸同源,LORF11B羧基端有592个氨基酸同源。VAC株虽然与Clone-03株相比又缺失了一个1 596 bp的序列,导致与LORF11B在羧基端仅有60个氨基酸序列同源。LORF11除了上述序列的插入或缺失外,各毒株之间还发生了许多非同义或同义替代,这些替代没有明显的规律可循(表2)。与CHv株相比,LH2011株的LORF11同源子在1 030位发生一个碱基缺失,但此区域并不在开放阅读框LORF11A或LORF11B内。
鸭瘟病毒的LORF11仅在禽疱疹病毒中有同源子,如马立克氏病病毒、火鸡疱疹病毒、传染性喉气管炎病毒等[9-11],研究结果表明马立克氏病病毒的LORF11与病毒的增殖与毒力相关[12],其他病毒的LORF11功能未见报道。鸭瘟病毒LORF11的功能尚不清楚。从图1A可以发现,与欧洲株不同的是中国强毒株和弱毒株均有一段142 bp的序列,提示这段序列可能与毒株的地域性相关,是中国源毒株的特异性序列。而与强毒株相比,无论Clone-03,还是VAC株均发生了大片段的序列缺失,提示这种变异很可能与病毒的毒力减弱相关,而Clone-03和VAC疫苗株仍能在鸡胚或鸡胚成纤维细胞和鸭体生长,说明这些片段的缺失没有对病毒的生长造成严重损害。
2.2 UL2序列分析
DEV的UL2是单纯疱疹病毒1型(Herpes simplex virus type 1,HSV-1)的UL2的同源基因,后者编码尿嘧啶DNA糖基化酶,是一个非必须基因,是对病毒复制发挥重要作用的一种酶[13-14]。图1显示了LH2011株及几种不同毒株的LORF11(A)和UL2(B)同源子序列的碱基插入或缺失。序列测定结果(图1B)显示,LH2011的UL2序列与2085株、N1株、N2株、N3株、LS株和CHv株鸭瘟强毒株完全一致(GenBank KC480259),而与VAC株、Clone-03株、Attenuated strain 1株和Attenuated strain 2株鸭瘟弱毒株差异显著。弱毒株均发生了一个528 bp的片段缺失,这段528 bp序列占UL2的一半以上,可能已导致UL2功能的丧失,提示这个片段缺失很可能与弱毒株的毒力减弱直接相关。图1B中所注2085株和VAC株的起始位点是GenBank中登录的序列标注的位点,经过进一步分析其上游序列,发现与其他毒株一致,因此可以认为这种起始位点的差异是由于预测软件的不同导致的。从所有这些序列对比中发现,LH2011株的UL2与Attenuated strain 1株相比发生了1个非同义替代导致第2位氨基酸由苏氨酸变为丙氨酸,与VAC株相比发生了1个非同义替代导致第24位氨基酸由谷氨酸变为缬氨酸。此外,除VAC株外,所有毒株在下游280 bp序列中的3个碱基缺失使整个ORF发生移码,导致了18个氨基酸的改变,因为这种改变在强毒株和弱毒株中均有出现,所以应与病毒的毒力和生长能力无关。

图1 LH2011株与不同鸭瘟病毒毒株的LORF11和UL2的对比Fig.1 Alignment of LORF11 homologues and UL2 between strain LH2011 and other DEV strains
2.3 LH2011株 UL47、UL41、UL12和 US10与欧洲强毒株2085株的比较
测序结果显示,LH2011株的UL47、UL41、UL12和US10序列与2085株的序列完全相同(KC480260-KC480263)。UL47在HSV-1中的同源基因是一个非必须基因,在病毒囊膜中富聚,与UL6共同加强α-TIF活性[15]。对比UL47序列(表2)发现,在第36位氨基酸上,LH2011株、2085株和CHv株3个强毒株为丙氨酸,而VAC株和Clone-03株2个弱毒株为缬氨酸,提示这个变异可能与病毒毒力的变化相关。在113位、148位和598位(599位)上,LH2011株与2085株一致,而CHv株、VAC株、Clone-03株均相同。
UL41的单纯疱疹病毒(HSV-1)同源基因是一个非必须基因,在病毒感染的早期发挥核糖核酸内切酶的作用[16]。对比UL41的序列可以发现在第44位氨基酸上,包括LH2011的3个强毒株为谷氨酸,而2个弱毒株为甘氨酸,提示这个变异可能与病毒的毒力变化有关。在125位和301(302)位氨基酸位点上,LH2011株与2085株一致,而 CHv株、VAC株和 Clone-03株相同(表 2)。
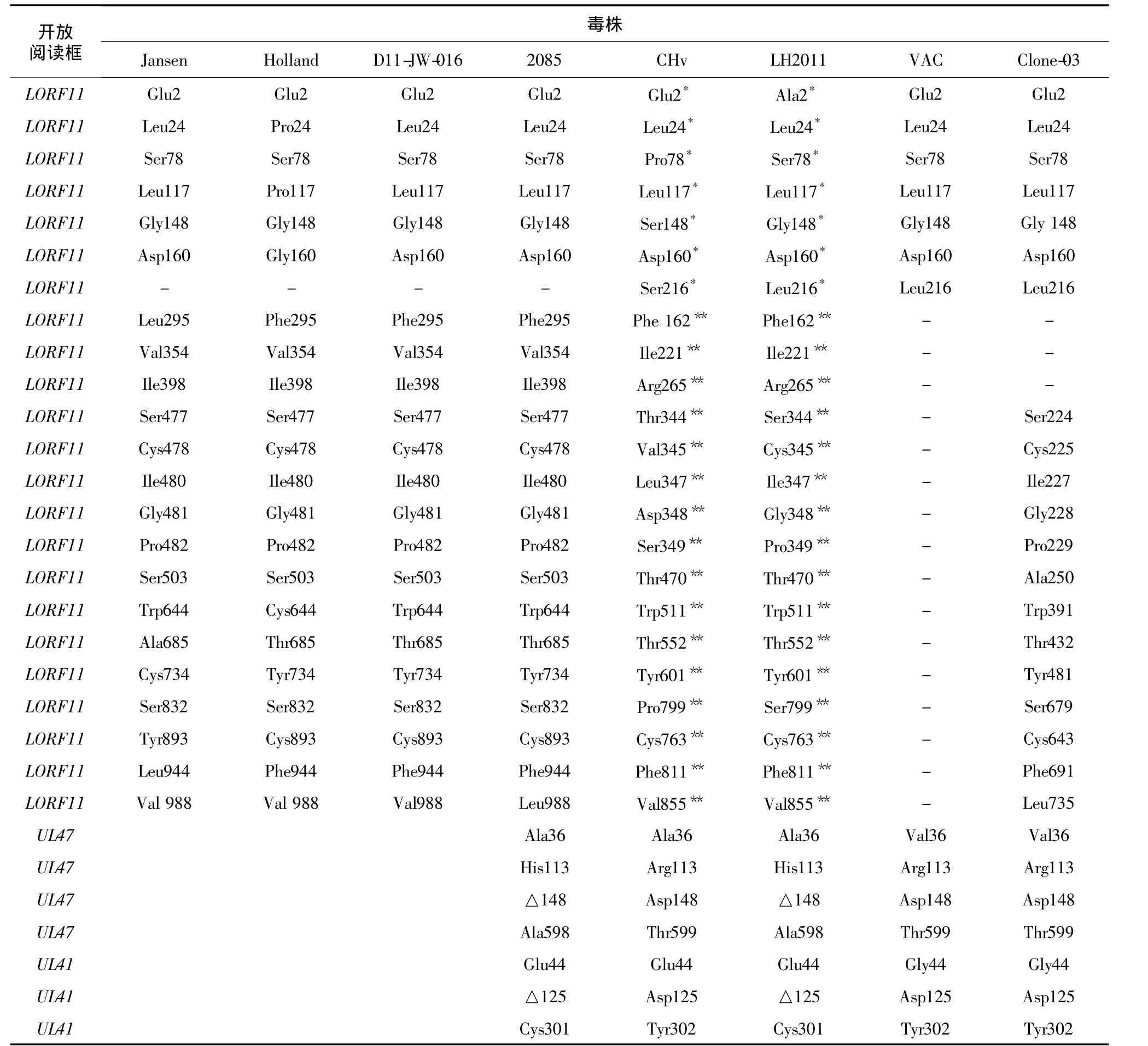
表2 LORF11、UL47和UL41的单个氨基酸差异对比Table 2 Single amino acids in LORF11,UL47 and UL41 of different strains
UL12的HSV-1同源基因是一个非必须基因,编码一种碱性核酸酶,参与病毒DNA复制[17]。LH2011株的UL12序列与CHv株和2085株一致,而与VAC株和Clone-03株2个弱毒疫苗株差别明显,在VAC株中,因243位胸腺嘧啶发生缺失使得其ORF向下游移码237 bp,造成氨基端丢失了79个氨基酸,被另2个氨基酸残基取代。在Clone-03株中,因1324位插入一个胞嘧啶,使UL12的ORF提前终止,导致羧基端116个氨基酸被另5个非同义的氨基酸替代。此结果进一步提示弱毒株中UL12编码蛋白质发生的变化可能与病毒的致病力减弱有关,而与病毒在鸡胚成纤维细胞上的生长性能无关[8]。
US10的HSV-1同源基因是一个非必须基因,编码一种囊膜磷蛋白质,但其作用不明[18]。LH2011株的US10序列与2085株和CHv株完全一致,但2个弱毒株在787位都发生了一个胸腺嘧啶的缺失,导致ORF发生移码,引起其下游35个氨基酸发生改变,而且整个ORF延长了11个氨基酸[8]。此差别发生在强、弱毒之间,提示这个变化可能与弱毒株的毒力减弱有关。
3 结论
LH2011 株的 ORF11、UL47、UL41、UL12、UL2 和US10表现出典型的强毒株的特征,其中LORF11、UL12、UL2和US10的变异仅在强毒株中被发现,且发生了大片段碱基序列的插入、缺失或移码,提示可能与鸭瘟病毒的致病力相关,尤其是LORF11和UL2在强、弱毒株中差异更加显著,应是鸭瘟病毒机理研究的重点。鸭瘟病毒细菌人工染色体(Bacterial artificial chromosome,BAC)技术的成功建立使得对病毒基因组进行缺失、突变和插入的分子生物学操作可以利用大肠杆菌原核系统中的大量操作工具,能大大提高研究效率,且基因不易发生变异[7,19-21],为开展鸭瘟病毒的机理研究和活载体疫苗的研制提供了强有力的研究工具,根据本研究的结果,可以首先对鸭瘟病毒的LORF11和UL2应用BAC技术进行基因敲除或插入,研究其在鸭瘟病毒致病力中的作用机理。
[1]KALETA E,KUCZKA A,KÜHNHOLD A,et al.Outbreak of duck plague(duck herpesvirus enteritis)in numerous species of captive ducks and geese in temporal conjunction with enforced biosecurity(in-house keeping)due to the threat of avian influenza a virus of the subtype asia h5n1[J].Dtsch Tierarztl Wochenschr,2007,114:3-11.
[2]LEIBOVITZ L,HWANG J.Duck plague on the american continent[J].Avian Dis,1968,12:361-378.
[3]WOBESER G.Experimental duck plague in blue-winged teal and canada geese[J].J Wildl Dis,1987,23:368-375.
[4]WOBESER G,DOCHERTY D.A solitary case of duck plague in a wild mallard[J].J Wildl Dis,1987,23(3):479-482.
[5]BUTTERFIELD W,DARDIRI A.Serologic and immunologic response of ducks to inactivated and attenuated duck plague virus[J].Avian Dis,1969,13(4):876-887.
[6]LIU J,CHEN P,JIANG Y,et al.A duck enteritis virus-vectored bivalent live vaccine provides fast and complete protection against h5n1 avian influenza virus infection in ducks[J].J Virol,2011,85(21):10989-10998.
[7]WANG J,OSTERRIEDER N.Generation of an infectious clone of duck enteritis virus and generation of a vectored dev expressing hemagglutinin of h5n1 avian influenza virus[J].Virus Res,2011,159(1):23-31.
[8]WANG J,HÖPER D,BEER M,et al.Complete genome sequence of virulent duck enteritis virus(dev)strain 2085 and comparison with genome sequences of virulent and attenuated dev strains[J].Virus Res,2011,160(1-2):316-325.
[9]AFONSO C,TULMAN E,LU Z,et al.The genome of turkey herpesvirus[J].J Virol,2001,75(2):971-978.
[10]BUCKMASTER A,SCOTT S,SANDERSON M,et al.Gene sequence and mapping data from marek’s disease virus and herpesvirus of turkeys:Implications for herpesvirus classification[J].J Gen Virol,1988,69(8):2033-2042.
[11]LEE S,MARKHAM P,MARKHAM J,et al.First complete genome sequence of infectious laryngotracheitis virus[J].BMC Genomics,2011,12:197.
[12]LEE L,SILVA R,CUI X,et al.Characterization of lorf11,a unique gene common to the three marek’s disease virus serotypes[J].Avian Dis,2007,51:851-857.
[13]BOGANI F,CHUA C,BOEHMER P.Reconstitution of uracil DNA glycosylase-initiated base excision repair in herpes simplex virus-1[J].J Biol Chem,2009,284:16784-16790.
[14]PYLES R,THOMPSON R.Evidence that the herpes simplex virus type 1 uracil DNA glycosylase is required for efficient viral replication and latency in the murine nervous system[J].J Virol,1994,68:4963-4972.
[15]MEREDITH D,LINDSAY J,HALLIBURTON I,et al.Posttranslational modification of the tegument proteins(vp13 and vp14)of herpes simplex virus type 1 by glycosylation and phosphorylation[J].J Gen Virol,1991,72(11):2771-2775.
[16]SMILEY J.Herpes simplex virus virion host shutoff protein:Immune evasion mediated by a viral rnase?[J].J Virol,2004,78(3):1063-1068.
[17]MARTINEZ R,SARISKY R,WEBER P,et al.Herpes simplex virus type 1 alkaline nuclease is required for efficient processing of viral DNA replication intermediates[J].J Virol,1996,70(4):2075-2085.
[18]YAMADA H,DAIKOKU T,YAMASHITA Y,et al.The product of the us10 gene of herpes simplex virus type 1 is a capsid/tegument-associated phosphoprotein which copurifies with the nuclear matrix[J].J Gen Virol,1997,78(11):2923-2931.
[19]MESSERLE M,CRNKOVIC I,HAMMERSCHMIDT W,et al.Cloning and mutagenesis of a herpesvirus genome as an infectious bacterial artificial chromosome[J].Proc Natl Acad Sci USA,1997,94:14759-14763.
[20]WARDEN C,TANG Q,ZHU H.Herpesvirus bacs:Past,present,and future[J].J Biomed Biotechnol,2011,2011:124595.
[21]TISCHER B,SMITH G,OSTERRIEDER N.En passant mutagenesis:A two step markerless red recombination system[J].Methods Mol Biol,2010,634:421-430.
——紫 苏

