双齿围沙蚕消化道共栖微生物菌群多样性的PCR-DGGE分析
张柏东,连彬,王斌,周一兵,何洁
(大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023)
双齿围沙蚕消化道共栖微生物菌群多样性的PCR-DGGE分析
张柏东,连彬,王斌,周一兵,何洁
(大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023)
将采自青岛市沿海潮间带的双齿围沙蚕Perinereis aibuhitensis(体质量为4~5 g)置于无菌海水中暂养24 h后,分别从5条双齿围沙蚕消化道中提取微生物基因组总DNA,应用细菌16S rDNA通用引物341f/ 534r进行细菌16S rDNA基因V3高变异区的PCR扩增,再将PCR产物进行变性梯度凝胶电泳 (DGGE),从而获得样品消化道共栖微生物群落特征的DNA指纹图谱。通过对指纹图谱半定量分析发现,采集的双齿围沙蚕消化道共栖微生物菌群多样性丰富,优势条带明显,不同个体间既存在共同的微生物种属,也有各自特异的种属。其中存在一条共同的优势条带,但优势条带含量存在个体间差异。分别对DGGE指纹图谱中公共条带序列进行测序比对,结果表明,产丙酸菌属Propionigenium分别为5个样品中的优势菌群,假交替单胞菌属Pseudoalteromonas广泛分布于双齿围沙蚕消化道中。研究表明,基于16S rDNA的PCRDGGE图谱技术是分析双齿围沙蚕及其他海洋沉积食性无脊椎动物消化道微生物菌群结构较为有效的手段。
双齿围沙蚕;消化道;微生物;PCR-DGGE技术
双齿围沙蚕Pernereis aibuhitensis隶属于环节动物门、多毛纲,是重要的海洋沉积食性无脊椎动物,其迁徙距离短,在中国河口沉积物和沿海滩涂有着广泛的地理分布[1]。双齿围沙蚕能够蓄积环境中的大量有机污染物并通过自身的生物转化作用代谢和分解污染物,沙蚕也可以改善养殖水体底质,修复环境,是海洋沉积环境早期污染生态风险评价的指示生物和生态毒理学研究中重要的模式生物[2-4]。消化道微生物群落不仅是无脊椎动物正常生长代谢的重要保障,同时也与环境的变化息息相关[5-6]。目前,国内外关于双齿围沙蚕消化道内共栖微生物菌群结构、分布特点和各种群之间关系的研究还很少,关于双齿围沙蚕消化道微生物的研究多集中于对蛋白酶高产菌株等具有生物活性菌株的分离鉴定及其酶学特性的研究[7-9]。因此,获取更多关于双齿围沙蚕消化道微生物菌群结构和分布特点的相关信息,对海洋环境的监测和治理具有重要意义。本研究中,作者通过提取双齿围沙蚕消化道定植菌总菌基因组DNA,利用PCR-DGGE方法获得消化道菌群的种类组成,为探讨双齿围沙蚕消化道中微生物菌群的生物多样性提供了技术支持。
1 材料与方法
1.1 材料
双齿围沙蚕采自山东青岛沿海潮间带同一滩涂区域,体质量为4~5 g。
PCR引物由生工生物工程 (上海)股份有限公司合成;TaqDNA聚合酶和PCR反应缓冲液购自宝生物工程 (大连)有限公司;DGGE相关试剂和GeneFinder核酸染料均购自生工生物工程 (上海)股份有限公司;细菌基因组DNA提取试剂盒购自北京博凌科为生物科技有限公司。PCR仪为Eppendorf Authorized Thermal Cider;DGGE电泳仪为Bio-Rad Dcode Universal Detection System。
1.2 方法
选取健康的5条双齿围沙蚕置于实验室无菌海水中暂养24 h,使其排出肠道内容物。用体积分数为10%的乙醇溶液麻醉后,将沙蚕置于超净工作台解剖盘中,用体积分数为75%的乙醇消毒体表,再经无菌水冲洗后,解剖取出沙蚕消化道样品备用。
1.2.1 基因组DNA的提取 无菌操作提取双齿围沙蚕新鲜肠道样品 (约200 mg)后,按照粪便基因组DNA快速提取试剂盒说明书进行沙蚕消化道总菌基因组DNA的提取。提取后的DNA用15 g/L的琼脂糖凝胶电泳检测后,置于-20℃下保存备用。
1.2.2 基因组DNA 16S rDNA V3区的扩增 以提取的双齿围沙蚕消化道细菌基因组DNA为模板,进行细菌16S rDNA V3区的PCR扩增。细菌通用引物序列为GC-341f(5'-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3')和534r(5'-ATTACCGCGGCTGCTGG-3')[10-11]。PCR反应体系:无菌双蒸水29.6μL,10×PCR缓冲液5μL,2.5 mmol/L dNTP 5μL,10 mmol/L PCR引物各3μL,5μg/μLTaq酶0.4μL,DNA模板3μL。试验以分离自双齿围沙蚕消化道的游海假交替单胞菌纯菌株作为阳性对照,以ddH2O替代样品DNA作为阴性对照。PCR反应程序:94℃下预变性5 min;94℃下变性30 s,53℃下退火30 s,72℃下延伸90 s,共进行30个循环,每个循环退火时间增加1 s;最后在72℃下延伸10 min。PCR产物用15 g/L的琼脂糖凝胶电泳进行检测。
1.2.3 DGGE凝胶电泳及切胶的测序分析 采用Bio-Rad公司的Dcode DGGE系统对PCR产物进行电泳分离。采用8%聚丙烯酰胺凝胶,变性梯度为35%~50%(100%的变性剂为100mL去离子水中含42 g尿素和40 g去离子甲酰胺),电泳缓冲液为1×TAE,在60℃、120 V条件下电泳12 h。电泳后,用Genefinder染色30 min,在超纯水中脱色15 min。使用Kodak Image Station 440紫外凝胶成像系统进行拍照。在紫外灯下,对DGGE指纹图谱中特异性条带进行切胶,切下的胶溶于50μL无菌双蒸水中过夜将DNA模板溶出,再通过不带GC夹子的引物341f和534r对溶出模板进行相同条件的PCR扩增,将扩增后的产物经检测后交付生工生物工程 (上海)股份有限公司进行双向测序,测序结果拼接后进行比对分析。
1.2.4 DGGE指纹图谱的分析 使用凝胶成像分析软件Kodak MI(Kodak GL2000,USA)和Bio-Dap分析软件,分别计算香浓-威纳多样性指数(H)、丰度(S)、均匀性指数(EH),以及各优势条带所占比例,对DGGE指纹图谱进行半定量分析。
2 结果与分析
2.1 基因组DNA16SrDNAV3区的扩增
分别以5条双齿围沙蚕消化道样品 (S1~S5)总菌基因组DNA为模板,对16S rDNA V3区进行扩增,PCR产物用10 g/L的琼脂糖凝胶电泳检测,发现均获得了特异性扩增片段 (图1),扩增出的条带片段大小在230 bp左右。

图1 16S rDNA V3区PCR扩增产物的琼脂糖凝胶电泳图谱Fig.1 Agarose gel electrophoresis of PCR am plified product of V3 region of the 16S rDNA genes
2.2 变性梯度凝胶电泳 (DGGE)
对S1~S5样品的16S rDNA V3区PCR扩增产物进行变性梯度凝胶电泳,获得DGGE指纹图谱如图2所示。可以看出,S1~S5样品的扩增条带数分别为22、21、18、18、19,均表现出较高的菌群多样性。其中条带1~10均为5条双齿围沙蚕样品的公共条带,条带14为S1、S2和S3号样品的公共条带,条带15为S2~S5样品的公共条带,说明不同双齿围沙蚕的肠道菌群结构有较多共同的微生物种属,而不同沙蚕个体间也存在着各自特异的种属,如条带11、12、13分别为S3、S4、S2样品的特异性种属。同时,5个样品中各自的优势条带明显并且一致,均为条带4,而优势条带信号强度有所不同,说明定植于双齿围沙蚕消化道的优势菌群的分布相对一致,但含量有所差异。
2.3 DGGE指纹图谱分析
对双齿围沙蚕消化道共栖菌群的DGGE指纹图谱进行分析,结果见表1。从表1可见:5组样品的丰度相近,但多样性指数和均匀性指数有所差异,表现在S3、S4样品的多样性指数和均匀性指数相对较低,而S2、S5样品的多样性指数和均匀性指数相对较高。
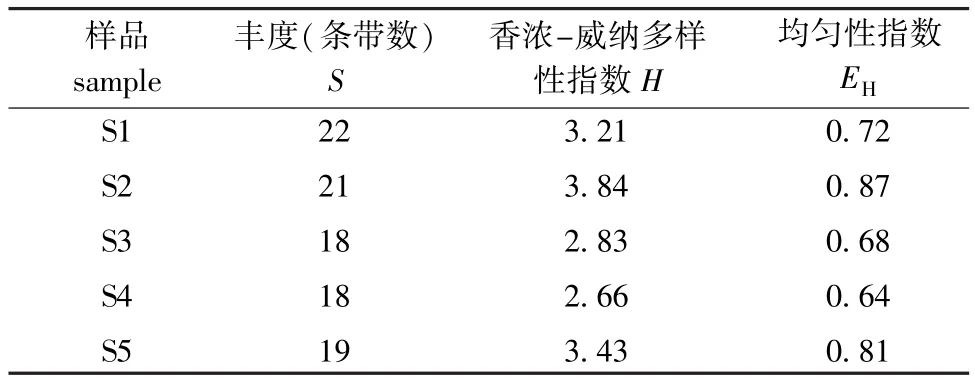
表1 特征指数计算结果Tab.1 Calculated results of characteristic index
2.4 消化道共栖细菌群落组成和优势菌群分析
从16S rDNA V3区PCR-DGGE指纹图谱中分别割胶回收了10个公共条带 (图2条带1~10),对回收得到的序列进行双向测序比对分析,得到样品的细菌群落组成,结果见表2。从表2可见,条带4为产丙酸菌属Propionigenium,分别为5个样品的优势菌群,但样品间相对含量不同。其中假交替单胞菌属Pseudoalteromonas菌群分离比率相对较高,说明假交替单胞菌属广泛分布于双齿围沙蚕消化道中。本研究中获得的亲缘关系最近的序列大部分来自海洋环境,包括近岸海水、海洋沉积物和海洋生物,很好地反映了双齿围沙蚕的生活和栖息环境特点。
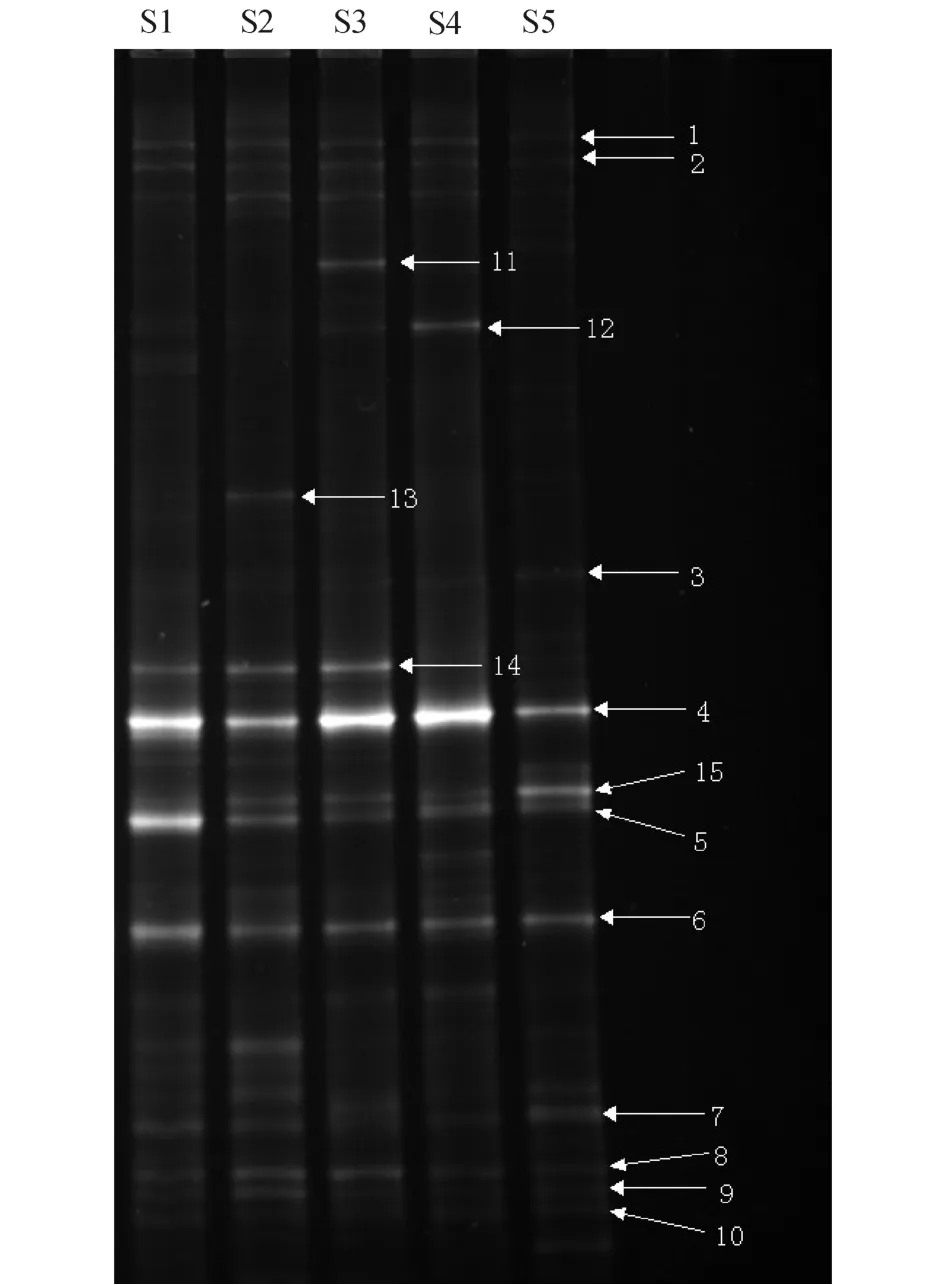
图2 不同肠道样品 (S1~S5)的DGGE指纹图谱Fig.2 DGGE profile of different intestinal samples (S1~S5)

表2 DGGE共性条带基因片段序列的比对结果Tab.2 Com parison of genom ic sequences in DGGE common bands by sequencing and Blast analysis
3 讨论
本研究中,以双齿围沙蚕消化道总菌基因组DNA为模板,经PCR反应后获得了5条16S rRNA基因V3区片段,琼脂糖凝胶电泳显示,各样品目的扩增片断亮度均匀一致,没有其他非特异性条带。通过对5条PCR产物片段进行变形梯度凝胶电泳,所得5个样品的DGGE条带清晰可见,优势条带明显,证实通过变性梯度凝胶电泳对于双齿围沙蚕消化道微生物菌群分离达到了良好的效果。5个样品含有较多的共同菌群,但个体间也存在特异的菌群,可能是沙蚕个体之间差异或空间异质性产生摄食环境的细微变化造成的。其中4号条带优势显著,但不同样品中亮度不同,说明个体间优势菌群的含量有所差别。沙蚕迁徙能力较弱,肠道菌群受摄食环境的影响较大[12-13],为了强化对有机物的腐化和矿化作用,沙蚕形成了适应微生物生长繁殖的消化道环境,保持了特定微生物良好的活性和稳定的菌群结构,从而维持了机体的正常生长代谢[14]。应用Kodak MI软件分析计算香浓-威纳多样性指数、丰度、均匀性指数,对DGGE指纹图谱进行进一步分析表明,S3、S4样品的多样性指数相对于S2、S5样品较低,香农-威纳多样性指数包含物种数和各种间个体分配的均匀性两个成分,5个样品的条带数差别不大,但S3、S4样品的均匀性指数明显低于S2、S5样品,造成S3、S4样品的多样性指数相对偏低的原因是条带间含量分布不均匀。
对DGGE图谱中10条公共条带进行测序比对后发现,5个样品中的优势菌群均为产丙酸菌属,其次为假交替单胞菌属。假交替单胞菌属很可能作为特定环境下双齿围沙蚕常在的共栖异养菌,有助于分解代谢有机碳化合物,维持沙蚕正常的生长代谢。同时,本研究中获得序列中亲缘关系最近的序列的来源大部分为海洋环境,其中与条带5和9最相近的序列分别分离自污损生物海绵[15]和发生水华的近岸海域,说明双齿围沙蚕消化道菌群的组成随栖息环境的变化会出现相应的调整和改变,因此,实时分析和监测双齿围沙蚕消化道菌群结构的组成,对于养殖水体和近海的水质评价及环境监测具有重要意义。
在对微生物菌群的分析中,传统的培养方法对于菌种有一定的选择性[16]。研究表明,只有10% ~50%的微生物菌群是可培养的[17-18],因而传统的培养方法会造成样品微生物多样性的评价不准确。PCR-DGGE技术是分析细菌群落多样性和监测微生物群落动态的一种重要工具,目前已成功应用于研究包括人类肠道菌群[19]在内的不同动物的消化道微生物生态组成[20-22],如马悦欣等[23]曾采用此技术研究了四角蛤蜊的细菌多样性。关于用沉积食性无脊椎动物作为环境质量评价指示生物,探讨其消化道微生物菌群与环境变化响应关系的研究多集中于土壤无脊椎动物[6,13],而对作为海洋沉积环境质量评价指示生物的双齿围沙蚕的相关研究还很少,本研究中,采用PCR-DGGE技术测定双齿围沙蚕消化道菌群,在一定程度上反映了其消化道常在菌的分布特点及菌群间的相对关系,而关于双齿围沙蚕消化道菌群随环境污染的动态变化,还需要通过更多定量的研究手段如原位荧光杂交(FISH)和荧光实时定量PCR(Real-time PCR)进行更深入的研究。
[1] Jørgensen A,Giessing A M B,Rasmussen L J,et al.Biotransformation of polycyclic aromatic hydrocarbons in marine polychaetes [J].Mar Environ Res,2008,65:171-186.
[2] Yuan X T,Chen A H,Zhou Y B,et al.The influence of cadmium on the antioxidant enzyme activities in polychaetePerinereis aibuhitensisGrube(Annelida:Polychaeta)[J].Chinese Journal of O-ceanology and Limnology,2010,28(4):849-855.
[3] 周一兵,陈雪,杨大佐,等.双齿围沙蚕CYP4基因的克隆及序列分析[J].大连海洋大学学报,2011,26(6):507-513.
[4] 李霞,宋贞坪,王福景,等.雌激素对双齿围沙蚕性比、生长和卵母细胞发育的影响[J].大连海洋大学学报,2011,26(2): 97-101.
[5] 葛莉莉.鱼类肠道菌群的研究概况[J].水利渔业,2006,26 (4):17-20.
[6] Knapp B A,Seeber J,Podmirseg S,et al.Insam molecular fingerprinting analysis of the gut microbiota ofCylindroiulus fulviceps(Diplopoda)[J].Pedobiologia,2008,52:325-336.
[7] 闫志勇,毕春霞,辛晓妮,等.1株高产蛋白酶嗜麦芽寡养单胞菌的分离鉴定及其酶学活性的研究[J].微生物学杂志,2010, 30(5):7-11.
[8] 闫志勇,王斌,宋旭霞,等.一株具有纤维蛋白溶解活性的海单胞菌的分离和鉴定[J].中国生物制品学杂志,2007,20(10): 717-732.
[9] 代玉梅,闫志勇,王斌,等.沙蚕消化道产蛋白酶菌D2株的筛选及其酶学性质[J].中国生物制品学杂志,2007,20(7):493-496.
[10] Baker PW,Harayama S.An analysis ofmicroorganisms in environments using denaturing gradient gel electrophoresis[J].Environmental Microbiology,2004,16:323-338.
[11] Muyzer G,De Waal E C,Uitterlinden A G.Profiling of complex microbial populationsby denaturing gradientgel electrophoresisanalysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied and EnvironmentMicrobiology,1993,59: 695-700.
[12] 高菲,孙慧玲,许强,等.刺参消化道内含物细菌群落组成的PCR-DGGE分析[J].中国水产科学,2010,17(4):672-679.
[13] Knapp BA,Podmirseg SM,Seeber J,etal.Diet-related composition of the gutmicrobiota ofLumbricus rubellusas revealed by a molecular fingerprinting technique and cloning[J].Soil Biology& Biochemistry,2009,41:2299-2307.
[14] Fonte S J,Winsome T,Six J.Earthworm populations in relation to soil organicmatter dynamicsandmanagement in California tomato cropping systems[J].Applied Soilecology,2009,41:206-214.
[15] 单红云,赫崇波,李晓冬,等.一种海绵动物对室内养殖海参危害的初步研究[J].水产科学,2008,27(12):645-647.
[16] Amann R,LudwigW,Schleifer K.Phylogenetic identification and in situ detection of individual microbial cells without cultivation [J].Microbiology and Molecular Biology Reviews,1995,59: 143-169.
[17] Holzapfel W,Haberer P,Snel J,et al.Overview of gut flora and probiotics[J].International Journal of Food Microbiology,1998, 41:85-101.
[18] Wilson K,Blitchington R.Human colonic biota studied by ribosomal DNA sequence analysis[J].Applied and Environmental Microbiology,1996,62:2273-2278.
[19] Vanhoutte T,Huys G,De Brandt E,etal.Temporal stability analysis of themicrobiota in human feces by denaturing gradient gel electrophoresis using universal and group-specific 16S rRNA gene primers[J].FEMSMicrobiol Ecol,2004,48:437-446.
[20] Reeson A F,Jankovic T,Kasper M L,et al.Application of 16S rDNA-DGGE to examine themicrobial ecology associated with a socialwaspVespula germanica[J].Insect Mol Biol,2003,12: 85-91.
[21] Tanaka H,Aoyagi H,Shina S,et al.Influence of the diet components on the symbioticmicro-organisms community in hindgut ofCoptotermes formosanusShiraki[J].Appl Microbiol Biotechnol, 2006,71:907-917.
[22] 李可俊,管卫兵,徐晋麟,等.PCR-DGGE对长江河口八种野生鱼类肠道菌群多样性的比较研究[J].中国微生态学杂志, 2007,19(3):268-272.
[23] 马悦欣,王颖,杨凤,等.应用PCR-DGGE技术研究四角蛤蜊的细菌多样性[J].大连海洋大学学报,2011,26(3):55-58.
PCR-DGGE analysis of immobilized microbial diversity in digestive tract of sand worm Perinereis aibuhitensis
ZHANG Bai-dong,LIAN Bin,WANG Bin,ZHOU Yi-bing,HE Jie
(Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province,Dalian Ocean University,Dalian 116023,China)
The totalmicrobial genomic DNA was isolated from intestines of5 healthy sand wormPerinereis aibuhitensiswith biomass of approximately 4-5 g collected from shoreline of Qingdao(Shandong Province,China)and held in sterile seawater for 24 hours by Fecal DNA Extraction Kit.The V3 region of the 16S rRNA genes(approximately 230 bp)in the intestinalmicroorganismswas amplified with specific primers setof GC-341f/534r,and then DGGE(Denaturing gradient gel electrophoresis)of amplified products generated with GC-341f/534r primer set was performed with the Bio-Rad Dcode TM mutation detection system.The DGGE band profiles of immobilized bacteria in the digestive tract of the sand worm were finally generated,which provided a snapshot of composition and diversity of the immobilized bacterial population in the digestive tract of the sand worm.PCR-DGGE DNA profiles of the V3 region gene of16S rDNA showed similar profiles of5 selected individualworms and themaximal diversity levelswere observed.Some common and special bandswere identified among 5 samples,and three common predominant bands on the DGGE gelwere found,with different relative contents in each predominantband.The sequencing and blasts of the DGGE common bands indicate that the predominant intestinalmicroflora wasPropionigeniumin the 5 samples andPseudoalteromonaswas widely distributed in the digestive tract of the sand worm.The findings indicate that PCR-DGGE analysis is an effective technique for analysis of immobilized intestinalmicrobial diversity in the sand worm.
Perinereis aibuhitensis;digestive tract;microorganism;PCR-DGGE
Q959;S94
A
2095-1388(2013)05-0413-05
2013-02-16
国家 “863”计划项目 (2006AA10Z410);国家海洋公益性行业科研专项 (200805069,201305002)
张柏东 (1986-),男,硕士研究生。E-mail:zhangbaidong1986@126.com
王斌(1962-),女,教授。E-mail:wangbin@dlou.edu.cn

