4株琼胶降解菌的分离、鉴定及产酶条件分析
牟宗娟,李贵阳,茅云翔,孔凡娜,莫照兰
(1.中国海洋大学 海洋生命学院,山东 青岛 266003;2.中国水产科学院 黄海水产研究所,海水养殖生物疾病控制与分子病理学实验室,山东 青岛 266071;3.中国科学院 海洋研究所 实验海洋生物学重点实验室,山东 青岛 266071)
琼胶酶能水解琼胶多糖产生具有多种生理活性的琼胶寡糖,广泛应用于医药、食品、化工领域。琼胶酶在生物学领域也有重要的用途,用于制备海藻单细胞和原生质体、回收DNA、研究海藻细胞壁多糖组成和结构等[1]。琼胶酶分为 α-琼胶酶(EC3.2.1)和 β-琼胶酶(EC3.2.1.81)两类,来源于海洋、淡水和陆地土壤微生物。不同种属细菌、同种细菌不同菌株甚至同株细菌可以产生不同的琼胶酶,其降解产物也有不同。如从假单胞菌和交替单胞菌产生的β-琼胶酶,降解产物分别为新琼二糖[2]和新琼四糖[3];假交替单胞菌属(Pseudoalteromonas sp.)细菌 CY24产生两种β-琼胶酶,其降解产物不同[4-5]。海洋细菌是产生琼胶酶的主要来源,海洋生态环境的多样性造就了海洋细菌的多样性,目前发现海洋细菌产生的琼胶酶在分子结构、大小、底物特异性和催化活性等方面都有独特的功能[1]。本研究拟从多种海藻和刺参中分离、筛选、鉴定琼胶降解细菌,优化高效产酶的培养条件,为了解琼胶酶降解菌多样性、发现新型琼胶酶打下基础。
1 材料与方法
1.1 琼胶降解菌的分离和筛选
将从青岛各海边采集的腐烂紫菜、海带、江蓠和从烟台东方海洋公司收集的刺参肠道分别放入无菌研钵中充分研磨匀浆,用无菌海水稀释匀浆液,取合适稀释度的匀浆液涂布于 2216E海水固体培养基上,于 28℃培养观察一周。挑取周围出现明显凹陷的菌落,在2216E海水平板划线纯化两次,得到的菌株在平板培养48 h后用Lugols碘液染色观察其菌落周围产生水解透明圈的情况。取产生明显透明圈的菌株保存于–80℃冰箱。
1.2 琼胶酶活力的测定
1.2.1 粗酶液的制备
取细菌单菌落接入 2216E液体培养基培养过夜后,按5%接种量转接到新鲜的100 mL2216E液体培养基内,于28℃、摇速120 r/min培养一定时间,测定OD540后,取10 mL培养液在4℃、6 000 r/min离心20 min,收集上清液用于细菌胞外酶酶活测定。收集细胞沉淀测定湿质量后,悬浮于 10 mL PBS(pH7.4,0.2 mol),在冰浴条件下超声破碎细胞: 200 W破碎3次,每次破碎时间10 s,间隔时间10 s ,细胞破碎液经4℃、12 000 r/min离心20 min,收集上清液用于胞内酶酶活的测定[6]。另取10 mL细菌培养液直接超声破碎、离心,上清用于总酶活的测定。每个实验重复3次。
1.2.2 琼胶酶活力测定
采用 DNS测定还原糖的方法测定琼胶酶酶活[7]。用D-半乳糖为标准品制作标准曲线。1 mL粗酶液与1 mL 0.3%的琼脂底物溶液(用pH 7.4,0.2 mol的 Na2HPO4-柠檬酸缓冲液配制)混合,40℃反应 30 min后,加入1.5 mL DNS,煮沸5 min,加入蒸馏水定容至25 mL,在520 nm处测定还原糖吸收值。在上述反应条件下,每分钟产生l g还原糖所需的酶量作为一个酶活力单位。相对酶活力为每mg细菌湿重产生的酶活力单位。
1.3 产酶条件的优化
1.3.1 最适培养基的选择
比较分离菌株在 2216E海水培养基,分离培养基[8],发酵培养基[9]的生长情况。
分离培养基: 琼脂 1.3 g,NaCl 2.5 g,MgSO4·7H2O 0.05 g,NaNO30.2 g,K2HPO41.0 g,FeSO4·7H2O 0.002 g,CaCl20.02 g,蒸馏水 100 mL,pH 7.5;发酵培养基: 琼脂 1.3 g,NaCl 2.5 g,蛋白胨 0.5 g,酵母膏 1 g,FeSO4·7H2O 0.01 g,蒸馏水100 mL,pH 7.5。
取细菌单菌落分别接入上述液体培养基培养过夜后,按5%接种量转接100 mL相应液体培养基,在28℃、摇速120 r/min条件下培养,在24、36、48、72 h测定菌悬液的 OD540、细菌质量及相对酶活力,根据相对酶活力大小确定最适培养基及最适培养时间。
1.3.2 最适培养条件的选择
利用1.3.1得到的最适培养基和培养条件,在28℃、摇速120 r/min条件下,进一步优化培养温度、初始pH和琼胶底物浓度。设置的培养温度为15℃、20℃、28℃、37℃,pH为5、7.5、9,培养基琼脂质量分数为0.1%、0.3%、0.5%、1%。在上述培养条件下测定各菌株的相对酶活力。
1.4 菌株鉴定
按常规方法对细菌进行革兰氏染色和形态观察,用API ID-32E试剂条(生物-梅里埃,法国)进行生理生化指标检测,根据伯杰氏细菌鉴定手册确定细菌的分类地位;按文献方法进行细菌16SrDNA序列扩增和序列比对分析[10]。利用软件Clustal X(V2.0)进行序列完全比对,比对参数中Output Files选项中选择NEXUS format。利用Mrmodeltest3.7估计核酸最优取代模型,应用于BI的构建。利用MrBayes3.1.2,采用GTR+I+G模型,构建贝叶斯树[11]。综合生理生化、16SrDNA序列比对聚类分析结果综合鉴定细菌。
1.5 琼胶酶基因的克隆和分析
根据文献设计 PCR引物扩增细菌琼胶酶基因agaB[12],正向引物 agaB-for: 5'-ccacttagcactagagcccgtaa-3',反向引物agaB-rev: 5'-tgtacctagcagattgcactccc-3'。在25 µL的PCR反应体系中含有: 10×PCR buffer 2.5 µL,dNTP(2.5 mmol)各 1 µL,引物(10 µmol)各 1 µL, Taq DNA 聚合酶(5U/UL)0.5µL,DNA 模板 1 µL。PCR 反应条件为: 95℃预变性4 min;94℃变性30 s,45℃复性45 s,72℃延伸3 min,30个循环;72℃温育10 min。用MEGA4软件建树分析。
1.6 数据分析
利用SPSS软件分析各组数据的平均值、显著性等。
2 结果与分析
2.1 菌株的筛选以及菌落形态观察
利用 2216E海水固体培养基从腐烂藻体以及刺参肠道内筛选到多株具有降解琼胶能力的菌株,可观察到在菌落处形成明显的凹陷,随培养时间延长,菌落周围出现可辨的透明圈。用Lugols碘液覆盖菌落周边区域,被降解的琼胶不能与碘着色而呈明显的透明区(图 1,A),未降解琼胶与碘结合呈棕褐色(图1,B)。根据培养48 h后菌落产生的透明圈半径初步判断酶活力大小,分别从紫菜、海带、江蓠、刺参肠道内筛选到 4株产琼胶酶酶活力最高的菌株,编号分别为QJ、HD、JL和HS,其透明圈半径(不包括菌落半径)大小均在0.7~0.8 cm。
在2216E平板上,HD、JL和HS菌株生长较快,24 h即可长出半径0.1~0.3 cm的菌落,可观察到菌落周围有明显的透明圈,48 h后菌落处琼胶出现凹陷,一周后平板琼胶完全分解、液化。QJ菌株在2216E的生长较缓慢,培养36 h的菌落半径约为0.1 cm,但可观察到菌落周围产生明显凹陷或透明圈,培养过程中未出现平板液化现象。
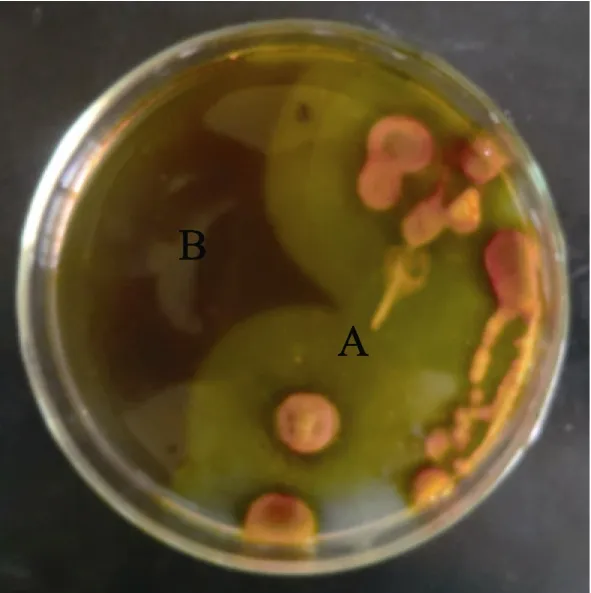
图1 用卢戈氏碘液染色的细菌平板Fig.1 Bacterial cultures stained with Lugol iodine
2.2 最佳产酶条件的确定
2.2.1 最适培养基的确定
将4株菌分别在液体的2216E海水培养基、分离培养基、发酵培养基中摇床培养,测定 24、36、48和72 h的OD540值以及相对酶活力。结果如图2所示,4株菌在3种培养基生长36 h的OD540值最大(P<0.05),相对酶活力最高 (P<0.05);4株菌在2216E培养基上生长最好,在36 h的相对酶活力在80 U/mg以上。结果还显示,菌株QJ在3种培养基的OD540低于其他3株菌,在发酵培养基中基本不生长;在静置培养时,4株细菌易形成菌膜,在摇床培养时有絮状出现。
结果表明,4株菌最适产酶培养基为2216E海水培养基,产酶最适培养时间为36 h。
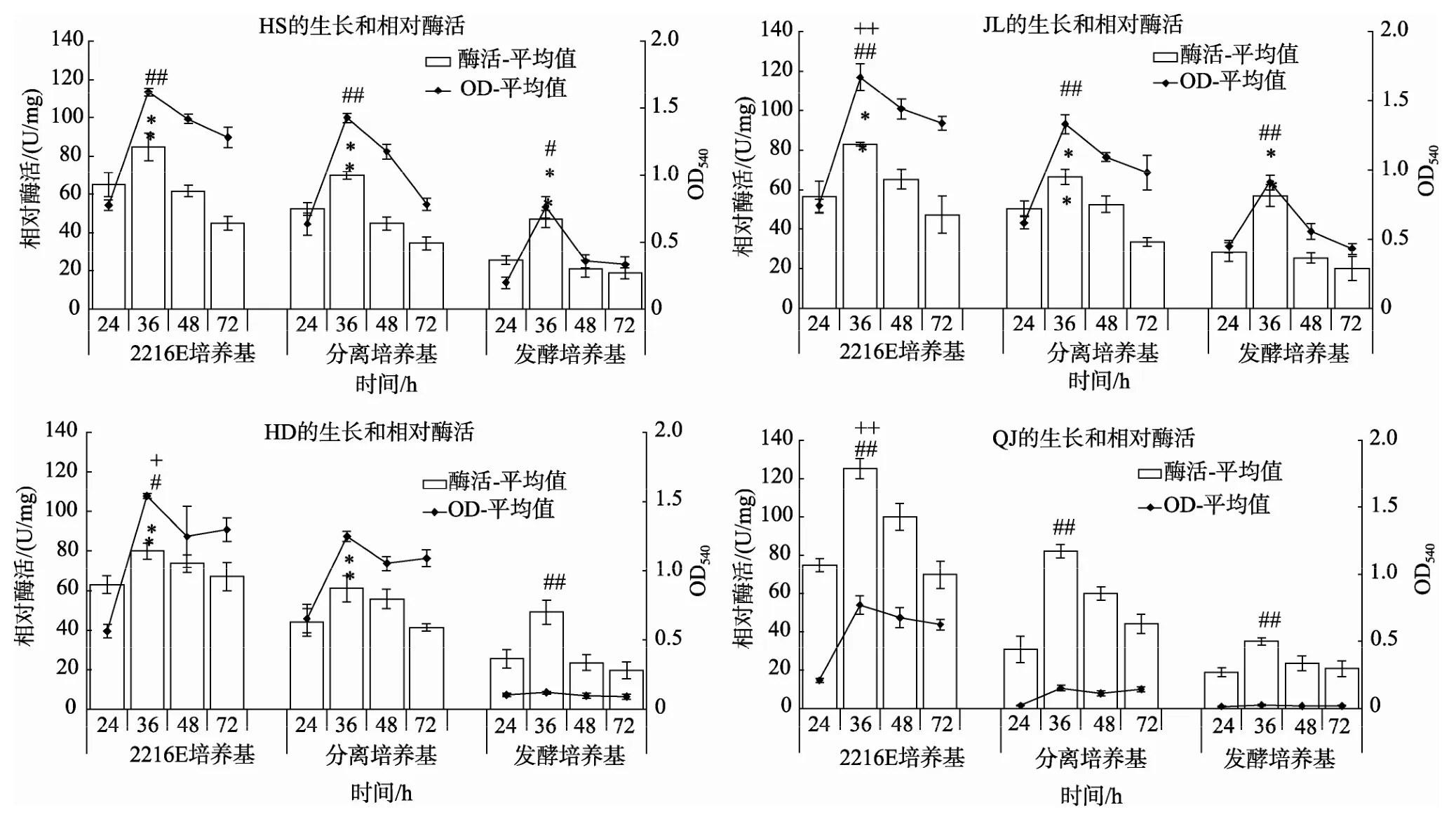
图2 琼胶降解菌在3种培养基的相对酶活和OD540Fig.2 The relative enzymatic activities and OD540values of the agarase-producing bacteria cultured in three different media
2.2.2 其他培养条件的优化
以 2216E液体培养基为基本培养基,120 r/min摇速培养细菌 36 h,确定培养温度、pH、琼胶底物浓度对细菌产酶的影响。如图 3所示,培养温度为28℃时,4株菌显示最高相对酶活(P<0.05)。培养基起始pH为7.5时,4株菌的相对酶活最高;除JL和HD在 pH7.5的相对酶活与 pH5.0时的相比有显著差异外(P<0.05),其他无明显差异(P>0.05)。培养基琼胶浓度为0.30%时,4株菌的相对酶活最高,除HD、JL、HS在0.30%琼胶的酶活与0.50%的酶活相比差异不显著外,其他均呈显著差异(P<0.05)。
综合上述结果,4株菌产酶的最佳培养条件:2216E液体培养基为基本培养基,培养基起始pH7.5,琼胶底物浓度0.30%,培养温度28℃,120 r/min摇床,培养时间36 h。
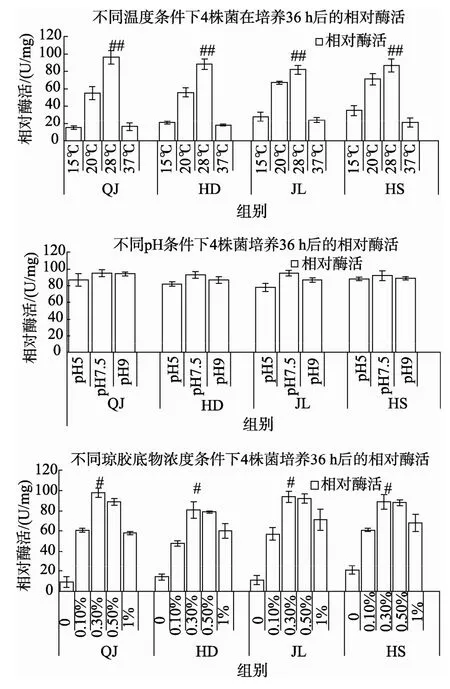
图3 细菌在不同温度、pH、底物浓度培养36 h的相对酶活力Fig.3 The relative enzymatic activities under different temperature,pH and substrate concentrations for 36 h
2.3 细菌细胞产酶部位的确定
制备 4株细菌胞外酶和胞内酶的粗酶液,测定酶活并比较大小。结果如图4所示,在4株细菌的细胞外检测到的相对酶活力显著高于胞内相对酶活力(P<0.01),胞外酶活占总酶活的90%以上。
2.4 琼胶降解菌菌株的鉴定
4株琼胶降解菌均为G-,菌落光滑、圆形、白色或奶油色。电镜下HD、JL和HS菌株的细菌形态为直或弯短杆状,有周生纤毛;QJ为长杆状,无周生或极生鞭毛。16SrDNA基因序列相似性比较结果显示,菌株HD、JL和HS与白色噬琼胶菌最相似,相似性均达到98%,QJ与Simiduia sp.、糖噬胞菌属和船蛆杆菌属最相似,相似性分别为94%、94%、93%。HD、JL、QJ、HS的 16SrDNA基因序列登录号为JX070038-JX070041。16SrDNA基因序列的聚类分析显示,HD、JL、HS与白色噬琼胶菌属聚为一类,QJ与糖噬胞菌属、船蛆杆菌属、假单胞菌属、海微菌属(Marinimicrobium sp.)等聚为一簇,但形成一个独立的分支(图5)。生理生化反应指标显示,菌株HD、JL和HS的生理生化反应指标一致,与文献描述的白色噬琼胶菌的特征相近;菌株QJ与 Simiduia sp.、糖噬胞菌属、船蛆杆菌属的特征有不同(表1)。综合生理生化特征和16SrDNA基因序列分析结果,将HD、JL和HS鉴定为白色噬琼胶菌;菌株QJ的分类地位待定。
2.5 琼胶酶基因的克隆
从JL、HD和HS基因组DNA扩增得到β-琼胶酶基因(登录号为 JX070035-JX070037),均含有一个2868bp的阅读框,三者之间的相似性达到98%~99%,与白色噬琼胶菌属和弧菌属菌株的 β-琼胶酶蛋白质序列相似性为 98%~99%,相关序列聚类分析如图 6所示。
3 讨论
本研究利用琼胶作为碳源,从青岛海域的腐烂海带、紫菜、江蓠和烟台的养殖刺参中分离到多株琼胶降解菌,经过比较菌落降解琼胶产生的凹陷直径大小,得到 4株具有较高琼胶降解能力的琼胶降解菌并进行了发酵条件的优化。目前多数的研究以含有较高蛋白胨和酵母膏的发酵培养基来培养琼胶降解菌,以获得高酶量[9],本研究结果表明以寡营养的 2216E培养基就能获得较高的酶量,可以减少培养成本。

图4 4株菌胞内外酶活的比较Fig.4 Comparison of intracellular and extracellular enzymes activities in the 4 strains
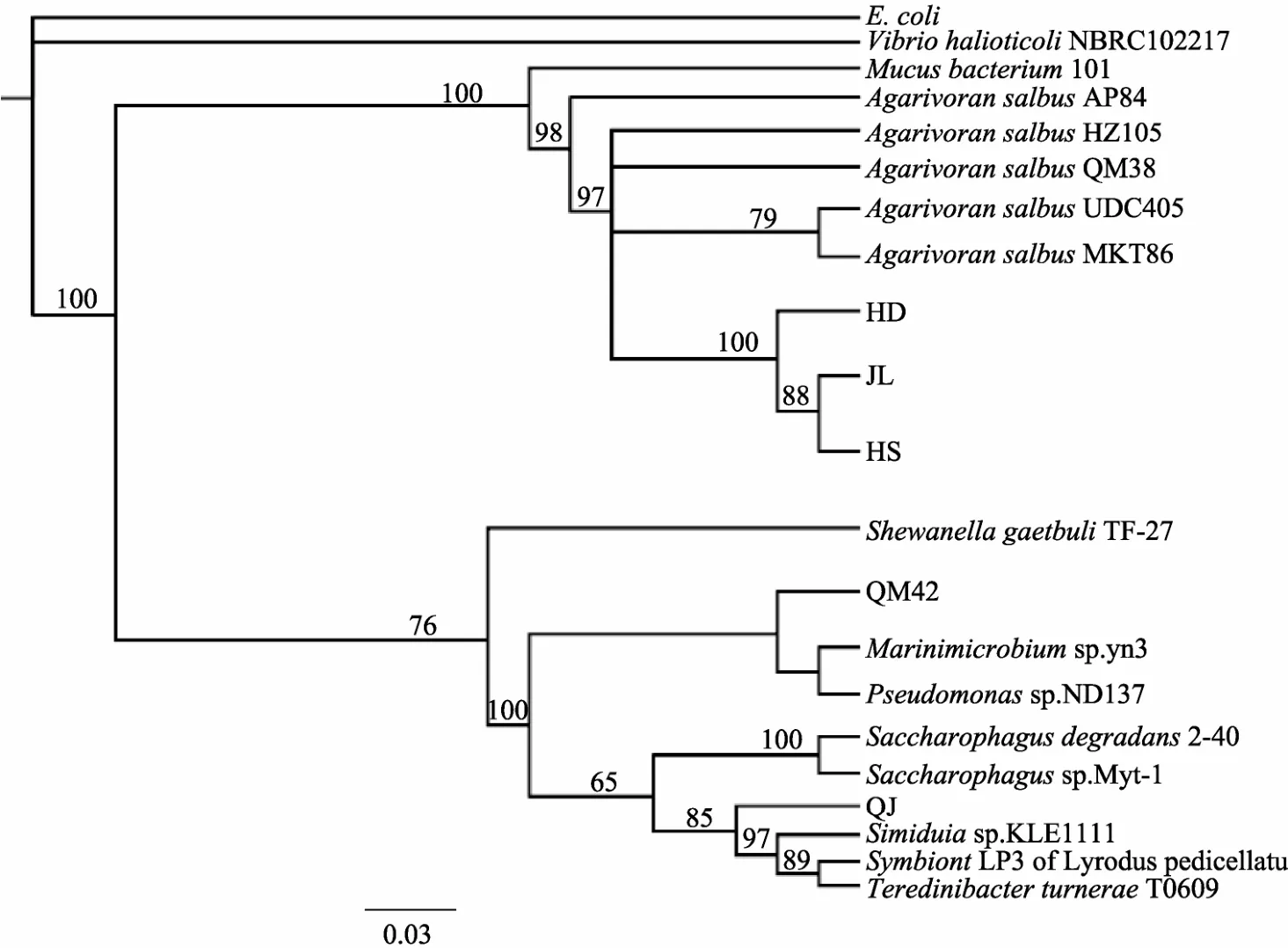
图5 琼胶降解菌的系统发育树分析Fig.5 Phylogenetic tree analysis diagram of the agarase-producing bacteria
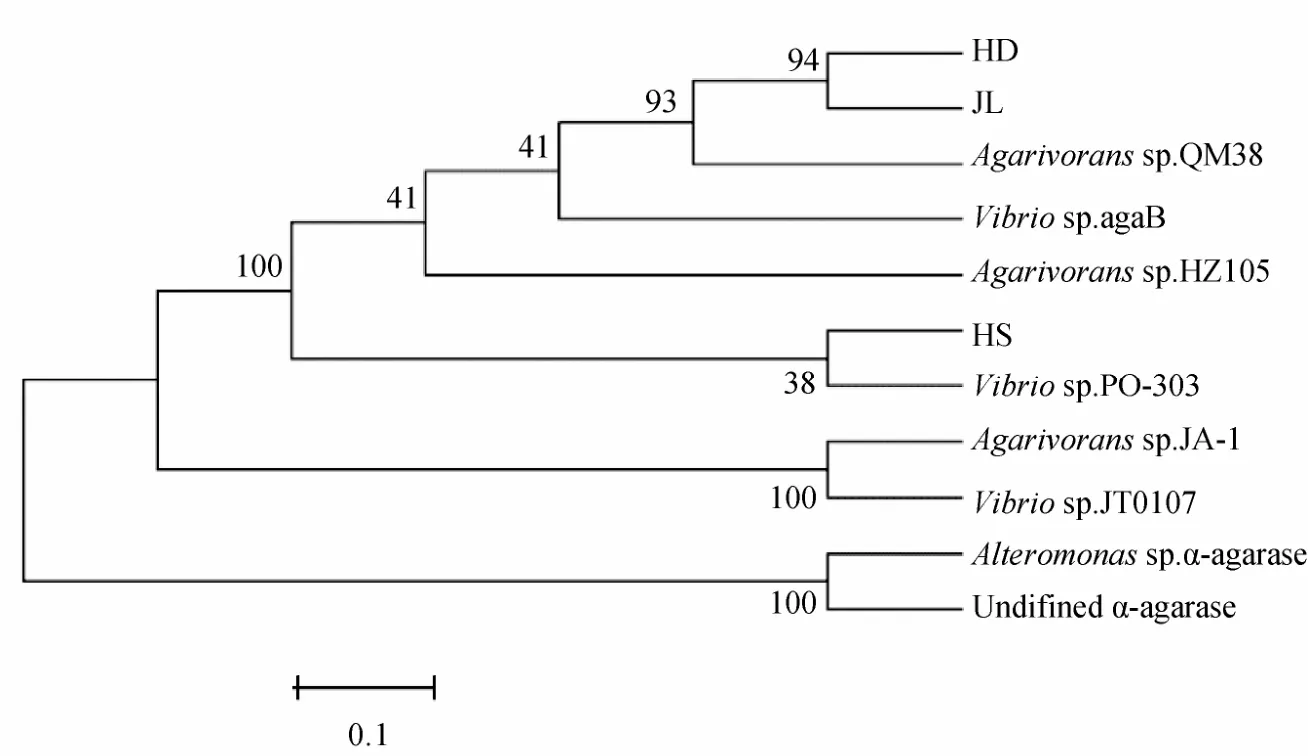
图6 琼胶酶基因的系统发育树分析Fig.6 Phylogenetic tree analysis diagram of the agarase genes
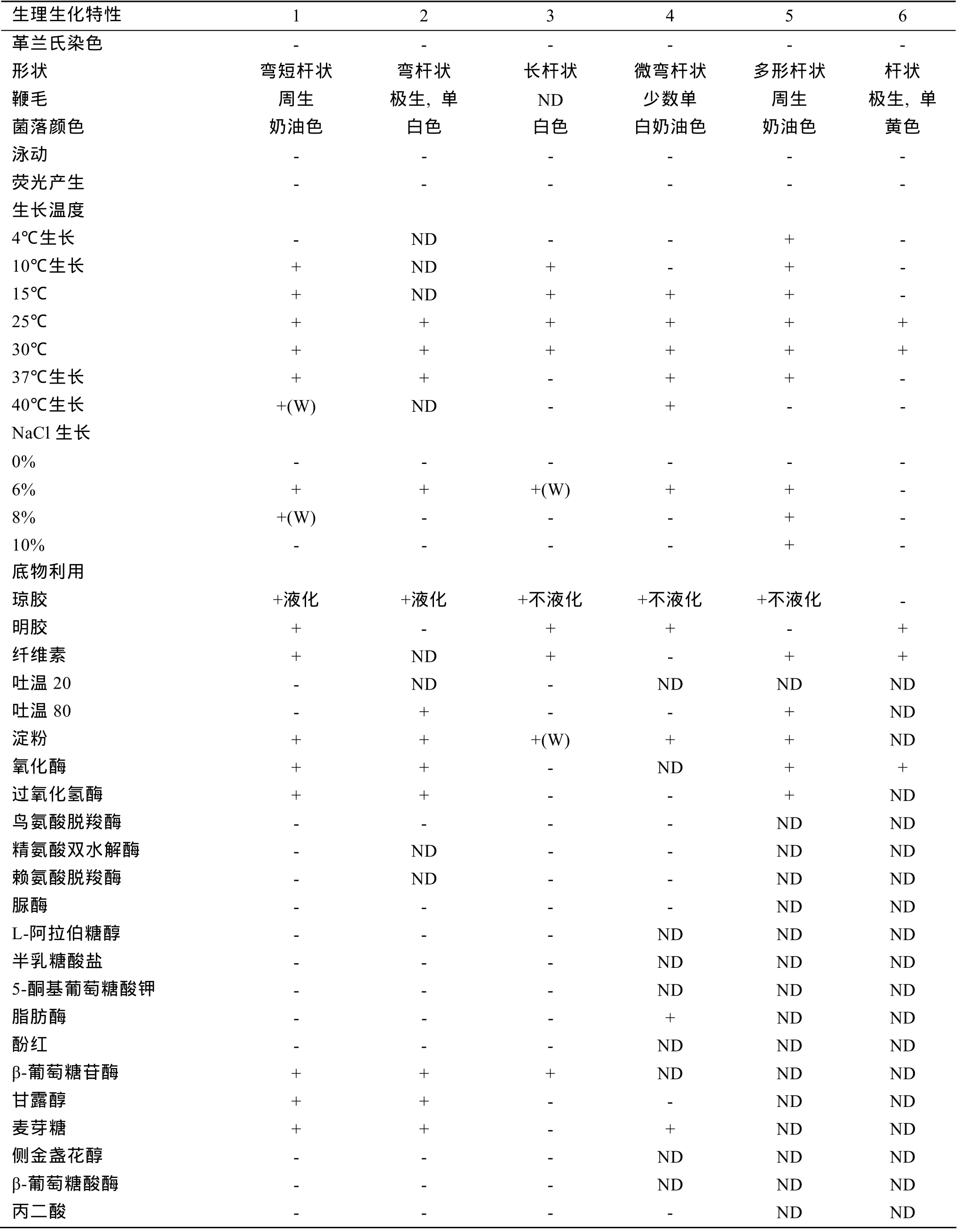
表1 4株琼胶降解菌及相关菌株生理生化性状Tab.1 Comparison of the physiological and biochemical characteristics between the agarase-producing bacteria and the other relative bacterial species
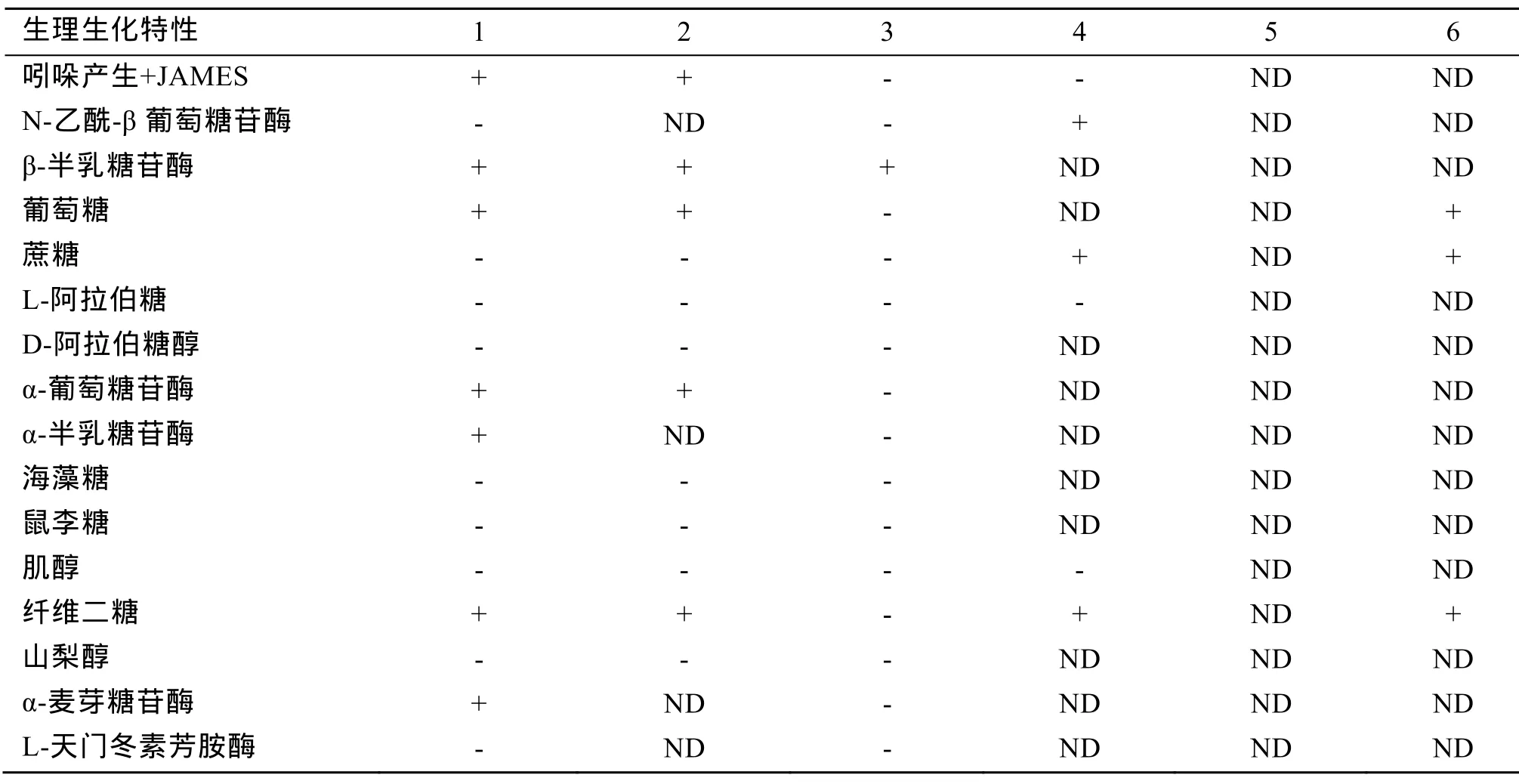
续表
细菌鉴定结果表明,菌株HD、JL和HS为白色噬琼胶菌,与一些学者的研究结果一致[13-16]。已从白色噬琼胶菌属分离纯化到胞外β-琼胶酶,其降解产物有的为新琼二糖[15,17]、有的为新琼四糖、六糖组成的琼胶寡糖[16,18];理化性质也有所不同,如 Long等[18]发现一种新的β-琼胶酶在较广的 pH范围内保持较高的活性。本研究从HD、JL和HS扩增得到β-琼胶酶基因,这些酶的特性有待进一步确定。
从紫菜分离得到的 QJ菌株的分类地位与Simiduia sp.、糖噬胞菌属、船蛆杆菌属等的亲缘关系较近,这些属的细菌都具有分解复杂多糖的特性,如船蛆杆菌能分解纤维素[19],Simiduia agarivorans能降解琼脂[20-21],糖噬胞菌属能分解多种复杂多糖,如褐藻胶、几丁质、海带多糖、淀粉、木聚糖等[21]。QJ菌株的生理生化特征与这些细菌有所不同,其分类地位需要进一步确定。
[1]Fu X T,Sang M K.Agarase: review of major sources,categories,purification method,enzyme characteristics and applications [J].Mar Drugs,2010,8: 200-218.
[2]Groleau D,Yaphe W.Enzymatic hydrolysis of agar:purification and characterization of beta neoagarotetraose hydrolase from Pseudomonas atlantica [J].Can J Microbiol,1977,23(6): 672-679.
[3]Morrice L M,Mclean M W,Williamson F B,et al.Beta-agarase Ⅰ and Ⅱ from Pseudomonas atlantic:purification and some properties [J].Eur J Biochem,1983,135(3): 553-558.
[4]Ma C,Lu X,Shi C,et al.Molecular cloning and characterization of a novel beta-agarase,AgaB,from marine Pseudoalteromonas sp.CY24 [J].J Biol Chem,2007,282: 3747-3754.
[5]Lu X Z,Chu Y,Wu Q Q,et al.Cloning,expression and characterization of a new agarase-encoding gene from marine Pseudoalteromonas sp.[J].Biotechnology Letters,2009,31(10): 1565-1570.
[6]Jean H E.The role of bacteria in the digestion of seaweed by the ablone Hallotls [J].Aquaculture,1997,155:377-386.
[7]Miller G L,Blum R,Glennon W E,et al.Measurement of carboxy methy Ieellulase activity [J].Analytical Biochmistry,1960,(2): 127-132.
[8]陈惠源,蔡俊鹏,刘江涛.海洋细菌产琼胶酶的条件优化 [J].现代食品科技,2005,21(3): 48-50.
[9]王晓燕,桑卫国,章宗铭.腐烂紫菜中琼胶酶高产菌株的筛选及鉴定 [J].食品科技,2009,34(7): 2-5.
[10]Woese C W R,Gutell R,Gupta R,et al.Detailed analysis of the higher-order structure of 16S-like ribosomal ribonucleic acids [J].Microbiology Reviews,1983,47(3):621-669.
[11]Ronquist F,Teslenko M,et al.MrBayes 3.2: efficient bayesian phylogenetic inference and model choice across a large model space [J].Systematic Biology,2012,61(3):539-542.
[12]Sugano Y,Terada I,Arita M,et al.Purification and charactetrization of a new agarase from a marine bacterium,Vibrio sp.strain JT0107 [J].Appl Environ Microbiol,1993,59(5): 1549-1554.
[13]杜宗军.海洋琼胶降解细菌的多样性研究与Agarivorans albus QM38 β-琼胶酶基因的克隆与表达[D].青岛: 中国海洋大学,2007.
[14]Kurahashi M,Yokota A.Agarivorans albusgen.nov.,sp.nov.,a gamma-proteobacterium isolated from marine animals [J].Int J Syst Evol Microbiol,2004,54:693-697.
[15]Fu X T,Lin H,Kim S M.Purification and characterization of a novel β-agarase,AgaA34,from Agarivorans albus YKW-34 [J].Appl Microbiol Biotechnol,2008,78:265-273.
[16]Du Z J,Wang J,Yang L J,et al.Identification of a marine agarolytic bacterium Agarivorans albus QM38 and cloning and sequencing its beta-agarase genes [J].Acta Oceanol Sin,2011,30(1): 118-124.
[17]Lee D G,Park G T,Kim N Y,et al.Cloning,expression,and characterization of a glycoside hydrolase family 50 β-agarase from a marine Agarivorans isolate[J].Biotechnol Lett,2006,28: 1925-1932.
[18]Long M X,Yu Z N,Xu X.A Novel β-Agarase with high pH stability from marine Agarivorans sp.LQ48 [J].Mar Biotechnol,2010,12: 62-69.
[19]Daniel L D,Wendy M,Noelle M T,et al.Teredinibacter turnerae gen.nov.,sp.nov.,a dinitrogen-fixing,cellulolytic,endosymbiotic c-proteobacterium isolated from the gills of wood-boring molluscs (Bivalvia:Teredinidae)[J].International Journal of Systematic and Evolutionary Microbiology,2002,52: 2261-2269.
[20]Wung Y S,Liu T Y,Lin S Y,et al.Simiduia agarivorans gen.nov.,sp.nov.,a marine,agarolytic bacterium isolated from shallow coastal water from Keelung,Taiwan [J].International Journal of Systematic and Evolutionary Microbiology,2008,58: 895-900.
[21]Nathan A E,Jose M G,Michael B H,et al.Saccharophagus degradans gen.nov.,sp.nov.,a versatile marine degrader of complex Polysaccharides [J].International Journal of Systematic and Evolutionary Microbiology,2005,55: 1545-1549.

