畜产动物过氧化物酶体增殖物激活受体基因的表达模式及功能研究进展
黄春红肖调义刘巧林胡 毅赵玉蓉
(1.湖南农业大学动物科学技术学院,长沙 410128;2.湖南文理学院生命科学学院,动物学湖南省高校重点实验室,常德 415000)
过氧化物酶体增殖物激活受体(peroxisome proliferator-activated receptors,PPARs),于 1990 年被英国科学家 Issemann等[1]首先发现,是一种新型的固醇类激素受体,也是配体依赖的核转录因子,属于核受体第1亚家族C群(NR1C)。目前,国内外研究人员对啮齿动物和人体PPARs基因的结构与多态性、组织中表达模式、影响表达的因素、信号传导途径、在脂类代谢调控等方面的功能进行了广泛和深入的研究。在啮齿动物和人体中的研究表明,PPARs是调控脂肪酸及其衍生物对相关基因表达的一组受体[2-4],PPARs也因此被称为脂肪酸感受器。PPARs主要通过作用于特定目标基因来调控脂肪酸的合成与分解代谢(β氧化和 ω 氧化)[5]、脂肪细胞的增殖与分化[6]、心肌的能量与脂肪平衡[7]、胎盘发育[8]、炎症反应[9-10]等过程,并影响胰岛素敏感性和糖类代谢[11]。PPARs信号传导途径已成为脂类代谢的3条重要的信号传导途径之一[12]。近年来,PPARs在畜产动物上功能的研究也逐渐开展了起来,并取得了相应的进展,现对PPARs基因在畜产动物组织中的表达模式及功能研究进行综述。
1 PPARs的亚型、结构与作用模式
PPARs具有过氧化物酶体增殖物激活受体α[PPARα,属于核受体第1亚家族 C群第 1位(NR1C1)]、过氧化物酶体增殖物激活受体 β[PPARβ,也称为过氧化物酶体增殖物激活受体δ(PPARδ),属于核受体第1亚家族 C群第2位(NR1C2)]和过氧化物酶体增殖物激活受体γ[PPARγ,属于核受体第1亚家族 C群第3位(NR1C3)]3 种亚型[13],其中 PPARβ 在鱼类以及PPARγ在哺乳动物中又包括多种亚型。PPARs的3种亚型由不同的基因编码,它们的功能及在组织中的表达也存在差异[14]。
PPARs的结构分成6个区,分别为 A、B、C、D、E、F(图 1)[15],其中 5'端 A/B 区称为配体非依赖的转录激活域,该区在3种亚型间的保守性很差,主要通过磷酸化作用来调节配体和受体间的亲和力,进而调节PPARs活性;C区称为DNA结合域(DNA biding domain,DBD),与其他转录因子一样,该区高度保守,包含2个锌指结构和1个α螺旋的DNA结合基序;D区为灵活的铰链区(hinge),主要连接DBD和配体结合域(ligand biding domain,LBD),参与 PPARs的构象变化;3'端E/F区为保守性较差的LBD。LBD中有1个Y型的疏水袋,PPARβ的配体袋较狭窄,一些相对较大的PPARα、PPARγ配体不能与之结合;受LBD氨基酸种类的影响,PPARα的 LBD较亲脂,PPARγ的 LBD较亲水,故饱和脂肪酸易于和PPARα结合,亲水的不饱和脂肪酸则是PPARγ良好的配体。PPARs通过 LBD与配体结合,将PPARs转化成能够结合DNA并调节基因表达的活性形式。关于PPARs配体的研究比较多,根据来源分为天然配体和人工合成配体2种,根据作用则有激动剂和拮抗剂2类;激动剂或拮抗剂与LBD结合后,PPARs能稳定地结合协同激动子或协同抑制子[16]。另外,E区也在核酸定位和受体二聚化过程中起作用,与其他的核受体超家族成员一样,二聚化是PPARs发挥活性所必需的。PPARs首先与其配体结合,当PPARs被配体激活后,与顺式维甲酸受体(RXR)结合形成异源二聚体并发生构象变化,再与位于靶基因启动子中的过氧化物酶体增殖物反应元件(peroxisome proliferator response element,PPRE)结合,调节靶基因的转录活性[17]。PPARs的作用模式见图2。

图1 PPARs结构示意图Fig.1 Structure diagram of PPARs[15]
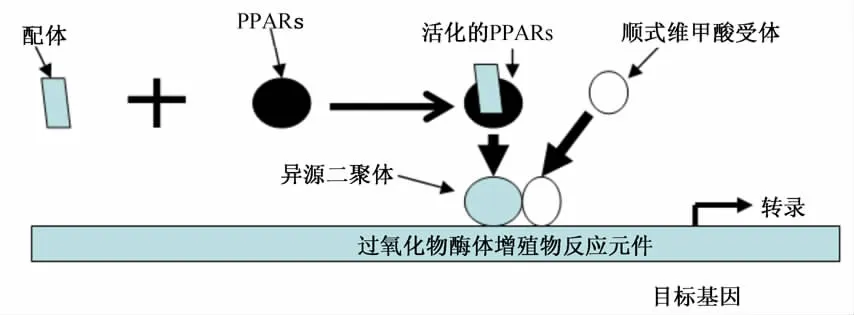
图2 PPARs作用模式示意图Fig.2 Function model diagram of PPARs[5]
2 畜产动物PPARs基因的组织表达及功能研究
2.1 猪PPARs基因的组织表达及功能研究
猪 PPARα、PPARβ研究的相对较少,PPARγ研究的相对较多。Sundvold等[18]对猪体10种组织PPARs基因的 Northern blot检测发现,PPARα基因高度表达于肝脏和肾脏,中度表达于心肌、骨骼肌和小肠。PPARα基因的多态位点在巴马小型猪、五指山小型猪、中国农大小型猪3个品系小型猪之间的分布存在差异,说明猪的品种不同PPARα基因的多态性也不同[19]。
PPARγ基因位于猪第 13号染色体上[20],具有γ1和γ2 2种亚型。PPARγ1基因在八眉猪的内脏脂肪、皮下脂肪、肌肉、肾脏、肝脏、心脏、肺脏和脾脏这8种组织中均有表达,其中在内脏脂肪和皮下脂肪组织中的表达量最高,在肺脏中的表达量最低;PPARγ2基因也高表达于八眉猪的内脏脂肪、皮下脂肪、脾脏和心脏,但在其他4种组织中的表达量均较低[21]。也有研究表明PPARγ基因限制性表达于猪的脂肪组织和脾脏[22]。PPARγ基因在二元杂交母猪(Ds)、三元杂交母猪(DDs)和三元杂交公猪(DDS)骨骼肌中均能有效表达,表达量与体脂和肌间脂肪代谢间呈一定关联度,但关联度受山猪血统和性别影响。例如,PPARγ基因在1/2山猪血统的母猪骨骼肌中有高表达,且能够促进脂肪酸的合成代谢,显著增加皮下和肌内大理石纹以及肌内脂肪含量,改善猪肉品质;PPARγ基因在1/4山猪血统的杂交公猪和母猪的骨骼肌中表达量则下降,脂肪沉积量显著减少[23]。PPARγ基因在猪的皮下前脂肪细胞中也有高表达,提示PPARγ基因除了影响猪体脂肪酸代谢外,在猪皮下前脂肪细胞分化过程中也起着非常重要的作用[24]。研究发现,约克夏猪、杜洛克猪、长白猪、金华猪、碧湖猪和嵊县花猪共6个品种猪的PPARγ2基因编码区第175 bp处存在 A175G单核苷酸多态性,根据A175G突变位点与背膘厚性状存在显著相关,推测PPARγ2可能是影响猪胴体性状的潜在分子育种标记[25]。约克夏猪、杜洛克猪、长白猪、汉普夏猪PPARγ基因BsrI位点的多态性显著影响其日增重、眼肌面积、大理石纹和肌肉嫩度,显示PPARγ基因的BsrI位点可以作为猪肉质性状的候选基因位点[26]。PPARγ基因表达量与初产母猪的体况,如体重和背膘厚、生殖激素水平、生殖激素受体的表达等有关,PPARγ基因可能通过调控下丘脑-垂体-卵巢轴促性腺激素释放激素受体(GnRH-R)、促卵泡生成素(FSH)、促黄体生成素(LH)、雌二醇(E2)等生殖激素基因的表达而影响初产母猪断奶后发情[27]。在大白猪和长白猪中,PPARγ基因有3个单核苷酸多态性(SNP)位点,分别位于5'端调控区的第220、243 bp和第6外显子的第147 bp处;进一步的研究发现,PPARγ基因SNP位点与猪的窝产仔数之间存在显著的相关性[28]。
PPARα、PPARβ和 PPARγ mRNA 除在成年母猪的胰腺组织中未检测到表达以外,在其他17种组织(子宫绒毛膜、皮下脂肪、卵巢、大肠、脾脏、大脑、肾上腺、心脏、肺、小肠、脊髓、子宫蜕膜、胃、肝脏、背最长肌、膀胱、肾脏)中均广泛表达,其中PPARα mRNA在子宫绒毛膜、皮下脂肪和卵巢中表达量较高;PPARβ mRNA在子宫绒毛膜、卵巢和大肠中表达量较高;PPARγ mRNA在背最长肌、皮下脂肪和卵巢中表达量较高。根据3种亚型PPARs mRNA在卵巢和/或子宫绒毛膜中都有较高的表达,推断PPARs可能与猪的繁殖性能相关[29]。母猪妊娠第12、18及25天的子宫内膜组织免疫组化结果显示,PPARβ基因主要在猪胚胎附植早期表达,且在多数细胞中均有表达,并以绒毛膜滋养层细胞中表达量最高;PPARα和PPARγ基因则主要在猪胚胎附植中、晚期表达,也在多种细胞中均有表达,说明PPARs可能通过参与猪胚胎附植来影响猪的繁殖性能[30]。
猪PPARs基因在体内许多组织和器官中均可表达,但是不同亚型在体内的高表达部位不同,其中PPARγ基因主要高表达于脂肪组织中。猪PPARγ基因通过调控体脂肪的代谢影响猪肉肌间脂肪含量、背膘厚和胴体性状,通过调控生殖激素的表达影响母猪的产后发情。PPARα、PPARβ和PPARγ 3种亚型在不同的胚胎附植阶段可能不同程度地影响猪的繁殖性能。
2.2 家禽PPARs基因的组织表达及功能研究
家禽PPARs的研究相对较少,且以 PPARα、PPARγ研究为主,目前所查阅的文献资料中尚未发现关于家禽PPARβ的报道。家禽PPARs的研究内容包括PPARs基因的克隆、组织表达、多态位点分析及功能研究几个方面。
基因克隆方面:据报道,鸭PPARα基因的cDNA全长1 430 bp,最长开放阅读框为1 407 bp,共编码468个氨基酸[31];鹅 PPARα和 PPARγ基因的 cDNA序列长度分别为1 407和1 428 bp[32]。组织表达方面:PPARα mRNA除在爱拔益加(AA)肉鸡的胸肌中无表达和在脾脏中表达量较低以外,在脑、肺脏、肾脏、心脏、小肠、胃、肝脏、脂肪组织中均有较高表达;PPARγ mRNA则在肝脏和肌肉中无表达,但在上述其他6种组织中均有表达,其中在脂肪组织中表达量最高;Northern blot检测结果则显示PPARα mRNA只在心脏、肝脏、肾脏和胃这4种组织中表达,以肝脏杂交信号最强;PPARγ mRNA只在脂肪和肾脏中表达,以脂肪组织杂交信号较强[33]。Diot等[34]的 Northern blot检测结果也显示鸡PPARα mRNA较高表达于肝脏、心脏、肾脏和尾脂腺,该研究结果与孟和等[33]所得结果相似。多态位点与功能研究方面:PPARα基因的第4外显子在安卡×固始鸡资源群中存在1个多态性位点,该位点的多态性显著影响鸡的腹脂重和腹脂率,即PPARα基因影响鸡体脂肪代谢,并可以作为分子标记应用于鸡脂肪性状的标记辅助选择[35]。PPARα基因在 AA肉鸡以及石岐杂鸡、北京油鸡、白耳鸡3个中国地方鸡品种中也检测到了单碱基突变位点,该突变位点显著影响鸡群的腹脂重及胴体性状,推测PPARα基因是影响鸡体脂肪代谢的主效基因或与控制该性状的主效基因连锁,因此可以作为分子标记用于鸡脂肪性状的标记辅助选择[36],该研究结果与田亚东等[35]所得结果相似。除了PPARα基因的表达可以影响家禽的腹脂重和胴体性状以外,家禽PPARγ基因的表达也影响脂肪沉积。PPARγ基因在高脂型肉鸡腹部脂肪中有高表达,在低脂型肉鸡腹部脂肪中则表现为低表达。PPARγ基因表达量下降后,鸡脂肪细胞的增殖能力增强,但分化能力减弱,这说明PPARγ基因可能是调控鸡脂肪细胞增殖与分化的关键因子[37]。另外,PPARγ基因在所有被检朗德鹅的腹部脂肪中均呈高度表达[38],进一步说明家禽PPARγ基因在家禽脂肪沉积中具有重要作用。
综上所述,家禽PPARα、PPARγ基因在不同组织和器官中的表达具有一定的特异性,二者均对脂肪代谢起调控作用,影响家禽体脂的沉积,进而影响腹脂率和胴体品质。
2.3 反刍动物PPARs基因的组织表达及功能研究
反刍动物PPARs的研究也以PPARγ为主,对PPARα和PPARβ的研究较少,这一点与猪类似。据报道,PPARα和PPARγ基因在广灵大尾羊和小尾寒羊不同部位的脂肪组织中都有表达,不同脂肪组织中PPARα基因的表达量存在显著差异,浅层脂肪组织中的表达量低于深层脂肪组织,品种、性别和月龄对PPARα基因表达量则无显著影响。PPARγ基因在不同脂肪组织中的表达量差异极显著,在不同性别中差异显著,但品种、月龄对PPARγ基因的表达量无显著影响[39]。PPARγ基因在湖羊不同部位肌肉中的表达量随月龄的变化而变化,具体体现为先上升后下降。PPARγ基因表达量影响湖羊羔羊生长早期的肌内脂肪含量,但部位不同影响程度不同,湖羊肌肉中PPARγ基因表达量与肌内脂肪含量呈不同程度的负相关[40]。PPARγ基因在雄性哈萨克羊肌肉中的表达量随着日龄的增加呈下降趋势,而肌内脂肪含量则随日龄的增加而增加,表明雄性哈萨克羊生长发育早期PPARγ基因的表达量与肌内脂肪含量呈负相关[41],该研究结果与郝称莉等[40]所得结果相似,并推测湖羊肌肉中PPARγ的调控作用可能不是发生在转录水平上,而可能是发生在蛋白质翻译水平上。不同亚型PPARs mRNA在绵羊妊娠第7、9、12、14、17 天子宫内的表达结果显示,PPARα mRNA在妊娠的第7~17天之间表达量下降,而 PPARβ mRNA表达量持续增强,PPARγ mRNA表达量呈规律性变化,表明PPARβ基因可能与绵羊繁殖性能相关[42]。
对牛PPARs基因的研究表明,西杂牛PPARγ2基因表达量与月龄和组织类型有关,表现为在肌肉和脂肪中的表达量均随着月龄的增加而持续上升,在24和36月龄时表达量急剧上升;在不同脂肪中的表达量都极显著高于不同类型肌肉,在腹部脂肪中的表达量又稍高于背部脂肪;但背最长肌肌内脂肪含量和剪切力与PPARγ2基因表达量无显著相关性,表明PPARγ2对西杂牛的肉质影响不大[43]。秦川牛、南阳牛、郏县红牛、鲁西牛、安格斯、夏南牛这6个品种牛的PPARα基因第7外显子的SNP位点是影响牛背膘厚和胴体长的主效数量性状基因座(QTN)或与之紧密连锁,推断该位点可作为肉牛选育的候选分子标记[44]。PPARα基因第5外显子的SNP位点突变则是影响肉牛背膘厚、眼肌面积和系水力的QTN或与之紧密连锁[45],故可作为肉牛产肉性状的标记辅助选择。荷斯坦奶牛PPARα基因的44 087(G/A)位点GA基因型乳蛋白率显著高于GG基因型,在炎热环境下GA基因型产奶量也显著高于其他基因型,提示该位点的GA基因型可能有利于提高中国荷斯坦奶牛的产奶性能,可以作为人工选择高产奶性能奶牛的分子标记[46]。
牛、羊PPARs基因的研究结果进一步说明PPARγ基因主要在哺乳动物的脂肪组织中表达。PPARα基因外显子的多态性不仅影响肉牛的背膘厚、眼肌面积和胴体性状,还影响奶牛的牛奶品质与产奶量,故可在肉牛和奶牛生产性能相关性状的选择方面充当分子标记。PPARγ基因主要影响生长早期羊的肌内脂肪含量,对牛的肉质影响不大,表明PPARγ对体脂肪代谢和肉质性状影响可能与动物种类和生长阶段有关。
2.4 鱼类PPARs基因的组织表达及功能研究
水产动物PPARs的研究主要集中在鱼类上,研究内容主要集中在基因克隆、组织表达和功能研究3个方面。相对于畜禽,鱼类PPARs基因的克隆研究得比较多(表1),但关于基因序列全长的报道并不多,许多研究只对某些物种的PPARs基因的部分片段进行了克隆,或者只是对某一物种PPARs的一种亚型进行了克隆。在组织表达方面,除了对表1中鱼类PPARs基因进行了研究以外,还对其他多种鱼类进行了研究,例如在斑马鱼(Danio rerio)中也检测到了PPARs的3种亚型,其中PPARα基因主要表达于肝脏实质细胞、肾脏近端小管、肠上皮细胞和胰腺组织;PPARβ基因则广泛表达于肝脏、肾近端和远端小管、肾小球、胰腺等众多组织中;PPARγ基因在胰腺、肠道和性腺中表达量则很微弱[58]。鲽鱼(Pleuronectes plates)和金头鲷(Sparus aurata)几乎所有的鱼体组织中均有PPARγ基因表达,其表达范围比在哺乳动物组织中的表达更加广泛;PPARα和PPARβ基因表达范围则相对较窄,与哺乳动物相近[59]。也有报道表明PPARα和PPARγ基因在草鱼黏液中未检测到表达[49]。以上报道均表明鱼类PPARs基因的表达也存在组织特异性。
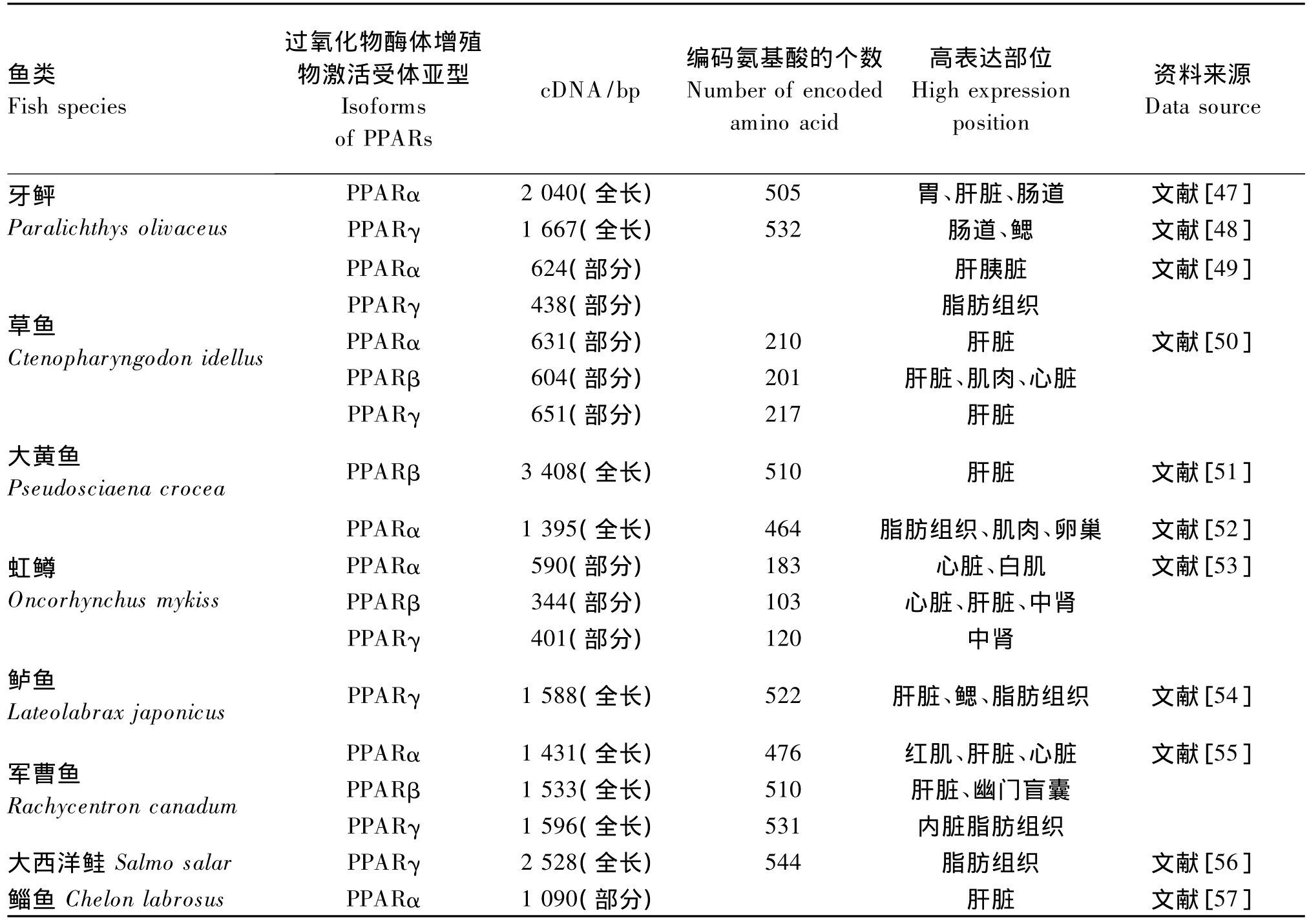
表1 鱼类PPARs基因的克隆与组织表达情况Table 1 Cloning and tissue expression characterization of PPARs genes in fish species
目前有关鱼类PPARs基因功能的研究报道较多。鱼类PPARs基因的主要功能是通过调控脂质代谢途径与脂质转运相关酶的表达来影响鱼体的脂质代谢[60]。PPARα基因表达上调可以刺激摄食高脂饲料鱼体内脂肪酸的β氧化过程,减少鱼体脂肪酸和甘油三酯的合成,并参与高度不饱和脂肪酸(HUFA)的合成调控[61]。鱼类 PPARγ基因的功能与哺乳动物可能存在区别,但基本类似,主要是调节脂类合成和脂肪生成相关蛋白基因的表达,在鱼体脂肪沉积过程中起关键作用。研究表明,经哺乳动物PPARα激动剂处理过的大西洋鲑[62]及非诺贝特处理过的虹鳟[63],肝细胞中辅酶A氧化酶(ACO)活性均有所提高,虹鳟肝细胞过氧化物体β氧化过程增强,但虹鳟肝脏线粒体中ACO和肉碱棕榈酰转移酶1(CTP1,长链脂肪酸β氧化过程的限速酶)活性不受影响。饲料中添加十四烷基硫乙酸能够活化PPARα基因,并显著提高大西洋鲑肝脏线粒体中脂肪酸的β氧化,但不影响ACO活性[64]。营养因素对PPARs基因及脂质代谢的研究表明,草鱼饲料中适量添加n-3 HUFA可以上调PPARα、脂蛋白脂酶(LPL)等脂代谢关键基因表达,降低体脂沉积,调控体脂在组织间分配[65]。饲料中添加共轭亚油酸可以显著提高大黄鱼肝脏中 PPARα、PPARγ 基因的表达[66],进而提高大西洋鲑肝脏中脂肪酸的β氧化[67],以及金头鲷肝脏中 ACO活性[68]。军曹鱼 PPARα和PPARγ基因的表达量都与脂质沉积具有显著的相关性,表明二者在肝脏、肌肉和内脏脂肪的脂肪代谢和储存中发挥着重要作用[55]。Leaver等[59]的研究表明,金头鲷和鲽鱼肝脏PPARα2基因的表达量取决于饲料的营养水平,并且禁食条件下肝脏PPARα2基因的表达量上调,摄食条件下表达量下调,推测PPARα2基因的表达可能受鱼类摄食和饲料营养水平的调控。同时,研究还发现不饱和脂肪酸与哺乳动物PPARα和PPARβ的特异配体分别可以激活鱼类PPARα和PPARβ基因表达,而脂肪酸和哺乳动物PPARγ的特异配体则不能激活金头鲷和鲽鱼PPARγ基因的表达,鱼类PPARγ基因可能被一些不确定的复合物激活,这些未知复合物很可能与脂肪酸有关。
3 小结
猪和反刍动物PPARs研究以PPARγ为主,对PPARα、PPARβ的研究较少。畜禽及鱼类PPARβ的研究均比较少,可能与PPARβ配体袋较狭窄,一些相对较大的PPARα、PPARγ配体不能与之结合有关。国内对反刍动物PPARs的研究较国外多,但在组织表达方面研究得相对较窄,涉及组织种类较少,对繁殖性状影响的研究也不多。家禽PPARs研究的内容相对较少,范围也比较窄。总的看来,畜产动物PPARs在脂类代谢调控方面的功能与啮齿动物和人类PPARs的研究结果相似,其中对脂肪代谢有影响的主要是PPARα和PPARγ。尽管人类对畜禽PPARs在脂类代谢、肉质性状、繁殖性状等候选基因或与之相关的分子标记方面已经做了一些基础的研究,但是在畜禽的研究不如对啮齿动物和人类的研究深入,有关PPARs在畜禽体内的详细调控机理以及具体的信号传导途径也不是很清楚,需进一步研究。与畜禽相比,水产动物PPARs功能的研究也较浅,深入研究鱼类PPARs在脂类代谢调控方面的功能以及机理,并寻找到鱼类脂肪性状的候选基因或与之相关的分子遗传标记,这在渔业生产实践中将具有非常重要的意义。
[1]ISSEMANN I,GREEN S.Activation of a member of the steroid hormone receptor superfamily by peroxisome proliferators[J].Nature,1990,347:645 - 650.
[2]GERMAIN P,STAELS B,DACQUET C,et al.Overview of nomenclature of nuclear receptors[J].Pharmacological Reviews,2006,58(4):685 -704.
[3]SAMPHTH H,NTAMBI J M.Polyunsaturated fatty acid regulation of genes of lipid metabolism[J].Annual Review of Nutrition,2005,25:317 -340.
[4]DESVERGNE B,WAHLI W.Peroxisome proliferatoractivated receptors:nuclear control of metabolism[J].Endocrinology Review,1999,20(5):649 -688.
[5]NAKAMURA M T,CHEON Y,LI Y,et al.Mechanisms of regulation of gene expression by fatty acids[J].Lipids,2004,39(11):1077 -1083.
[6]BRAISSANT O,WAHLI W.Differential expression of peroxisome proliferatoractivated receptor-alpha,-beta,and-gamma during rat embryonic development[J].Endocrinology,1998,139:2748 -2754.
[7]QINGLIN Y,YUHAO L.Roles of PPARs on regulating myocardial energy and lipid homeostasis[J].International Journal of Molecular Medicine,2007,85:697-706.
[8]MICHALIK L,DESVERGNE B,WHALI W.Peroxisomeproliferator-activated receptorsand cancers:complexstories[J].Nature Reviews Cancer,2004,4(1):61-70.
[9]RICOTE M,LI A C,WILSON T M,et al.The peroxisome proliferator-activated receptor-gamma is a negative regulator of macrophage activation[J].Nature,1998,391:79 -82.
[10]JIANG C,TING A T,SEED B.PPAR-gamma agonists inhibit production of monocyte inflammatory cy-tokines[J].Nature,1998,391:82 - 86.
[11]DESVERGNE B,WHALI W.Peroxisome proliferator activated receptors:nuclear control of metabolism[J].Endocrinology Review,1999,20:649 -688.
[12]李龙江,余瑜.脂代谢的信号转导途径[J].儿科药学杂志,2005,11(1):7 -9.
[13]BRAISSANT O,FOUFELLE F,SCOTTO C,et al.Differential expression of peroxisome proliferator-activated receptors(PPARs):tissue distribution of PPAR-alpha,-beta,and-gamma in the adult rat[J].Endocrinology,1996,137:354 -366.
[14]ELBRECHT A,CHEN Y,CULLINAN C A,et al.Molecular cloning,expression and characterization of human peroxisome proliferator activated receptors γ1 and γ2[J].Biochemical and Biophysical Research Communicatons,1996,224:431 -437.
[15]ERIC B,JEANO C G,RURH R.Advances in understanding the regulation of apoptosis and mitosis by peroxisome proliferator-activated receptors in preclinical models relevance for human health and disease[J/OL].Comparative Hepatological,2003(2):1 - 8.[2003 - 01 -31].http://www.ncbi.nlm.nih.gov/pmc/articles/PMC151270/.
[16]叶卫雄,严瑾.PPARs配体的研究进展[J].浙江中医药大学学报,2009,33(4):606 -610.
[17]JPENBERG A,JEANNIN E,WAHLI W,et al.Polarity and specific sequence requirements of peroxisome proliferator activated receptors(PPAR)/retinoid X receptor heterodimer binding to DNA:a functional analysis of malic enzyme gene PPAR response element[J].Journal of Biological Chemistry,1997,137:272-281.
[18]SUNDVOLD H,GRINDFLEK E,LIEN S.Tissue distribution of porcine peroxisome proliferator activated receptor α:detection of an alternatively spliced mRNA[J].Gene,2001,273:105 -113.
[19]赵玉琼,姚春涛,刘亚千,等.我国特有三个小型猪品系PPARα基因的多态性分析[J].中国比较医学杂志,2008,18(10):41 -44.
[20]GRINDFLEK E,SUNDVOLD H,LIEN S,et al.Rapid communication:physical and genetic mapping of the peroxisome proliferator receptor gamma(PPAR gamma)gene to porcine chromosome 13[J].Journal of Animal Science,2000,78(5):1391 -1392.
[21]王博,吴江维,杨公社.PPARγ在八眉猪不同组织中的表达差异[J].畜牧兽医学报,2008,39(3):273 -277.
[22]GRINDFLEK E,SUNDVOLD H,KLUNGLAND H,et al.Characterization of porcine peroxisome proliferator-activated receptor C1 and C2:detection of breed and age difference in gene expression [J].Biochemical and Biophysical Research Communications,1998,249:713-718.
[23]龚兰,金邦荃,章熙霞,等.杂交山猪骨骼肌PPARγ、RARα基因与脂代谢关系的研究[J].食品科学,2011,32:62 -67.
[24]刘海峰,阎振鑫,董涵,等.猪前脂肪细胞诱导分化过程中 PPARγ、DECR1和 ECHS1基因表达模式[J].四川农业大学学报,2010,28(2):215 -218.
[25]陈哲,王公金,于建宁,等.PPARγ2基因外显子1多态性对猪胴体性状的影响[J].江苏农业学报,2012,28(2):332 -336.
[26]REBECCA E,STEVEN M,KEITH I,et al.An investigation into the genetic controls of pork quality[J].National Swine Improvement Federation,2000,77:846-852.
[27]KONG L J,WANG A G,FU J L,et al.Peroxisome proliferator-activated receptor c is involved in weaning to estrus of primiparous sows by regulating the expression of hormone genes in hypothalamus-pituitary-ovary axis[J].Asian and Australian Journal of Animal,2007,20(3):340 -350.
[28]GUIYING W,LUJIN K,PENG H,et al.Effect of polymorphism in the peroxisome proliferator-activated receptor gamma gene on litter size of pigs[J].Molecular Biology Reports,2011,38:1807 -1812.
[29]KONG L J,WANG A G,FU J L,et al.Study on the characteristics of tissue expression of peroxisome proliferators-activated receptors in pigs[J].Chinese Journal of Animal Science,2006,42(19):1 -4.
[30]李京励,孔路军,王爱国,等.猪PPARs基因在胚胎附植期子宫内膜表达特性研究[J].中国畜牧杂志,2012,48(11):15 -17.
[31]马云,王云云,张晓婷,等.鸭PPARα基因结构及功能的生物信息学分析[J].浙江大学学报:农业与生命科学版,2011,37(4):371 -379.
[32]孟和,李辉,王宇祥.鹅PPARα基因全长 cDNA的克隆和序列分析[J].遗传,2004,26(4):469 -472.
[33]孟和,李辉,王宇祥.鸡PPARs基因组织表达特性的研究[J].遗传学报,2004,31(7):682 -687.
[34]DIOT C,DUAIRE M.Characterization of a cDNA sequence encoding the peroxisome proliferat or-activated receptor α in the chicken[J].Poultry Science,1999,78:1198-1202.
[35]田亚东,亢娟娟,孙桂荣,等.PPARα基因对安卡×固始鸡资源群胴体品质的遗传效应分析[J].华北农学报,2010,25(6):14 -18.
[36]孟和,王桂华,王启贵,等.鸡PPARα基因单核苷酸多态与脂肪性状相关的研究[J].遗传学报,2002,29(2):119-123.
[37]王丽,那威,王宇祥,等.鸡PPARγ基因的表达特性及其对脂肪细胞增殖分化的影响[J].遗传,2012,34(5):454-464.
[38]苏胜彦,李齐发,刘振山,等.朗德鹅填饲后不同组织PPARγ基因mRNA表达量差异的初步研究[J].畜牧兽医学报,2008,39(7):879 -884.
[39]林婄婄,高中元,袁亚男,等.PPARα和PPARγ基因在不同脂尾型绵羊脂肪组织中的发育性表达研究[J].畜牧兽医学报,2012,43(9):1368 -1376.
[40]郝称莉,李齐发,乔永,等.湖羊肌肉组织 H-FABP和PPARγ基因表达水平与肌内脂肪含量的相关研究[J].中国农业科学,2008,41(11):3776 -3783.
[41]HUANG Z G,XIONG L,LIU Z S,et al.The developmental changes and effect on IMF content of H-FABP and PPARγ mRNA expression in sheep muscle[J].Acta Genetica Sinica,2006,33:507 -514.
[42]CAMMAS L,REINAUD P,BORDAS N,et al.Developmental regulation of prostacyclin synthase and prostacyclin receptors in the ovine uterus and conceptus during the peri-implantation period[J].Reproduction,2006,131(5):917 -927.
[43]李健.西杂牛 PPARγ2、PGC-1a、MEF2C基因表达量及其与肌内脂肪含量、嫩度的相关分析[D].硕士学位论文.雅安:四川农业大学,2010.
[44]樊月圆,富国文,昝林森.过氧化物酶体增殖激活受体α(PPARα)基因突变对牛的背膘厚和胴体长的影响[J].家畜生态学报,2012,33(1):14 -18.
[45]樊月圆,昝林森,王洪宝,等.牛过氧化物酶体增殖激活受体α(PPARα)基因第5外显子多态性与胴体、肉质性状的相关性[J].农业生物技术学报,2010,18(4):713 -718.
[46]方文良,黄金明,王长法,等.中国荷斯坦牛 PPARα基因多态性研究及其与耐热性能的关联分析[J].中国畜牧兽医,2010(8):165-170.
[47]HYUN K C,HEE J K,HVE Y K,et al.Characterization of paralichthys olivaceus peroxisome proliferatoractivated receptor-a gene as a master regulator of flounder lipid metabolism[J].General and Comparative Endocrinology,2012,175:39 -47.
[48]HYUN K C,HEE J K,BO H N,et al.Molecular cloning and characterization of olive flounder(Paralichthys olivaceus)peroxisome proliferator-activated receptor γ[J].General and Comparative Endocrinology,2009,163:251 -258.
[49]林亚秋,吉红,郑玉才.草鱼PPARα和PPARγ基因的克隆与组织表达差异[J].水产学报,2011,30(2):94-97.
[50]陈亮,梁旭方,瞿春梅,等.草鱼过氧化物酶体增殖物激活受体(PPARs)基因cDNA序列的克隆及其组成型表达[J].暨南大学学报:自然科学版,2011,32(1):80-87.
[51]钱伦,钱云霞,童丽娟.大黄鱼PPARβ全长 cDNA的克隆和组织表达[J].生物学杂志,2010,27(6):1-4.
[52]贾成霞,张照斌,张清靖.虹鳟PPARα基因克隆、序列分析及其组织表达分布[J].中国水产科学,2012,19(4):707 -714.
[53]CARLA B P,PEDRO R,EDUARDO R,et al.Identification and organ expression of peroxisome proliferator activated receptors in brown trout(Salmo trutta f.fario)[J].Biochimica et Biophysica Acta,2005,1731:88-94.
[54]钱云霞,杨孙孝,梁洪,等.鲈PPARγ基因的克隆、组织表达及其抗体制备[J].水产学报,2010,34(8):1156-1164.
[55]TSAI M L,CHEN H Y,TSENG M C,et al.Cloning of peroxisome proliferators activated receptors in the cobia(Rachycentron canadum)and their expression at different life-cycle stages under cage aquaculture[J].Gene,2008,425(12):69 -78.
[56]ANDERSEN Q,EIJSINK V G,THOMASSEN M.Multiple variants of the peroxisome proliferator-activated receptor(PPAR)γ are expressed in the liver of Atlantic salmon(Salmosalar)[J].Gene,2000,255:411-418.
[57]DAMIEN R,IBON C,MIREN P,et al.Cloning and expression pattern of peroxisome proliferator-activated receptor a in the thicklip grey mullet Chelon labrosus[J].Marine Environmental Research,2006,62:113 -S117.
[58]IBABE A,GRABENBAUER M,BAUMGART E,et al.Expression of peroxisome proliferator-activated receptors in zebrafish(Danio rerio)[J].Histochemistry of Cell Biology,2002,118:231 -239.
[59]LEAVER M J,BOUKOUVALA E,ANTONOPOULOU E,et al.Three peroxisome proliferator-activated receptorisotypes from each of two species of marine fish[J].Endocrinology,2005,146(7):3150 -3162.
[60]DESVERGNE B,JJPENBERG A,DEVEHAND P R,et al.The peroxisome proliferators-activated receptors at the cross-road of diet and hormonal signaling[J].Journal of Steroid Biochemistry and Molecular Biology,1998,65(1):65 -74.
[61]NAKAMURA M T,CHEON Y,LI Y,et al.Mechanisms of regulation of gene expression by fatty acids[J].Lipids,2004,39(11):1077 -1083.
[62]RUYTER B O,ANDERSEN A K,FARRANTS T,et al.Peroxisome proliferator activated receptors in Atlantic salmon(Salmo salar):effects on PPAR transcription and acyl-CoA oxidase activity in hepatocytes by peroxisome proliferators and fatty acids[J].Biochimica et Biophysica Acta,1997,1348:331 -338.
[63]DU Z Y,DEMIZIEUX P,DEGRACE J,et al.Alteration of 20∶5n-3 and 22∶6n-3 fat contents and liver peroxisomal activities in fenofibrate-treated rainbow trout[J].Lipids,2004,39:849 - 855.
[64]MOYA-FALCON C E,HYATTUM T N,TRAN M S,et al.Phospholipid molecular species,beta-oxidation,desaturation and elongation of fatty acids in Atlantic salmon hepatocytes:effects of temperature and 3-thia fatty acids[J].Comparative Biochemistry and Physiology B:Biochemistry & Molecular Biology,2006,145:68 -80.
[65]JI H,LI J,LIU P.Regulation of growth performance and lipid metabolism by dietary n-3 highly unsaturated fatty acids in juvenile grass carp,Ctenopharyngodon idellus[J].Comparative Biochemistry and Physiology,2011,159:49 -56.
[66]赵占宇.共轭亚油酸(CLA)对大黄鱼脂肪代谢、免疫、肉品质及PPAR基因表达的影响[D].博士学位论文.杭州:浙江大学,2008.
[67]LEAVER M J,TOCHER A,OBACH L,et al.Effect of dietary conjugated linoleic acid(CLA)on lipid composition,metabolism,and gene expression in Atlantic salmon(Salmo salar)tissues[J].Comparative Biochemistry and Physiology A:Molecular&Integrative Physiology,2006,145:258 -267.
[68]DIZ A D,MENOYO S,PEREZ-BENAVENTE J,et al.Conjugated linoleic acid affects lipid composition,metabolism,and gene expression in gilthead sea bream(Sparus aurata L)[J].The Journal of Nutrition,2007,137:1363-1369.

