DNA平衡离子凝聚的动态光散射分析*
林 瑜 杨光参 王艳伟
(温州大学物理与电子信息工程学院,温州 325035)
(2012年12月19日收到;2013年2月2日收到修改稿)
1 引言
DNA作为一种生物大分子,与中性高分子和简单电解质相比,它是一种有着不同特性的聚电解质[1,2].当溶解于极性溶剂时,DNA将离解成高带电量的聚离子,且周围布满了许多小的平衡离子.由于DNA附近不同化合价的平衡离子分布不均匀,导致DNA的长程静电相互作用和熵发生变化,从而改变DNA的构型.
通常人们利用平衡离子凝聚理论[3,4],蒙特卡洛模拟[5]或者通过解泊松-玻尔兹曼方程[6]来研究聚电解质周围的平衡离子分布,这些理论研究结果表明平衡离子在聚电解质表面凝聚形成一种薄层结构.其中Manning的平衡离子凝聚理论通常用于描述在溶液中小离子与聚电解质的非特异性结合.强带电的线性聚电解质将吸附它周围的平衡离子,吸附的平衡离子中和了聚电解质上的电荷.当溶液里只存在一种化合价为Z的平衡离子,根据Manning理论[2]可知聚电解质上的电荷被中和的部分与原来的电荷之比θ可写为

DNA是遗传信息的载体,细胞中的DNA必须凝聚成特定结构装载到细胞核中.因此,DNA凝聚在人工基因转移与转染过程[7,8]、基因治疗[9,10]和基因重组[11,12]等方面有潜在的应用.从Manning的平衡离子凝聚理论可知,平衡离子能中和DNA上的大部分电荷,但DNA上还有部分未被中和的电荷,DNA分子之间还存在着静电排斥力.如何克服这个剩余静电排斥力,Shklovskii[13]研究小组提出了一种新的理论解释.他们认为,由于强的横向排斥效应,平衡离子在DNA表面形成了一种类似Wigner晶体结构的强关联流体结构.吸附在DNA表面的平衡离子屏蔽了DNA的电荷,从而使得DNA间的排斥力减小,当DNA之间的静电排斥力小于静电吸引力时,DNA发生凝聚.多价平衡离子在DNA表面形成的强关联流体结构使得平衡离子的结合能大于kBT,这导致了更多的平衡离子吸附在DNA表面.当多价平衡离子的浓度达到一个临界值时,吸附在DNA表面的平衡离子的总电荷在绝对值上将大于DNA的电荷,使得DNA的净电荷的符号发生了反转,这种现象就叫做DNA的电荷反转.
在过去的几十年里,有很多关于DNA和平衡离子相互作用的理论和实验研究[14-16].如Vuleti等[1]利用荧光关联光谱(FCS)和介电光谱研究了在非常低的单价盐浓度(csalt<0.05 mM,1 M=1 mol/L)的水溶液中棒状聚电解质上的有效电荷.他们验证了Manning的凝聚和电导模型适用于描述处于稀溶液中的棒状聚电解质.Ma等[17]通过凝胶电泳实验测量了DNA(1000—5000 bp)在不同结构和不同化合价的平衡离子的溶液情况下的有效电荷.他们的实验结果与两种平衡离子共存的理论结果相符合.Besteman等[18]利用动态光散射(DLS)和磁镊技术研究了多价平衡离子导致DNA发生电荷反转.实验结果表明对于四价平衡离子,DNA的电泳迁移率随着平衡离子浓度的增大由负变为正,即DNA的电荷发生反转.而对于三价平衡离子,DNA的电泳迁移率随着离子浓度的增大逐渐减小但始终为负.在理论上,Luan等[19]通过分子动力学模拟研究了平衡离子导致的DNA电荷反转.他们发现对于三价或者四价平衡离子,随着平衡离子浓度的增大,DNA的迁移率发生反转.但是对于单价或者二价平衡离子,随着平衡离子浓度的增大,DNA的迁移率逐渐减小但不会反转.
然而,对于自由溶液中DNA的电泳迁移率与不同化合价的平衡离子之间的关系还没有系统的实验研究.本文利用DLS技术系统地研究了DNA的电泳迁移率与平衡离子(化合价1—4)浓度的关系.同时,利用原子力显微镜(AFM)研究了DNA分子构型随不同化合价平衡离子的变化.
2 实验过程
2.1 实验材料
实验选用的噬菌体λ-DNA(原始浓度是500 ng/µL)购买于 New England Biolabs公司,使用前无需提纯.实验选用的单价和二价平衡离子分别为NaCl和MgCl2,二者均购买于Inalco-America公司.纯水(18.2 MΩ·cm)是经密理博超纯化系统去离子与净化处理.多价离子三氯六氨络合钴([Co(NH3)6]3+)和精胺([C10N4H30]4+)购买于Sigma公司.
2.2 DLS实验的样品制备
动态光散射仪器采用Malvern公司的Zetasizer NanoZS设备,光源是氦氖气体激光(λ=633 nm),探测角为90°,利用M3-PALS技术测量电泳迁移率.实验中所需的缓冲液为Tris溶液(10 mM Tris,pH=8.0),用该缓冲液配制不同浓度的平衡离子溶液.在不同浓度的平衡离子溶液中加入DNA,使得DNA的最终浓度为1 ng/µL.混合液在室温条件下培育10 min,然后取1 mL的混合液注入Zeta电位毛细管样品池,置于动态光散射的样品槽中进行测量.
2.3 AFM实验的样品制备与扫描
实验仪器选用日本岛津的SPM-9600原子力显微镜,工作模式为轻敲模式.实验中采用1 Hz的扫描速度采集图像,图像像素是512×512.通过AFM本身的off-line软件分析DNA图像的高度、宽度等信息.将云母片切成1 cm×1 cm,新解离即用.实验所用的缓冲液为1×TE溶液(10 mM Tris-HCl,1 mM EDTA,pH=8.0),用该缓冲液配制不同浓度的平衡离子与DNA的混合溶液,其中DNA的浓度为1 ng/µL.混合液在室温条件下培育10 min,然后用移液器取20µL的混合液滴在新解离的云母表面,室温培育5 min后,每次用20µL的超纯水冲洗,共冲洗10—15次,去除云母表面未吸附的DNA分子和杂质,接着用氮气吹干,放于干燥箱1—2 h后扫描.所有的实验结果至少重复三次,确保实验结果的准确性.
3 结果与讨论
3.1 单价和二价平衡离子与DNA的相互作用
DNA可看成一种线性的强带电的聚电解质,且每个磷酸根基团都带有一个单位的负电荷,当溶于极性溶剂中,平衡离子将在DNA周围凝聚成一个薄层.根据Stern(斯特恩)模型,这个薄层应该分成两个部分:第一部分包括吸附在表面的一层离子,形成了一个内部紧密的双电层,称为Stern层;第二部分为Gouy-Chapman扩散层.按照Stern模型,DNA分子在运动时,应该与Stern层不可分离,似乎切动面就是Stern面.但是由于固体表面吸附的离子仍然保持者溶剂化(至少在扩散层的一侧),故DNA分子运动除了与吸附的平衡离子一起外,还会带着一薄层溶剂化的液体,因此实际运动的切动面应该在Stern面的更右侧一点,这个切动面上的电势就称为ζ(Zeta)电势或动电势.
当溶液处于电场中,在外加电场的作用下,DNA分子向着与自己的电荷相反的电极方向迁移,而平衡离子与之作相对运动,这种现象称为电泳.DNA的电泳迁移率µ由下式给出:
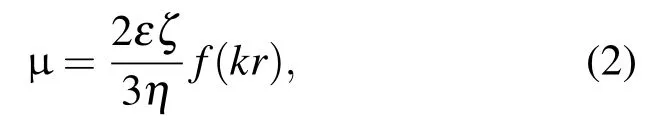
其中ζ为Zeta电势,ε为溶剂的介电常数,η为溶剂的黏度系数,f(kr)为Henry函数.通过DLS实验我们可以直接测量DNA分子的Zeta电位,如图1所示,利用(2)式可以得到DNA的电泳迁移率.
在外加电场的作用下,假设DNA分子的电泳迁移率只与DNA上的有效电荷有关[17],则根据Manning的平衡离子凝聚理论可得缓冲液中只包含单价平衡离子(下标a)时的电泳迁移率与缓冲液中只包含Z价平衡离子(下标z)时的电泳迁移率之比为
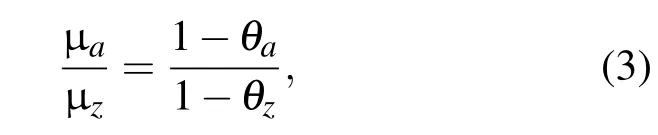
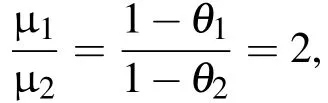
本文利用DLS技术分别测量了在缓冲液中只包含平衡离子Na+或Mg2+的情况下DNA的电泳迁移率随平衡离子浓度的变化,如图2所示.从图2中可以发现当平衡离子的浓度c<5 mM时,DNA的电泳迁移率随着平衡离子浓度的增大而逐渐减小,且平衡离子的化合价越大,DNA的电泳迁移率减小得越快.当c≥5 mM时,DNA的电泳迁移率逐渐趋于稳定,且DNA分子的电泳迁移率µ1:µ2≈ 2:1,即缓冲液中只包含 Na+或 Mg2+时的电泳迁移率之比约为2:1,实验所得结果与Manning的理论值相符合.对于单价或者低浓度的二价的平衡离子,它们只是中和DNA双螺旋结构上的磷酸根基团所带的负电荷,而无法使DNA发生凝聚.通过AFM实验观察到,在低浓度的二价平衡离子的条件下DNA在溶液中是以自由舒展的形态存在,如图4(a)所示.正是由于低浓度的二价离子无法导致DNA凝聚,所以二价平衡离子常用于DNA分子的AFM成像.

图1 DNA的Zeta电位分布图
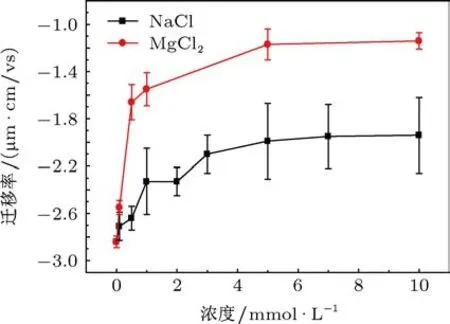
图2 DNA的电泳迁移率随平衡离子浓度的变化(NaCl用黑色方块线表示,MgCl2用红色圆点线表示,DNA的浓度为1 ng/µL)
3.2 三价平衡离子与DNA的相互作用
众所周知,多价阳离子能导致DNA的形态发生变化.由于DNA双螺旋结构上带负电荷的磷酸根基团之间的静电排斥作用,DNA在溶液中是以自由舒展的形式存在.而这些多价阳离子能中和DNA双螺旋结构上的磷酸根基团所带的负电荷,从而使磷酸根基团间的静电排斥力减小,导致DNA的形态发生改变.当多价阳离子的浓度大于临界值时,DNA分子将凝聚成特定的紧密结构.本文通过DLS实验研究了DNA的电泳迁移率与三价平衡离子[Co(NH3)6]3+浓度的变化关系.

图3 DNA的电泳迁移率随平衡离子浓度的变化(NaCl用黑色方块线表示,[Co(NH3)6]3+用红色圆点线表示,DNA的浓度为 1 ng/µL)

当DNA溶液中加入三价离子,这些三价离子结合在磷酸根基团和氮基位点上使得DNA上的负电荷大部分被中和[21].通过观察AFM图像,可知[Co(NH3)6]3+可使DNA分子发生凝聚,凝聚结构为花状结构,如图4(b)所示.根据文献[22]可知,当平衡离子的化合价Z≥3时,DNA发生凝聚,这表明多价离子的化合价越高,越容易使DNA发生凝聚[22].在溶液中多价离子导致DNA凝聚,此时的DNA电泳迁移率不仅与DNA上的有效电荷有关,而且可能与DNA的结构有关.由于在理论上我们假设DNA的迁移率只与它的有效电荷有关,而不考虑DNA的结构的变化,所以我们得出的实验结果与理论数据有明显偏离.

图4 (a)MgCl2成像DNA的AFM图像;(b)[Co(NH3)6]3+导致DNA凝聚的AFM图像,DNA,MgCl2和[Co(NH3)6]3+的浓度分别为 1 ng/µL,3.5 mM,和50µM
3.3 DNA的电荷反转
在水溶液中,一个带电的粒子(高分子)结合足够多的平衡离子,使得其净电荷的符号发生逆转,这个现象称为电荷反转.在实验中采用精胺([C10N4H30]4+)作为四价的平衡离子,随着精胺浓度的增大,由于电荷中和作用DNA的电泳迁移率逐渐减小,如图5所示.当平衡离子浓度等于1 mM时,DNA 的电泳迁移率 µ1:µ4≈ 5:1,即缓冲液中只包含单价离子或四价离子时DNA的迁移率之比是5:1.根据Manning的凝聚理论,DNA的电泳迁移率 µ1:µ4应为 4:1,显然理论值与实验结果有明显偏离.可能的原因是DNA在四价平衡离子的作用下发生了凝聚,而理论计算并没有考虑DNA的结构发生了变化所带来的影响.从图5中可以发现当精胺的浓度为5 mM时,DNA的电泳迁移率由负变正.随着精胺浓度的增大,越来越多的平衡离子结合到DNA的表面,当结合在DNA表面的四价平衡离子的总电荷大于裸DNA的电荷的绝对值,此时DNA-[C10N4H30]4+复合物的净电荷由负变为正,DNA发生电荷反转.然而,Manning的平衡离子凝聚理论无法解释DNA的电荷反转现象.

图5 DNA的电泳迁移率随平衡离子浓度的变化(NaCl用黑色方块线表示,精胺(spermine)用红色圆点线表示,DNA的浓度为 1 ng/µL)
4 结论
本文系统地研究了不同化合价的平衡离子与DNA之间的相互作用.动态光散射的实验结果表明当溶液中只存在单价平衡离子或者二价平衡离子时,其实验结果与Manning的平衡离子凝聚理论结果相符合.当溶液中的平衡离子的化合价Z≥3时,DNA发生凝聚,此时DNA的电泳迁移率不仅与DNA上的有效电荷有关,还可能与DNA的结构有关,所以测得的实验结果大于理论值.当平衡离子的化合价Z=4时,DNA的电荷发生反转,这主要是由于吸附在DNA表面的多价离子的总电荷大于裸DNA的电荷的绝对值.因此,对于一般情形,自由溶液中的聚电解质的构型和离子关联效应与聚电解质迁移过程密切相关.关于电荷反转部分的详细研究正在进行中.
[2]Ouyang Z C 2003 Physics 32 728(in Chinese)[欧阳钟灿2003物理32 728]
[3]Manning G S 1978 Quart.Rev.Biophys.11 179
[4]Manning G S,Ray J 1998 J.Biomol.Struct.Dyn.16 461
[5]Mills P,Anderson C F,Record Jr M T 1985 J.Phys.Chem.89 3984
[6]Mohanty U,Ninham B W,Oppenheim I 1996 Pro.Natl.Acad.Sci.93 4342
[7]Raspaud E,Durand D,Livolant F 2005 Biophys.J.88 392
[8]Hud N V,Vilfan I D 2005 Annu.Rev.Biophys.Biomol.Struct.34 295
[9]Thomas T,Thomas T 2001 J.Cell.Mol.Life Sci.58 244
[10]Hougaard D M 1992 Int.Rev.Cytol.138 51
[11]Camerini-OteroR D,Hsieh P 1993 Cell73 217
[12]Sikorav J L,Church G M 1991 J.Mol.Biol.222 1085
[13]Nguyen T,Grosberg A Y,ShklovskiiB 2000 J.Chem.Phys.113 1110
[14]Ran S Y,Sun B,LiM 2007 Physics 36 228(in Chinese)[冉诗勇,孙博,李明2007物理36 228]
[15]Hou X M,Zhang X H,WeiK J,JiC,Dou S X,Wang W C,LiM,Wang P Y 2010 Physics 39 108(in Chinese)[侯锡苗,张兴华,魏孔吉,季超,窦硕星,王渭池,李明,王鹏业2010物理39 108]
[16]JiC,Zhang L Y,Dou S X,Wang P Y 2011 Acta Phys.Sin.60 098703(in Chinese)[季超,张凌云,窦硕星,王鹏业 2011物理学报 60 098703]
[17]Ma C,Bloom field V A 2004 Biopolymers 35 211
[18]Besteman K,Van Eijk K,Lemay S 2007 Nat.Phys.3 641
[19]Luan B,Aksimentiev A 2010 Soft Matter 6 243
[20]HsiaoP Y,Luijten E 2006 Phys.Rev.Lett.97 148301
[21]Ouameur A A,Tajmir-RiahiH A 2004 J.Biol.Chem.279 42041

