成骨不全症血清中骨相关microRNAs的初步筛查
王子强,鲁艳芹,任秀智,王延宙,李志良,徐超,韩金祥
1 山东省医学科学院 山东省医药生物技术研究中心,山东 济南 250062
2 天津医院,天津 300211
3 山东省省立医院,山东 济南 250033
4 武清区人民医院,天津 301700
生物技术与方法
成骨不全症血清中骨相关microRNAs的初步筛查
王子强1,鲁艳芹1,任秀智2,王延宙3,李志良4,徐超1,韩金祥1
1 山东省医学科学院 山东省医药生物技术研究中心,山东 济南 250062
2 天津医院,天津 300211
3 山东省省立医院,山东 济南 250033
4 武清区人民医院,天津 301700
旨在初步筛选出成骨不全症 (Osteogenesis imperfecta,OI) 患者血清中差异表达的骨相关microRNAs(miRNAs),通过探讨其在该疾病中的可能作用,为进一步研究成骨相关miRNA的功能及miRNA在成骨不全症诊断中的应用奠定基础。首先用geNorm和NormFinder等程序挑选出适于血清miRNAs定量检测的内参基因,然后利用实时荧光定量PCR技术对miRanda,Targetscan和Pictar软件以及文献报道筛选出的骨形成相关miRNAs进行定量检测,最后用配对t检验统计学方法筛查出差异表达的骨相关miRNAs。结果显示6个候选内参基因在不同年龄、性别和用药情况的成骨不全症患者和健康个体血清中的表达稳定性良好 (表达稳定值M<1.5),后经标准化因子配对差异 (Pairwise variations) 分析确定最适内参数目为 4个 (配对差异值V4/5=0.133<0.15),分别是 miR-16、Let-7a、snRNAU6、miR-92a。通过在 16个成骨不全症患者以及 8个健康对照个体的血清中进一步检测 miR-16、Let-7a、snRNAU6和 miR-92a,发现其表达稳定性依然良好 (M<1.5)。对100余种骨相关 miRNAs进行实时荧光定量检测,发现 11种 miRNAs在成骨不全症患者血清中有差异表达(P<0.05),并且生物信息学分析显示这些差异表达的miRNAs 很可能在成骨不全症的发生发展中起重要作用。以上实验结果表明成骨不全症患者血清中存在多种差异表达的骨相关miRNAs,而且这些miRNAs很可能成为一种用于成骨不全症血清学检查及诊断的生物标志物。
成骨不全症,血清miRNAs,内参基因,生物标志物
Abstract:We screened differential expression bone-related microRNAs (miRNAs) in serum of patients with osteogenesis imperfect (OI). First, we selected the reference gene (s) fit for quantitative detection of serum miRNAs by using geNorm and several other programmes. Then real-time fluorescent quntitative PCR was used to detect the expression level of bone-related miRNAs gained by means of miRanda, Targetscan and Pictar softwares caculation and reading literature.Then, the results were analyzed with the matched t test. All 6 candidate reference genes had a stable expression level in serum of healthy controls and patients with different characters, and the optimal number of reference genes is 4 (miR-16,let-7a, snRNAU6, miR-92a) after Pairwise Variations analysis (V4/5=0.133<0.15). For validating the universality of expression stability, we detected the relative expression value of miR-16, let-7a, snRNAU6 and miR-92a in another 8 healthy controls and 16 patients with OI and the result revealed that the expression of 4 genes remained stable (M<1.5).After measuring serum levels of more than 100 bone-related miRNAs in patients with real-time qPCR, 11 miRNAs showed differential expression, and bioinformatic analysis suggested these altered expressional mioRNAs had possibilities to participate in the process of OI. So the experiment indicated that there existed many differential expression bone-related miRNAs in serum of patients with OI, and these miRNAs had potentials to be promising biomarkers for serologic tests and diagnosis of OI.
Keywords:osteogenesis imperfecta, serum miRNAs, reference gene, biomarker
成骨不全症是一种在骨形成过程中以成骨细胞增殖分化异常和破骨细胞活性异常为特征的罕见异常性疾病。研究发现,双磷酸盐药物可以通过调节某些 miRNAs的表达水平来促进成骨细胞的增殖和抑制破骨细胞活性,进而缓解症状[1]。然而对于miRNAs的异常表达是否参与了成骨不全症的发生发展过程以及血清学检查miRNAs能否作为对成骨不全症的诊断依据,至今未有定论。
miRNAs是一种长约 22 nt的非编码单链RNA,通过和靶基因mRNA 3¢非翻译区序列互补结合抑制其翻译,有人推测人类约有1/3的基因受miRNAs的调控。随着对miRNAs功能的深入研究,发现miRNAs不仅可以参与肿瘤的发生发展过程,而且对骨的发生过程有重要的调节作用[2]。另外,miRNAs不仅可以调节细胞内基因的表达水平,也可以在外周血中发挥其生物学作用。目前已知,血清miRNAs在体内免疫以及一些血液性疾病中都发挥重要作用[3-4],并且,其表达水平与疾病的发生发展阶段有紧密联系[5],提示血清 miRNAs很有可能成为一种新型的疾病生物标志物用于疾病预测与诊断。
为了深入研究 miRNAs在成骨不全症发生发展中的作用,本研究通过检测成骨不全症患者血清中差异表达的骨相关miRNAs,试图找到一种或几种能表征该疾病的标志miRNAs,为研究骨相关miRNAs的功能以及miRNAs在成骨不全症的作用奠定基础。
1 材料与方法
1.1 试剂
Trizol LS试剂购自Invitrogen公司;miRcute miRNA cDNA第一链合成试剂盒和 miRcute miRNA荧光定量检测试剂盒购自 TIANGEN公司;引物序列参照 QIAGEN 公司网站(https://www.qiagen.com/geneglobe/default.aspx)推荐序列并由华大基因合成。
1.2 仪器
NanoDrop2000c紫外/分光光度计购自Thermo Fisher Sientific公司;LightCycler 480购自Roche公司。
1.3 血液样本
病例组 (Patients group,PG) 血液样本采自天津医院和山东省省立医院确诊的 22名成骨不全症患者的清晨空腹静脉血,男女性别比例8∶3,年龄为 (9.54±3.65) 岁。10名对照组 (Control group,CG) 的血液样本取自本市大佛小学志愿者的清晨空腹静脉血,男女比例 6∶4,年龄为(8.20±0.42) 岁。全部血液样本均在2 h内完成离心与分装。
1.4 血清总RNA提取
抽取的新鲜血液样本经1 900×g离心10 min后,取200 µL上清于新的预冷离心管中。向离心管中加入750 µL Trizol LS试剂,充分混匀后室温静置5 min。加入200 µL三氯甲烷,漩涡振荡30s后再室温静置5 min。4 ℃、12 000×g离心15 min,取其上清于另一预冷离心管中,并向其管中加入600 µL异丙醇,上下颠倒混匀后,室温静置 10 min。4 ℃、12 000×g离心 10 min,弃液相,沉淀用75%乙醇洗涤2次后溶于15 µL去RNA酶水中。RNA样本质量由2%琼脂糖凝胶和NanoDrop2000c紫外/分光光度计进行评估。
1.5 反转录及实时荧光定量PCR
按照miRcute miRNA cDNA第一链合成试剂盒操作方法,对各样品总RNA进行反转录,反转录产物可直接用于下游反应或−20 ℃贮藏备用。实时荧光定量 PCR反应是根据 SYBR Green I嵌合荧光法的原理利用miRcute miRNA荧光定量检测试剂盒完成的。反应体系为:2×miRcute miRNA预混合液5 µL;上游引物0.2 µL;下游引物0.2 µL;miRNA第一链cDNA 1 µL;DEPC-H2O 3.6 µL。总反应体系为 10 µL,每个样品重复3次。反应条件:94 ℃预变性2 min;94 ℃变性 20s,60 ℃退火 20s,72 ℃延伸 20s,共45个循环。将LightCycler 480读取的CT值利用 Q=EΔCT,计算出各基因的相对表达量 Q,后经内参基因标准化因子处理用于数据分析,其中E为PCR扩增效率。
1.6 内参基因的选择及验证
选择在性别、年龄、用药情况差异较大的6名OI患者和2名健康个体人作为研究对象,选取 miR-16、miR-92a、miR-638、Let-7a、snRNAU6以及18S rRNA作为候选内参基因,其中miR-16、miR-638、snRNAU6和 18S rRNA是外周血miRNAs实时定量检测常用的内参,miR-92a和Let-7a是在前期预实验中发现的较稳定表达的基因。然后应用实时荧光定量 PCR技术以及geNorm和NormFinder等程序对6种候选内参基因进行表达稳定性分析,选出最优内参基因组合,最后在另外16个OI患者和8个健康对照个体血清中验证其表达稳定性,并计算出内参基因的标准化因子。
1.7 骨相关miRNAs的差异表达筛查
选取在成骨细胞和破骨细胞增殖分化过程中起重要作用的相关蛋白,后用 miRanda、Targets和 Pictar软件找到能和靶蛋白 mRNA 3¢非编码区互补结合的miRNAs,取其交集,加之文献已经报道的在骨发生过程中有调节功能的miRNAs确定待检骨相关 miRNAs。选取没有药物治疗史的8名成骨不全症患者血清和以性别、年龄相配比的 8名健康人血清作为实验研究对象,利用荧光定量 PCR技术对待检骨相关miRNAs进行定量检测,最后用配对t检验统计学方法筛选出OI患者血清中差异表达的骨相关miRNAs。
2 结果与分析
2.1 内参基因的选择及验证
geNorm程序对内参基因表达稳定性分析时,通常建议基因表达稳定值M<1.5即可认为其表达稳定性较好,并且 M 值越小,稳定性越高。本实验经geNorm程序分析显示,6个候选内参基因的M值都小于1.5,且其表达稳定性从大到小依次为snRNAU6/miR-92a (0.5228)、miR-16(0.6783)、Let-7a (0.7215)、18S rRNA (0.7708)、miR-638 (0.8617) (图1)。最适内参数目分析结果显示V4/5=0.133<0.15 (geNorm程序默认0.15为取舍值),即最适合内参数目为 4个,分别为snRNAU6、miR-92a、miR-16 和 Let-7a (图 2)。用normFinder、Bestkeeper、delta CT软件程序分析基因表达稳定性显示结果和geNorm程序分析结果一致,并且用geNorm软件程序对筛选出的4个内参在另外16个成骨不全症患者和8个健康对照个体血清中的表达稳定性进行验证,结果显示,4个内参基因表达稳定性依然良好 (图3)。
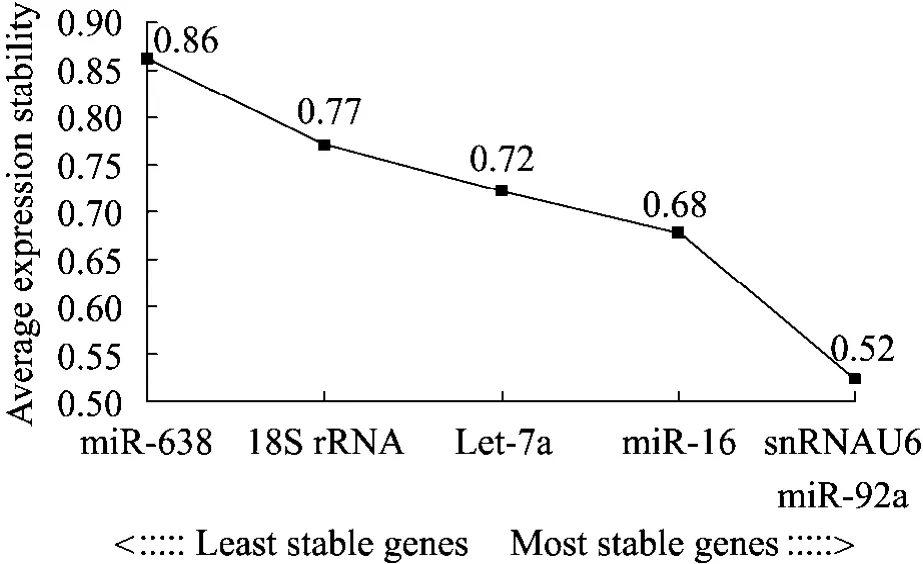
图1 6个候选内参基因的表达稳定值Fig.1 Expression stability values of 6 candidate reference genes.

图2 最适内参基因的数目Fig.2 Determination of the optimal number of reference genes for normalization.
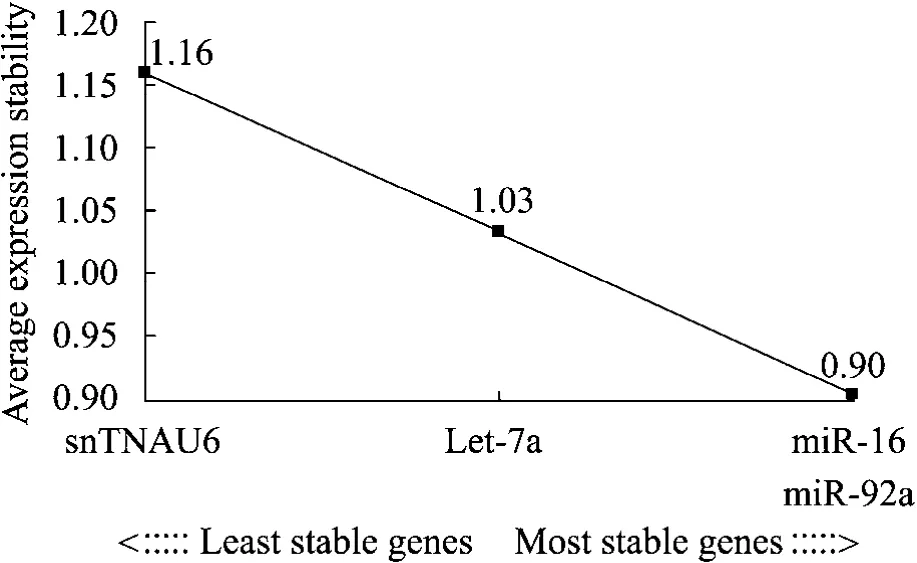
图3 4个内参基因在8个健康个体和16个成骨不全症患者血清中的表达稳定值Fig.3 Expression stability values of 4 reference genes in serum of 8 healthy controls and 16 patients with OI.
2.2 骨相关miRNAs的差异表达筛查
对116种骨相关miRNAs进行实时荧光定量检测,发现有11种miRNAs (miR-34c,miR-29a,miR-29b,miR-489,miR-133a,miR-145,miR-210,miR-1297,miR-26a,miR-30e,miR-21) 在成骨不全症血清中出现差异表达 (P<0.05),其中miR-26a、miR-30e、miR-21为上调 (图 4)。通过比较的差异倍数,发现miR-29a和miR-133a变化幅度最大,分别是下调了12.80和6.31倍,变化最小的是下调了0.75倍的miR-145,除此之外,其余miRNAs的变化倍数均在1.5~4之间。另外,miR-29a在病例组血清中的相对表达量普遍低于对照组 (图 5),而 miR-26a普遍高于对照组(图 6)。
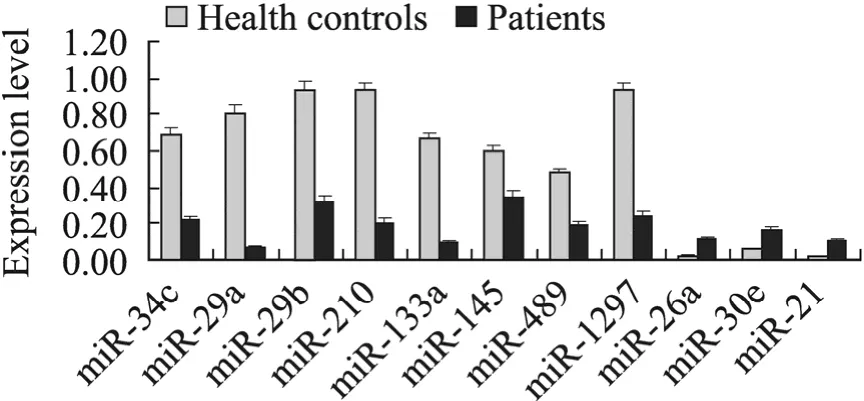
图4 11个差异表达的骨相关miRNAs在健康个体和成骨不全症患者血清中的表达情况Fig.4 Differential expression levels of eleven serum miRNAs in healthy controls and patients with OI.
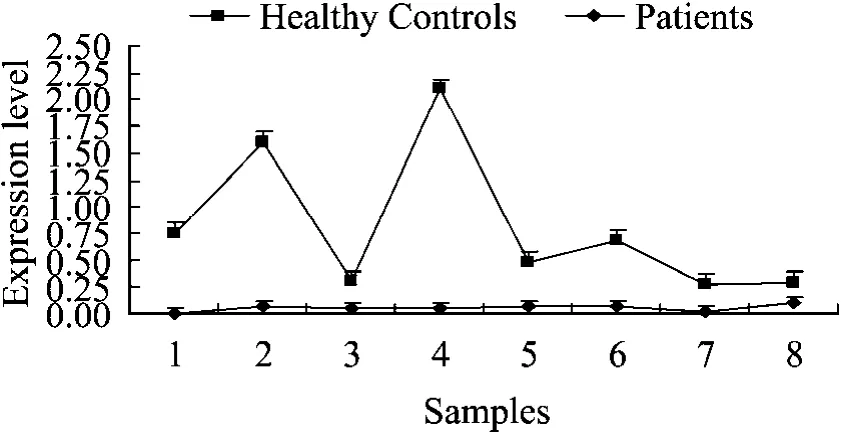
图5 miR-29a在健康个体和成骨不全症患者血清中的表达情况Fig.5 Expression level of serum miR-29a in healthy controls and patients with OI.
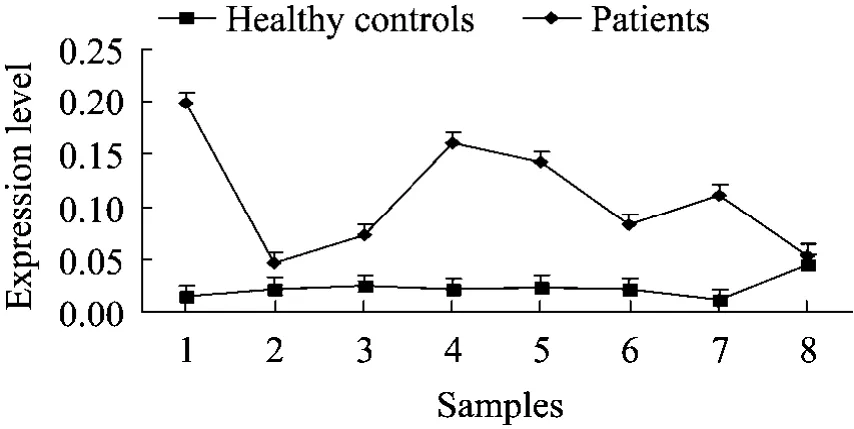
图6 miR-26a在健康个体和成骨不全症患者血清中的表达情况Fig.6 Expression levels of serum miR-26a in healthy controls and patients with OI.
3 讨论
实时荧光定量 PCR是一种能高效地实时检测某种基因 mRNA表达水平的技术,目前已在实验室普遍使用,但也存在诸多问题,最突出的问题就是内参基因的选择。研究显示,没有一种基因是稳定表达的,单一利用一种看家基因对实验数据进行标准化,可以造成几十倍以上的差异[6]。本实验利用Vandesompele在2002年编写的一款geNorm程序筛选出4种较高表达稳定性的基因作为实时荧光定量检测的内参基因。为验证geNorm程序分析结果的准确性以及4种内参基因稳定稳定的普遍性,本实验又通过normFinder、Bestkeeper、Delta CT method 等程序以及扩大样本量的方法对4种基因进行了进一步的分析,结果显示,snRNAU6、miR-92a、miR-16和 Let-7a在实验组和对照组的血清中都有较好的表达稳定性,为后续试验数据的准确性和可靠性提供了有力的证据。四个内参基因中,miR-16是外周血检测miRNAs最常用的内参基因,迄今应用于多种疾病的研究,如胃癌[7]、前列腺癌[8]、乳腺癌[9]和肝癌[10]等癌症。snRNAU6是一种在细胞内稳定表达的核糖体 RNA,近年来也发现在多种疾病的外周血中能稳定表达,如结肠直肠癌的血浆,乳腺癌的血清口腔鳞状细胞癌的血浆以及膀胱癌的尿液中[10]。但未见 miR-92a和Let-7a的相关报道。
在筛选出的11种miRNAs中,有些因在多种肿瘤的血清中都能检测到其差异表达而被称为“肿瘤miRNA”,如miR-21,已被多种肿瘤疾病[11-16]作为潜在的生物标志物用于临床疾病预测及诊断,另外几种 miRANs (miR-210,miR-133a,miR-26a,miR-29a,miR-34c) 也多在一些肿瘤[17-25]的血清中检测到差异变化。虽然没有在骨相关疾病血清中存在差异表达的相关报道,但有不少关于这些miRNAs在细胞中影响骨形成过程的研究。已知miR-29b可以促进成骨细胞分化,其机制是和组蛋白去乙酰化酶4,转化生长因子 β3,人连环蛋白-β互动蛋白1等成骨细胞分化抑制基因的 3¢非编码端互补结合并抑制其表达[26],另外,miR-29a[27]和 miR-210[28]也可分别通过抑制基因Dikkopf-1和1B型激活素A受体的翻译来促进成骨细胞分化。在抑制成骨细胞分化方面,miR-34c[29]、miR-133a[30]和miR-489[31]是分别通过抑制转录因子runx2,钙结合蛋白grancalcin、和转录因子runx2的基因表达来实现的。其他几种 miRNAs,除了 miR-21有促进破骨细胞分化的作用外,其余未见相关报道。但是经miRanda、PicTar、TargetScan三种软件分析靶基因发现,miR-145、miR-30e和miR-1297不仅可以影响一些经典的骨相关信号通路如Wnt、ERK/MEK、Hedgehog去调节骨形成过程,还可以直接作用于细胞的增殖与凋亡,所以预测这些骨相关 miRNAs在细胞中也可能存在差异表达变化并且变化程度和血清中的相符合。
总之,本研究筛选出了4种在成骨不全症患者和健康个体血清中都能稳定表达的内参基因snRNAU6、miR-92a、miR-16和 Let-7a,并且通过生物信息学分析认为初步筛查出的 11种差异表达的骨相关 miRNAs很有可能作为一种生物标志物用于成骨不全症的诊断与研究。
REFERENCES
[1] Zhou Q, Zhao ZN, Cheng JT, et al. Ibandronate promotes osteogenic differentiation of periodontal ligament stem cells by regulating the expression of microRNAs. Biochem Biophys Res Commun,2011, 404(1): 127−132.
[2] Liu CJ. MicroRNAs in skeletogenesis. Frontiers in Biosciences, 2009, 14: 2757−2764.
[3] Shaked I, Meerson A, Wolf Y, et al. MicroRNA-132 potentiates cholinergic anti-inflammatory signalingby targeting acetylcholinesterase. Immunity, 2009,31(6): 965−973.
[4] Schmidt WM, Spiel AO, Jilma B, et al.In vivoprofile of the human leukocyte microRNA response to endotoxemia. Biochem Biophys Res Commun,2009, 380(3): 437−441.
[5] Moltzahn F, Olshen AB, Baehner L, et al.Microfluidic-based multiplex qRT-PCR identifies diagnostic and prognostic microRNA signatures in the sera of prostate cancer patients. Cancer Res,2011, 71(2): 550−560.
[6] Vandesompele J, De Preter K, Pattyn F, et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multipleinternal control genes. Genome Biol, 2002,3(7): RESEARCH 0034.
[7] Cortez MA, Bueso-Ramos C, Ferdin J, et al.MicroRNAs in body fluids-the mix of hormones and biomarkers. Nat Rev Clin Oncol, 2011, 8(8):467−477.
[8] Mahn R, Heukamp LC, Rogenhofer S, et al.Circulating microRNAs (miRNA) in serum of patients with prostate cancer. Urology, 2011, 77(5):1265.e9−1265.e16.
[9] Zhu W, Qin W, Atasoy U, et al. Circulating microRNAs in breast cancer and healthy subjects.BMC Res Notes, 2009, 2(1): 89.
[10] Bihrer V, Waidmann O, Friedrich-Rust M, et al.Serum microRNA-21 as marker for necroinflammation in hepatitis C patients with and without hepatocellular carcinoma. PLoS ONE,2011, 6(10): e26971.
[11] Liu R, Chen X, Du Y, et al. Serum MicroRNA expression profile as a biomarker in the diagnosis and prognosis of pancreatic cancer. Clin Chem,2012, 58(3): 610−618.
[12] Xu J, Wu C, Che X, et al. Circulating MicroRNAs,miR-21, miR-122, and miR-223, in patients with hepatocellular carcinoma or chronic hepatitis. Mol Carcinog, 2011, 50(2): 136−142.
[13] Lawrie CH, Gal S, Dunlop HM, et al. Detection of elevated levels of tumour-associated microRNAs in serum of patients with diffuse large B-cell lymphoma. Br J Haematol, 2008, 141(5): 672−675.
[14] Asaga S, Kuo C, Nguyen T, et al. Direct serum assay for microRNA-21 concentrations in early and advanced breast cancer. Clin Chem, 2011, 57(1):84−91.
[15] Zhang HL, Yang LF, Zhu Y, et al. Serum miRNA-21: elevated levels in patients with metastatic hormone-refractory prostate cancer and potential predictive factor for the efficacy of docetaxel-based chemotherapy. Prostate, 2011,71(3): 326−331.
[16] Wang ZX, Bian HB, Wang JR, et al. Prognostic significance of serum miRNA-21 expression in human non-small cell lung cancer. J Surg Oncol,2011, 104(7): 847−851.
[17] Li T, Cao H, Zhuang J, et al. Identification of miR-130a, miR-27b and miR-210 as serum biomarkers for atherosclerosis obliterans. Clin Chim Acta, 2011, 412(1/2): 66−70.
[18] Gunel T, Zeybek YG, Akcakaya P, et al. Serum microRNA expression in pregnancies with preeclampsia. Genet Mol Res, 2011, 10(4):4034−4040.
[19] Kuwabara Y, Ono K, Horie T, et al. Increased microRNA-1 and microRNA-133a levels in serum of patients with cardiovascular disease indicate myocardial damage. Circ Cardiovasc Genet, 2011,4(4): 446−454.
[20] Xie L, Chen X, Wang L, et al. Cell-free miRNAs may indicate diagnosis and docetaxel sensitivity of tumor cells in malignant effusions. BMC Cancer,2010, 10(1): 591.
[21] Roderburg C, Urban GW, Bettermann K, et al.Micro-RNA profiling reveals a role for miR-29 in human and murine liver fibrosis. Hepatology, 2011,53(1): 209−218.
[22] Wu Q, Lü ZH, Li HL, et al. Next-generation sequencing of microRNAs for breast cancer detection. J Biomed Biotechnol, 2011, 2011:597145.
[23] Wang LG, Gu J. Serum microRNA-29a is apromising novel marker for early detection of colorectal liver metastasis. Cancer Epidemiol,2012, 36(1): e61−e67.
[24] Fu Y, Yi Z, Wu X, et al. Circulating microRNAs in patients with active pulmonary tuberculosis. J Clin Microbiol, 2011, 49(12): 4246−4251.
[25] Zhao C, Dong J, Jiang T, et al. Early second-trimester serum miRNA profiling predicts gestational diabetes mellitus. PLoS ONE, 2011,6(8): e23925.
[26] Pavlides S, Tsirigos A, Migneco G, et al. The autophagic tumor stroma model of cancer: role of oxidative stress and ketone production in fueling tumor cell metabolism. Cell Cycle, 2010, 9(17):3485−3505.
[27] Li Z, Hassan MQ, Jafferji M, et al. Biological functions of miR-29b contribute to positive regulation of osteoblast differentiation. J Biol Chem, 2009, 284(23): 15676−15684.
[28] Kapinas K, Kessler C, Ricks T, et al. MiR-29 modulates Wnt signaling in human osteoblasts through a positive feedback loop. J Biol Chem,2010, 285(33): 25221−25231.
[29] Mizuno Y, Tokuzawa Y, Ninomiya Y, et al.miR-210 promotes osteoblastic differentiation through inhibition of AcvR1b. FEBS Lett, 2009,583(13): 2263−2268.
[30] Zhang Y, Xie RL, Croce CM, et al. A program of microRNAs controls osteogenic lineage progression by targeting transcription factor Runx2.Proc Natl Acad Sci USA, 2011, 108(24):9863−9868.
[31] Schoolmeesters A, Eklund T, Leake D, et al.Functional profiling reveals critical role for miRNA in differentiation of human mesenchymal stem cells. PLoS ONE, 2009, 4(5): e5605.
Screening of bone-related microRNAs in serum of patients with osteogenesis imperfect
Ziqiang Wang1, Yanqin Lu1, Xiuzhi Ren2, Yanzhou Wang3, Zhiliang Li4, Chao Xu1, and Jinxiang Han1
1Shandong Medicinal Biotechnology Center,Shandong Academy of Medical Sciences,Jinan250062,Shangdong,China
2Tianjin Hospital,Tianjin300211,China
3Shandong Provincial Hospital,Jinan250033,Shangdong,China
4Wuqing People's Hospital,Tianjin301700,China
王子强, 鲁艳芹, 任秀智, 等. 成骨不全症血清中骨相关microRNAs的初步筛查. 生物工程学报, 2012, 28(10): 1245−1252.
Wang ZQ, Lu YQ, Ren XZ, et al. Primary screening of bone-related microRNAs in serum of patients with osteogenesis imperfecta. Chin J Biotech, 2012, 28(10): 1245−1252.
Received:March 17, 2012;Accepted:April 17, 2012
Supported by:National Key Technology R&D Program (No. SQ2011SF12C03081).
Corresponding author:Jinxiang Han. Tel: +86-531-82919888; E-mail: samshjx@sina.com
国家科技支撑计划 (No. SQ2011SF12C03081) 资助。