SSR标记在家蚕×野桑蚕后代群体分析中的应用
彭云武,楚 渠
(安康学院,陕西 安康 725000)
野桑蚕 (Bombyx mandarina)是一种与家蚕亲缘关系最近的野生资源,蕴含着丰富的遗传信息,是家蚕品种选育的宝贵材料[1]。1999年后,SSR分子标记在家蚕资源的遗传多样性分析、基因作图、基因定位、品种鉴定和标记辅助育种等方面进行了应用[2-6]。在农作物方面,钟昌松等[7]用 SSR标记对普通玉米和特用玉米进行聚类分析,王海燕等[8]利用SSR标记分析云南、西藏和新疆小麦的遗传多样性,王凯华等[9]用 SSR标记分析甘蓝型油菜种质遗传多样性,邱红波等[10]用SSR标记对贵州玉米种质进行遗传多样性分析,宗绪晓等[11]利用SSR标记分析中国豌豆地方品种遗传多样性,吴晓雷等[12]用SSR标记研究大豆属种间亲缘进化关系,张真真等[13]对矮化佛手的遗传差异进行SSR分析。作者用SSR标记对家蚕 ×野桑蚕后代群体进行亲缘关系与遗传多态性的研究,为野桑蚕资源利用和家蚕杂交育种亲本的选配及杂种后代的预选提供理论指导。
1 材料和方法
1.1 材料
供试的26份材料 (表1)中有家蚕品种2个,分别是874和873;野桑蚕品种4个,分别是CS6、Bh1、Bh2、Tb2;栗蚕材料1个;其余的为家蚕×野桑蚕的品种组合。采用在公开期刊上发表的应用于家蚕的SSR引物序列[14](表2-3),引物由北京六合华大基因科技股份有限公司合成。TaqDNA聚合酶和dNTP购自北京鼎国生物公司。

表1 供试材料的类型及来源

表2 SSR(Chr 1-15)引物的名称、序列及大小

表3 SSR(Chr 16-28)引物的名称、序列及大小
1.2 方法
1.2.1 基因组DNA的提取
采用改良的CTAB法进行提取。在65℃水浴锅中预热CTAB提取液;每供试材料取双翅在液氮中迅速研磨,将粉末状材料转入2 mL离心管中,加入预热的CTAB提取液 (每克样品加入3~5 mL的提取液),65℃保温30~60 min,每隔10 min轻轻颠倒混匀;12 000 r·min-1离心5 min,取上清转入新离心管;加入等体积酚/氯仿 (1∶1),充分混匀,12 500 r·min-1离心10 min,取上清转入新离心管;加入等体积氯仿,充分混匀,12 500 r·min-1离心10 min,取上清转入新离心管;重复加入酚/氯仿和氯仿的步骤;加入2/3体积的异丙醇混匀,室温放置,沉淀15 min;12 000 r·min-1离心6 min,弃上清;将沉淀用70%的乙醇漂洗1次,室温下12 500 r·min-1离心 2 min,弃上清,重复洗1次;提取产物取2~3 μL用2.0%琼脂糖凝胶电泳检测,其余置于-20℃保存备用。
1.2.2 引物检测及退火
以1号样本为模板,分别选择 45.8,49.1,53.7,62.5和65℃作为退火温度,根据检测结果和经验给出建议退火温度,并从100对SSR引物中筛选出能够有效扩增的引物为初筛合格引物。再选取其中扩增效果好、条带差异明显、重复性好、信号强、背景清晰的引物在26份不同类型材料间进行SSR-sPCR反应。
1.3 PCR反应和电泳
PCR反应在PCR仪上进行,反应体系为20 μL,其中 ddH2O 7.2 μL,2 × PCRMix 10 μL,正向引物 0.3 μL,反向引物 0.3 μL,DNA 2 μL,Taq酶 0.2 μL PCR扩增程序:94℃ 4 min→ (94℃35 s;62.5℃35 s;72℃50 s)×35个循环→72℃10 min→4℃后取出扩增产物。反应完成后每个反应体系中加入10 μL装载染料。电泳缓冲液为1×TBE,电流50 mA,电泳时间为75 min。
1.4 数据统计分析
每一对SSR引物电泳后将在特定位置出现扩增带,将凝胶成像系统中成像图片扫描并数扩增带数。同一标记在不同材料中扩增出来的条带,在某一位置有带存在记为1,没有的则记为0。用浙江大学开发的DPS v7.05版统计软件中的子程序多元分析,选聚类分析,再选0,1数据系统聚类,聚类距离选Nei-Li(Czekanoeki);聚类方法选类平均法 (UPGMA),得出遗传距离和聚类树形图。
2 结果与分析
2.1 引物筛选
以1号样本为模板,引物检测的结果是扩增好的81对,扩增不太好的11对,没有扩增的8对。经琼脂糖检测获得69对扩增比较好的引物进行PAGE检测 (图1)。

图1 引物的PAGE检测结果
2.2 DNA的多态性分析
采用69对引物对表1所列品种的基因组DNA进行扩增,共获得2 529个多态性片段,平均每对引物扩增出等位片段36.6个 (图2)。等位片段数最多的为S0105,共有113个,等位片段数最少的为S1706和 S2707,均为21个。各品种的扩增带从23~115条不等。由此可见不同引物、不同品种之间的多态性各不相同。

图2 引物S2215的扩增情况
2.3 亲缘关系的聚类分析
用69对引物对26个材料的DNA进行PCR扩增,将各引物的扩增结果转化为0,1数据,利用浙江大学的DPS v7.05版统计软件中的Nei-Li(Czekanoeki)方法计算各品种间的遗传距离,并进行聚类分析,得到如图3所示的树状。
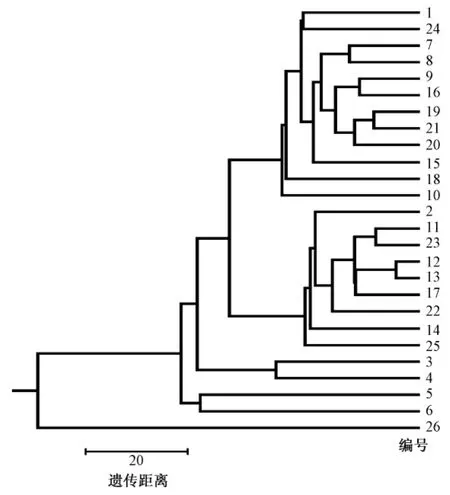
图3 家蚕、野桑蚕及其它们杂交后代SSR分析的遗传关系
研究结果显示,26个材料中最先聚在一起的是Ak11051(野蚕斑)和Ak11051(普斑),即幼虫斑纹不同的同一品种,说明所选SSR标记引物足以区分亲缘关系很近的不同材料。26个试验材料之间的遗传距离最小的为0.110 9,最大为1.918 3,表明供试材料具有丰富的遗传多样性。
根据SSR分子标记聚类分析可知,供试的26个材料分为明显的4个大类群:第1大类群为栗蚕;第2大类群有Bh1和CS6野桑蚕材料;第3大类群Tb1和Tb2野桑蚕材料,表明同一区域内的野桑蚕的亲缘关系较近;第4大类群又按品种间亲缘关系的远近分为2大亚类,即与家蚕873遗传性状相近的聚为1个亚类、与家蚕874遗传性状相近的聚为另1个亚类。
3 小结与讨论
杂交育种是选育新品种的主要途径,也是近代家蚕育种工作的重要方法。本研究所用材料是家蚕×野桑蚕杂交所得的后代群体,变异类型丰富,为培育家蚕新品种提供了良好的物质基础。
本研究选用了69对家蚕SSR引物对26种供试材料基因组DNA进行了扩增。聚类结果表明,SSR标记可以完全鉴别出本试验中的家蚕的两大系统,这与郭秋红等[15-16]用 SSR标记对家蚕品种进行亲缘关系分析得到的结果基本一致。
白河野桑蚕与汉滨野桑蚕各聚一类,试验结果印证了白河野桑蚕是一个与其他野桑蚕材料遗传差异大的野桑蚕[17]。所以,微卫星标记能较为客观地反映不同地方野桑蚕的遗传关系。
[1]向仲怀.中国蚕种学 [M].成都:四川科学技术出版社,1995.
[2]李竞,房守敏,刘文明,等.SSR标记在中国野桑蚕和家蚕的遗传多样性分析中的应用 [J].蚕业科学,2005,31(3):251-256.
[3]Li M W,Shen L,Xu A Y,et al.Genetic diversity among the silkworm(Bombyx moriL.,Lep.,Bombycidae)germplasm revealed by microsatellites [J].Genome,2005,48:802-810.
[4]侯成香,李木旺,张月华,等.利用SSR标记进行家蚕部分品种资源的指纹图谱分析 [J].中国农业科学,2006,39(10):2124-2131.
[5]房守敏.基于 SSR-GeneScan技术的家蚕起源与进化研究[D].西南农业大学,2005.
[6]沈利,李木旺,李明辉,等.家蚕微卫星标记的筛选及其在遗传多样性分析中的应用 [J].蚕业科学,2004,30(3):230-236.
[7]钟昌松,徐利远,余桂蓉,等.普通玉米和特用玉米的SSR标记聚类 [J].核农学报,2009,23(1):42-48,74.
[8]王海燕,王秀娥,陈佩度,等.利用SSR标记分析云南、西藏和新疆小麦的遗传多样性 [J].遗传学报,2007,34(7):623-633.
[9]王凯华,张文英,王会,等.国内外甘蓝型油菜种质SSR标记遗传多样性分析 [J].中国农学通报,2011,27(19):144-149.
[10]邱红波,戴保威,彭中华.利用SSR标记对贵州玉米种质进行遗传多样性分析 [J].浙江农业学报,2011,23(4):667-670.
[11]宗绪晓,关建平,王述民,等.中国豌豆地方品种SSR标记遗传多样性分析 [J].作物学报,2008,34(8):1330-1338.
[12]吴晓雷,贺超英,陈受宜,等.用SSR分子标记研究大豆属种间亲缘进化关系 [J].遗传学报,2001,28(4):359-366.
[13]张真真,王俊军,郭卫东.矮化佛手遗传差异的SSR分析[J].浙江农业科学,2009(1):67-71.
[14]Miao Xuexia,Xu Shijie,Li Minghu,et al.Simple sequence repeat-based consensuslinkagemap ofBombyxmori[J].PNAS November 8,2005,102(45):16303-16308.
[15]郭秋红,李木旺,李明辉,等.利用SSR标记鉴定家蚕不同系统的品种初探 [J].蚕业科学,2005,31(3):257-260.
[16]钱荷英,李木旺,张月华,等.用SSR标记进行家蚕日系品种资源指纹图谱的构建及亲缘关系分析 [J].蚕业科学,2005,31(4):422-428.
[17]彭云武,楚渠,杨金宏,等.安康野桑蚕遗传多样性分析[J].西北农业学报,2010,19(11):24-28.

