产ESBLs革兰阴性杆菌中16SrRNA甲基化酶基因的检测
高 辉,王 杨,黄云昆,朱雯梅,徐 敏
(昆明市延安医院 检验科,云南 昆明 650051)
产ESBLs革兰阴性杆菌中16SrRNA甲基化酶基因的检测
高 辉,王 杨,黄云昆,朱雯梅,徐 敏
(昆明市延安医院 检验科,云南 昆明 650051)
产 超 广 谱 β-内 酰 胺 酶 (extended-spectrumβlactamases,ESBLs)的革兰阴性杆菌是医院感染的重要病原菌,表现为多重耐药,给临床抗感染治疗带来了极大的困扰。近年来研究发现,一类由质粒介导的16SrRNA甲基化酶可使细菌的30S核糖体16SrRNA不与氨基糖苷类药物结合,从而导致细菌对氨基糖苷类药物出现高水平耐药,有研究显示16SrRNA甲基化酶基因常与ESBLs基因位于同一质粒上,可在不同菌种间水平传播或同种间克隆传播从而造成多重耐药性的传播。本研究对临床标本中分离的56株产ESBLs革兰阴性肠杆菌进行了16SrRNA甲基化酶基因筛查,以了解其流行情况,为进一步研究昆明地区该耐药基因的分布情况奠定基础,为提高临床抗感染治疗效果提供科学依据。
1 材料与方法
1.1 菌株来源
2010年1月-2010年10月昆明市延安医院(三甲综合医院)临床标本中分离的ESBLs阳性非重复性革兰阴性肠杆菌56株,包括大肠埃希式菌、肺炎克雷伯菌、产酸克雷伯菌、奇异变形杆菌。药敏质控菌株为大肠埃希菌ATCC25922,ESBLs表型确认试验质控菌株为肺炎克雷伯菌ATCC700603。
1.2 药敏纸片和培养基
药敏纸片中亚胺培南、美洛培南、环丙沙星、为英国Oxoid公司产品,其余药敏纸片为北京天坛药物生物技术开发公司产品。药敏试验用M-H培养基,为英国Oxoid公司产品。
1.3 细菌鉴定与药敏试验
用法国生物梅里埃VITEK32细菌鉴定仪进行细菌鉴定,药敏试验采用K-B琼脂扩散法,按美国临床实验室标准化协会(CLSI2009)的标准判读药敏试验结果。
1.4 ESBLs检测
采用CLSI推荐的双纸片扩散法,使用CTX和CTX/CA、CAZ和CAZ/CA两对纸片进行ESBLs表型确认试验,严格按照CLSI推荐的操作和结果判断标准进行,以肺炎克雷伯菌ATCC700603为ESBLs阳性对照。
1.5 提取细菌基因组DNA
用细菌基因组提取试剂盒提取细菌DNA,按试剂盒说明书操作,试剂盒由天根生物技术有限公司生产。
1.6 基因检测
用Biometra Personal Cycler PCR仪扩增6种16SrRNA甲基化酶基因和TEM型β-内酰胺酶基因,Taq PCR MasterMix购自北京博迈德科技发展有限公司,PCR扩增引物由北京百泰克生物技术有限公司合成,靶基因引物序列见表1[1]。TEM基因PCR扩增体系为:2×Taq Master Mix 25μl,模板0.5μl,P1引物2μl,P2引物2μl,无菌去离子水补足至50μl。热循环参数为:94℃2min,然后94℃60s→55℃60s→72℃60s,循环35个周期,最后72℃10min。6种16SrRNA甲基化酶基因PCR扩增体系均为:2×Taq Master Mix 25μl,模板0.5 μl,P1引物2μl,P2引物2μl,无菌去离子水补足至50μl。热循环参数均为:95℃预变性3min,然后95℃60s→55℃60s→72℃60S。循环30次,72℃延伸10min。产物经1.5%琼脂糖凝胶电泳,EB染色,Bio-Rad Laboratories 6000凝胶成像系统观察结果,并摄像保存。出现与阳性对照分子相当的目的条带为阳性,armA基因阳性对照DNA由浙江大学医学院俞云松教授惠赠。
2 结果
2.1 β-内酰胺酶基因PCR检测结果
56株表型确认产ESBLs革兰阴性肠杆菌科细菌均检测出TEM基因,其PCR产物电泳结果见图1。
2.2 16SrRNA甲基化酶基因PCR检测结果
56株产ESBLs革兰阴性肠杆菌科细菌进行armA、rmtA、rmtB、rmtC、rmtD、npmA 基因扩增均为阴性,其PCR产物电泳结果见图2。

表1 PCR引物序列
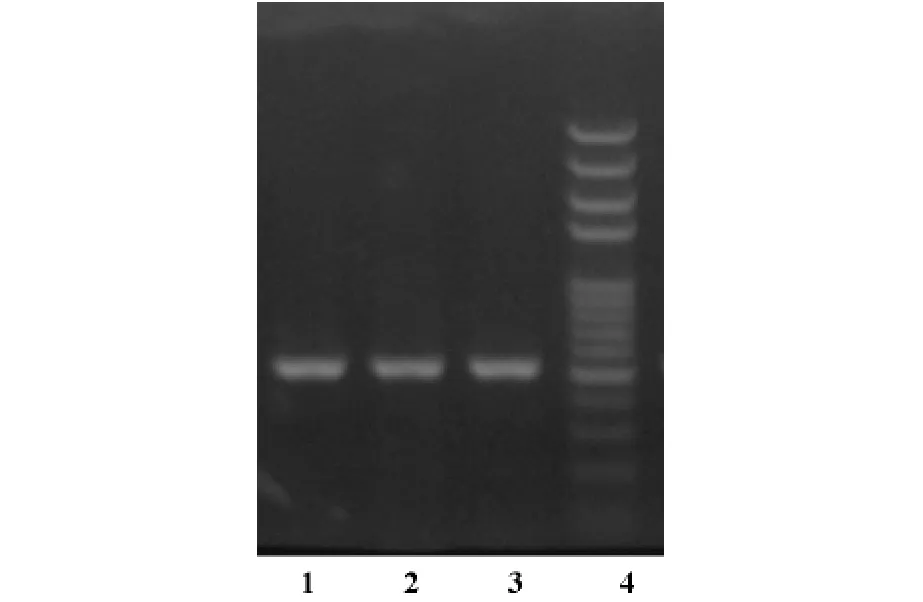
图1 TEM基因扩增产物电泳图

图2 16SrRNA甲基化酶基因扩增产物电泳图
3 讨论
文献报道,昆明地区产ESBLs的耐药菌株有较高的检出率[2]。本研究通过表型确证试验确证的56株产ESBLs革兰阴性杆菌均检测出TEM型β-内酰胺酶基因。目前已发现了160种TEM型衍生酶,其中大部分是 ESBLs[3-5]。本研究中的产 ESBLs菌株具体为何种TEM亚型尚待测序分析。
产ESBLs的菌株表现为多重耐药,常常对临床所用的氨基糖苷类药物同时耐药。普遍认为细菌对氨基糖苷类药物耐药的主要机制是细菌产生了可以水解氨基糖苷类药物的酶,但这些水解酶不能水解所有的氨基糖苷类药物,如阿贝卡星。近年研究发现,一类由质粒介导的16SrRNA甲基化酶能使药物作用靶位16SrRNA G1405上的N-7位鸟苷变为7-甲基鸟苷或将A1408上的腺嘌呤N-1位甲基化,致使细菌的30S核糖体16SrRNA与氨基糖苷类药物的亲和力下降,从而出现对氨基糖苷类药物的高水平耐药,其 MIC值往往高达512-1024μg/ml[6-10],因此,产16SrRNA 甲基化酶可使细菌泛氨基糖苷类耐药。有研究发现16SrRNA甲基化酶基因与β-内酰胺酶基因存在连锁关系,常与碳青霉烯酶或者ESBLs编码基因连锁传播[11],通过质粒介导,以转化、转导、结合、传递等方式在同种或不同种属菌株间转移和传播,造成院内感染流行,对氨基糖苷类和β-内酰胺类抗菌药物的临床应用产生严重的影响。
到目前为止,在革兰阴性杆菌中已发现armA、rmtA、rmtB、rmtC、rmtD和npmA 6种16SrRNA甲基化酶基因。在一些欧美国家都有质粒介导16S rRNA甲基化酶临床分离株引起感染的报道[6,8,12-14]。研 究 表 明,北 美 洲 主 要 以 armA 和rmtB为主,欧洲以armA为主,拉丁美洲以rmtD为主[14],不同地区16SrRNA甲基化酶的流行率和流行基因型存在差别。2004年中国台湾地区分离的肺炎克雷伯菌armA和rmtB的阳性率分别为0.4%和0.04%[8]。国内不同地区医院16SrRNA甲基化酶基因检出率也各有不同,杭州沈晓强等报道在6省市临床分离的447株产ESBLs的菌株中筛选到1株产armA型16SrRNA甲基化酶的产酸克雷伯菌,吴蓉等报道在上海普陀医院临床分离的53株对庆大霉素和/或阿米卡星耐药的革兰阴性杆菌中筛选出10株产16SrRNA甲基化酶的菌株[1,11]。
本院近年来革兰阴性杆菌对庆大霉素的耐药率持续在50%-60%,对阿米卡星的耐药率则维持在8%-10%,监测我院16SrRNA甲基化酶基因的携带情况对临床有重要意义。本研究结果显示56株产ESBLs革兰阴性肠杆菌均未检出16SrRNA甲基化酶基因,与国内其他地区的检测结果存在差别[1,11],导致菌株耐药基因携带率不同的原因可能与菌株来源(地理位置)和各地区医院抗菌药物的使用习惯不同有关,也与本研究的样本仅限于产ESBLs革兰阴性肠杆菌科细菌有关。虽然如此,仍应重视对该耐药基因的早期筛查和监控,本课题组也将扩大样本范围及样本量继续追踪调查本地区耐药基因的流行情况。
[1]吴 蓉,张 隆,戴俊华,等.革兰阴性杆菌16SrRNA甲基化酶基因检测及作用研究[J].检验医学,2010,25(6):423.
[2]刘晓莉,杨 旭,单 斌,等.2002年昆明地区临床分离菌的耐药性监测及分析[J].中国抗感染化疗杂志,2004,4(4):202.
[3]钟运华,何 林,周克元,等.超广谱β-内酞胺酶研究进展[J].国际检验医学杂志,2008,29(3):252.
[4]张 玲,黄留玉.革兰阴性杆菌产超广谱β-内酰胺酶研究进展[J].中华医院感染学杂志,2008,18(6):897.
[5]Varsha G.An update on newerβ-lactamases[J].Indian J Med Res,2007,11:417.
[6]Galinland M,CourvMin P.Lambert T.Plasmid mediated highlevel resistance to aminoglycosides in Enterobacteriaceae due to 16SrRNA methylation[J].Antimierob Agents Chemother,2003,47:2565.
[7]Yokoyama K,Doi Y,Yamane K,et al.Acquisition of l6SrRNA methylase gene in Pseudomonas aeruginosa[J].Lancet,2003,362:1888.
[8]Yah JJ,Wu JJ,Ko WC,et al.Plasmid-mediated 16SrRNA methylases conferring high-level aminoglycoside resistance in Escherichia coli and Klebriella pneumoniae isolates from two Taiwanese hospitals[J].J Antimicrob Chemother,2004,54:1007.
[9]Wachino J,Yamane K,Shibayama K,et al.Novel plasmid-mediated 16SrRNA methylase,RmtC,found in a proteus mirabilis isolate demonstrating extraordinary high-level resistance against various aminoglycosides[J].Antimicrob Agents Chemother,2006,50(1):178.
[10]Yokoyama K,Doi Y,Yamane K,et al.Acquisition of 16SrRNA methylase gene in Pseudomonas aeruginosa[J].Lancet,2003,362:1888.
[11]沈晓强,周 华,季淑娟,等.产ESBLs肠杆菌科细菌中16S rRNA甲基化酶基因的研究[J].中华临床感染病杂志.2010,3(3):142.
[12]Bogaerts P,Galimand M,Bauraing C,et al.Emergence of ArmA and RmtB aminoglycoside resistance 16SrRNA methylases in Belgium[J].J Antimicrob Chemother,2007,59:459.
[13]Doi Y,Adams JM,Yamane K,et al.Identification of 16SrRNA Methylase-producing Acinetobacter baumanannii clinical strains in north America[J].Antimicrob Agents Chemother,2007,51:4209.
[14]Fritsche TR,Castanheira M,Miller GH,et al.Detection of methyltransferases conferring high-1evel resistance to aminoglycosides in enterobacteriaceae from Europe,North America,and Latin America[J].Antimicrob Agents Chemother,2008,52 1843.
1007-4287(2012)10-1910-03
2011-03-07)

