IgA肾病组蛋白H3K4三甲基化和DNA甲基化基因修饰的相关性*
齐素文,涂植光△,戴 勇,睢维国
(1教育部临床检验诊断学重点实验室,重庆医科大学检验医学院,重庆 400016;2中国人民解放军181医院全军肾移植与透析治疗中心,广西 桂林 541002)
表观遗传机制对免疫系统的正常发育和功能发挥具有重要作用,外界因素的影响使表观遗传在免疫反应中出现不平衡可致基因异常表达,免疫功能紊乱,从而导致自身免疫性疾病的发生。表观遗传的两个主要机制为DNA甲基化和组蛋白修饰,进而对染色质结构的改变、基因转录和复制产生重要影响。DNA甲基化和组蛋白修饰之间也存在相互作用[1,2]。为此,我们采用染色质免疫共沉淀联合芯片(chromatin immunoprecipitation linked to microarrays,ChIP-chip)技术对 IgA肾病(IgA nephropathy,IgAN)患者的组蛋白H3赖氨酸4三甲基化进行研究,采用甲基化 DNA免疫共沉淀荧光定量 PCR(methylated DNA immunoprecipitation-quantitative PCR,MeDIP-qPCR)方法检测阳性基因的DNA甲基化水平,以探讨IgAN中组蛋白H3K4三甲基化和DNA甲基化的相关性,从基因水平研究IgAN的发病机制。
材料和方法
1 临床资料
收集2008年10月~2010年7月在中国人民解放军181医院肾病中心收治的IgA肾病患者40例,参考HAAS分级法[3]进行诊断。其中女性25例,男性15 例,年龄(48.6 ±9.3)岁,HAAS III级20 例、IV级10例、V级10例。正常对照组40例,女性25例,男性15例,年龄(45.2 ±10.6)岁。本研究计划经重庆医科大学伦理会批准,所有受试对象签署了知情同意书。
2 主要试剂
淋巴细胞分离液(Cedarlane),12K CpG岛芯片(UHN Microarray Centre),组蛋白H3赖氨酸4三甲基化抗体(Millipore);整个基因组扩增试剂盒(Qiagen),蛋白酶 K(Epigentek),5-甲基胞嘧啶抗体(Upstate),磁珠偶联Ⅱ抗(Upstate),QIA快速PCR纯化试剂盒(Invitrogen),EpiQuikTMH3K4三甲基化定量试剂盒(Epigentek),Sss I甲基转移酶(New England Biolabs);固体闪烁剂 MicroScint 20(Millipore);[3H]-S-腺苷甲硫氨酸([3H]-SAM)(Sigma)。
3 单个核细胞分离
每例患者和健康者空腹抽取10 mL外周静脉血,使用肝素钠抗凝。外周血单个核细胞(peripheral blood mononuclear cells,PBMCs)的分离采用 Ficoll密度梯度离心法,按照淋巴细胞分离液试剂盒的产品说明进行操作。收集得到的单个核细胞分装成3份,1份用于染色质免疫共沉淀芯片实验,1份加1 mL Trizol用于总RNA的提取,1份用于DNA甲基化实验。于-80℃保存备用。
4 ChIP-chip
采用试剂盒对受试对象进行总体H3K4三甲基化水平检测。ChIP-chip步骤基本参照文献[4]并略作修改。在室温条件下,用1%甲醛处理PBMCs 10 min,使组蛋白和DNA发生交联,再用0.125 mol/L甘氨酸在室温下处理5 min终止交联反应。用预冷1×PBS 10 mL清洗细胞2次后,300 μL溶解缓冲液(10 mmoL/L Tris-HCl pH 8.0;100 mmoL/L NaCl;1 mmoL/L乙二胺四乙酸pH 8.0;0.1%脱氧胆酸钠和蛋白酶抑制剂)重悬细胞,并在冰上孵育30 min,超声处理细胞悬液4 min。随后在4℃下离心10 min,弃上清,获得PBMCs片状碎屑物溶解液,分装为3份,第1份与H3K4三甲基化抗体进行免疫沉淀反应;第2份和第3份分别作为内参(input contro1)和阴性对照(negative control),在第1份溶解液和阴性对照中加入50 μL磁珠并在4℃孵育1 h,使用低盐、高盐、氯化锂溶液和TE缓冲液洗涤,随后加热65℃,5 h解交联、RNase A和proteinase K消化,酚氯仿抽提DNA。然后进行DNA扩增并对扩增产物进行纯化。Cy3或Cy5荧光基团标记ChIP DNA和Input DNA,作为芯片杂交的荧光探针与12K CpG岛芯片进行杂交,使用GenePix 4000B芯片扫描仪(Axon Instruments)扫描芯片,使用Genespring分析软件分析原始数据。
5 CHIP-qPCR
ChIP步骤与上述 ChIP-chip一样。采用ABI7700 real-time PCR仪对免疫沉淀的DNA、input control和negative control进行扩增。设计阳性基因扩增引物序列(Primer 5.0软件设计)。FCRL4基因:5'-TTGGAGTAATAGTGCCCAGCA -3',5'-TAGTCCCCAGAAGCCTCAGT - 3',286bp;SERPINB7基因:5'-TACAAGCTAGTTTTACTTTTTTGTTAAGCA - 3',240 bp;5'- TCAAGTACTCAAAAGTGCCTCAT-3';PTPRN2基因:5'-GTGACATTACAGAGCCCCGT - 3',5' - TTATTCCCCAGAGCGCCTTT-3',180 bp;MBP 基因:5'-AAGCAAACAGCTTCACAAATTCTC -3',5'- ACTTAATTCTGAGTAAGCAAGACATACAAC - 3',240 bp。测定标准曲线和熔解曲线,确保扩增的准确性和专一性,用Ct(threshold cycle)值表示样品中模板的相对含量,采用 2-ΔΔCt法[5]进行分析。
6 荧光qRT-PCR
用Trizol试剂提取RNA并测定RNA浓度值,采用逆转录试剂盒合成cDNA。设计阳性基因和内参基因(GAPDH)扩增引物序列。FCRL4基因:5'-CTCTGACTTGCAATGGATTTCAG - 3',5' - CCAAGTATATTTCACAGCAGTCAA - 3',139 bp;SERPINB7基因:5'-TCCATTTTGCAATGGCCT -3',5'- TATCAGAAAAAACTCTTTTCAGTTGAGAC - 3',270 bp;PTPRN2基因:5'- GCCAGAAGGTTCCGGCAAT -3',5'- CAGGTAGGTTTTCGGGAGGT -3',170 bp;MBP 基因:5'- CACACCAAGGGAGACCATGT - 3',5'-CCTTTTTGGCCCTTTGGTCC - 3',300 bp;GAPDH 基因:5'- CCATGGAGAAGGCTGGGG - 3',5'-CAAAGTTGTCATGGATGACC -3',250 bp。测定标准曲线和熔解曲线,确保扩增的准确性和专一性,用Ct值表示样品中模板的相对含量,采用 2-ΔΔCt法[5]进行分析。
7 MeDIP-qPCR
采用Sss I甲基转移酶法(methyl accepting assay/methyl group acceptance assay)对受试对象进行总体DNA甲基化水平分析[6]。具体操作步骤如下:基因组 DNA 1 μL,Sss I酶(4 ×106U/L)1 μL,10 × NE buffer 22 μL,[3H]- SAM(1.6 mmol/L)12 μL,三蒸水14 μL,混匀,37℃孵育 1 h。然后65℃孵育20 min终止反应。将反应混合液加到96孔板,洗去未结合DNA的[3H]-SAM;取出板底座,每孔加入闪烁液100 μL,在Microbeta液闪仪上检测每个孔的每分钟脉冲数(counts/min),计算整体基因组甲基化率。MeDIP-qPCR具体实验步骤如下:吸取1.5 mL的裂解缓冲液至冷冻的细胞管中,用移液器吹打混匀,冰上放置10 min,800×g、4℃离心5 min,弃上清,用600 μL的裂解缓冲液重悬沉淀,混匀,加入20 g/L的蛋白酶K溶液5 μL,混匀,56℃消化至溶液呈澄清状。冷却至室温,加入5 μL 5 g/L RNase消化30 min,转入新的1.5 mL EP管。加入等体积的酚 -氯仿溶液(酚∶氯仿∶异戊醇 =25∶24∶1),剧烈振荡15 s,室温静置 5 min,12000×g、4℃离心 10 min,将上清转入新的1.5 mL EP管。加入20 μL 5 mol/L 的 NaCl、1 mL 无水乙醇,冰上放置 10 min,12000×g、4℃离心10 min,弃上清。用75%乙醇清洗沉淀2次 ,室温放置自然风干,200 μL纯水溶解沉淀得到DNA样品。超声处理DNA样品,加入5'-甲基胞嘧啶抗体,4℃、垂直混匀器上孵育过夜;加入磁珠偶联Ⅱ抗,4℃、垂直混匀器上孵育2 h;用1 mL预冷的溶液[2 mmol/L EDTA,20 mmol/L Tris(pH 8.0),1%Triton X - 100,0.1%SDS,150 mmol/L NaCl]洗涤3-5 min,然后将EP管放到磁力架上,放置10 min直至磁珠全部被吸附到管壁上,弃上清;洗脱的DNA纯化后对各样品的目的基因和对照(input)分别进行real-time PCR反应。设计阳性基因扩增引物序列如下,FCRL4基因:5'-AGTTTGATTAATATGGCGAAATTTC - 3',5'- TAACCAAAAAACTCCTAACCTCGTA -3',141 bp;SERPINB7 基因:5'-TTTGTTAGAAATGTAAATGTTGGTC - 3',5'- ATAATCTCGATCTCCTAACCTCGTA -3',107 bp;MBP 基因:5'-TTTGGTTATGAGGAAGAGAATTTAC -3',5'-TCTAAAACTACCACACATTACCGAT -3',200 bp;PTPRN2基因:5'-TGATTTAGATTATAAATTAATTATTCG - 3',5'- ATAATAAAAAACGAACGCGAA -3',137 bp。测定标准曲线和熔解曲线,确保扩增的准确性和专一性,用Ct值表示样品中模板的相对含量,采用 2-ΔΔCt法[5]进行分析。
8 统计学处理
SPSS 13.0 统计软件分析采用 2-ΔΔCt法处理得出的数据。每部分的荧光定量PCR均进行3次实验,相对定量数据采用均数±标准差()表示,两组数据比较采用t检验。
结 果
1 CpG岛芯片数据分析
与健康对照组相比,IgAN患者组总体的H3K4三甲基化水平升高,甲基化比率为0.626±0.050 vs 0.351 ±0.024(P <0.05),见图 1。使用 GenePix 4000B芯片扫描仪配套软件计算所有基因的Cy3/Cy5值(ratio值),筛选Ratio值>2的所有数据项,获得组蛋白H3K4三甲基化(H3K4me3)靶点图谱。结果显示321条探针H3K4me3出现显著差异,其中154条探针显示为H3K4me3水平升高,167条探针显示H3K4me3水平下调,共涉及到83个基因。表1中列举了20个H3K4me3水平上调或下降的基因。比较IgAN患者与健康者之间组蛋白H3K4三甲基化的差异,根据ChIP-chip的结果,我们选择2个H3K4三甲基化水平增高的基因(FCRL4和SERPINB7)和2个H3K4三甲基化水平降低的基因(PTPRN2和MBP)进行下一步的研究。
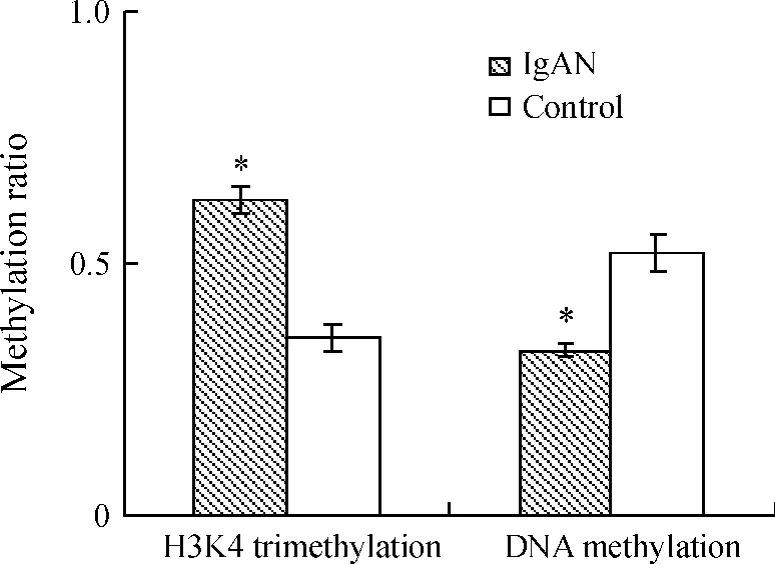
Figure 1.The levels of histone H3 lysine 4 trimethylation and DNA methylation in IgAN patients.*P <0.05 vs control.图1 IgAN患者总体H3K4三甲基化和DNA甲基化水平
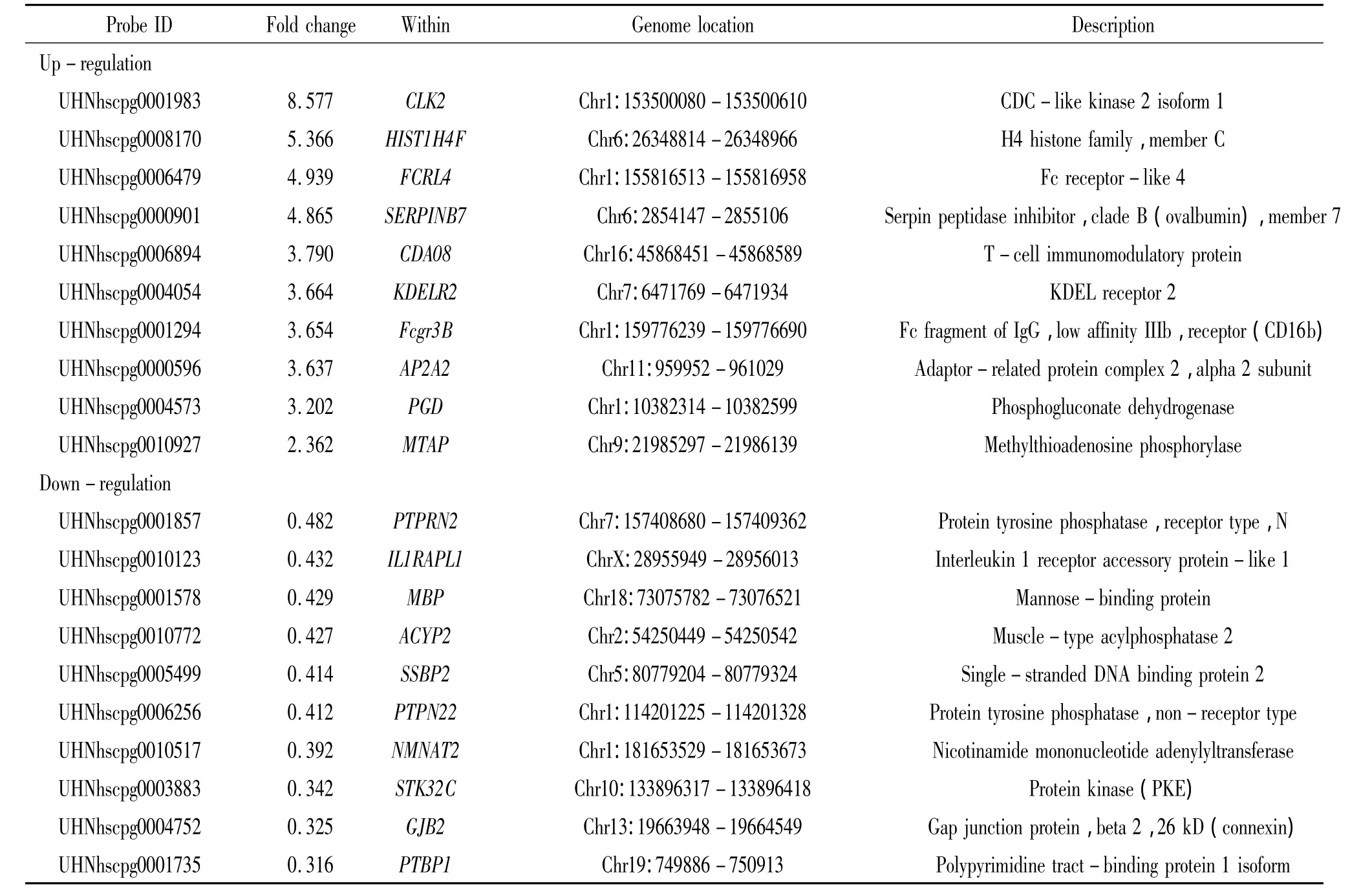
表1 CpG岛芯片IgA肾病患者与健康对照之间2倍以上和以下改变的H3K4me3基因Table 1.The 20 selected genes with H3K4me3 alterations between IgAN patients and healthy subjects
2 ChIP-qPCR验证结果
为了证实CpG岛芯片结果的准确性,我们运用荧光定量PCR对免疫沉淀的DNA进行分析。如表2所示,IgAN患者4个基因的荧光定量PCR结果与正常对照之间存在显著差异(P<0.05),与芯片分析结果相一致。
表2 4个阳性基因ChIP-qPCR验证结果Table 2.The validation results of ChIP-qPCR from the 4 selected genes(.n=40)

表2 4个阳性基因ChIP-qPCR验证结果Table 2.The validation results of ChIP-qPCR from the 4 selected genes(.n=40)
*P <0.05 vs control.
Group FCRL4 SERPINB7 PTPRN2 MBP IgAN 1.72 ±0.03* 2.13 ±0.01* 0.52 ±0.01* 0.31 ±0.03*Control 1.00 ±0.00 1.00 ±0.00 1.00 ±0.00 1.00 ±0.00
3 阳性基因mRNA的表达
IgAN患者4个阳性基因的mRNA表达与正常对照之间存在显著差异(P<0.05),见表3。H3K4三甲基化增高的2个基因mRNA表达增加;反之,H3K4三甲基化降低的基因mRNA表达也随之下降。
表3 4个阳性基因mRNA表达差异Table 3.The mRNA expression of the 4 selected genes(.n=40)

表3 4个阳性基因mRNA表达差异Table 3.The mRNA expression of the 4 selected genes(.n=40)
*P <0.05 vs control.
Group FCRL4 SERPINB7 PTPRN2 MBP IgAN 2.04 ±0.05* 1.87 ±0.02* 0.23 ±0.01* 0.48 ±0.05*Control 1.00 ±0.00 1.00 ±0.00 1.00 ±0.00 1.00 ±0.00
4 DNA甲基化水平检测结果
与健康对照组相比,IgAN患者组总的DNA甲基化水平降低,甲基化比率为 0.326±0.016 vs 0.518±0.037(P<0.05),见图1。IgAN 患者4个阳性基因DNA甲基化水平与正常对照之间存在显著差异(P<0.05),FCRL4基因DNA甲基化水平降低,另外3个基因DNA甲基化水平增高,见表4。
表4 4个阳性基因DNA甲基化水平差异Table 4.The DNA methylation levels of the 4 selected genes(.n=40)

表4 4个阳性基因DNA甲基化水平差异Table 4.The DNA methylation levels of the 4 selected genes(.n=40)
*P <0.05 vs control.
Group FCRL4 SERPINB7 PTPRN2 MBP IgAN 0.32 ±0.04* 1.96 ±0.04* 2.56 ±0.03* 2.04 ±0.02*0.00 Control 1.00 ±0.00 1.00 ±0.00 1.00 ±0.00 1.00 ±
讨 论
DNA甲基化和组蛋白修饰是表观遗传的两种主要修饰形式,在进化过程中组蛋白甲基化和DNA甲基化两者在机能上经常联系在一起。组蛋白H3K4甲基化可激活基因转录,而DNA甲基化则可抑制基因的转录[7,8]。
IgAN是以IgA为主的免疫球蛋白弥漫沉积于肾小球系膜区的一种自身免疫性疾病,我们的实验结果显示IgAN病人总体H3K4三甲基化水平升高,基因组DNA甲基化水平降低。我们选择4个与IgAN致病可能存在关联的基因,2个H3K4三甲基化水平增高的基因为FCRL4和SERPINB7,2个H3K4三甲基化水平降低的基因PTPRN2和MBP,探讨组蛋白H3K4三甲基化与DNA甲基化之间的关联。
FCRL4(Fc receptor-like 4)基因编码一种免疫球蛋白Fc段受体,它通过激活细胞因子,加剧IgAN的发展[9]。本研究发现PBMCs中FCRL4基因的组蛋白H3K4三甲基化水平有显著增强,DNA甲基化水平降低,mRNA表达水平增高。
PTPRN2(protein tyrosine phosphatase,receptor type,N polypeptide 2)是一种酪氨酸磷酸酶蛋白,可通过抑制酪氨酸激酶而降低肾脏足细胞表达血管内皮生长因子(vascular endothelial growth factor,VEGF),避免肾脏排出过多的蛋白[10]。本研究发现,PTPRN2基因H3K4三甲基化水平降低,DNA甲基化水平增高,mRNA表达也显著降低,提示低浓度的PTPRN2不足以抑制酪氨酸激酶的活性。
甘露醇结合蛋白(mannose-binding protein,MBP)是一种属于免疫系统的防御分子,是人体与外环境的首道防线,Zhang等[11]研究发现,在反复发生感染、怀疑有免疫调理缺陷的动物模型中,MBP血清水平显著低于正常对照组。我们在IgAN病人中发现,MBP基因H3K4三甲基化水平降低,DNA甲基化水平增高,同时mRNA表达也显著降低,提示有可能因此导致IgAN病人体内MBP蛋白含量减少,免疫功能紊乱。
上述3个基因中,组蛋白H3K4位点三甲基化修饰和DNA甲基化呈现协同作用。FCRL4基因H3K4位点三甲基化水平增高,DNA甲基化降低,均促进基因mRNA表达;而MBP和PTPRN2基因H3K4位点三甲基化水平降低,DNA甲基化增高,均抑制基因mRNA 表达。Irvine等[12]和 Okitsu等[13]的研究认为组蛋白在H3K4位点的双甲基化和DNA甲基化存在协同作用,从我们的研究结果来看,三甲基化的H3K4位点和DNA甲基化之间同样存在协同作用。
然而,SERPINB7(serpin peptidase inhibitor,clade B,member 7)基因的H3K4位点三甲基化和DNA甲基化呈现出矛盾的结果。SERPINB7是丝氨酸蛋白酶抑制剂进化枝B的第7位成员,为近年发现的特异性表达于肾脏的基因。研究表明,该基因表达产物可以与纤溶酶共价结合而抑制其活性,继而抑制细胞外基质(extracellular matrix,ECM)降解,参与肾脏纤维化[14]。该基因在正常的肾小球系膜细胞有微弱的表达,而在增殖性肾小球疾病表达明显增强,本研究结果显示IgAN患者 SERPINB7基因H3K4三甲基化水平、mRNA表达均显示为增高,与戴春等[15]的报道相符合。但是,SERPINB7基因DNA甲基化的结果也是明显增高。可见,SERPINB7基因mRNA表达与组蛋白H3K4me3甲基化修饰作用相同,而与DNA甲基化作用相反。我们推测在IgAN患者PBMCs内组蛋白甲基化和DNA甲基化修饰的相互作用过程中,H3K4三甲基化修饰作用可能处于优势地位;也有可能有其它的后天因子参与了SERPINB7基因修饰。这也说明了DNA和组蛋白甲基化修饰效应结果的复杂性,在不同的基因中,甲基化和组蛋白甲基化在基因表达过程中所起的关键作用可能不同。
本研究选择4个与IgAN发病密切相关的基因,研究组蛋白H3K4三甲基化水平和DNA甲基化水平,结果表明这2种共价修饰都可能参与了IgAN的发病机制,但其最终机制仍需要将来通过大量的基因检测来证实。
[1]Arai E,Kanai Y.Genetic and epigenetic alterations during renal carcinogenesis [J].Int J Clin Exp Pathol,2010,4(1):58-73.
[2]袁 敏,沈 南,唐元家,等.DNA甲基化与系统性红斑狼疮[J].中国病理生理杂志,2009,25(11):2217-2220,2224.
[3]Haas M.Histology and immunohistology of IgA nephropathy[J].J Nephrol,2005,18(6):676 -680.
[4]Wu H,Ji H.JAMIE:joint analysis of multiple ChIP-chip experiments[J].Bioinformatics,2010,26(15):1864-1870.
[5]Livak KJ,Schmittgen TD.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod[J].Methods,2001,25(4):402 -408.
[6]杨淋清,纪卫东,陶功华,等.结晶型硫化镍诱发细胞恶性转化过程中基因组总体DNA甲基化的改变[J].中华预防医学杂志,2010,44(7):622 -625.
[7]Shindo N.Histone modification:a new era of targeting epigenetics[J].Rinsho Ketsueki,2009,50(4):282 - 288.
[8]杨祖立,康 亮,黄美近,等.结直肠癌细胞中脾酪氨酸激酶基因甲基化状态和表达的关系[J].中国病理生理杂志,2008,24(9):1720 -1725.
[9]Kanamaru Y,Arcos- Fajardo M,Moura IC,et al.Fc alpha receptor I activation induces leukocyte recruitment and promotes aggravation of glomerulonephritis through the FcR gamma adaptor[J].Eur J Immunol,2007,37(4):1116-1128.
[10]Bukowski RM.Tyrosine kinase inhibitors in advanced renal cell carcinoma:the evolving treatment paradigm[J].Clin Genitourin Cancer,2009,7(1):9 -10.
[11]Zhang L,Ye F,He Y,et a1.Establishment of a mouse IgA nephropathy model with the MBP-20-peptide fusion protein[J].Anat Rec(Hoboken),2010 ,293(10):1729-1737.
[12]Irvine RA,Lin IG,Hsieh CL.DNA methylation has a local effect on transcription and histone acetylation[J].Mol Cell Bid,2002,22(19):6689 -6696.
[13]Okitsu CY,Hsieh CL.DNA methylation dictates histone H3K4 methylation [J].Mol Cell Biol,2007,27(7):2746-2757.
[14]Li YJ,Du Y,Li CX,et a1.Family - based association study showing that immunoglobulin A nephropathy is associated with the polymorphisms 2093C and 2180T in the 3'untranslated region of the megsin gene[J].J Am Soc Nephrol,2004,15(7):1739 -1743.
[15]戴 春,郭德森.基因干扰对人肾小球系膜细胞megsin基因表达及α平滑肌肌动蛋白和转化生长因子β1生成的影响[J].中华肾脏病杂志,2008,24(2):140-141.

