几株酵母菌的分子系统学鉴定
王泽举,王汝瑱,孙 悦,杨 莹,刘延琳
几株酵母菌的分子系统学鉴定
王泽举,王汝瑱,孙 悦,杨 莹,刘延琳*
(西北农林科技大学葡萄酒学院,陕西省葡萄与葡萄酒工程技术研究中心,陕西 杨凌 712100)
通过对酵母菌5.8S核糖体RNA的ITS区域PCR扩增及限制性酶切片段多态性(RFLP)的图谱分析以和26S rDNA D1/D2区及5.8S-ITS区的序列分析,共鉴定13株分离自新疆鄯善地区的葡萄酒相关酵母菌。5.8S-ITS区的4种内切酶(HaeⅢ、Hin6Ⅰ、HinfⅠ、AluⅠ)的酶切分析产出4种特异性图谱。根据26S rDNA D1/D2区的序列分析和BLAST比对,将供试菌株鉴定为4个属4个种,分别为Saccharomyces cerevisiae、Candida zemplinina、Hanseniaspora uvarum、Pichia kluyveri var. kluyveri;而根据5.8S-ITS-RFLP及5.8S-ITS序列分析,将供试菌种鉴定为Saccharomyces cerevisiae、Candida zemplinina、Hanseniaspora uvarum、Issatchenkia terricola 4个属4个种。3种方法对供试的10个菌株的鉴定结果一致,而对另外3株的鉴定结果不同。
葡萄酒相关酵母;分子系统学;鉴定;5.8S-ITS-RFLP;26S rDNA D1/D2序列分析;5.8S-ITS序列分析
传统的酵母菌株鉴定方法建立在菌株的形态、生理特性和生物化学特性质差异之上。然而这些特征会受培养条件的影响,在某种程度上具有不确定性,为确保鉴定结果的可靠性,鉴定一株菌经常需要完成50~100项实验[1]。尽管酵母菌的培养及菌落的观察极其简单,但其费时费力且重复性差的弊端却不可避免。与之相比,分子鉴定以其所提供的简便、准确与多种选择性的方法成为一种重要的鉴定手段。近年来,新的鉴定方法主要建立在核酸序列上。26S、18S、5.8S、5S核糖体是较为保守的片段,其所带的信息是菌种鉴定的依据。其中,ITS-5.8S(内转录区)位于小亚基和大亚基rRNA基因之间,该区可分为ITS1区和ITS2区。5.8S-ITS的限制性内切酶消化产生的片段大小和数目存在多态性,对于每一个种ITS-5.8S rDNA的限制性内切酶组合来说,所产生的片段是特异性的,作为该种菌ITS-5.8S rDNA所特有的“指纹”(即片段差异的多态性),可依此进行种的水平上的归类[2]。26S rDNA在对大量未知菌种的快速鉴定和系统发育分析中显示了很大的优势[3]。Kurtzman等[4]在对将近500株子囊酵母的26S rDNA D1/D2区的测序及系统发育分析中发现同一种内不同菌株间D1/D2区的核苷酸差异不超过1%。依此为依据,通过26S rDNA D1/D2区测序并进行核酸序列数据库查询,可方便地将酵母菌鉴定到种。
本研究通过最常用的3种分子系统学方法,即5.8SITS-RFLP分析、26S rDNA D1/D2区序列分析、5.8S-ITS区序列分析及GenBank查询和BLAST比对,对分离自新疆鄯善葡萄产区的酵母菌进行分类鉴定,同时比较几种分子系统学方法在鉴定结果上的异同。
1 材料与方法
1.1 材料
自新疆鄯善葡萄酒产区,从自然发酵葡萄醪分离获得酵母菌。在形态和生理生化鉴定基础上,选取13株代表性类型进行分子系统学鉴定。
1.2 方法
1.2.1 酵母菌分离
从新疆鄯善葡萄自然发酵醪中分离获得酵母菌株,菌株分离使用YPD培养基(10g/L酵母浸粉、20g/L蛋白胨、20g/L葡萄糖、20g/L琼脂,自然pH值)。
1.2.2 酵母DNA提取
采用石英砂破壁法[5]。
1.2.3 5.8S-ITS区和26S rDNA D1/D2基因序列的PCR扩增
按Guillamon等[6]的反应程序、使用引物ITS1 (5′-TC CGTAGGTGAACCTGCGG-3')和ITS4 (5′-TCCTC CGCTTATTGATATGC-3')[7]进行5.8S-ITS区的PCR扩增。测序结果查询GenBank数据库,采用BLAST在线工具进行序列相似性比较。
利用引物NL1和NL4扩增菌株的26S rDNA D1/D2基因序列[6],将测序产物送交生工生物(上海)有限公司测序。测序生物结果查询GenBank数据库,采用BLAST在线工具进行序列相似性比较。
1.2.4 限制性内切酶消化
纯化后的PCR扩增产物分别用限制性内切酶HaeⅢ、HinfⅠ、Hin6Ⅰ和AluⅠ(MBI产品)按产品说明书消化;30g/L琼脂糖凝胶电泳检测酶切片段多样性。
2 结果与分析
2.1 5.8S-ITS-RFLP分析
由引物ITS1和ITS4扩增的核糖体rRNA的重复单元包括两个非编码的内转录区ITS1和ITS2及5.8S rRNA基因。PCR产物的片段长度多样性也显示了种属间的差异[8]。在所鉴定的菌株中酿酒酵母的PCR产物长度为820~880bp,葡萄汁有孢汉逊酵母为760~800bp,伊萨酵母为400~480bp,毕赤酵母为420bp,假丝酵母为400~420bp。
5.8 S-ITS的 PCR产物由限制性内切酶消化所得的电泳图谱如图1所示。一般同种酵母具有相同的酶切位点数目,所得的酶切片段数目和大小相同[9]。通过酶切图谱分析,被测菌株可分别归为4个不同类型:Ⅰ类包括A24-3-1、A27-3-2、A24-2-2、A27-3-5;Ⅱ类包括A9-5-3、A5-5-5、A9-4-7、A13-1-3;Ⅲ类包括A6-1-6、A27-3-4、A27-4-1;Ⅳ类包括A9-3-2、A11-1-1。
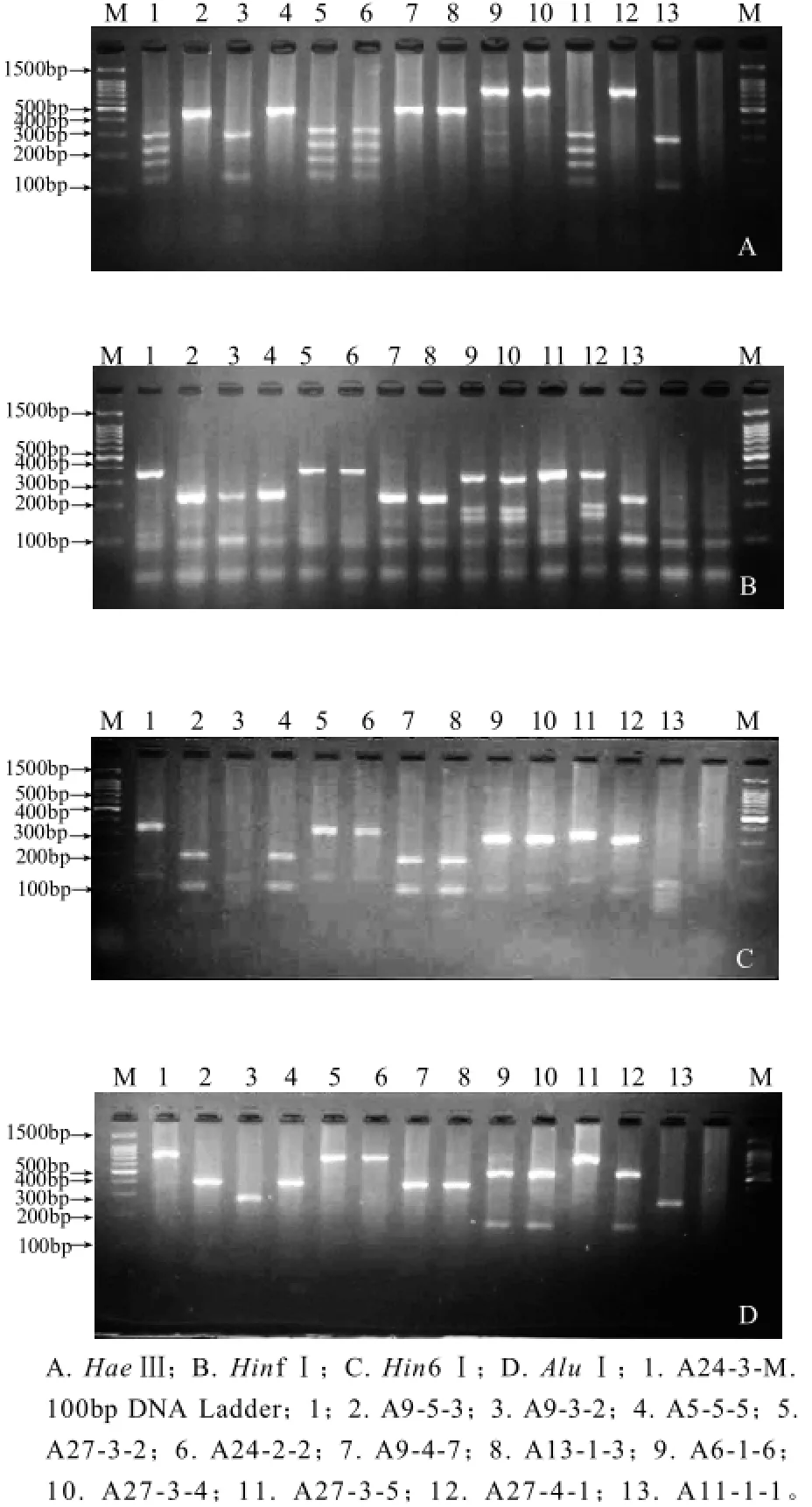
图1 鄯善地区菌株4种限制性内切酶消化的凝胶电泳图Fig.1 Restriction enzyme digestion analysis of PCR amplified 5.8S-ITS region from 13 yeast strains
PCR产物由4种限制性内切酶消化(HaeⅢ、HinfⅠ、Hin6Ⅰ和AluⅠ)消化片段见表1(100bp以下不可视片段未记录)。与已知文献[2,6,9]及5.8S-ITS-RFLP数据库(http:// yeast-id.com/)比对得出:菌株A24-3-1、A27-3-2、A24-2-2和A27-3-5可鉴定为Saccharomyces cerevisia,菌株A9-5-3、A5-5-5、A9-4-7和A13-1-3可鉴定为Candida zemplinina;菌株A6-1-6、A27-3-4和A27-4-1可鉴定为Hanseniaspora uvarum;菌株A9-3-2和A11-1-1可鉴定为Issatchenkia terricola。

表1 鄯善地区菌株限制性酶切片段长度Table 1 Lengths (in bp) of the 5.8S-ITS region of yeast strains from Shanshan amplified by PCR and the fragments obtained after digestion with four restriction endonucleases

表2 被测序菌株26S rDNA D1/D2片断大小及与相关菌株的序列相似性Table 2 26S rDNA D1/D2 fragment size of the sequenced strains and the identity with related yeasts
2.2 26S rDNA D1/D2区的测序及分析
根据26S rDNA D1/D2区的序列测定和GenBank查询及BLAST比对,将供试菌株鉴定到种的水平,包括3株Candida zemplinina、5株Hanseniaspora uvarum、4株Saccharomyces cerevisia、1株Pichia kluyveri var. kluyveri(表2)。
比较表1、2的结果,可以发现5.8S-ITS-RFLP的鉴定结果与26S rDNA的D1/D2区序列分析的结果存在一些差异:它们对供试的10株菌的鉴定结果一致,而对另外3株的鉴定结果不同(表3)。其中,对于菌株A9-4-7,前者的鉴定结果为H.uvarum,而后者将其鉴定为C.zemplinina;对于菌株A11-1-1和A9-3-2,前者的鉴定结果均为I. terricola,后者将其分别鉴定为P.kluyveri var. kluyveri和H.uvarum。
对鉴定结果存在分歧的3个菌株A9-4-7、A11-1-1和A9-3-2,继续进行5.8S-ITS测序和BLAST比对,结果均同5.8S-ITS-RFLP分析的鉴定结果相一致(表4),而与26S rDNA的D1/D2区序列分析结果不同。
由于菌株A9-4-7的5.8S-ITS的PCR产物为410bp,而H.uvarum的为800bp,因而不能接受26S rDNA D1/D2区的测序和鉴定结果;根据5.8S-ITS区的测序和序列比对结果,A9-4-7可确认为C. zemplinina。而菌株A11-1-1和A9-3-2的分类归属,尚需进一步鉴定。

表4 新疆鄯善地区酵母菌株5.8S-ITS测序结果Table 4 5.8S-ITS fragment size of the sequenced strains and their identity with related yeasts

表3 3种分子系统学鉴定方法的结果比较Table 3 Comparison of identification results by the three different methods
3 讨 论
5.8 S-ITS- RFLP分析是一种无需进行测序就可获得较为精确的鉴定结果的分析方法,而且简单易行,目前多用于菌种种间的区分。Montrocher等[8]利用rDNA扩增产物的RFLP分析和ITS序列分析方法研究了属于狭义酿酒酵母组(S.cerevisiae、S.bayanus、S.paradoxus、S.pastorianus)的菌株之间的系统发育关系,两种分析方法显示了非常相似的结果。两种方法都将这些菌株区分为较为明显的两大系统发育类群,即S.cerevisiae群和S.bayanus群,证实了S.cerevisiae和S.bayanus是两个定义明确的分类群。然而,另外两个现今被普遍接受的归入狭义酿酒酵母组的S.paradoxus和S.pastorianu,不能明显地与S.cerevisiae和S.bayanus区分开来,而且,无论是对于PCR-RFLP还是在ITS序列分析中S.paradoxus在狭义酿酒酵母组中处于最外层的位置。
de Llanos Frutos等[10]利用此技术结合序列分析对假丝酵母属进行了分子生物学鉴定,并且建立了包含有假丝酵母属的75个种共109株酵母菌的数据库。Fernadez-Espinar等[2]对酵母属菌种进行了鉴定,结果表明这是一种可进行菌种鉴定快捷而准确的方法。las Heras-Vazquez等[11]利用此技术结合序列分析对从橘子及其果汁自然发酵过程中分离到的酵母菌初步进行鉴定,鉴定出了Candida tropicalis、Clavispora lusitaniae、Hanseniaspora uvarum、Pichia anomala、Pichia fermentans、Rhodotorula mucilaginosa、Saccharomyces cerevisiae、Saccharomyces unisporus和Trichosporon asahii等酵母,结和形态学、生理学和生物化学特性分析表明该方法可以快速、准确地进行酵母菌的鉴定。
限制性内切酶的选择很关键,选用不同的内切酶会产生不同的酶切图谱。Naumova等[12]指出Hae Ⅲ对于区分酵母属间酵母具有很好的效果,他们在对Saccharomyces属的基因差异研究中指出Hae Ⅲ可以很好的将S. cerevisiae从S. paradoxus和S. bayanus中区分出来。Fernadez-Espinar等[2]使用了9种酶对酵母属(Sacch aromyces)的酵母进行了5.8S-ITS区的RFLP分析,也指出HaeⅢ对于区分酵母属间酵母具有很好的效果,对S.cerevisiae、S.paradoxus、S.pastorianus、S. castelli、S.dairenensis、S.exiguus、S.kunashirensis、S.martiniae、S.rosinii、S.servazzii等都可产生不同的限制型酶切片段。但无法区分S.bayanus与S.paradoxus。
HinfⅠ、Hin6Ⅰ在假丝酵母的分类中有广泛的应用,但de Llanos Frutos等[10]在研究中指出,5.8S rDNA的RFLP不能区分假丝酵菌的有性态和无性态。在德克酵母属中,5.8S rDNA的RFLP分析不能很好的区分该属酵母,因为在个别种的序列上仅有一个碱基的差异。
Hin6Ⅰ与HinfⅠ在葡萄酒相关酵母的RFLP分析鉴定中具有比较显著的多态性,这在本研究中得到证实,但HaeⅢ不能有效区分供试的非酵母属酵母,这在本实验中也得到证实;AluⅠ的多态性也较差,不能用来区分所有实验菌株。
虽然通过对酶切片段大小的估计和文献查询及数据库比对(5.8S-ITS-RFLP数据库http://yeast-id.com/),可以将供试酵母菌快速鉴定到种。但5.8S-ITS-RFLP数据库还未涵盖酵母的所有种,实际工作中对酶切片段大小的估计也往往存在误差,使得近缘种的区分不易进行。
相比之下,随着DNA序列分析技术的日趋成熟和简易化,序列分析方法已经被广泛应用于酵母菌的分类鉴定以及系统学研究。其中,rRNA基因(rDNA)及其转录间区(ITS)的序列是最为常用的,包括18S rDNA、26S rDNA的D1/D2、ITS、5.8S rDNA。这些序列均己经公布于GenBank/EMBL/DDBJ等国际核酸序列数据库,为酵母菌的分类鉴定带来了极大的便利。
酵母菌小亚基rRNA存在不同保守程度序列的镶嵌现象(从高度保守到半保守区到高可变区),使得该分子可以用来衡量较远的亲缘关系,也适宜于较近的亲缘关系。这个区域大约有1800bp,含有不同变异率的区段,可以用于种、属或更高等级分类群之间的系统关系研究。James等[13]根据18S rDNA基因序列,对Saccharomyces属及相关的子囊菌酵母(包括Kluyveromyces、Torulaspora、Zygosaccharomyces)进行了分子系统学分析,结果显示在分子系统树上狭义酿酒酵母组内的4个种形成一个独立的分支,表明其非常近缘的系统演化关系,但该属内的其他种却分散在不同的分支之中,并与Kluyveromyces、Torulaspora、Zygosaccharomyces属的某些种聚合在一起,分子系统学研究表明,以形态和生理生化特性界定的Saccharomyces属及相关的子囊菌酵母属均是异源性的非自然属,应作进一步的调整。
ITS(internal transcribed spacer)区为核糖体内转录间隔区,位于18S rDNA和26S rDNA之间,包括ITS1和ITS2两个片断,中间包含一个5.8S rDNA片段。通常,在分析ITS区域序列时也将5.8S rDNA一起进行考虑,但5.8S rDNA相对保守,从中获取的信息量很少[14]。ITS区域与rDNA转录区相比具有较高的变异率,对于亲缘关系较近的菌株间的区分是比较有效的[8]。Takashima[15]、Sugita[16-17]等研究表明ITS区域的差异大于1%时就可能代表不同的种,然而对于不同的属而言,情况会有所不同。
ITS区序列常常与其他序列相结合,应用于酵母菌分类的系统学研究中。Sipiczki[18]利用ITS和大亚基序列分析,对一株贵腐酒中的耐寒耐渗透性菌C. zemplinina进行了鉴定,结果分别显示了8% 和 8.1%的差异率,分析认为这是一种新种Candida zemplinina sp. nov.。
26S rDNA的Dl/D2区域位于大亚基的5'端,序列长度在600bp左右,该段区域具有较高的变异率,可以用于亲缘关系较近的菌株之间的分类研究。Peterson等[3]研究了核糖体大亚基5'端可变区D1/D2区域的序列,他们的工作显示同一菌种在 Dl/D2区域一般有少于1%的替代率,而不同种的菌株有比这个数字更大的值,因此为菌株的鉴定提供了一个经验性方法。Kurtzman等[4]利用D1/D2序列分析全面评价了囊括整个子囊菌酵母的500多种酵母菌的系统发育关系,数据显示用该区域的序列足以确定大部分菌种的发育地位,其结果预测了55个当今被确认的分类群是早期描述的菌种的异名。他们认为酵母菌同一个种的不同菌株其碱基差异一般不超过1%,而属于不同种的菌株其核甘酸序列差异一般较大,根据这一标准,可以将绝大部分种区分开。Fell等[19]根据Dl/D2区域对担子菌酵母做了系统学研究,它们的研究结果也得出了相似的结果。目前,一般认为Dl/D2区碱基差异在1%以上就代表了不同的种,尽管区分紧密相关的种时需要运用ITS区,但大多数种都可以用D1/D2区序列分析进行区别。
van Keulen等[20]根据26S rDNA的D1/D2区序列及酶切分析研究了Lake Erie地区霞多丽、雷司令、灰比诺自然发酵过程中分离的酵母菌,研究发现该方法可以在属的水平上对酵母菌进行很好地区分,成功的鉴定出了Torulaspora delbrueckii、S. bayanus,并获得了与H.uvarum、P.kluyveri、C.stellata、S.cerevisiae相关的菌种。
目前,所有已描述的酵母菌种的大亚基rDNA D1/D2区序列都可以在核酸序列数据库上查询到,因此,D1/D2区序列通常用于种的水平上的鉴定。总的来说,大亚基D1/D2区序列分析结果与标准表型分析结果相一致,但有时具有相同D1/D2区序列的菌株,杂交遗传学和标准特征却表明它们不属于同一个种。
其中,26S rDNA的D1/D2区序列已建成了完备的分析数据库,用其进行酵母菌种的鉴定更为方便准确,其应用也最为广泛[19,21]。虽然26S rDNA D1/D2区的序列分析,可以有效的将一些未知菌株鉴定到种,但是对于一些特殊菌种也存在一定的不足,需要辅以其他的鉴定方法,比如18S rDNA测序[13]、ITS区测序或者5.8S rDNA限制性酶切分析等[9],甚至要做一些复杂的生理生化检测才能鉴定到种。Scorzetti等[21]研究表明,C.magnus、F. magnus和F. floriforme 的26S rRNA D1/D2区基因序列是完全相同的,只通过这段基因序列分析无法区分这3个种,但是在5.8S-ITS区序列存在很大的差异,通过5.8S-ITS区序列分析可以被很好的区别。
本研究通过最常用的3种分子系统学方法,即5.8SITS的RFLP分析、26S rDNA D1/D2区序列分析、5.8SITS区序列分析及GenBank查询和BLAST比对,对分离自新疆鄯善葡萄产区的13株酵母菌进行了分类鉴定,同时比较了它们在鉴定结果上的异同。结果发现5.8S-ITSRFLP的结果与5.8S-ITS序列分析的鉴定结果相符合;而5.8S-ITS-RFLP及5.8S-ITS分析与26S rDNA的D1/D2区序列分析结果存在一些差异,3种方法对供试的10个菌株的鉴定结果一致,而对另外3株的鉴定结果不同。对于菌株A9-4-7的鉴定,否定了26S rDNA的D1/D2区序列分析认为其为H.uvarum的结果,因为该菌株5.8SITS区PCR产物的大小与H.uvarum的相差太大;而综合5.8S-ITS区PCR产物的大小及5.8S-ITS-RFLP分析和5.8S-ITS序列分析,将菌株A9-4-7鉴定为C.zemplinina,这也在经典分类鉴定的结果中得到了证实(数据未显示)。
对于菌株A11-1-1和A9-3-2,鉴于几种分类鉴定结果的分歧,它们的分类地位尚不能确定,还需佐以经典分类方法和/或其他的分子生物学方法进行确认。
本研究对酵母菌鉴定中最常用的3种分子系统学方法的鉴定结果进行比较得出:对酵母菌的鉴定,不能过分依赖于分子系统学的结果;如果三者的鉴定结果能相互印证,则可以确认鉴定的结果,而如果相互之间的结果不相符合,则需根据具体的情况确认,必要时再佐以经典分类方法和/或其他的分子生物学方法。
[1]LIN C C S, FUNG D Y. Conventional and rapid methods for yeast identification[J]. Crit Rew Microbiolo, 1987, 14(4): 273-289.
[2]FERNADEZ-ESPINA M T, ESTEVE-ZARZOSO B, QUEROL A, et al. RFLP analysis of the ribosomal internal transcrived spacers and the 5.8S rRNA gene region of the genus Saccharomyces: a fast method for species identification and the differentiation of flor yeasts[J]. Antonie Van Leeuwenhoek, 2000, 78: 87-97.
[3]PETERSON S W, KURTZMAN C P. Ribosomal RNA sequence divergence among sibling species of yeasts[J]. Mycologia, 1991, 83(4): 397-402.
[4]KURTZMAN C P, ROBNETT C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences[J]. Antonie Van Leeuwenhoek, 1998, 73 (4): 331-371.
[5]周小玲, 沈微, 饶志明, 等. 一种快速提取真菌染色体DNA的方法[J]. 微生物学通报, 2004, 31(4): 89-92.
[6]GUILLAMON J M, SABATE J, BARRIO E, et al. Rapid identification of wine yeast species based on RFLP analysis of the ribosomal internal transcribed spacer (ITS) region[J]. Archives of Microbiology, 1998, 169 (5): 387-392.
[7]WHITE T J, BRUNS T, LEE S, et al. A guide to methods and applications[M]// INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR protocols. San Diego: Academic Press, 1990: 315-322.
[8]MONTROCHER R, VERNER M C, BRIOLAY J, et al. Phylogenetic analysis of the Saccharomyces cerevisiae group based on polymorphisms of rDNA spacer sequences[J]. Int J Syst Bacteriol, 1998, 48: 295-303.
[9]ESTEVEN-ZARZOSO B, BELLTCH C, URUBURU F, et al. Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers[J]. Int J Syst Bacteriol, 1999, 49: 329-337.
[10]de LLANOS FRUTOS R, FERNANDEZ-ESPINAR M T, QUEROL A. Identification of species of the genus Candida by analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers[J]. Antonie Van Leeuwenhoek, 2004, 85(3): 175-185.
[11]las HERAS-VAZQUEZ F J, MINGORANCE-CAZORLA L, CLEMENTE-JIMENEZ J M, et al. Identification of yeast species from orange fruit and juice by RFLP and sequence analysis of the 5.8S rRNA gene and the two internal transcribed spacers[J]. FEMS Yeast Research, 2003, 3(1): 3-9.
[12]NAUMOVA E S, ZHOLUDEVA M V, MARTYNENKO N N, et al. The molecular genetic differentiation of cultured Saccharomyces strains[J]. Microbiology, 2005, 74(2): 179-187.
[13]JAMES S A, CAI J, ROBERTS I N, et al. A phylogenetic analysis of the genus Saccharomyces based on 18S rRNA gene sequencer: description of Saccharomyces kunashirensis sp. nov. and Sacchromyces martiniae sp. nov. [J]. Int J Syst Bacterial, 1997, 47(2): 453-460.
[14]VALENTE P, RAMOS J P, LEONCINI O. Sequencing as a tool in yeast molecular taxonomy[J]. Can J Microbiol, 1999, 45: 949-958.
[15]TAKASHIMA M, NAKASE T. Four new species of the genus Sporobolomyce isolated from leaves in Thailand[J]. Mycoscience, 2001, 41: 357-369.
[16]SUGITA T, CAFETE-GIBAS C F, TAKASHIMA M, et al. Three new species of Bullera isolated from leaves in the Ogasawara Islands[J]. Mycoscience, 1999, 40: 491-501.
[17]SUGITA T, NISHIKAWA A, IKEDA R, et al. Identification of medically relevant Trichosporon species based on sequences of internal transcribed spacer regions and construction of a database for Trichosporon identification [J]. J Clin Microbiol, 1999, 37: 1985-1993.
[18]SIPICZKI M. Candida zemplinina sp. nov., an osmotolerant and psychrotolerant yeast that ferments sweet botrytized wines[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53: 2079-2083.
[19]FELL J W, BOEKHOUT T, FONSECA A, et al. Biodiversity and systematics of basidiomycetous yeasts as determined by large-subunit rDNA D1/D2 domain sequence analysis[J]. Int J Syst Evol Microbiol, 2000, 50(3): 1351-1371.
[20]van KEULEN H, LINDMARK D G, ZEMAN K E, et al. Yeasts present during spontaneous fermentation of Lake Erie Chardonnay, Pinot Gris and Riesling[J]. Antonie van Leeuwenhoek, 2003, 83: 149-154.
[21]SCORZETTI G, FELL J W, FONSECA A, et al. Systematics of basidiomycetous yeasts: a comparison of large subunit D1/D2 and internal transcribed spacer rDNA regions[J]. FEMS Yeast Res, 2002, 2(4): 495-517.
Molecular Phylogenic Identification of Wine-Related Yeasts
WANG Ze-ju,WANG Ru-zhen,SUN Yue,YANG Ying,LIU Yan-lin*(College of Enology, Northwest A&F University, Shaanxi Engineering Research Center for Viti-Viniculture, Yangling 712100, China)
In this study, thirteen wine-related wild yeast strains from Shanshan, Xijiang were identified by PCR-RFLP (restriction fragment length polymorphism) and sequence analysis of the 5.8S-ITS region and the 26S rDNA D1/D2 domains. The PCR amplified 5.8S-ITS region were digested with four restriction endonucleases, Hae Ⅲ, Hin6Ⅰ, HinfⅠand AluⅠ to obtain 4 specific patterns. According to the sequence analysis of the 26S rDNA D1/D2 domains and BLAST alignment, the 13 yeast strains were identified as Saccharomyces cerevisiae, Candida zemplinina, Hanseniaspora uvarum and Pichia kluyveri var. kluyveri, while they were identified as Saccharomyces cerevisiae, Candida zemplinina, Hanseniaspora uvarum and Issatchenkia terricola according to the PCR-RFLP and sequence analysis of the 5.8S-ITS region. The same identification results for 10 of the 13 isolated yeast strains were obtained using the three methods, whereas inconsistent identification results were obtained for other three.
wine-related yeasts;molecular phylogeny;identification;5.8S-ITS-RFLP;26S rDNA D1/D2 sequence;5.8S-ITS sequence
Q93-331
A
1002-6630(2012)15-0195-06
2011-07-01
国家现代农业(葡萄)产业技术体系项目(CARS-30-jg-3)
王泽举(1981—),男,硕士研究生,研究方向为酿酒微生物。E-mail:zejuwang@163.com
*通信作者:刘延琳(1966—),女,教授,博士,研究方向为葡萄-葡萄酒工程及酿酒微生物。E-mail:yanlinliu@nwsuaf.edu.cn

