热处理辣椒酱贮藏过程中残留微生物分析
丁 文,汪先丁,高 鹏,杨 虎,赵 敏,孙 群,*
(1.四川大学生命科学学院,生物资源与生态环境教育部重点实验室,四川 成都 610064;2.四川省原子能研究院,四川 成都 610101)
热处理辣椒酱贮藏过程中残留微生物分析
丁 文1,汪先丁1,高 鹏2,杨 虎1,赵 敏1,孙 群1,*
(1.四川大学生命科学学院,生物资源与生态环境教育部重点实验室,四川 成都 610064;2.四川省原子能研究院,四川 成都 610101)
四川郫县辣椒酱多采用热处理控制微生物生长以延长货架期,但热处理后残留微生物仍会导致辣椒酱腐败。辣椒酱经80℃、30min热处理后室温贮藏120d期间检测其中菌落总数、大肠菌群、霉菌和酵母数,分别采用16S rDNA和ITS序列鉴定分离的细菌和真菌菌株,并运用PCR-DGGE法分析贮藏终点辣椒酱中细菌群落结构。结果表明:热处理后辣椒酱中微生物减少了90%以上,残留微生物主要为芽孢杆菌(Bacillus sp.),在贮藏期内微生物数量则呈先上升后下降的趋势;PCR-DGGE检测发现贮藏终点辣椒酱中主要残留细菌为乳酸菌(Pediococcus sp.和Lactobacillus sp.)和蜡样芽孢杆菌(Bacillus cereus)。因此,热处理可使辣椒酱中部分微生物致死、亚致死或转为“存活但非可培养” (viable but non-culturable,VBNC)状态,但残留的芽孢杆菌(Bacillus sp.)和乳酸菌可能导致辣椒酱的腐败。有效控制芽孢杆菌将是延长辣椒酱货架期的关键因素。
热处理;货架期;菌落总数;PCR-DGGE;VBNC
四川拥有享誉全国的四大菜系之一——川菜,而辣味无疑是川菜中的重要风味之一。四川郫县地区不仅出产著名的郫县豆瓣,也有优质辣椒酱生产。郫县辣椒酱选用优良品种的红辣椒,经切碎、搅拌、盐封后发酵1~2月生产而成,产品具有独特的风味,不仅供四川本地食用,也有销往全国各地,甚至出口日本等国。然而,自然发酵的郫县辣椒酱中微生物数量及其活性均较高,且辣椒酱盐分一般不会太高(10%~15%),因而在后期贮藏前需要采用适当手段(如80℃热处理30min)进行微生物控制以保证其货架期。但实际生产中热处理效果有时不尽人意,依然存在货架期不长的问题,所以对热处理后辣椒酱中残留微生物进行准确分析显得十分重要,从而才能有的放矢地采用适当抑菌剂或改进灭菌方式,从而延长辣椒酱的货架期。
对于食品中微生物的研究,传统上采用培养法统计微生物数量并分离得到纯培养菌株。研究发现,辣椒表面可分离得到Lactobacillus χylosus、Pediococcus acidilactici、Pediococcus pentosaceus、Rhodotorula mucilaginosa等[1],而米曲霉、酱油曲霉、黑曲霉、鲁氏酵母和盐水四联球菌等则是湖南永丰辣酱自然发酵过程中的主要优势菌[2]。但是,传统培养法往往在培养条件上受到限制,影响了分离培养的结果。近年来,随着分子生物学方法的发展,聚合酶链式反应-变形梯度凝胶电泳(PCR-DGGE)技术逐渐应用于食品中微生物的检测。高秀芝等[3-4]运用PCR-DGGE技术研究发现Lactococcus lactis、Bacillus licheniformis、Bacillus pumilus等为传统豆酱发酵过程中的优势菌;张琦等[5]则对郫县豆瓣自然发酵过程中的细菌群落结构变化进行了研究分析,从中发现了Staphylococcus χylosus、Lactobacillus plantarum、Weissella confusa等。杨虎等[6]曾运用培养法和16S rDNA-ARDRA分析研究了冷藏鸡肉胴体中的细菌多样性,但同时运用培养法和非培养法研究辣椒或辣椒酱尚未见报道。通过分子生物学方法和传统分离培养法的结合,应能更加全面地反映辣椒酱中的微生物数量及群落结构。
本实验通过对郫县辣椒酱进行热处理,研究其贮藏过程中微生物的数量变化,分离鉴定其中的可培养微生物,并结合PCR-DGGE技术进一步分析微生物群落结构,以期揭示热处理对辣椒酱中微生物数量和种类的影响,以及贮藏中导致腐败的主要残留微生物的种类,从而为延长辣椒酱货架期提供一定的理论依据。
1 材料与方法
1.1 材料、试剂与仪器
辣椒酱由四川郫县豆瓣厂提供。
0.85 %灭菌生理盐水、1×TE Buffer (pH7.6)、1× TAE Buffer (pH8.5);聚丙烯酰胺、尿素、甲酰胺(均为分析纯) 上海捷倍思基因技术有限公司;TaKaRa TaqTMDR100AM 日本TaKaRa公司;2×Taq PCR MasterMix KT201 天根生化科技(北京)有限公司;Primer EU27F、1492R、ITS1、ITS4 美国Invitrogen公司。
DZKW-4电子恒温水浴锅 北京中兴伟业公司;S1000TMThermal Cycler PCR反应仪、Universal Hood II凝胶成像系统、DCODE Universal Mutation Detection SystemTM通用突变检测系统 美国Bio-Rad公司。
1.2 培养基
平板计数琼脂(PCA)培养基[7]、结晶紫中性红胆盐琼脂(VRBA)培养基[8]、孟加拉红培养基[9]、PDA和PDB培养基[10]。
1.3 方法
1.3.1 辣椒酱的处理与分装
将盛有(300±0.1)g辣椒酱的500mL烧杯使用保鲜膜和双层报纸封口,于80℃恒温水浴30min并迅速冷却以模拟实际工艺,期间监测辣椒酱中心温度变化。另设未处理组。两组辣椒酱均分装入无菌PE袋(约15g/袋)。1.3.2热处理辣椒酱微生物计数
在第1、7、14、20、40、60、120天各取3袋热处理辣椒酱,另在第1天取3袋辣椒酱作为未处理组。无菌条件下从每袋称取10g辣椒酱,加入90mL 0.85%灭菌生理盐水,180r/min振摇30min,制成10倍稀释液,并梯度稀释成100倍和1000倍的样品稀释液。菌落总数、大肠菌群计数、霉菌和酵母计数分别参照文献[7-9]。
1.3.3 热处理辣椒酱微生物种类分析
1.3.3.1 微生物分离与基因组DNA的提取
根据菌落形态及预实验时菌落总数的结果,选择从热处理辣椒酱第1、20、60、120天的菌落总数测定平板上挑取单菌落,划线纯化后选择代表性菌株富集培养,取1.5mL培养液采用改良的CTAB法[11]提取基因组DNA作为后续PCR反应模板。
1.3.3.2 细菌16S rDNA序列扩增
选用细菌16S rDNA通用引物Eu27F (5'-GAG AGT TTG ATC CTG GCT CAG-3')和1492R (5'-CTA CGG CTA CCT TGT TAC GA-3')进行扩增。PCR反应体系(50μL):10×PCR Buffer 5μL,2.5mmol/L dNTP 4μL,25mmol/L MgCl24μL,Taq DNA聚合酶 0.25μL,引物各1μL,ddH2O 30.75μL,模板DNA 4μL。反应程序:94℃ 5min;94℃ 60s,50℃ 60s,72℃ 90s,30个循环;72℃ 10min[6]。扩增产物用1%琼脂糖电泳检测。
1.3.3.3 真菌ITS序列扩增
选用引物ITS1 (5'-TCC GTA GGT GAA CCT GCG G-3')和ITS4 (5'-TCC TCC GCT TAT TGA TAT GC-3')。PCR反应体系(50μL):10×PCR Buffer 5μL,2.5mmol/L dNTP 4μL,25mmol/L MgCl23μL,Taq DNA聚合酶0.25μL,引物各1μL,ddH2O 31.75μL,模板DNA 4μL。反应程序:95℃ 5min;95℃ 60s,55℃ 60s,72℃ 90s,30个循环;72℃ 10min。扩增产物用1%琼脂糖电泳检测。
1.3.3.4PCR-DGGE分析细菌群落
在第120天取适量辣椒酱制成10倍稀释液,取1.5mL匀液500r/min离心2min,取上清液10000r/min离心5min,收集沉淀经改良的CTAB法[11]提取宏基因组DNA。
采用Nested PCR法。先用EU27F和1492R扩增细菌16S rDNA,反应程序同1.3.3.2节。然后用带GC clamp (CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG G)的通用引物GC-338f (5'-GC clamp-ACT CCT ACG GGA GGC AGC AG-3')和518r (5'-ATT ACC GCG GCT GCT GG-3'),经降落PCR扩增16S rDNA V3区,反应程序:94℃ 5min;94℃ 30s,60~55℃ 30s (每个循环降低0.5℃),72℃ 30s,10个循环;94℃ 30s,55℃30s,72℃ 30s,25个循环;72℃ 10min[5]。PCR反应体系均为(50μL):2×Taq PCR MasterMix 25μL,引物各1μL,ddH2O 19μL,模板DNA 4μL。16S rDNA扩增产物用1%琼脂糖电泳检测;16S rDNA V3区扩增产物用2%琼脂糖电泳检测。
细菌16S rDNA V3区PCR产物DGGE电泳、染色及图像分析参考文献[5]。切胶回收代表性条带,加入40μL TE Buffer 4℃浸泡过夜,取上清为模板,用不含GC clamp的引物338f和518r再次扩增16S rDNA V3区,反应程序同上,反应体系(50μL):10×PCR Buffer 5μL,2.5mmol/L dNTP 4μL,25mmol/L MgCl24μL,Taq DNA聚合酶0.5μL,引物各1μL,ddH2O 29.5μL,模板DNA 5μL。
1.3.3.5 序列分析
PCR产物测序后提交NCBI (http://www.ncbi.nlm.nih. gov/)进行Blast相似序列检索,并于GenBank获得序列登录号(Accession No.)。
1.4 统计分析
使用Excel 2003 SP3和SPSS Statistics 19.0软件进行数据分析。使用ANOVA分析微生物数量变化的差异显著性。
2 结果与分析
2.1 热处理辣椒酱微生物计数结果
辣椒酱中心温度在热处理过程中呈上升趋势,处理结束时达到最高温度57℃。热处理前后及贮藏过程中辣椒酱的菌落总数变化见图1。热处理后辣椒酱菌落总数由(5.25±0.05)(lg(CFU/g))降为(4.06±0.03) (lg(CFU/g)),说明80℃热处理30min有效杀灭了辣椒酱中超过90%的微生物。菌落总数在处理后第7天达到贮藏期内的最大值(4.44±0.04) (lg(CFU/g)),相比第1天增加了约0.4 (lg(CFU/g)) (P<0.05)。这可能是因为热处理后虽有降温处理但不够彻底,且样品堆积,从而导致了辣椒酱中残留微生物的生长。但热处理使辣椒酱中微生物钝化或产生了其他不可修复的损伤,从而抑制了微生物大量增殖,因此菌落总数在第7~40天期间维持在一定水平,并从第60天开始下降,至第120天仅为(2.94±0.10) (lg(CFU/g)),相比第1天下降了1.12 (lg(CFU/g)) (P<0.05)。菌落总数在贮藏后期的下降,可能是由于微生物逐渐死亡或是转为“存活但非可培养”(viable but non-culturable,VBNC)[12]状态。
另一方面,热处理后辣椒酱中真菌数由72CFU/g降为不可检出,说明热处理对辣椒酱中真菌也有杀灭作用,而未灭菌处理的辣椒酱中真菌数量很少,推测真菌可能不是发酵中的功能菌群,也不是热处理辣椒酱贮藏过程中的腐败菌。
大肠菌群在热处理前后均未检出,产品符合农业行业标准NY/T 1070—2006《辣椒酱》。
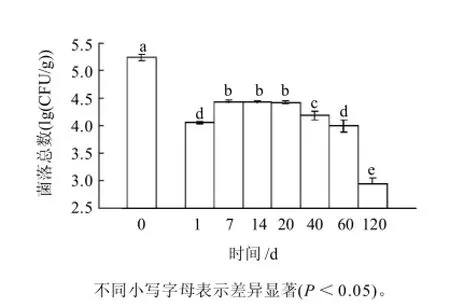
图1 热处理辣椒酱贮藏过程中的菌落总数变化Fig.1 Total bacterial count in chili sauce before and after heat treatment
2.2 热处理辣椒酱微生物的分离鉴定

表1 辣椒酱分离细菌16S rDNA序列分析Table 1 16S rDNA sequence analysis of bacteria isolated from chili sauce
从热处理辣椒酱中分离纯化得到6株可培养细菌,16S rDNA鉴定结果见表1。可以看出,从热处理辣椒酱中分离的可培养细菌均为芽孢杆菌属(Bacillus sp.),推测可能是由于Bacillus sp.细菌可形成芽孢从而逃避了热处理的损害,从而大量存活。Bacillus sp.细菌可能是环境菌种,同时也具有一定的发酵和抑菌作用。有研究指出,细菌型豆豉发酵曲种主要是Bacillus subtilis,并且Bacillus subtilis和Bacillus amyloliquefaciens可产生具有显著溶栓作用的豆豉纤溶酶[13]。高雅等[14]曾从自然发酵的郫县豆瓣中分离得到一株Bacillus subtilis (L4),该菌能有效抑制产毒黄曲霉的生长及毒素的合成。热处理辣椒酱中未检出真菌,但自处理前辣椒酱中分离得到1株酵母菌F1。经ITS序列分析比对,F1与菌株Rhodotorula mucilaginosa PYCC 4349 (AF444584)相似性达100%,序列提交GenBank并获得登录号JF338814。推测辣椒酱中的Rhodotorula mucilaginosa可能来自于原料辣椒[2]。相比其他微生物,Bacillus sp.细菌在热处理压力下具有更佳的存活能力,其数量在贮藏期内占据着优势地位,具有潜在的致腐可能。但是,本研究采用的PCA培养基和培养条件(37℃、48h、非厌氧)只适合部分细菌生长,对于营养要求复杂、生长缓慢等生长条件要求严苛的细菌可能无法成功培养。此外,经热处理作用,部分微生物细胞可能处于VBNC状态,具有潜在的生长能力及危害性[15],但也未能进一步验证和检出。这些因素都影响了微生物分离鉴定的结果。
2.3 辣椒酱贮藏终点细菌群落PCR-DGGE分析
第120天时辣椒酱细菌群落PCR-DGGE指纹图谱见图2,DGGE条带经测序比对,序列鉴定结果表2。

表2 第120天辣椒酱细菌群落DGGE条带比对分析Table 2 Identification of DGGE bands of bacteria isolated from chili sauce at day 120 of storage
由表2可知,辣椒酱贮藏终点第120天时的细菌群落主要为乳酸菌(Pediococcus sp.和Lactobacillus sp.)。乳酸菌可能对于辣椒酱的独特风味形成具有一定的影响,并可通过形成低pH值的环境抑制部分有害菌的生长。据报道,Lactobacillus brevis是自然发酵东北酸菜中的优势菌[16];Lactobacillus acidipiscis则曾在泰国豆酱中被发现[17],而细菌群落中的Bacillus cereus则具有潜在的致腐性及致病性[18]。
与从辣椒酱中分离培养的细菌鉴定结果相比,DGGE图谱中各条带所代表的细菌种类更多。这一方面可能是由于在热处理辣椒酱中Bacillus sp.细菌数量上处于优势地位,在分离培养过程中抑制了Pediococcus sp.和Lactobacillus sp.细菌生长,致使未获得乳酸菌纯培养菌株;另一方面也可能是由于Pediococcus sp.和Lactobacillus sp.细菌在热处理辣椒酱中处于死亡或是VBNC状态而无法通过培养法检测,仅能通过PCR-DGGE证明其存在于辣椒酱中。有研究指出,由于基于DNA的PCR-DGGE可能因死细胞的存在而产生误差,而rRNA的合成与细胞的生长紧密相关,所以基于RNA的分析方法无疑更能提供关于微生物群落存活与代谢活性的有用信息。Han Yanqing等[19]对比了基于rDNA和rRNA的PCR-DGGE结果,显示RNA-DGGE图谱具有更高的微生物多样性。
但另一方面,本研究的DGGE图谱中仅出现1个条带,经鉴定为Bacillus sp.细菌(Bacillus cereus,相似度99%),多样性小于培养法,这可能是由于使用通用引物338f和518r扩增16S rDNA V3区对于辨识Bacillus sp.细菌的检测灵敏度不高,使得检测存在一定的缺陷。Kim等[20]曾在研究中通过在16S rDNA扩增之后增加一轮选择性PCR (引物pB和1492R),然后选用引物Ec1055和GC-Ec1392扩增16S rDNA V9区,用于DGGE特异性检测Bacillus sp.细菌,取得了很好的效果。
3 结 论
本实验使用培养法和非培养法(PCR-DGGE)研究了热处理辣椒酱中的残留微生物,结果显示,热处理对辣椒酱中微生物的控制取得了一定成效,杀灭了辣椒酱中90%以上的微生物;但是,热处理辣椒酱中的某些残留微生物(如Bacillus cereus)仍然具有潜在的致腐性和致病性,控制残留的芽孢杆菌才能更有效地延长辣椒酱的货架期。
使用传统的培养法检测辣椒酱中的微生物,存在着培养基等条件上的限制,可能导致优势菌(如芽孢杆菌)在检测中大量生长;此外,某些微生物可能在热处理后转变为VBNC状态而无法通过培养法检出,这也使得培养法检测结果往往不够全面。而非培养法(PCR-DGGE)除了检测出芽孢杆菌之外,还检出了辣椒酱发酵过程中残留的乳酸菌。因此,相比传统上单独使用培养法,非培养法(PCR-DGGE)与培养法的联合应用,能够更加全面地反映并准确监测辣椒酱中的微生物种类,可为选用适
当的抑菌剂或改进灭菌方式提供科学依据。
[1]刘丽娜. 辣椒表面微生物区系的研究[D]. 重庆: 西南大学, 2007.
[2]苏东林, 张忠刚, 陈亮, 等. 永丰辣酱自然发酵过程中主要优势菌及化学成分的动态变化分析[J]. 食品科学, 2009, 30(17): 212-216.
[3]高秀芝, 王小芬, 李献梅, 等. 传统发酵豆酱发酵过程中养分动态及细菌多样性[J]. 微生物学通报, 2008, 35(5): 748-753.
[4]高秀芝, 王小芬, 李献梅, 等. 传统发酵豆酱与商品豆酱养分及微生物多样性比较[J]. 食品科学, 2008, 29(3): 222-226.
[5]张琦, 汪先丁, 杨虎, 等. 郫县豆瓣自然发酵过程中细菌群落结构的变化[J]. 食品与发酵科技, 2010, 46(6): 16-18; 35.
[6]杨虎, 向文良, 张弛, 等. 培养和非培养法分析冷藏鸡肉胴体中的细菌多样性[J]. 微生物学通报, 2010, 37(10): 1451-1456.
[7]中华人民共和国卫生部. GB/T 4789.2—2010 食品安全国家标准食品微生物学检验 菌落总数测定[S]. 北京: 中国标准出版社, 2010.
[8]中华人民共和国卫生部. GB/T 4789.3—2010 食品安全国家标准食品微生物学检验 大肠菌群计数[S]. 北京: 中国标准出版社, 2010.
[9]中华人民共和国卫生部. GB/T 4789.15—2010 食品安全国家标准食品微生物学检验 霉菌和酵母计数[S]. 北京: 中国标准出版社, 2010.
[10]东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 370.
[11]程志学, 陈清华, 于远, 等. 适合AFLP分析用的节瓜基因组DNA提取方法的研究[J]. 分子植物育种, 2009, 7(2): 420-424.
[12]LIAO Hongmei, ZHANG Liyun, HU Xiaosong, et al. Effect of high pressure CO2 and mild heat processing on natural microorganisms in apple juice[J]. International Journal of Food Microbiology, 2010, 137 (1): 81-87.
[13]汪孟娟, 陈廷涛, 姜淑英, 等. 豆豉发酵过程中的微生物和功能性组分研究动态[J]. 中国微生态学杂志, 2010, 22(1): 81-84.
[14]高雅, 丁文, 张琦, 等. 传统发酵豆瓣中产毒黄曲霉高效拮抗菌的筛选[J]. 微生物学通报, 2010, 37(3): 369-374.
[15]ROWAN N J. Viable but nonculturable forms of food and waterborne bacteria: Quo Vadis?[J]. Trends in Food Science & Technology, 2004, 15(9): 462-467.
[16]张鲁冀, 孟祥晨. 自然发酵东北酸菜中乳杆菌的分离与鉴定[J]. 东北农业大学学报, 2010, 41(11): 125-131.
[17]TANASUPAWAT S, THONGSANIT J, OKADA S, et al. Lactic acid bacteria isolated from soy sauce mash in Thailand[J]. Journal of General and Applied Microbiology, 2002, 48(4): 201-209.
[18]李志明, 郭洁, 崔希勇, 等. 黄豆酱类食品的微生物状况分析[J]. 中国调味品, 2008, 33(12): 75-76; 87.
[19]HAN Yanqing, XU Xinglian, JIANG Yun, et al. Inactivation of food spoilage bacteria by high pressure processing: evaluation with conventional media and PCR-DGGE analysis[J]. Food Research International, 2010, 43(6): 1719-1724.
[20]KIM T W, LEE J H, KIM S E, et al. Analysis of microbial communities in doenjang, a Korean fermented soybean paste, using nested PCR-denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2009, 131(2/3): 265-271.
Analysis of Microbial Survival in Heat-Treated Pixian Chili Sauce during Storage
DING Wen1,WANG Xian-ding1,GAO Peng2,YANG Hu1,ZHAO Min1,SUN Qun1,*
(1. Key Laboratory of Bio-resource and Bio-environment, Ministry of Education, College of Life Science, Sichuan University, Chengdu 610064, China;2. Sichuan Institute of Atomic Energy, Chengdu 610101, China)
Heat treatment is often used to inhibit microbial growth in Pixian chili sauce for extending its shelf-life, but some microorganisms are still likely to survive, causing Pixian Chili Sauce to spoil during storage. In this study, Pixian chili sauce treated at 80℃ for 30 min was enumerated for total aerobic bacteria, coliforms, moulds and yeasts during storage at room temperature for 120 days. Bacterial and fungal strains isolated from chili sauce were identified by 16S rDNA and ITS sequence analysis respectively, and PCR-DGGE technique was used to analyze the bacterial community at the end of the storage period. The total number of bacteria in chili sauce decreased more than 90% after heat treatment at 80 ℃ for 30 min, and the majority of surviving microbes were Bacillus sp. During the storage period of 120 days, the total number of bacteria in heat treated chili sauce increased at first and then decreased. The dominant surviving bacteria in chili sauce at the end of the storage period, as detected by PCRDGGE, were Pediococcus sp., Lactobacillus sp. and Bacillus cereus. These results demonstrate that partial microorganisms in chili sauce are killed or presented in VBNC (viable but non-culturable) state after heat treatment, and surviving Bacillus sp. and lactic acid bacteria may be the reason for the spoilage of heat-treated chili sauce. Consequently, effectively controlling the survival Bacillus sp. and lactic acid bacteria is the key to extend the shelf-life of chili sauce.
heat treatment;shelf-life;aerobic bacteria count;PCR-DGGE;viable but non-culturable (VBNC)
Q939.99
A
1002-6630(2012)03-0146-05
2011-04-18
国家科技人员服务企业行动项目(2009GJF00039);四川省科技支撑计划项目(2010NZ0051);国际原子能机构(IAEA)支持项目(16356/R0)
丁文(1986—),男,硕士研究生,研究方向为食品微生物和食品安全。E-mail:davidting200518@yahoo.com.cn
*通信作者:孙群(1967—),女,教授,博士,研究方向为食品微生物学和微生物技术。E-mail:qunsun@scu.edu.cn

