多种抑癌基因在食管鳞状细胞癌中的甲基化状态及其临床意义*
王长春 毛伟敏 凌志强
浙江省肿瘤医院 浙江省肿瘤研究所肿瘤分子遗传实验室
浙江省胸部肿瘤(肺、食管、乳腺)诊治技术研究重点实验室(310022)
肿瘤的表观遗传学研究已成为近年肿瘤研究的热点问题。有研究发现多种肿瘤包括胃癌、食管癌中可见基因启动子区5’CpG岛异常甲基化[1,2],食管癌前病变Barrett食管中抑癌基因APC基因启动子区亦存在高甲基化[3]。大量研究已证实抑癌基因甲基化与食管癌相关,但多种抑癌基因启动子区5’CpG岛甲基化与肿瘤家族史的研究鲜见报道。本研究通过检测食管鳞状细胞癌(ESCC)患者中多种抑癌基因的甲基化状态,旨在探讨其与肿瘤家族史和预后的相关性,为进一步研究肿瘤易感家族提供依据。
对象与方法
一、研究对象
选取2010年2~7月浙江省肿瘤医院76例经病理检查确诊的ESCC患者,均为首诊病例,术前未接受过放、化疗以及其他任何治疗。其中男61例,女15例,年龄40~76岁,中位年龄60岁。食管上/中段病变46例,下段30例;高/中分化60例,低分化16例。有肿瘤家族史(定义为两代以内有直系亲属曾患有恶性肿瘤)者16例,其中食管癌5例,结肠癌2例,胰腺癌2例,肺癌2例,淋巴瘤2例,白血病、胃癌和肾癌各1例;无肿瘤家族史者60例。按照国际抗癌联盟(UICC)和美国肿瘤联合会(AJCC)2009第7版TNM分期标准,Ⅰ期5例,Ⅱ期30例,Ⅲ期40例,Ⅳ期1例。本研究方案经浙江省肿瘤医院伦理委员会批准同意,患者均签署知情同意书。
二、方法
1.组织DNA提取:手术切除标本后,由专人负责取组织标本。肿瘤组织标本取自癌巢,癌旁正常组织取自距肿瘤边缘5 cm处,切取后立即置于液氮-76℃保存。采用苯酚/氯仿法提取基因组DNA(杭州化学试剂有限公司),置于-20℃保存待测。
2.DNA亚硫酸氢盐修饰:以紫外分光光度计(型号NanoDrop ND-1000)测定DNA浓度和纯度,A260nm/A280nm比值≥1.8的DNA判定为合格样本。基因组DNA行亚硫酸氢盐修饰和纯化,具体步骤按EpiTect Bisulfite Kits(Qiagen)说明书操作。
3.MSP 检测:参照 APC、RARβ2、CDH1、p16INK4a和RASSF1A基因序列,分别设计5’CpG岛特异性甲基化和非甲基化引物,所有引物由Invitrogen公司合成(见表1)。PCR反应体系20μl,反应条件:95℃预变性 10 min;95℃变性 15 s,60℃退火1 min,72℃延伸1 min,共40个循环;72℃延伸10 min[2]。采用2-△CT法定量分析各抑癌基因的甲基化状态。以体外经SssⅠ甲基转移酶处理过的人类基因组DNA作为阳性对照。
三、统计学分析
采用SPSS 17.0统计软件。计数资料的比较采用χ2检验;生存分析采用Kaplan-Meier分析,终点事件为肿瘤复发或死亡,观察无进展生存期(progression-free survival,PFS)。P<0.05 为差异有统计学意义。
结 果
一、基因启动子区甲基化状态
ESCC患者肿瘤组织APC、RARβ2、CDH1、p16INK4a、RASSF1A的甲基化率分别为44.7%、72.4%、72.4%、86.8%、55.3%,均显著高于相应癌旁正常组织,差异均有统计学意义(P=0.000)(见表 2)。

表2 ESCC患者肿瘤组织和癌旁正常组织中各抑癌基因甲基化率的比较 n(%)
二、甲基化与肿瘤家族史
16例有肿瘤家族史的ESCC患者中,肿瘤组织 APC、RARβ2、CDH1、p16INK4a、RASSF1A的甲基化率分别为75.0% (12/16)、100% (16/16)、93.8%(15/16)、93.8%(15/16)、100%(16/16);60 例无肿瘤家族史者中,甲基化率分别为36.7%(22/60)、65.0%(39/60)、66.7%(40/60)、85.0%(51/60)和 43.3%(26/60),两组相比差异均有统计学意义(P<0.05)。

表1 各抑癌基因甲基化特异性PCR反应引物和片段长度
三、生存分析
66例ESCC患者有随访结果,其中18例复发或死亡,另有10例失访。经Kaplan-Meier分析,CDH1和RASSF1A非甲基化患者的平均生存期均明显高于甲基化患者(P=0.015、P=0.016),其余 3种抑癌基因甲基化与否对预后的影响无明显差异(见表 3、图 1)。
讨 论
肿瘤的病因和发病机制复杂,其中抑癌基因起重要调控作用。APC基因涉及调节细胞黏附、细胞迁移乃至细胞凋亡,其调控方式是与微管结合,调节胞液中β-catenin蛋白水平;RARβ2是一种抑癌基因,定位于染色体3p24,通过在靶细胞中转化成视黄酸而发挥作用,其启动子区甲基化可引起癌症的发生[4];p16基因主要通过调节细胞周期来参与肿瘤的发生,其调节机制可能通过Rb基因依赖途径实现[5];CDH1基因参与同型细胞间黏附;RASSF1A除具有调节细胞周期的作用之外,还可促进凋亡和调节微管稳定性[6]。
业已证实抑癌基因在食管癌的发生、发展中起重要作用[7],其表遗传沉默与肿瘤侵袭、生长、新生血管生成、转移行为密切相关,可能是食管癌发生、发展以及治疗后复发的重要因素之一,并影响患者的总体生存率。多项研究证实ESCC组织中存在多种抑癌基因的高甲基化。且在癌变过程中,不同组织类型的基因甲基化状态亦不相同[8]。Zare等[9]发现44.4%的ESCC患者发生APC启动子区高甲基化,其两年生存率更低,而正常组织中未发生甲基化;Kim等[10]的研究结果显示50例ESCC患者中,APC、RARβ2、p16、RASSF1A 的 甲 基 化 率 分 别 为46%、34%、10%和 14%;Kuroki等[11]的研究发现ESCC细胞株和 ESCC组织中均可见 RARβ2、RASSF1A高甲基化,且肿瘤组织的甲基化率高于癌旁组织;Lee等[12]发现251例ESCC患者中p16、RARβ2、CDH1 的甲基化率分别为 52%、25%和43%,且CDH1高甲基化可作为TNM分期Ⅰ期患者的复发指标;Taghavi等[13]发现ESCC患者肿瘤组织p16INK4a甲基化率显著高于相邻癌旁组织。本研究中,ESCC 患者肿瘤组织中 APC、RARβ2、CDH1、p16INK4a、RASSF1A的甲基化率分别为44.7%、72.4%、72.4%、86.8%、55.3%,均显著高于相应癌旁正常组织。由此可见,各种抑癌基因甲基化可能在ESCC的发生、发展中起重要作用。
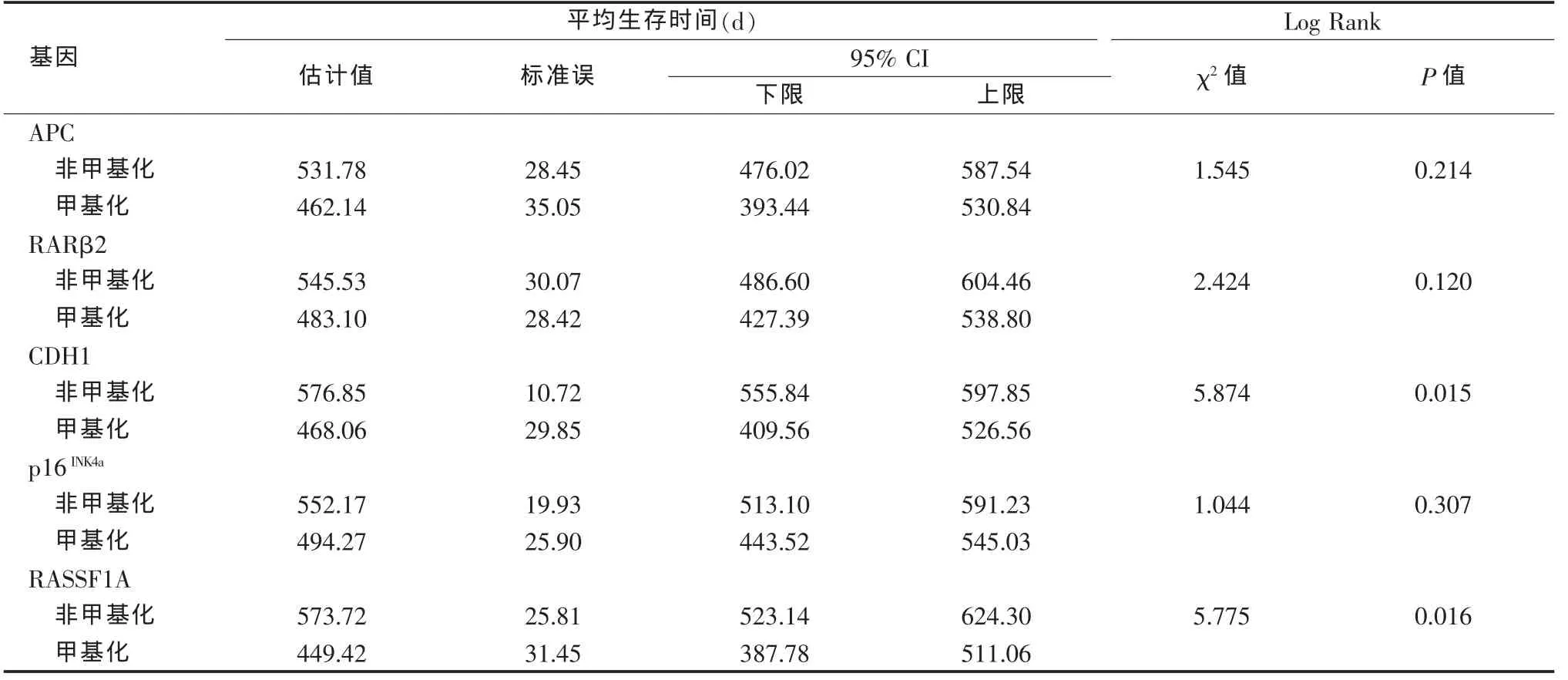
表3 ESCC患者各抑癌基因甲基化Kaplan-Meier分析

图1 CDH1和RASSF1A甲基化和非甲基化患者的生存曲线
Abbaszadegan等[14]在ESCC患者家族成员中发现,p16启动子区异常甲基化率为64.3%,而健康志愿者为0,说明p16异常甲基化可作为高危ESCC家族成员肿瘤的早期鉴别标记。本研究发现ESCC患者 APC、RARβ2、CDH1、RASSF1A 基因甲基化与肿瘤家族史显著相关,而p16INK4a基因甲基化与肿瘤家族史无关。
目前抑癌基因甲基化与ESCC患者预后关系的报道少见。本研究中,CDH1和RASSF1A甲基化患者的生存期明显低于非甲基化患者,但因随访时间过短,故不能肯定其临床意义,尚需进一步随访并重新分析才能得出可信的结果。
由此可见,对具有肿瘤家族史的健康家庭成员行多种抑癌基因甲基化检测,并密切随访甲基化阳性者,可在一定程度上早期诊断和治疗ESCC等疾病。但该结论尚需扩大样本量进一步验证。
1 张斌,曹俊,陈敏,等.死亡相关蛋白激酶基因高甲基化在胃癌形成过程中作用的初步研究.胃肠病学,2008,13(12):737-740.
2 郑秋青,凌志强,李沛,等.RASSF1A在食管鳞癌组织中的表达、甲基化状态及其与预后之间的关系.世界华人消化杂志,2010,18(29):3134-3139.
3 宋明全,常英,朱金水.质子泵抑制剂治疗对Barrett食管APC基因启动子区甲基化的影响.胃肠病学,2009,14(9):536-539.
4 许旭,孙敬岩,顾林,等.从表观遗传学角度探讨RAR-β2基因在乳腺癌发生中的作用.中国肿瘤临床,2010,37(23):1326-1329,1333.
5 孙树汉主编.遗传与疾病.第1版.北京:人民卫生出版社,2009.157-179.
6 周绍荣,徐美荣,陈云.RASSF1A基因甲基化与肿瘤关系的研究进展.实用癌症杂志,2008,23(5):540-542.
7 Raja S,Godfrey TE,Luketich JD.The role of tumor suppressor genes in esophageal cancer.Minerva Chir,2002,57(6):767-780.
8 Yang HH,Hu N,Wang C,et al.Influence of genetic background and tissue types on global DNA methylation patterns.PLoS One,2010,5(2):e9355.
9 Zare M,Jazii FR,Alivand MR,et al.Qualitative analysis of Adenomatous Polyposis Coli promoter: hypermethylation,engagementand effectson survivalof patients with esophageal cancer in a high risk region of the world,a potential molecular marker.BMC Cancer,2009,9:24.
10 Kim YT,Park JY,Jeon YK,et al.Aberrant promoter CpG island hypermethylation of the adenomatosis polyposis coli gene can serve as a good prognostic factor by affecting lymph node metastasis in squamous cell carcinoma of the esophagus.Dis Esophagus,2009,22(2):143-150.
11 Kuroki T,Trapasso F,Yendamuri S,et al.Allele loss and promoter hypermethylation of VHL, RAR-beta,RASSF1A,and FHIT tumorsuppressorgeneson chromosome 3p in esophageal squamous cell carcinoma.Cancer Res,2003,63(13):3724-3728.
12 Lee EJ,Lee BB,Han J,et al. CpG island hypermethylation of E-cadherin (CDH1)and integrin alpha4 is associated with recurrence of early stage esophageal squamous cell carcinoma.Int J Cancer,2008,123(9):2073-2079.
13 Taghavi N,Biramijamal F,Sotoudeh M,et al.p16INK4ahypermethylation and p53,p16 and MDM2 protein expression in Esophageal Squamous Cell Carcinoma.BMC Cancer,2010,10:138.
14 Abbaszadegan MR,Raziee HR,Ghafarzadegan K,et al.Aberrant p16 methylation,a possible epigenetic risk factor in familial esophageal squamous cell carcinoma.Int J Gastrointest Cancer,2005,36(1):47-54.

