簇毛麦
——用于小麦改良的一种野生植物
赵万春,董 剑,陈其皎,李晓燕,高 翔,石引刚,陈良国
(西北农林科技大学农学院, 陕西 杨凌 712100)
小麦(Triticumaestivum)是世界上广泛种植的粮食作物,全世界35%~40%的人口以它为主要粮食来源。我国的作物品种资源比较丰富,在中国国家作物种质库中保存作物种质资源39万余份,其中小麦种质资源45 500多份[1],但生产上使用的品种遗传来源较为单一。为解决这个问题,最有效的办法之一是把小麦不同遗传背景的优良性状基因整合到新品种中,不断增加新品种的遗传类型。要达到这一目的,就必须有足够多的小麦遗传资源。目前,把小麦野生近缘属的有益基因导入普通栽培小麦已成为小麦品种改良重要而有效的途径之一。
簇毛麦属小麦族,簇毛麦属(Dasypyrum,又名为Haynaldiavillosa),为一年生或多年生异株授粉二倍体植物(2n=2x=14,Sears[2]将簇毛麦基因组命名为VV)。它具有抗条锈(Pucciniastriiformis)、叶锈(P.recondita)、秆锈病(P.graminis)[3-6]、白粉病(Erysiphegraminisf.ssp.tritici)[6-7]和小麦梭条花叶病毒(Polymyxagraminis)[9]等多种小麦主要病虫害的特性,同时具有耐寒、分蘖力强、生长繁茂、多小花、蛋白质含量高和耐盐抗旱等特性[10-11]。因而它常被作为重要的基因资源之一,应用于小麦品种改良。
本研究从簇毛麦染色体及其组型和带型、簇毛麦与小麦属的亲缘关系、簇毛麦与小麦属的杂交以及簇毛麦在普通小麦改良中的应用等方面,对其研究进展进行回顾总结,以期为更好地利用簇毛麦提供理论依据。
1 簇毛麦染色体的命名及其组型和带型
簇毛麦染色体的名称V是经过几度易名统一而来。Sears最早将簇毛麦染色体设计为1Ha~7Ha[12],根据C分带模式,Friebe等[13]重新将簇毛麦染色体分别命名为A~G,对应于Sears的1Ha~7Ha,Liu等[14]部分同意Sears的结论,并用符号V1~V7暂时命名簇毛麦染色体。根据小麦与簇毛麦代换系的基因组间的代换关系和对附加系和代换系的同工酶分析结果,Liu等[12]用1V~7V代替了先前设计的簇毛麦染色体V1、V7、V5、V6、V3、V2和V4。
簇毛麦共有7对染色体,其中5对为近中着丝粒染色体,2对为亚中着丝粒染色体。1对染色体具有随体。簇毛麦所有染色体具有清晰的C带,多数带型显现于染色体端部或亚端部,根据它们的带型特征,每对染色体可以与小麦染色体区分开来[2-3,13,15-16]。然而,只有6对簇毛麦染色体呈现N带,而且其带型特征与小麦染色体的N带相似,所以,不能用N带来区分簇毛麦和小麦染色体[17]。
2 簇毛麦与小麦属的亲缘关系
簇毛麦染色体与小麦属染色体存在着部分同源关系,这种同源关系可以通过观察外源染色体与特定小麦染色体间的配对水平或将外源染色体代换到小麦遗传背景来研究其遗传补偿作用等多种方法确定。Sears[2]发现簇毛麦V基因组与小麦的A、B、D 基因组之间几乎没有染色体的配对,认为它们亲缘关系较远。陈佩度等[18]对簇毛麦×节节麦(Aegilopssquarrosa,2n=14,DD) 的杂种F1花粉母细胞减数分裂中期I(PMCM I)构型研究,认为V染色体组与D染色体组亲缘关系较近。Blanco[19]通过圆锥小麦(T.turgidum)与簇毛麦杂种F1的PMCM I 构型分析,结合染色体C-分带结果,证明了V 染色体组与A组的亲缘关系较B组近,而且发现簇毛麦自然群体中有促进部分同源染色体配对的基因,其水平相当于缺失Ph基因。由此推断小麦的A、B、D基因组中,D染色体组与V染色体组亲缘关系最近,其次是A染色体组,而B染色体组较远。
3 簇毛麦与小麦属的杂交
早在20世纪30年代,Sando已进行了小麦属与簇毛麦属的远缘杂交[10]。簇毛麦与普通小麦的杂交能力显著低于簇毛麦与四倍体小麦的杂交能力,六倍体小麦与簇毛麦直接杂交非常困难[20]。尽管如此,通过秋水仙素处理和胚胎培养已成功获得簇毛麦与普通小麦的杂种和双二倍体。然而,八倍体的双二倍体(AABBDDVV)比六倍体的双二倍体(AABBVV)繁殖力低,生长势弱,因此,后者常被用作簇毛麦与普通小麦杂交的桥梁[21]亲本。
基于普通小麦与簇毛麦杂交种中V染色体与A染色体和V染色体与B染色体配对观察,Minelli等[22]认为簇毛麦和乌拉尔图小麦(T.uratu,小麦A基因组的供体),拟斯卑尔脱山羊草(Aegilopsspeltoides,小麦B基因组的供体)和早先源自于簇毛麦的A.squarrosa(D基因组)属于同一线系种族。因为这种关系,簇毛麦属中的部分物种,主要为簇毛麦,已与小麦属的二倍体、四倍体、六倍体物种成功杂交[10,23]。
4 簇毛麦在普通小麦改良中应用
4.1普通小麦-簇毛麦异源附加系、代换系和易位系的创制 簇毛麦有益基因导入小麦,大都是以双二倍体为中间材料,通过杂交及回交,以及组织培养等途径获得异附加系、异代换系及易位系实现的。Sears[2,24]和Hyde[3]通过野生二粒小麦-簇毛麦双二倍体与中国春杂交和回交,获得了6个二体附加系(1V,2V,4V,5V,6V,7V)和7个单体附加系。Chen等[7]和Liu等[12]以基本相似的途径先后获得了2V~7V 等6个异附加系和5个异代换系。
已经鉴定出的小麦与簇毛麦的代换系(涉及簇毛麦的1V~6V染色体)有1V/1A和1V/1B[2]、DS2V到DS6V[25]、6A/6V[26]和6D/6V[27]。但是,异附加系和异代换系不仅表现型上有许多簇毛麦的不良性状,还可能出现部分同源染色体的缺失、重复以及被导入外源染色质的代尝性能和对原有遗传平衡的影响等问题,因此不能有效地用于小麦的改良。
在各类异染色体系中,以遗传稳定的整臂互补易位系的育种利用价值较好。迄今,选育出的中国春-簇毛麦整臂互补易位系有T6AL·6V#2S[7]、T4DL·4V#2S[9]、T1DL·1V#3S和T1DS·1V#3L[28-29];此外笔者参加创制了10个新易位系:T2BS·2V#3L,T3DS·3V#3L,T3DL·3V#3S,T4DL·4V#3S,T4DS·4V#3L,T5DL·5V#3S,T6AS·6V#3L,T6AL·6V#3S, T7DS·7V#3L和 T7DL·7V#3S[30-31]。
4.2普通小麦遗传背景中簇毛麦染色体的鉴定 随着分子遗传学、细胞遗传学和分子生物学的发展,小麦中外源遗传物质的鉴定方法被丰富和深化,出现了传统的形态标记、细胞学标记、生化标记、分子标记和原位杂交等方法相结合鉴定小麦遗传背景中簇毛麦染色体的特点。
4.2.1形态标记 簇毛麦的黑芒(6VS)、护颖脊背刚毛(2VS)和穗轴易断性(3VS)等性状可用作检测小麦中是否含有该性状所属簇毛麦染色体或染色体臂的形态标记[2,7]。
4.2.2细胞学标记 目前,小麦染色体经过染色体分带,尤其是Giemsa C-分带后,每条染色体都能产生特征性带纹[32]。而大多数小麦亲缘物种的染色体经过C-分带之后能产生与小麦明显不同的特征带型[33-34]。所以,通过染色体C-分带技术可以鉴定出被导入普通小麦的亲缘物种的染色体或染色体片段,甚至可确定其具体身份[7,35]。
根据簇毛麦染色体C-分带研究结果,簇毛麦具有丰富的末端带和亚端带,与普通小麦的染色体较易区分开来[2-3,15-16]。但是,当外源染色体片段缺失可诊断的带型时,染色体的分带技术却不能提供有用的信息。同时,在小麦遗传背景下带型的多态性有时会干扰外源染色体的鉴定。
4.2.3生化标记 Montebove等[36]对小麦-簇毛麦二体添加系进行种子贮藏蛋白电泳分析,将编码高分子量谷蛋白亚基的基因定位于1V上。簇毛麦1V~7V 均找到了一至几个相应的生化标记,主要为同工酶及非酶蛋白两类[36-38]。虽然还没有更多的研究表明这些标记与特定的有益性状连锁,但是,利用已知的生化标记可以有效地识别小麦背景中的簇毛麦染色体。
4.2.4分子标记 物种专化的DNA 重复序列、RAPD标记、RFLP标记、SSR标记和STS标记可以用来鉴定导入小麦遗传背景的簇毛麦染色体。
研究发现,簇毛麦1V~6V染色体近末端分布有1个380 bp重复序列[39-40]和pHv62重复序列[41]。由于这2个序列属于V基因组特有,一般不能与普通小麦、黑麦等其他DNA序列杂交,所以它可以被用作探针来检测不同小麦属背景的簇毛麦染色体[39]。
Qi等[42]、王振英等[43]分别获得了对6VS具有专化性的RAPD标记,在小麦育种中用它作为分子标记来检测和筛选簇毛麦的抗白粉病显性基因Pm21。刘守斌等[44]筛选出一个簇毛麦基因组特异性RAPD 片段并建立了簇毛麦基因组特异性PCR标记。
Qi等[45]用一套小麦属第6部分同源群DNA的RFLP标记鉴定和表征了小麦-簇毛麦6V染色体附加系的3个缺体系,定位了6V染色体的断点,发现6VS和6VL染色体臂的RFLP标记。Yildirim等[46]用RFLP标记将抗眼斑病的基因PchDv定位在4V的长臂上。Qi等[47]用RFLP进一步支持了早先经形态学、同工酶和C-分带建立的簇毛麦和小麦染色体之间的部分同源关系。Li等[7]利用RFLP标记鉴定出了3个小麦与簇毛麦的T6DL/6VS易位系。
Zhang等[48]选出了分别可用来追踪簇毛麦1V~7V染色体的SSR引物wmc49(1BS)、wmc25(2BS)、gdm36(3DS)、gdm145(4AL)、wmc233(5DS)、wmc256(6AL)和gwm344(7BL),这些簇毛麦染色体特异的微卫星标记可用来追踪普通小麦背景中的簇毛麦染色体。
Chen等[27]检测出一个对簇毛麦染色体6VS专化的800 bp PCR-STS标记,用作抗小麦瘿螨(Aceriatosichella)的标记选择。Cao等[49]开发出了1个共显性分子标记NAU/xibao15902,可以用来标记筛选6VS上的Pm21基因,同时又能区分小麦的6AS、6BS和6DS。王春梅等[50]从105对小麦第1部分同源群染色体的EST-STS引物中得到5个簇毛麦1V染色体的EST-STS标记。笔者也成功开发出了4个EST-STS标记,且成功应用于中国春-簇毛麦易位系的创制[28-31]。
4.2.5原位杂交 原位杂交是鉴定外源染色体片段的最为直观有效的方法,其主要优点在于杂交信号在细胞分裂的任何时期都可以观察到,但用基因组DNA作探针只能检测到外源染色体是否存在,不能了解被检测外源染色体的具体身份。因此,异源易位系的鉴定往往需要染色体分带与基因组原位杂交两种技术结合进行[51]。目前,以标记的簇毛麦基因组DNA为探针的基因组原位杂交(GenomicalinsituHybridization,GISH)技术在小麦-簇毛麦双二倍体、附加系、代换系和易位系[6,22,28-31,52]的鉴定中得到广泛应用。陈佩度等[7,52]应用分子原位杂交技术,鉴定出发生易位的簇毛麦染色体片段,还鉴定了小麦-簇毛麦附加系、代换系,证明了6AL/6VS易位断点靠近着丝粒。Chen等[27]利用GISH技术鉴定出了6D/6V异代换系。Zhang等[8]在小麦-簇毛麦代换系与普通小麦品种杨麦5号杂交的后代群体中,鉴定出了带有4VS端体系和含有1条或2条T4VS-4DL整臂易位系。Minelli等[22]利用GISH研究发现带有簇毛麦1V、3V和5V染色体或它们的部分片断的小麦品系遗传不稳定,表明杂交种中簇毛麦的染色体结构发生了变化。Zhao等[29]、Liu等[30]利用分子标记、GISH结合C-分带技术创制出了一套新的中国春-簇毛麦整臂互补易位系。
4.3簇毛麦的有益性状及其在普通小麦改良中的利用 簇毛麦拥有许多有益性状(基因),主要包括种子贮藏蛋白、抗病虫性、特有的形态性状和同工酶等,并已明确决定这些性状的基因所位于的染色体(表1,表2)。
4.3.1种子贮藏蛋白 Montebove等[36]和Shewry等[53]最早进行簇毛麦贮藏蛋白的研究。在簇毛麦的1V、 4V和6V染色体上,发现有醇溶谷蛋白。簇毛麦的1V染色体上有多个复合基因位点:与小麦的Glu-A1,Glu-B1和Glu-D1位点近等的簇毛麦高分子量谷蛋白基因(Glu-V1),与小麦的Gli-1位点部分同源的贫硫(ω-型)和富硫(γ-型)醇溶蛋白基因(Gli-V1)和低分子量聚合醇溶蛋白基因(Glu-V1)[38,53]。簇毛麦中Gli-V1、Gli-V2和Gli-V3三个基因位点分别位于1VS、6VS和4VL上,Shewry等[53]发现这3个位点分别编码7、2和2个Gli亚基。由于储藏蛋白与面团的流变性质直接有关,因此,簇毛麦在这些位点上的多态性,为改良小麦的加工品质提供了有用的等位基因。
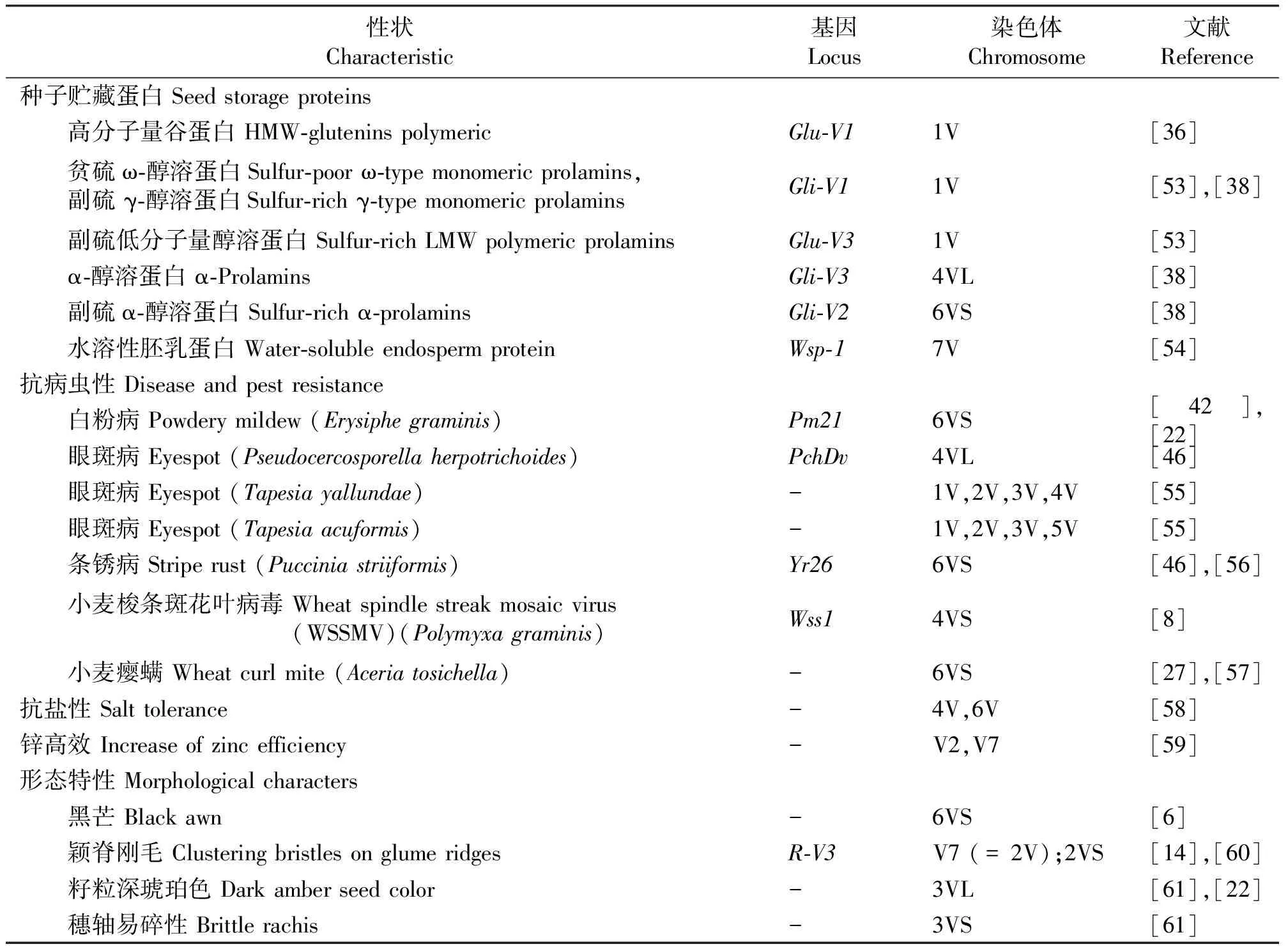
表1 簇毛麦某些性状基因的染色体位置Table 1 Chromosome localization of Dasypyrum villosum traits

表2 利用小麦-簇毛麦染色体附加系检测到的簇毛麦同工异构酶Table 2 Dasypyrum villosum co-worker isomerase detected by using addition lines
De Pace等[68]研究表明,含有簇毛麦1V染色体的小麦-簇毛麦附加系、代换系和易位系的籽粒蛋白质含量和SDS-沉淀值高于其小麦亲本的蛋白含量和SDS-沉淀值。存在于簇毛麦4V和6V染色体上的麩盶贮藏蛋白位点Gli-V2和Gli-V3对增加蛋白质的品质有贡献,但它们并不改进小麦的品质。相反簇毛麦1V染色体(存在Glu-V1和Gli-V1/Gli-V3位点)对小麦的品质具有较大的正效应。然而,由于未创制出含有簇毛麦1VS、1VL的小麦材料,簇毛麦的高分子量谷蛋白基因(Glu-V1)、醇溶蛋白基因(Gli-V1)和低分子量聚合醇溶蛋白基因(Glu-V3)究竟位于1V染色体的哪条臂还不清楚。
簇毛麦的7V染色体上存在水溶性胚乳蛋白基因Wsp-1,它与小麦第7部分同源染色体长臂上的基因同源[54]。
4.3.2抗病抗逆性 含簇毛麦6V染色体的品系抗白粉病、抗小麦条锈、秆锈、叶锈病和抗小麦瘿螨。附加系6V(6A)和6V(6D)、易位系T6AL·6VS[6]、易位系T6DL·6VS[7]和代换系6D/6V[69]均抗白粉病,并且将抗白粉病基因Pm21定位于6VS。Yildirim等[70]鉴定结果表明,含有簇毛麦6VS染色体臂的小麦材料抗小麦条锈病,并将该抗病新基因命名为Yr26。陈佩度等[56]连续3年的鉴定结果也证明了含有簇毛麦6V或6VS染色体臂的小麦材料对小麦条锈病具有高度抗性,然而,Chen等[4]认为抗条锈病基因Yr26与簇毛麦6VS染色体臂无关系。易位系T6ASW·6V#3L含有来之于簇毛麦的抗小麦秆锈病Ug99的基因Sr52[31]。小麦-簇毛麦的6V附加系和6VS易位系同时也抗小麦瘿螨[4,27]。
4V染色体携带抗眼斑病的基因,此病由Pseudocercosporellaherpotrichoides[46,71]和Tapesiayallundae[72]两种病菌引起。抗前一病菌的显性基因PchDv位于4VL的末端区[46],抗后一种病菌的基因存在于1V、2V和3V染色体上。而抗另一种眼斑病病菌Tapesiaacuformis的基因位于1V、2V、3V和5V染色体上。这表明簇毛麦抗两种Tapesia属病菌的遗传基础不同[72]。Zhang等[9]创建的小麦-簇毛麦T4VS·4DL易位系农艺性状好,且抗小麦梭条斑花叶病毒(WSSMV),因此,证明在簇毛麦的4VS上含有抗小麦梭条斑花叶病毒的基因。
Zhong和Dvora′k[58]发现4V和6V这两条染色体上带有与耐盐性显著正相关的基因位点。V7(=2V)染色体和V2(=6V)染色体一起携带可增强锌效率的基因[59]。
4.3.3形态特性 Chen等[7]将簇毛麦的黑芒定位于6VS上。Liu等[12]发现在2V(=V7)染色体上存在控制颖片背脊丛生刚毛的基因,徐川梅[60]后来又进一步将该性状定位于2VS上。籽粒暗琥珀色基因R-V3位于3V染色体长臂和易碎穗轴显性基因定位于3V的短臂上[61]。这些性状可用作鉴定簇毛麦染色体/臂的标记性状。
4.3.4同工酶位点 生化标记被用于小麦育种和簇毛麦群体部分同源关系以及遗传多样性研究。利用小麦-簇毛麦染色体附加系,检测到几个同工异构酶(表2)。Qualset等[73]对13个同工酶研究发现14个位点分布于簇毛麦所有染色体上,并鉴定了多数位点的等位基因形态。根据De Pace簇毛麦自身的同工酶Got-2(2个等位基因)和Est-1(4个等位基因)存在多态性[74],而这些同工酶位点是否和有利于性状连锁有待进行鉴定。
5 小结
综上所述,簇毛麦属于小麦近缘野生种,可为小麦改良提供丰富的有益基因资源。通过创建小麦-簇毛麦易位系,在将簇毛麦的优良抗病性基因转移给小麦的研究与应用方面已取得了显著成效。尤其是位于6VS上的抗白粉病基因Pm21,具有抗谱广、抗性强的特点,该基因在小麦抗白粉病育种中已得到广泛利用,并取得了显著成效。簇毛麦及其衍生系对来自美国、德国和中国的84个小麦白粉病菌菌系表现出高度的免疫和高抗,比供试的18个已知基因的小麦品种抗性强,抗谱广;抗性基因在陕7859、冀麦30、81086A、D311、墨75、宛7107等小麦品种的遗传背景下均能很好地表达,呈显性遗传[69]。此外,位于4VS上的抗小麦梭条斑花叶病毒基因Wss1、位于4VL上的抗眼斑病基因PchDv和位于6VL上的抗小麦秆锈病Ug99的基因Sr52已经被成功地转移到小麦遗传背景中,成为小麦育种中的重要抗源。而对于抗条锈病基因Yr26是否与6VS有关还有争议,因此对簇毛麦的抗条锈病基因的染色体定位有待进一步确证。
而对簇毛麦高、低分子量谷蛋白亚基基因(Glu-V1、Glu-V3)、醇溶蛋白基因(Gli-V1)、抗旱耐盐和锌高效等有益性状的研究尚不够深入,相关性状的基因或染色体片断在小麦中的遗传机理和表达效应还不清楚,因此创制新的小麦-簇毛麦易位系,对进一步充分挖掘和利用簇毛麦的宝贵基因资源十分重要。譬如,利用分子标记和C-分带和基因组原位杂交(GISH)等细胞学技术创制、筛选和鉴定2个新的小麦-簇毛麦整臂互补易位系T1VS·1DL和1DS·T1VL,这将对利用簇毛麦的1V染色体上高分子量谷蛋白亚基基因(Glu-V1)、醇溶蛋白基因(Gli-V1)和低分子量聚合醇溶蛋白基因(Glu-V3)进行小麦品质改良具有重要意义。
总之,对簇毛麦许多潜在的有益性状和基因资源的开发利用前景仍然十分广阔。
[1] 刘旭,黎裕,曹永生,等.中国禾谷类作物种质资源地理分布及其富集中心研究[J].植物遗传资源学报,2009,10(1):1-8.
[2] Sears E R.Adition of the genome ofHaynaldiavillosatoTriticumavestivum[J].American Journal of Botany,1953,40:168-174.
[3] Hyde B B.Addition of individualHaynaldiavillosachromosomes to hexaploid wheat[J].American Journal of Botany,1953,40:174-182.
[4] Chen Q,Conner R L,Li H,etal.Expression of resistance to stripe rust,powdery mildew and the wheat curl mite inTriticumaestivum·Haynaldiavillosalines[J]. Canadian Journal of Plant Science,2002,82(2):451-456.
[5] 陈静,邓光兵,余懋群,等.小麦-簇毛麦端体附加系中6VS 的细胞遗传学行为及其抗性遗传研究[J].四川农业大学学报,2001,19(1):1-5.
[6] Blanco A,Simeone R,Resta P.The addition ofDasypyrumvillosum(L.) Candargy chromosomes to durum wheat (TriticumdurumDesf.)[J].Theoretical and Applied Genetics,1987,74:328-333.
[7] Chen P D,Qi L L,Zhou B,etal.Development and molecular cytogenetic analysis of wheat-Haynaldiavillosa6VS/6AL translocation lines specifying resistance to powdery mildew[J].Theoretical and Applied Genetics,1995,91:1125-1128.
[8] Li H,Chen X,Xin Z Y,etal.Development and identification of wheat-HaynaldiavillosaT6DL·6VS chromosome translocation lines conferring resistance to powdery mildew[J].Plant Breeding,2005,124:203-205.
[9] Zhang Q,Li Q,Wang X,etal.Development and characterization of aTriticumaestivum-Haynaldiavillosatranslocation line T4VS·4DL conferring resistance to wheat spindle streak mosaic virus[J].Euphytica,2005,145:317-320.
[10] Gradzielewska A.The genusDasypyrum-part 1.The taxonomy and relationships withinDasypyrumand with Triticeae species[J].Euphytica,2006,152:429-440.
[11] 孔凡晶,陈孝.簇毛麦基因组及其在小麦改良中的应用研究进展[J].麦类作物学报 2001,21(2):85-87.
[12] Liu D J,Chen P D,Raupp W J.Determination of homoeologous groups ofHaynaldiavillosachromosomes[A].In:Proceedings of the 8th International Wheat Genetic Symposium[C].Bejing,China:Chinese Agricultural Scientech Press,1995:181-185.
[13] Friebe B,Cermeno M C,Zeller F J.C-banding polymorphism and the analysis of nucleolar activity inDasypyrumvillosum(L.) candargy,its added chromosomes to hexaploid wheat and the amphiploidTriticumdicoccum-D.villosum[J].Theoretical and Applied Genetics,1987,73:337-342.
[14] Liu D J,Chen P D,Pei G Z,etal.Transfer ofHaynaldiavillosachromosomes intoTriticumaestivum[A].In:Miller T E,Koebner R M D.Proceedings of the 7th International Wheat Genetic Symposium[C].Cambridge,England:the 7th International Wheat Genetic Symposium,1988,1:355-361.
[15] 张自立,董广元.簇毛麦的染色体组型和带型[J].遗传,1984,6(1):19-20.
[16] 傅体华,任正隆.簇毛麦染色体的形态学及Giemsa-C带的多态性研究Ⅰ.簇毛麦染色体的核型及C-带核型[J].四川农业大学学报,2001,19(4):400-403.
[17] 董凤高,陈佩度,刘大钧.簇毛麦染色体的改良C-分带[J].遗传学报,1991,18(6):525-528.
[18] 陈佩度,刘大钧.普通小麦与簇毛麦杂种后代的细胞遗传学研究[J].南京农学院学报,1982(4):1-16.
[19] 〗Blanco A.Chromosome pairing variation inTriticumturgidum(L.)×Dasypyrumvillosum(L.)Candargy hybrids and genome affinities[A].In:Proceedings of the 7th.International Wheat Genetic Symposium[C].Cambridge,UK:the 7th International Wheat Genetic Symposium,1988:63-67.
[20] Halloran G M.Hybridization ofHaynaldiavillosawithTriticumaestivum[J].Australian Journal of Botany,1966,14:355-359.
[21] Bothmer von R,Claesson L.Production and meiotic pairing of intergeneric hybrids ofTriticum×Dasypyrumspecies[J].Euphytica,1990,51:109-117.
[22] Minelli S,Ceccarelli M,Mariani M,etal.Cytogenetics ofTriticum-Dasypyrumhybrids and derived lines[J].Cytogenetic and Genome Research,2005,109:385-392.
[23] Gradzielewska A.The genusDasypyrum-part 2.Dasypyrumvillosum——a wild species used in wheat improvement[J].Euphytica,2006,152:441-454.
[24] Sears E R.Genetic control of chromosome pairing in wheat[J].Annual Review of Genetics,1976,10:31-51.
[25] Qi L L,Chen P D,Liu D J,etal.Development of translocation lines ofTriticumaestivumwith powdery mildew resistance introduced fromHaynaldiavillosa[A]. In:Proceedings of the 8th International Wheat Genetic Symposium[C].Bejing,China:Chinese Agricultural Scientech Press,1995:333-337.
[26] 马渐新,周荣华,贾继增.用基因组原位杂交与RFLP标记鉴定小麦、簇毛麦抗白粉病代换系[J].遗传学报,1997,24:447-452.
[27] Chen Q,Conner R L,Laroche A.Molecular characterization ofHaynaldiavillosachromatin in wheat lines carrying resistance to wheat curl mite colonization[J].Theoretical and Applied Genetics,1996,93:679-684.
[28] 赵万春.普通小麦-簇毛麦整臂互补易位系T1DS·1VL和T1DL·1VS的创制、鉴定和性状评估[D].杨凌:西北农林科技大学,2010.
[29] Zhao W C,Qi L L,Gao X,etal.Development and characterization of two newTriticumaestivum-DasypyrumvillosumRobertsonian translocation lines T1DS·1V#3L and T1DL·1V#3S and their effect on grain quality[J].Euphytica,2010,175: 343-350.
[30] Liu C,Qi L L,Liu W X,etal.Development of a set of compensatingTriticumaestivum-DasypyrumvillosumRobertsonian translocation lines[J].Genome,2011,54: 836-844.
[31] Qi L L,Pumphrey M O,Bernd Friebe,etal.A novel Robertsonian translocation event leads to transfer of a stem rust resistance gene (Sr52) effective against race Ug99 fromDasypyrumvillosuminto bread wheat[J]. Theoretical and Applied Genetics,2011,123:159-167.
[32] Gill B S.Standard karyotype and nomenclature system for description of chromosome bands and structural aberrations in wheat[J].Genome,1991,34:830-839.
[33] Gill B S.Giemsa C-banding technique for cereal chrosome[J].Cereal Research Communication,1974,2:87-94.
[34] Jiang J,Gill B S.Sequential chromosome banding andinsituhybridization analysis[J].Genome,1993,36:792-795.
[35] Friebe B,Heun M.C-banding pattern and powdery mildew resisitance ofTriticumovatumand fourT.aestivum-T.ovatumchromosome addition lines[J].Theoretical and Applied Genetics,1989,78(3):417-424.
[36] Montebove L,De Pace C,Jan C C,etal.Chromosomal location of isozyme and seed storage protein genes inDasypyrumvillosum(L.) Candargy[J].Theoretical and Applied Genetics,1987,73:836-845.
[37] Liu C J,Gale M D.The chromosomal location of genes encoding NADH dehydrogenase isosymes in hexaploid wheat and related species[J].Genome,1991,34(1):44-51.
[38] Blanco A,Resta P,Simeone R,etal.Chromosomal location of seed storage protein genes in the genome ofDasypyrumvillosum(L.) Candargy[J].Theoretical and Applied Genetics,1991,82:358-362.
[39] De Pace C,Delre V,Scarascia Mugnozza G T,etal.Molecular and chromosomal characterization of repeated and single-copy DNA sequences in the genome ofDasypyrumvillosum[J].Hereditas,1992,116:55-65.
[40] Frediani M,Colonna N,Cremonini R,etal.Redundancy modulation of nuclear DNA sequences inDasypyrumvillosum[J].Theoretical and Applied Genetics,1994,88:167-174.
[41] Li W,Liu D,Chen P,etal.Cloning of repeated sequences ofHaynaldiavillosaDNA for detection of its chromatin in wheat[A].In:Proceedings of the 8th International Wheat Genetic Symposium[C].Bejing,China:Chinese Agricultural Scientech Press,1995: 817-822.
[42] Qi L L,Cao M,Chen P D,etal.Identification,mapping,and application of polymorphic DNA associated with resistance genePm21 of wheat[J].Genome,1996,39:191-197.
[43] 王振英,赵红梅,洪敬欣,等.簇毛麦6VS上4个新分子标记的鉴定及与抗白粉病基因Pm21的连锁分析[J].作物学报,2007,33(4):605-611.
[44] 刘守斌,唐朝晖,尤明山,等.簇毛麦染色体组特异性RAPD标记的筛选、定位和应用[J].遗传学报,2002,29(5):453-457.
[45] Qi L L,Wang S L,Chen P D,etal.Identification and physical mapping of threeHaynaldiavillosachromosome-6V deletion lines[J].Theoretical and Applied Genetics,1998,97:1042-1046.
[46] Yildirim A,Jones S S,Murray T D.Mapping a gene conferring resistance toPseudocercosporellaherpotrichoideson chromosome 4V ofDasypyrumvillosumin a wheat background[J].Genome,1998,41:1-6.
[47] Qi L L,Chen P D,Liu D J,etal.Homoeologous relationships ofHaynaldiavillosachromosomes with those ofTriticumaestivumas revealed by RFLP analysis[J].Genes and Genetic System,1999,74:77-82.
[48] Zhang W,Gao A L,Zhou B,etal.Screening and applying wheat microsatellite markers to trace individualHaynaldiavillosachromosomes[J].Acta Genetica Sinica,2006,33(3):236-243.
[49] Cao A Z,Wang X E,Chen Y P,etal.A sequence-specific PCR marker linked withPm21 distinguishes chromosomes 6AS,6BS,6DS ofTriticumaestivumand 6VS ofHaynaldiavillosa[J].Plant Breeding,2006,125(3):201-205.
[50] 王春梅,别同德,陈全战,等.簇毛麦6V染色体短臂特异分子标记的开发和应用[J].作物学报,2007,33(10):1595-1600.
[51] Friebe B.Characterization of rust-resistance wheat-Agropyroninternediumderivatives by C-banding,insituhybridization and isozyme analysis[J].Theoretical and Applied Genetics,1992,83:775-782.
[52] 陈佩度,周波,齐莉莉,等.用分子原位杂交(GISH)鉴定小麦-簇毛麦双倍体、附加系、代换系和易位系[J].遗传学报,1995,22(5):380-386.
[53] Shewry P R,Parmar S,Pappin D J C.Characterization and genetic control of the prolamins ofHaynaldiavillosa:Relationship to cultivated species of the Triticeae (rye,wheat and barley)[J].Biochemical Genetics,1987,25:309-325.
[54] Liu C J,Chao S,Gale M D.Wsp-1,a set of genes controlling water-soluble proteins in wheat and related species[J].Genetics Research,1989,54(3):173-181.
[55] Uslu E,Miller T E,Rezanoor N H,etal. Resistance ofDasypyrumvillosumto the cereal eyespot pathogens,TapesiayallundaeandTapesiaacuformis[J]. Euphytica,1998,103:203-209.
[56] 陈佩度,刘大钧,齐莉莉,等.一个小麦条锈病新抗源及其抗谱初步测定[J].植物病理学报,2001,31(1):31-36.
[57] Li H J,Conner R L,Chen Q J,etal. Different reactions to the wheat curl mite and wheat streak mosaic virus in various wheat-Haynaldiavillosa6V and 6VS lines[J].Plant Disease,2002, 86(4):423-428.
[58] Zhong G Y,Dvora′k J.Evidence for common genetic mechanisms controlling the tolerance of sudden salt stress in the tribe Triticeae[J].Plant Breeding,1995,114(4):297-302.
[59] Schlegel R,Cakmak I,Torun B,etal.Screening for zinc efficiency among wheat relatives and their utilization for alien gene transfer[J].Euphytica,1998,100:281-286.
[60] 徐川梅.普通小麦-簇毛麦2V染色体端体异附加系和易位系的选育与鉴定及分子原位杂交技术的应用[D].南京:南京农业大学,2007.
[61] Urbano M,Resta P,Benedetelli S,etal.ADasypyrumvillosum(L.) Candargy chromosome related to homoeologous group 3 pf wheat[A].In:Miller T E,Koebner R M D.Proceedings of the 7th International Wheat Genetic Symposium[C].Cambridge,UK:the 7th International Wheat Genetic Symposium,1988,1:355-361.
[62] De Pace C,Benedettelli S,Qualset C O,etal.Biochemical markers inTriticum·Dasypyrumamphiploidsand derived disomic addition lines[A].In:Miller T E,Koebner R M D.Proceedings of the 7th International Wheat Genetic Symposium[C].Cambridge,England,1988:503-509.
[63] Liu C J,Gale M D.Est-7, a set of genes controlling green tissue esterases in wheat and related species[J].Theoretical and Applied Genetics,1990,79(6):781-784.
[64] Koebner R M D,Martin P K.Chromosomal control of the aminopeptidases of wheat and its close relatives[J].Theoretical and Applied Genetics,1989,78:657-664.
[65] Benedettelli S,Hart G E.Genetic analysis of Triticeae Shikimate dehydrogenase[J].Biochemical Genetics,1988,26:287-301.
[66] Koebner R M D.Genetic control of dipeptidase in the Triticeae[J].Theoretical and Applied Genetics, 1987,74:387-390.
[67] Koebner R M D,Miller T E,Snape J W,etal.Wheat endopeptidase:Genetic control, polymorphism, intrachromosomal gene location, and alien variation[J].Genome,1988,30:186-192.
[68] De Pace C,Snidaro D,Ciaffi M,etal.Introgression ofDasypyrumvillosumchromatin into common wheat improves grain protein quality[J].Euphytica,2001,117:67-75.
[69] 陈孝,施爱农,尚立明.簇毛麦对不同白粉病菌系的抗性反应及其在小麦背景下的表达[J].植物病理学报,1997,27(1):17-22.
[70] Yildirim A,Jones S S,Murray T D,etal.Evaluation ofDasypyrumvillosumpopulations for resistance to cereal eyespot and stripe rust pathogens[J].Plant Disease,2000,84(1):40-44.
[71] Murray T D,De la Pena R C,Yildirim A,etal.A new source of resistance toPseudocercosporellaherpotrichoidescause of eyespot disease of wheat located on chromosome 4V ofDasypyrumvillosum[J].Plant Breeding,1994,113(4):281-286.
[72] Mohammad P,Hossain M A,Khan F,etal.Leaf blight disease tolerance/resistance in diploid relatives of wheat[J].Sarhad Journal of Agriculture,1999,15(4):311-316.
[73] Qualset C O,Zhong G Y,De Pace C,etal.Population biology and evaluation of genetic resources ofDasypyrumvillosum[A].In:Damania A B.Biodiversity and Wheat Improvement[M].Wiley,Chichester,UK,1993:227-233.
[74] De Pace C.Genetic variability in natural populations ofDasypyrumvillosum(L.) Candargy[D].Davis,USA:University of California-Davis,1987.

