HKT蛋白与植物耐盐性研究进展
赵常玉,李 剑,张金林,王锁民
(草地农业系统国家重点实验室 兰州大学草地农业科技学院,甘肃 兰州 730020)
土壤盐渍化严重影响作物产量,降低农业生产力[1]。盐渍生境中,过多的Na+破坏植物体内离子平衡、引起生物膜失活、新陈代谢活性下降,最终导致植物生长受到抑制甚至死亡[2]。Na+是土壤溶液中的大量元素,浓度范围在0.4~150 mmol·L-1,浓度较低时,可以促进某些植物的生长,是有益元素[3-6],但浓度较高时,大多数植物的生长受到严重抑制[7-8]。K+是植物所必需的大量元素之一,与Na+有相似的水合半径。盐胁迫时,非选择性阳离子通道或转运蛋白不能将二者区分,大量Na+的吸收抑制了植物根系对K+的摄入,植物体内K+/Na+降低,造成盐害,因此维持细胞质中高的K+/Na+是植物应对胁迫的有效措施[9-10]。高亲和性钾离子转运蛋白HKT(High-affinity K+Transporter)能维持植物体内离子平衡,增强植物耐盐性。本研究就HKT蛋白的最新研究进展进行综述,以期阐述其与植物耐盐性的关系。
1 HKT蛋白种类和特性
Schachtman和Schroeder[11]首次从小麦(Triticumaestivum)中克隆了HKT蛋白基因TaHKT2;1,其开放阅读框(ORF)编码534个氨基酸,分子量58.9 KD,具有10~12个跨膜区,位于第7号染色体上,原位杂交显示TaHKT2;1主要在根皮层和叶维管组织细胞中表达。继小麦TaHKT2;1之后,先后在拟南芥(Arabidopsisthaliana)、桉树(Eucalyptuscamaldulensis)、水稻(Oryzasativa)、冰叶日中花(Mesembryanthemumcrystallinum)、盐地碱蓬(Suaedasalsa)等植物中发现了HKT基因[12-16],与大肠杆菌(Escherichiacoli) TrkH、超嗜热菌(Aquifexaeolicus)KtrB、粟酒裂殖酵母(Schizosaccharomycespombe)Trk1,2等共同构成一个超级K+转运蛋白基因家族[17-18]。根据异源表达系统上的离子选择性特征差异和氨基酸序列同源性分析,Platten等[19]将HKT蛋白分为两类:第1类以拟南芥AtHKT1;1为代表,包括盐地碱蓬SsHKT1;1、水稻OsHKT1;5和冰叶日中花McHKT1;1等,通常为Na+转运蛋白,有的也能转运K+[13,15];第2类以小麦TaHKT2;1为代表,包括水稻OsHKT2;1、大麦(Hordeumvulgare)HvHKT2;1等,为K+-Na+共转运蛋白,双子叶植物缺少此类蛋白基因(图1)。HKT基因通常含有两个内含子,且第1类上的内含子明显比第2类大,而一粒小麦(Triticummonococcum)TmHKT1;4-A2只有一个内含子[20]。这种命名法可以分析不同物种相同基因和相同物种不同基因在进化和功能上的区别与联系[19]。
高等植物HKT蛋白的氨基酸数目多在500个左右。有的HKT蛋白基因是单拷贝,或是一个小基因家族,通常受盐胁迫和K+饥饿诱导,可以在根、茎、叶和花等不同组织中表达[21-23](表1)。拒盐型盐生植物小花碱茅(Puccinelliatenuiflora)PutHKT2;1在盐胁迫时,根中表达量增加,地上部变化不大[24],而盐地碱蓬SsHKT1;1叶中表达量高于根中[16,21]。不同物种基因组中HKT基因数目差异很大,拟南芥只有1个AtHKT1;1[12],而Garciadeblás等[25]在水稻品种日本晴(Nipponbare)基因组中发现7个可能编码Na+转运蛋白的HKT基因(另2个为假基因)。

图1 高等植物HKT蛋白氨基酸序列同源性分析[19]Fig.1 Phylogenetic analysis of HKT proteins of higher plants[19]
2 HKT蛋白结构与功能
Durell等[26]认为原核生物含有1个MPM(Membrane-Pore-Membrane),包括2个跨膜螺旋和1个P环,植物HKT蛋白由KcsA类K+通道亚基经复制加倍及融合进化而来,所以包含4个MPM,此模型已在小麦、水稻和拟南芥中得到证实[27-28](图2)。HKT蛋白4个MPM高度保守,并且含有1个甘氨酸-酪氨酸-甘氨酸(GYG)基序,研究表明,HKT蛋白存在与K+和Na+选择性吸收相关的多个位点,且多位于P环或离P环很近的区域[27]。第1类HKT蛋白第1个P环过滤器位置处有1个丝氨酸残基,其余3个P环上为甘氨酸残基,构成S-G-G-G类型;第2类HKT蛋白第1个P环处的丝氨酸残基被甘氨酸代替,其余3个P环处仍为甘氨酸残基,构成G-G-G-G类型,水稻OsHKT2;1例外[14,29](图3)。

表1 部分高等植物HKT蛋白基因分子特性Table 1 Molecular characteristics of HKT of higher plants

图2 HKT类蛋白拓扑结构示意图[17,27-28,34]Fig.2 Diagrammatic representation of HKT-like protein topological structure[17,27-28,34]
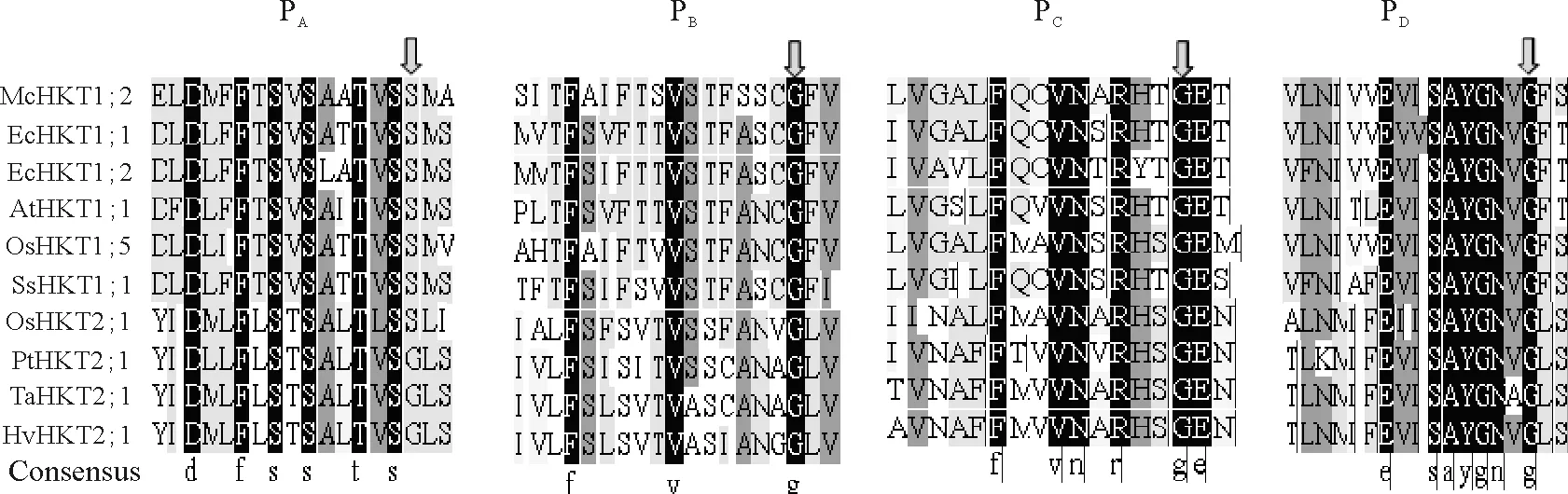
图3 一些高等植物HKT的4个P环保守序列[34]Fig.3 Conservative sequences of four P-loop of higher plants[34]
Rubio等[30]认为小麦TaHKT2;1中存在1个高亲和性K+结合位点和1个高亲和性Na+结合位点,Km分别为3和175 μmol·L-1。为确定这些位点,Diatloff等[31]采用定点突变和酵母(Saccharomycescerevisiae)突变体(K+吸收缺陷)异源表达方法,发现小麦TaHKT2;1一段16个氨基酸保守序列在Na+转运方面发挥着重要作用。Rubio等[32]发现小麦TaHKT2;1突变体N365S通过缓解对高亲和性K+吸收的抑制、减少低亲和性Na+吸收,从而降低酵母体内Na+/K+,显著提高盐敏感型酵母(Na+ATPase缺失)的耐盐性。Liu等[18]对小麦TaHKT2;1蛋白上可能参与K+、Na+选择性吸收相关位点做了进一步研究,认为跨膜区3和4之间的高电荷P环上可能存在这些位点,环中片段L149-E180缺失后转入酵母中,提高了酵母体内K+/Na+,增强了其耐盐性。Mäser等[29]认为植物HKT蛋白存在4个P环,若第1个P环中过滤器处为甘氨酸残基,通常为K+-Na+的共转运蛋白;若第1个P环中过滤器处为丝氨酸残基,通常为Na+转运蛋白。拟南芥AtHKT1;1与小麦TaHKT2;1嵌合体试验表明,在拟南芥AtHKT1;1中将Ser-68突变为甘氨酸后发现,增大了K+的通透性;在小麦TaHKT2;1中将Gly-91突变为丝氨酸,K+通透性受到抑制。但第2类HKT蛋白的甘氨酸残基并不是决定K+选择性吸收的唯一因素,因为一些第1类HKT蛋白同样具有K+转运特性,且赖氨酸和精氨酸残基对于K+吸收可能也有作用[33]。
3 HKT与植物耐盐性
3.1小麦HKT与其耐盐性 Epstein等[35]认为高等植物中存在两个K+吸收系统:1)当K+浓度较低时,主要由K+载体起作用的高亲和性吸收系统;2)当K+浓度较高时,主要由K+通道起作用的低亲和性吸收系统。小麦TaHKT2;1的发现为此假说提供了直接证据[11]。Schachtman和Schroeder[11]采用功能互补法,从小麦幼根cDNA文库中筛选到能互补酵母K+吸收缺陷的TaHKT2;1,其编码蛋白对几种单价阳离子的选择性顺序为K+>Cs+>Rb+>Na+>NH4+。Gassmann等[36]证明TaHKT2;1属于K+-Na+同向转运蛋白。当Na+浓度较低时,介导两种离子吸收;当Na+浓度较高、K+浓度较低时,介导低亲和性Na+吸收,K+吸收受到抑制。因此小麦TaHKT2;1的转运特性与外界离子情况密切相关。
Laurie等[37]将反义的TaHKT2;1转入小麦后发现,转基因植株内源TaHKT2;1强烈下调,在100 mmol·L-1NaCl浓度下,转基因植株22Na+内流明显低于对照组;在200 mmol·L-1NaCl浓度下,转基因株系根部的Na+浓度更低,转基因株系耐盐性提高,由此认为小麦TaHKT2;1在整株水平上的生理功能是介导Na+吸收。
小麦含有A、B和D三个基因组,硬粒小麦(T.turgidum)含有A和B两个基因组,前者比后者具有更高的耐盐性,因此推测小麦基因组D中可能含有耐盐性位点[38-39]。Dubcovsky等[39]从小麦4D染色体中分离到的Kna1能控制K+和Na+向地上部选择性运输,维持体内高的K+/Na+,提高其耐盐性。Munns等[40]从硬粒小麦耐盐品系149中分离到两个与Na+外排相关的位点,Nax1和Nax2。James等[41]研究表明,Nax1能够从根木质部和叶鞘中卸载Na+,防止Na+在叶片中过度积累,Nax2仅在根部发挥作用。Huang等[20]采用图位克隆法从一粒小麦(T.monococcum)中分离到两个可能的Na+转运蛋白基因TmHKT1;4-A1(TmHKT7-A1) 和TmHKT1;4-A2 (TmHKT7-A2),TmHKT1;4-A2主要在根部和叶鞘中表达,参与Na+在根部和叶鞘木质部中的卸载,被认为是Nax1的候选基因。Byrt等[42]发现TmHKT1;5-A是Nax2的候选基因。
3.2拟南芥AtHKT1;1与其耐盐性 Uozumi等[12]从拟南芥(A.thaliana)中克隆到AtHKT1;1,在非洲爪蟾卵母细胞(Xenopuslaevisoocytes)中表达分析表明,AtHKT1;1为专一的Na+转运蛋白,外界K+浓度不影响其转运特性;拟南芥AtHKT1;1在酵母突变体G19(Na+-extruding ATPase基因缺失)中表达后,发现G19对盐更敏感,但小麦TaHKT2;1在G19中表达后降低了其盐敏感性;AtHKT1;1不能互补酵母突变体CY162(缺失Trk1和Trk2)的K+吸收缺陷,但能使大肠杆菌(Escherichiacoli)突变体(无K+吸收功能,KAT1被点突变)LB2003在低K+培养基上生长。
Rus等[43]研究发现,AtHKT1;1的突变(athkt1-1,athkt1-2)能抑制拟南芥sos3-1(salt overly sensitive)的盐敏感性,HKT1;1的缺失提高了sos3-1的耐盐性,而且改善了sos3-1的缺K+表型,双突变体sos3-1hkt1-1幼苗的耐盐性提高,由此认为,AtHKT1;1介导根部Na+吸收。但Berthomieu等[44]认为AtHKT1;1不参与根部Na+吸收,因为突变体sas2-1(AtHKT1;1功能缺失)的Na+内流比野生型还高20%。
Munns[45]提出假设,地上部过多的Na+随韧皮部汁液流转运至根中,限制过多Na+在地上部积累对于提高植物耐盐性非常重要。Berthomieu等[44]研究发现,在盐胁迫下,突变体sas2-1韧皮部汁液中Na+的含量显著降低,Na+在叶中大量积累,但根中Na+含量降低,定位研究表明,AtHKT1;1主要在各器官的韧皮部组织中表达,由此认为AtHKT1;1参与Na+从地上部到根部的长距离运输,将地上部过多的Na+装载到韧皮部汁液中,防止过多Na+在地上部富集。
生理学研究表明,无机离子(如K+和Na+)被装载到根木质部中,在蒸腾拉力作用下向上运输,之后被转移到叶木质部薄壁细胞中,接着离子在薄壁细胞中卸载,经共质体途径进入韧皮部,重新返回根中[46-49]。Sunarpi等[50]采用GUS染色和免疫定位技术证明,AtHKT1;1主要在木质部薄壁细胞中表达,在韧皮部组织中仅有少量表达,因而提出AtHKT1;1从根木质部汁液中卸载Na+到周围薄壁细胞中,防止过多Na+上运至地上部。Davenport等[51]采用22Na同位素示踪法进行验证表明,AtHKT1;1主要介导Na+从木质部中的卸载。Møller等[52]将AtHKT1;1在拟南芥根中柱区域超表达后发现,与对照相比,转基因植株地上部Na+含量下降37%~64%,因为AtHKT1;1将更多的Na+卸载到木质部薄壁细胞中,提高了拟南芥的耐盐性。
AtHKT1;1对木质部K+没有直接调控作用。一种假设认为,AtHKT1;1将Na+卸载到木质部周围薄壁细胞中,随着Na+不断积累,引起薄壁细胞质膜去极化,激活外整流K+通道蛋白(KOR)或非选择性离子外整流通道蛋白(NOR)活性,K+被装载到木质部向地上部运输[34,46,48]。Gaymard等[53]从拟南芥中分离到K+外整流通道蛋白基因AtSKOR,异源表达系统结果和突变体atskor试验显示,AtSKOR介导K+从木质部薄壁细胞向木质部的装载。
Zhang等[54]研究表明,枯草芽孢杆菌(Bacillussubtilis)GB03能调节AtHKT1;1在组织中的特异性表达,提高拟南芥的耐盐性,在100 mmol·L-1盐胁迫下,GB03上调AtHKT1;1在地上部的表达量,而根中表达量降低。认为AtHKT1;1在盐胁迫时根中表达量下调,可以减少Na+进入根中,地上部表达量增加,可以使更多的Na+装载到韧皮部中,随韧皮部汁液运输到根中,降低Na+在地上部的积累,从而提高了耐盐性。
综上所述,当AtHKT1;1基因被敲除后,突变体植株会随着Na+含量的不断升高出现生长缓慢,地上部严重萎黄[29,44,50,55-57],说明AtHKT1;1在维持拟南芥体内K+和Na+稳态平衡中发挥重要作用。
3.3水稻OsHKT与其耐盐性 Horie等[14]在水稻中克隆到OsHKT2;1,在爪蟾卵母细胞和酵母中表达分析表明,OsHKT2;1对Na+有强选择性[14,24-25],但也介导K+的转运[58-59],不能互补低K+条件下酵母突变体的K+吸收功能缺陷。
Horie等[4]研究发现,在缺钾和低盐条件下,突变体oshkt2;1与野生型相比,oshkt2;1大大降低了根部Na+内流。进一步研究发现,OsHKT2;1主要在根部皮层和内皮层表达,因此认为OsHKT2;1可以介导根部有益Na+吸收,部分行使K+功能,减缓缺K+对植株的伤害。
Horie等[14]在耐盐品种Pokkali中分离到OsHKT2;2,其与小麦和大麦HKT2;1亲缘关系相近[19],为Na+-K+共转运蛋白,能互补低K+条件下酵母突变体的K+吸收功能缺陷,表达模式与OsHKT2;1相似,其表达受K+饥饿诱导。Kader等[60]研究表明,在150 mmol·L-1盐处理下,OsHKT2;2转录水平上调,且在叶韧皮部中表达。但Garciadeblás等[25]认为,在水稻品种日本晴和Pokkali中,OsHKT2;2都是假基因。
Ren等[28]从水稻中分离出与耐盐相关的遗传位点SKC1(Shoot K+Content),候选基因为OsHKT1;5。在爪蟾卵母细胞中表达分析表明,OsHKT1;5为Na+转运蛋白,主要在木质部薄壁细胞中表达。在盐处理条件下,根中表达量显著高于地上部,且随着胁迫时间的延长,根中OsHKT1;5表达量上调,而地上部变化不大。OsHKT1;5可以将过多的Na+从木质部中卸载到周围薄壁细胞中,降低木质部汁液中Na+含量,防止向地上部转运,间接使得K+向地上部运输,从而提高地上部的K+/Na+,在Na+长距离运输过程中发挥重要作用[61-62]。
3.4其他植物HKT与其耐盐性 Fairbairn等[13]从桉树中分离到EcHKT1;1和EcHKT1;2,它们编码的蛋白都能互补大肠杆菌突变体TK2463在缺K+条件下的K+吸收功能缺陷,在爪蟾卵母细胞中介导K+和Na+共转运。由于HKT蛋白对TEA+和Cs+不敏感,但对Ba2+非常敏感[13,25,63],Wang等[64]研究发现,在25 mmol·L-1NaCl处理下,Ba2+显著降低海滨碱蓬(S.maritima)根部Na+净吸收和22Na+内流,而TEA+和Cs+对其没有影响,因此推测HKT蛋白可能介导海滨碱蓬在低盐浓度下低亲和性Na+吸收。Shao等[16]从盐地碱蓬中克隆到SsHKT1;1,其表达受K+饥饿和盐胁迫诱导,主要在叶中表达,根中表达量相对较少,可能参与盐地碱蓬体内离子稳态平衡,对其耐盐性有重要作用。Chen等[65]将大豆(Glycinemax)GmHKT1;1在烟草(Nicotianatabacum)中超表达后发现,GmHKT1;1可以调节转基因植株根和叶的K+、Na+转运,对离子稳态平衡有重要影响,提高了烟草的耐盐性。Su等[15]从冰叶日中花中克隆到McHKT1;1,其编码蛋白在酵母中为K+转运蛋白,在爪蟾卵母细胞中对离子的选择性为Rb+>Cs+>(K+=Na+=Li+)。大麦HvHKT2;1与HvHKT2;2都参与植株体内离子稳态平衡,与其耐盐性也有重要关系[66-68]。耐盐型芦苇(Phragmitesaustralis)与盐敏感型芦苇相比,能维持体内更低的Na+和更高的K+含量[69]。Takahashi等[70]从芦苇中克隆到PhaHKT2;1-n,PhaHKT2;1-e和PhaHKT2;1-u,它们可能对维持芦苇体内离子稳态平衡有重要影响。
4 展望
土壤盐渍化危害作物生长,影响粮食产量。基因工程技术和分子育种对解决这一难题有重要帮助。HKT蛋白是一种与植物耐盐性密切相关的Na+或K+-Na+转运蛋白,能将植物木质部中过多的Na+卸载到其周围薄壁细胞中,降低地上部Na+含量,并维持体内K+稳态平衡。基于目前的研究现状,今后对HKT蛋白的研究应放在以下几点:1)通过比较甜土植物和盐生植物的差异,探求盐生植物耐盐的分子机理,发掘更多的HKT基因;2)利用分子生物学技术,进一步确认HKT蛋白参与耐盐的分子机制;3)采用基因工程手段,将筛选得到的HKT基因转入到作物中,培育新的转基因耐盐品种,对于增强盐渍生境下的粮食产量将具有重要的促进作用。
[1] Horie T,Hauser F,Schroeder J I.HKT transporter-mediated salinity resistance mechanisms in Arabidopsis and monocot crop plants[J].Trends in Plant Science,2009,14(12):660-668.
[2] Hasegawa P M,Bressan R A,Zhu J K,etal.Plant cellular and molecular responses to high salinity[J].Annual Review of Plant Physiology and Plant Molecular Biology,2000,51:463-499.
[3] 张宏飞,王锁民.高等植物Na+吸收、转运及细胞内Na+稳态平衡研究进展[J].植物学通报,2007,24(5):561-571.
[4] Horie T,Costa A,Kim T H,etal.Rice OsHKT2;1 transporter mediates large Na+influx component into K+-starved roots for growth[J].EMBO Journal,2007,26:3003-3014.
[5] 李三相,周向睿,王锁民.Na+在植物中的有益作用[J].中国沙漠,2008,28(3):485-490.
[6] 马清,楼洁琼,王锁民.Na+对渗透胁迫下霸王幼苗光合特性的影响[J].草业学报,2010,19(3):198-203.
[7] Rains D W,Epstein E.Sodium absorption by barley roots,its mediation by mechanism 2 of alkali cation transport[J].Plant Physiology,1967,42(3):319-323.
[8] Flowers T J,Läuchli A.Sodium versus potassium,substitution and compartmentation[A].In: Läuchli A,Bieleski R L.Inorganic Plant Nutrition[M].Berlin:Springer-Verlag,1983:651-681.
[9] 陈敏,彭建云,王宝山.整株水平上Na+转运体与植物的抗盐性[J].植物学通报,2008,25(4):381-391.
[10] Tyerman S D,Skerrett M,Garrill A,etal.Pathways for the permeation of Na+and Cl-into protoplasts derived from the cortex of wheat root[J].Journal of Experimental Botany,1997,48:459-480.
[11] Schachtman D P,Schroeder J I.Structure and transport mechanism of a high-affinity potassium uptake transporter from higher plants[J].Nature,1994,370:655-658.
[12] Uozumi N,Kim E J,Rubio F,etal.TheArabidopsisHKT1 gene homolog mediates inward Na+currents inXenopuslaevisoocytes and Na+uptake inSaccharomycescerevisiae[J].Plant Physiology,2000,122(4):1249-1260.
[13] Fairbairn D J,Liu W,Schachtman D P,etal.Characterisation of two distinct HKT1-like potassium transporters fromEucalyptuscamaldulensis[J].Plant Molecular Biology,2000,43(4):515-525.
[14] Horie T,Yoshida K,Nakayama H,etal.Two types of HKT transporters with different properties of Na+and K+transport inOryzasativa[J].Plant Journal,2001,27(2):129-138.
[15] Su H,Balderas E,Vera-Estrella R,etal.Expression of the cation transporter McHKT1 in a halophyte[J].Plant Molecular Biology,2003,52(5):967-980.
[16] Shao Q,Zhao C,Han N,etal.Cloning and expression pattern ofSsHKT1 encoding a putative cation transporter from halophyteSuaedasalsa[J].DNA Sequence,2008,19(2):106-114.
[17] Durell S R,Guy H R.Structural models of the KtrB,TrkH,and Trk1,2 symporters based on the structure of the KcsA K+channel[J].Biophysical Journal,1999,77(2):789-807.
[18] Liu W,Schachtman D P,Zhang W.Partial deletion of a loop region in the high affinity K+transporter HKT1 changes ionic permeability leading to increased salt tolerance[J].Journal of Biological Chemistry,2000,275(36):27924-27932.
[19] Platten J D,Cotsaftis O,Berthomieu P,etal.Nomenclature for HKT transporter,key determinants of plant salinity tolerance[J].Trends in Plant Science,2006,11(8):372-374.
[20] Huang S,Spielmeyer W,Lagudah E S,etal.A sodium transporter (HKT7) is a candidate forNax1,a gene for salt tolerance in durum wheat[J].Plant Physiology,2006,142:1718-1727.
[21] 邵群,丁同楼,韩宁,等.高亲和K+转运蛋白(HKT)与植物抗盐性[J].植物生理学通讯,2006,42(2):175-181.
[22] 林婵娟,许海霞,赵一丹,等.植物HKT转运蛋白研究进展[J].分子植物育种,2008,6(6):1153-1159.
[23] Zhang J L,Flowers T J,Wang S M.Mechanisms of sodium uptake by roots of higher plant[J].Plant and Soil,2010,326:45-60.
[24] Ardie S W,Xie L,Takahashi R,Liu S,etal.Cloning of a high-affinity K+transporter genePutHKT2;1fromPuccinelliatenuifloraand its functional comparison withOsHKT2;1 from rice in yeast andArabidopsis[J].Journal of Experimental Botany,2009,60(12):3491-3502.
[25] Garciadeblás B,Senn M E,Bauelos M A,etal.Sodium transport and HKT transporters: the rice model[J].Plant Journal,2003,34(6):788-801.
[26] Durell S R,Hao Y,Nakamura T,etal.Evolutionary relationship between K+channels and symporters[J].Biophysical Journal,1999,77(2):775-788.
[27] Kato Y,Sakaguchi M,Mori Y,etal.Evidence in support of a four transmembrane-pore-transmembrane topology model for theArabidopsisthalianaNa+/K+translocating AtHKT1 protein,a member of the superfamily of K+transporters[J].Proceedings of the National Academy of Sciences USA,2001,98(11):6488-6493.
[28] Ren Z H,Gao J P,Li L G,etal.A rice quantitative trait locus for salt tolerance encodes a sodium transporter[J].Nature Genetics,2005,37(10):1141-1146.
[29] Mäser P,Hosoo Y,Goshima S,etal.Glycine residues in potassium channel-like selectivity filters determine potassium selectivity in four-loop-per-subunit HKT transporters from plants[J].Proceedings of the National Academy of Sciences USA,2002,99(9):6428-6433.
[30] Rubio F,Gassmann W,Schroeder J I.Sodium-driven potassium uptake by the plant potassium transporter HKT1 and mutations conferring salt tolerance[J].Science,1995,270(5242):1660-1663.
[31] Diatloff E,Kumar R,Schachtman D P.Site directed mutagenesis reduces the Na+affinity of HKT1,an Na+energized high affinity K+transporter[J].Federation of European Biochemical Societies Letters,1998,432(1):31-36.
[32] Rubio F,Schwarz M,Gassmann W,etal.Genetic selection of mutations in the high affinity K+transporter HKT1 that define functions of a loop site for reduced Na+permeability and increased Na+tolerance[J].Journal of Biological Chemistry,1999,274(11):6839-6847.
[33] Kato N,Akai M,Zulkifli L,etal.Role of positively charged amino acids in the M2D transmembrane helix of ktr/Trk/HKT type cation transporters[J].Channels,2007,1:161-171.
[34] Hauser F,Horie T.A conserved primary salt tolerance mechanism mediated by HKT transporters: a mechanism for sodium exclusion and maintenance of high K+/Na+ratio in leaves during salinity stress[J].Plant Cell and Environment,2010,33(4):552-565.
[35] Epstein E,Rains D W,Elzam O E.Resolution of dual mechanisms of potassium absorption by barley roots[J].Proceedings of the National Academy of Sciences USA,1963,49(5):684-692.
[36] Gassmann W,Rubio F,Schroeder J I.Alkali cation selectivity of the wheat root high-affinity potassium transporter HKT1[J].Plant Journal,1996,10(5):869-882.
[37] Laurie S,Feeney K A,Maathuis F J,etal.A role for HKT1 in sodium uptake by wheat roots[J].Plant Journal,2002,32(2):139-149.
[38] Gorham J,Wyn Jones R G,Bristol A.Partial characterization of the trait for enhanced K+-Na+discrimination in the D genome of wheat[J].Planta,1990,180:590-597.
[39] Dubcovsky J,Santa Maria G,Epstein E,etal.Mapping of the K+/Na+discrimination locusKna1 in wheat[J].Theoretical and Applied Genetics,1996,92(3/4):448-454.
[40] Munns R,Rebetzke G J,Husain S,etal.Genetic control of sodium exclusion in durum wheat[J].Australian Journal of Agricultural Research,2003,54(7):627-635.
[41] James R A,Davenport R J,Munns R.Physiological characterisation of two genes for Na+exclusion in durum wheat:Nax1 andNax2[J].Plant Physiology,2006,142:1537-1547.
[42] Byrt C S,Platten J D,Spielmeyer W,etal.HKT1;5-like cation transporters linked to Na+exclusion loci in wheat,Nax2 andKna1[J].Plant Physiology,2007,143:1918-1928.
[43] Rus A,Yokoi S,Sharkhuu A,etal.AtHKT1 is a salt tolerance determinant that controls Na+entry into plant roots[J].Proceedings of the National Academy of Sciences USA,2001,98(24):14150-14155.
[44] Berthomieu P,Conéjéro G,Nublat A,etal.Functional analysis ofAtHKT1 inArabidopsisshows that Na+recirculation by the phloem is crucial for salt tolerance[J].EMBO Journal,2003,22(9):2004-2014.
[45] Munns R.Comparative physiology of salt and water stress[J].Plant Cell and Environment,2002,25(2):239-250.
[46] Wegner L H,Raschke K.Ion channels in the xylem parenchyma of barley roots.A procedure to isolate protoplasts from this tissue and a patch-clamp exploration of salt passageways into xylem vessels[J].Plant Physiology,1994,105(3):799-813.
[47] Lacan D,Durand M.Na+-K+exchange at the xylem/symplast boundary (Its significance in the salt sensitivity of soybean)[J].Plant Physiology,1996,110(2):705-711.
[48] Wegner L H,De Boer A.Properties of two outward-rectifying channels in root xylem parenchyma cells suggest a role in K+homeostasis and long-distance signaling[J].Plant Physiology,1997,115(4):1707-1719.
[49] De Boer A,Volkov V.Logistics of water and salt transport through the plant:Structure and functioning of the xylem[J].Plant Cell and Environment,2003,26(1):87-101.
[50] Sunarpi,Horie T,Motoda J,etal.Enhanced salt tolerance mediated by AtHKT1 transporter-induced Na+unloading from xylem vessels to xylem parenchyma cells[J].Plant Journal,2005,44(6):928-938.
[51] Davenport R J,Muoz-Mayor A,Jha D,etal.The Na+transporterAtHKT1;1 controls retrieval of Na+from the xylem inArabidopsis[J].Plant Cell and Environment,2007,30(4):497-507.
[52] Møller I S,Gilliham M,Jha D,etal.Shoot Na+exclusion and increased salinity tolerance engineered by cell type-specific alteration of Na+transport inArabidopsis[J].The Plant Cell,2009,21(7):2163-2178.
[53] Gaymard F,Pilot G,Lacombe B,etal.Identification and disruption of a plant shaker-like outward channel involved in K+release into the xylem sap[J].Cell,1998,94(5):647-655.
[54] Zhang H M,Kim M S,Sun Y,etal.Soil bacteria confer plant salt tolerance by tissue-specific regulation of the sodium transporter HKT1[J].Molecular Plant-Microbe Interaction,2008,21(6):737-744.
[55] Gong J M,Waner D A,Horie T,etal.Microarray-based rapid cloning of an ion accumulation deletion mutant inArabidopsisthaliana[J].Proceedings of the National Academy of Sciences USA,2004,101(43):15404-15409.
[56] Horie T,Horie R,Chan W Y,etal.Calcium regulation of sodium hypersensitivities ofsos3 andathkt1 mutants[J].Plant and Cell Physiology,2006,47(5):622-633.
[57] Corratgé-Faillie C,Jabnoune M,Zimmermann S,etal.Potassium and sodium transport in non-animal cell: the Trk/Ktr/HKT transporter family[J].Cellular and Molecular Life Science,2010,67:2511-2532.
[58] Golldack D,Su H,Quigley F,etal.Characterization of a HKT-type transporter in rice as a general alkali cation transporter[J].Plant Journal,2002,31(4):529-542.
[59] Jabnoune M,Espeout S,Mieulet D,etal.Diversity in expression patterns and functional properties in the rice HKT transporter family[J].Plant Physiology,2009,150:1955-1971.
[60] Kader M A,Seidel T,Golldack D,etal.Expression ofOsHKT1,OsHKT2,andOsVHAare differentially regulated under NaCl stress in salt-sensitive and salt-tolerant rice (OryzasativaL.) cultivars[J].Journal of Experimental Botany,2006,57(15):4257-4268.
[61] Lin H X,Zhu M Z,Yano M,etal.QTLs for Na+and K+uptake of the shoots and roots controlling rice salt tolerance[J].Theoretical and Applied Genetics,2004,108:253-260.
[62] Cotsaftis O,Plett D,Johnson A A,etal.Root-specific transcript profiling of contrasting rice genotypes in response to salinity stress[J].Molecular Plant,2011,4(1):25-41.
[63] Liu W,Fairbairn D J,Reid R J,etal.Characterization of two HKT1 homologues fromEucalyptuscamaldulensisthat display intrinsic osmosensing capability[J].Plant Physiology,2001,127:283-294.
[64] Wang S M,Zhang J L,Flowers T J.Low-affinity Na+uptake in the halophyteSuaedamaritime[J].Plant Physiology,2007,145:559-571.
[65] Chen H,He H,Yu D.Overexpression of a novel soybean gene modulating Na+and K+transport enhances salt tolerance in transgenic tobacco plants[J].Physiologia Plantarum,2011,141:11-18.
[66] Wang T B,Gassmann W,Rubio F,etal.Rapid up-regulation ofHKT1,a high-affinity potassium transporter gene,in roots of barley and wheat following withdrawal of potassium[J].Plant Physiology,1998,118:651-659.
[67] Huang S,Spielmeyer W,Lagudah E S,etal.Comparative mapping ofHKTgenes in wheat,barley,and rice,key determinants of Na+transport,and salt tolerance[J].Journal of Experimental Botany,2008,59(4):927-937.
[68] Qiu L,Wu D,Ali S,etal.Evaluation of salinity tolerance and analysis of allelic function ofHvHKT1 andHvHKT2 in Tibetan wild barley[J].Theoretical and Applied Genetics,2011,122:695-703.
[69] Takahashi R,Nishio T,Ichizen N,etal.Salt-tolerant reed plants contain lower Na+and higher K+than salt-sensitive reed plants[J].Acta Physiologiae Plantarum,2007,29(5):431-438.
[70] Takahashi R,Liu S,Takano T.Cloning and functional comparison of a high-affinity K+transporter gene PhaHKT1 of salt-tolerant and salt-sensitive reed plants[J].Journal of Experimental Botany,2007,8(15/16):4387-4395.

