胡杨甜菜碱醛脱氢酶基因的功能分化
刘佳琪,杨雪,李迪,杨海灵
1 北京林业大学生物科学与技术学院,北京 100083
2 中国科学院植物研究所 系统与进化植物学国家重点实验室,北京 100093
甜菜碱是一类季铵化合物,普遍存在于细菌、藻类、动物和植物中[1]。植物甜菜碱可以通过不同过程保护细胞免受胁迫压力的损害,包括参与细胞渗透调节,清除自由基,保护细胞膜完整性,稳定生物大分子结构与功能,保护细胞内组织免受脱水损害等。甜菜碱是以胆碱为底物经两步酶催化合成,首先由胆碱单加氧酶 (Choline rnonooxygenase,CMO) 催化胆碱形成甜菜碱醛,然后由甜菜碱醛脱氢酶 (Betaine aldebyde dehydrogenase,BADH,E.C.1.2.1.8) 催化形成甜菜碱,其中BADH是合成甜菜碱的关键酶[2]。许多研究发现将BADH基因转入烟草[3]、胡萝卜[4]、水稻[5]、棉花[6]等植物中,转基因植株的抗旱、耐盐、抗冻性都得到一定程度的提高,比如在胡萝卜中过量表达 BADH基因显著增强了植株的抗盐能力;在棉花中转入菠菜BADH基因明显增强其抗冻性能[4],说明了转基因植株在低温下细胞膜的通透性能较好,这可能是甜菜碱对维持细胞膜的结构有一定程度的保护作用[6]。因此,这些研究表明 BADH在植物的抗逆反应中发挥着重要作用。
甜菜碱脱氢酶属于醛脱氢酶 (ALDH) 超基因家族中的一个亚家族[7],迄今为止,BADH基因已从拟南芥、菠菜、甜菜、碱蓬、水稻、小麦、豌豆等草本植物中得到分离和鉴定[7-10],而木本植物BADH基因的研究报道较少。胡杨Populus euphratica是杨柳科杨属中古老的一个种,具有耐盐碱、抗旱、抗寒、抗风沙、适应性强等特点,是我国西北干旱盐碱荒漠地带能形成纯林的唯一高大乔木树种[11]。由于胡杨独特的抗逆能力使其能在干旱、盐碱化、多风沙的恶劣环境中生长,这对维持该地区脆弱的生态平衡具有非常重要的作用[12]。本研究从胡杨中克隆得到2个BADH基因,对该基因的基因结构、系统进化、表达模式、所编码蛋白的结构与生化功能进行了详细的研究,为深入揭示BADH基因在植物逆境胁迫中的作用机理奠定基础。
1 材料与方法
1.1 BADH基因序列的鉴定与命名
根据已知拟南芥ALDH10A8和ALDH10A9蛋白序列 (GenBank Accession No. AAM13070,AAK44148),利用TBLASTN检索GenBank中毛果杨Populus trichocarpa的基因组数据库,获得的2个与拟南芥ALDH10A8和ALDH10A9均具有较高相似性的序列。获得序列经过Pfam判读,确定该序列编码的蛋白具有BADH结构域特征。我们将这2组序列分别命名为PtBADH1 (GenBank Accession No. XM_002318594) 和 PtBADH2 (GenBank Accession No. XM_002322111)。
1.2 胡杨BADH基因的克隆
胡杨苗取自新疆。取新鲜胡杨顶芽组织0.1 g,按照Aurum Total RNA Kit (BIO-RAD) 试剂盒说明书步骤提取胡杨顶芽组织总 RNA,并以总RNA为模板,通过RNA PCR Kit (AMV) Ver.3.0反转录试剂盒 (TaKaRa),将总RNA反转录合成cDNA。根据PtBADH1和PtBADH2基因序列分别设计引物 PtBADH1-CL1/CL2和PtBADH2-CL1/CL2 (表1)。引物由Invitrogen公司合成。以cDNA为模板,利用2对引物分别进行PCR扩增。PCR产物经1%琼脂糖凝胶电泳检测,回收纯化目的条带,纯化产物连接到pGEM-T载体上。将构建好的重组质粒转化到大肠杆菌JM109感受态细胞中。挑选多个阳性克隆进行双向测序,测序分析使用载体通用引物。
1.3 系统发生关系分析
PeBADH1和PeBADH2的蛋白序列与其他植物ALDH序列通过Clustal X 1.83 进行氨基酸序列比对,然后经BioEdit手动校对。利用MEGA V.4.0的neighbour-joining (NJ) 模型构建进化树,Bootstrap值为1 000。
1.4 胡杨BADH基因的组织表达分析
为了研究 BADH基因在胡杨不同组织中和不同胁迫下的表达分布,分别对1年生胡杨进行了NaCl和H2O2胁迫处理,NaCl胁迫处理条件是:150 mmol/L NaCl 处理7 d,而H2O2胁迫处理条件是:0.5% H2O2处理12 h,然后提取不同处理的顶芽、叶、韧皮部、茎和根等组织的总RNA,并反转录合成cDNA。根据PeBADH1和PeBADH2的基因序列分别设计特异性引物PeBADH1-SP1/SP2和PeBADH2-SP1/SP2 (表1)。在PCR反应中以含有目的基因的pGEM-T载体作为阳性对照,以不加任何cDNA模板的PCR反应液作为阴性对照,并以胡杨Actin基因作为内标。PCR反应程序是:94 ℃ 预变性5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min,26个循环;最后72 ℃延伸5 min。PCR产物进行琼脂糖凝胶电泳检测,为了进一步验证所扩增到的是否为目的基因,对PCR产物进行测序。
1.5 胡杨BADH蛋白表达载体的构建、融合蛋白的表达与纯化
根据PeBADH1和PeBADH2的cDNA序列设计表达引物PeBADH1-EX1/EX2和PeBADH2-EX1/EX2 (表1)。通过定向克隆技术将PeBADH1和PeBADH2基因的编码序列亚克隆到带6×His标签的pET30a表达载体中,转化大肠杆菌Tuner感受态细胞中。将测序验证正确的重组表达菌于含卡那霉素的LB培养基中培养过夜,以1∶100稀释后扩大培养至A600为0.5~0.6,加入IPTG至终浓度0.1 mmol/L,37 ℃诱导4 h。诱导表达后的菌液于4 ℃、6 500×g离心10 min收获菌体,用缓冲液A (20 mmol/L咪唑,0.5 mol/L NaCl,20 mmol/L Na3PO4,pH 7.5) 重悬,冰上超声破碎裂解。然后4 ℃、10 000×g离心30 min,取少量原菌液、超声破碎离心后的上清液和沉淀,经SDS-PAGE检测是否具有目的蛋白,SDS-PAGE检测采用10%的分离胶,5%的浓缩胶。蛋白质纯化用 Ni亲和柱 (Amersham pharmacia biotech)。首先用缓冲液A平衡Ni亲和柱,将超声破碎后离心上清液上样,然后以缓冲液 A平衡,最后用缓冲液B (0.5 mol/L咪唑,0.5 mol/L NaCl,20 mmol/L Na3PO4,pH 7.5) 洗脱目的蛋白,收集目的蛋白洗脱峰进行下一步酶学活性分析。

表1 本研究所用的引物Table 1 Primers used in this study
1.6 胡杨BADH蛋白的酶学活性和热力学稳定性测定
植物 BADH基因是植物醛脱氢酶 (ALDH)超家族的一个亚类型,本实验对ALDH常见底物乙醛的活性测定参照Black的方法进行测定[13]。以乙醛为底物,室温下50 mmol/L Tris (pH 8.5)、1 mmol/L NAD+、100 mmol/L KCl、2% TritonX-100为反应缓冲液,将纯化得到的BADH蛋白酶分别在25 ℃到75 ℃之间每5 ℃设定一个温度梯度下保温10 min,在保温结束后检测酶对底物乙醛的催化活性。
1.7 胡杨BADH蛋白结构模拟
选取豌豆AMADH蛋白 (PDB:3iwj) 的X衍射晶体结构为模板,以Align 2D程序 (InsightII的Homology模块) 进行序列比对,运用InsightII的 Modeler模块构建蛋白质三维结构,产生 10个结构,每个结构产生 10个不同的环区结构,优化水平为高等,以Profiles-3D对各结构进行评价,选取最优结构。
2 结果与分析
2.1 胡杨BADH基因的序列与系统发育关系分析
使用RT-PCR方法,以胡杨顶芽cDNA为模板,克隆获得 PeBADH1和 PeBADH2的cDNA序列 (GenBank Accession No. JN816361和JN816362)。PeBADH1的cDNA编码503个氨基酸的蛋白,预测分子量为 54.93 kDa。PeBADH2的 cDNA编码 503个氨基酸的蛋白,预测分子量为54.90 kDa。PeBADH1蛋白序列与毛果杨PtBADH1的蛋白序列仅有4个氨基酸差异,而 PeBADH2蛋白序列与毛果杨 PtBADH2的蛋白序列有9个氨基酸差异(图1)。

图1 植物BADH蛋白的序列比对分析Fig. 1 Sequence alignments of plant BADH proteins. Conserved residues in all plant BADH proteins are marked in black. The NAD+-binding sites of plant BADH proteins are marked with black arrows. This sequence alignment was created using the following sequences: PeBADH1 (GenBank Accession No. JN816361), PeBADH2 (GenBank Accession No. JN816362), PtBADH1 (GenBank Accession No. XM_002318594), PtBADH2 (GenBank Accession No. XM_002322111), PsBADH (GenBank Accession No. CAC48392), AtBADH (GenBank Accession No. AAM13070), OsBADH (GenBank Accession No. BAC76608), SoBADH (GenBank Accession No. P17202), AcBADH (GenBank Accession No. AAM19157), SlBADH (GenBank Accession No. AAL33906), BvBADH (GenBank Accession No. P28237), TaBADH (GenBank Accession No. AAL05264).
为了研究胡杨BADH基因与其他植物BADH基因的序列相似性,我们选取多个代表不同进化历史的植物BADH基因为研究对象,包括双子叶植物拟南芥Arabidopsis thaliana、菠菜Spinacia oleracea、甜菜 Beta vulgaris、碱蓬 Suaeda liaotungensis、滨藜atriplex centralasiatica、豌豆Pisum sativum,单子叶植物水稻Oryza sativa、小麦Triticum aestivum等植物。序列比对分析发现所有BADH蛋白高度保守 (图1),其序列相似性范围是 70%~92%,其中 PeBADH1和PeBADH2蛋白的序列相似性最高为 92%,而小麦BADH蛋白与豌豆BADH蛋白的序列相似性最低为70%。
BADH基因被证明是ALDH基因超家族的成员[7],植物ALDH基因超家族的其他亚家族类型,包括ALDH2、ALDH3、ALDH7和GAPDH亚家族的蛋白序列也加入到系统发育分析中。系统发育树显示PeBADH1和PeBADH2与所有BADH聚成一枝,支持率达到100% (图2),这支持本研究克隆的2个基因为BADH基因。
2.2 胡杨BADH蛋白三维结构模建
图3A和图3B展示了模拟的2个胡杨BADH蛋白的三维结构。优化后的构象用Profile-3D进行氨基酸残基的兼容性考察,Profile-3D分析结果如图3C所示,从图中可以看到绝大部分氨基酸残基得分都是正值,位于合理的范围。结构比较发现PeBADH1与PeBADH2的结构高度保守(图3D)。胡杨PeBADH1与PeBADH2蛋白都具有 3个结构域:辅酶 NAD+结合结构域(NAD-binding domain),催化结构域 (Catalytic domain) 和寡聚反应结构域 (Oligomerization domain) (图3A和3B)。其中第1~132与151~230位氨基酸是辅酶NAD+结合结构域,这个结构域特异结合辅酶尼克酰胺腺嘌呤二核苷酸;第231~480位氨基酸是催化结构域,这个结构域能特异结合不同的反应底物;而第 481~490与133~150位氨基酸是寡聚反应结构域,由2个长β折叠和1个短β折叠组成的。
2.3 胡杨BADH基因的组织表达模式
通过RT-PCR研究胡杨BADH基因在正常生长、盐胁迫、氧化胁迫下不同组织 (根、茎、叶、顶芽和韧皮部) 中的表达分布 (图 4)。RT-PCR扩增后对 PCR产物进行测序,结果显示在正常生长、盐和H2O2胁迫下,PeBADH1和PeBADH2基因在韧皮部中不表达。在正常生长情况下,PeBADH1和PeBADH2基因在茎、叶、顶芽中都表达;不同之处在于PeBADH2基因的表达丰度比PeBADH1高,同时PeBADH2基因在根中表达较弱,而PeBADH1基因在根中未检测到表达。盐胁迫明显地诱导根中PeBADH1基因的表达,而抑制了PeBADH1和PeBADH2基因在其他组织中的表达。H2O2胁迫抑制了PeBADH1和PeBADH2基因在茎和顶芽中的表达。这些结果显示PeBADH1与PeBADH2存在表达模式的分化。
2.4 胡杨 BADH蛋白表达、纯化及酶活性分析
将构建的表达载体 pET30a/PeBADH1和pET30a/PeBADH2分别转化到大肠杆菌Tuner感受态细胞中,经IPTG诱导后发现PeBADH1和PeBADH2在大肠杆菌Tuner中高效表达 (图5)。通过镍柱亲和层析纯化后,获得纯化的重组蛋白,SDS-PAGE结果显示蛋白分子量与预测的PeBADH1和PeBADH2蛋白分子量大小一致。
BADH是植物ALDH超基因家族成员,由于植物 ALDH对底物多种脂肪醛和芳香醛具有广泛活性,特别是大部分ALDH对乙醛具有很高的活性,因此我们分别测定了 PeBADH1和 PeBADH2对乙醛的活性,对乙醛的催化活性的测定参照 Black[13]的方法,PeBADH1和PeBADH2的酶活性分别为0.073 μmol/(min·mg)和0.107 μmol/(min·mg),PeBADH2的酶活性是PeBADH1的1.5倍,这说明胡杨BADH蛋白具有醛脱氢酶活性。
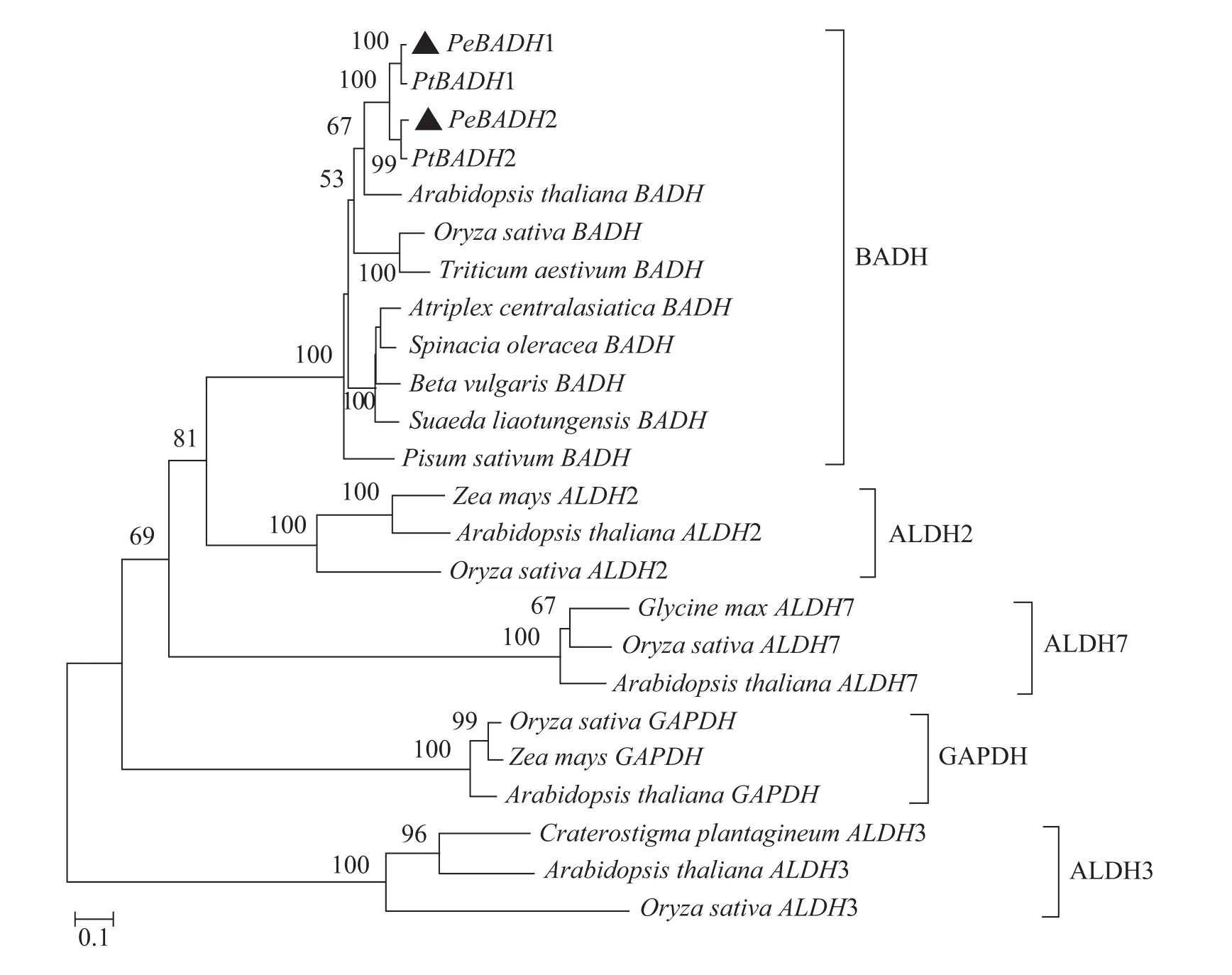
图2 植物BADH基因的系统发育分析Fig. 2 Phylogenetic tree showing relationships between PeBADH and other classes of plant ALDH. The affiliations of the plant BADH sequences used in the tree reconstruction are specified in the legend of Fig. 1. The affiliations of the other plant ALDH sequences are the following: Oryza sativa ALDH3 (GenBank Accession No. AAD35089), Arabidopsis thaliana ALDH3 (GenBank Accession No. AAL59944), Craterostigma plantagineum ALDH3 (GenBank Accession No. CAC84900), Zea mays GAPDH (GenBank Accession No. Q43272), Oryza sativa GAPDH (GenBank Accession No. AAM00227), Arabidopsis thaliana GAPDH (GenBank Accession No. AAK59790), Arabidopsis thaliana ALDH7 (GenBank Accession No. AAK55676), Oryza sativa ALDH7 (GenBank Accession No. AAG43027), Glycine max ALDH7 (GenBank Accession No. AAP02957), Oryza sativa ALDH2 (GenBank Accession No. BAA96794), Arabidopsis thaliana ALDH2 (GenBank Accession No. AAM27003), Zea mays ALDH2 (GenBank Accession No. AAK58370).

图3 胡杨PeBADH1和PeBADH2蛋白三维结构模拟Fig. 3 Structure modelling of PeBADH1 and PeBADH2 proteins. (A,B) Structures of PeBADH1 and PeBADH2. (C) Evaluation of the structure of PeBADH1 and PeBADH2 by Profile-3D program. (D) Structural comparison of PeBADH1 (green) and PeBADH2 (red).

图4 胡杨PeBADH1和PeBADH2基因的表达模式Fig. 4 Expression of the PeBADH1 and PeBADH2 genes in various tissues of P. euphratica. NC, NA and HO indicate the tissue under normal growth conditions and following NaCl and H2O2 treatments, respectively.
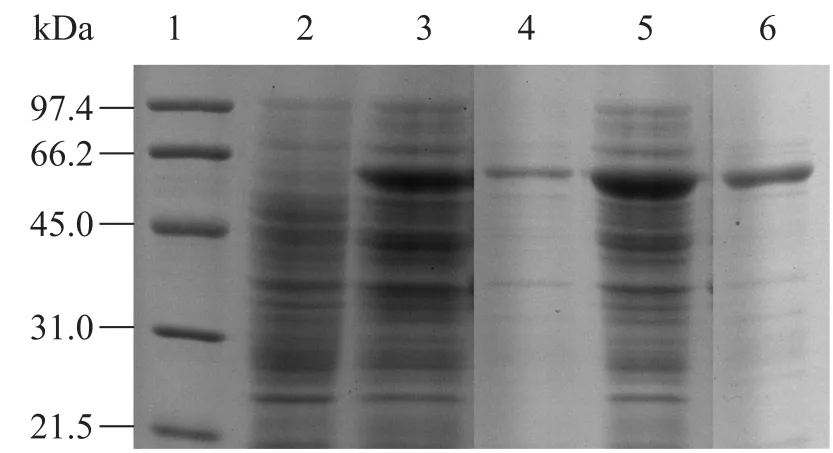
图5 胡杨PeBADH1和PeBADH2蛋白的表达与纯化Fig. 5 Overexpression and purification of BADH proteins from P. euphratica. 1: molecular mass markers; 2: total cellular extract from E. coli; 3, 5: total cellular extracts from induced bacteria containing PeBADH1 and PeBADH2, respectively; 4, 6: the purified PeBADH1 and PeBADH2, respectively.
2.5 胡杨BADH蛋白的热力学稳定性分析
PeBADH1和PeBADH2蛋白热力学稳定性结果如图 6所示。结果显示 PeBADH1和PeBADH2蛋白的热力学稳定性具有较大差异。PeBADH1在40 ℃时还保持着最高活性的85%,而PeBADH2仅保持着最高活性的25%。在50 ℃时,PeBADH1保持着最高活性的60%,而PeBADH2仅保持着最高活性的 10%。这说明PeBADH1是一个热力学稳定性较高的酶,在50 ℃以下都是稳定的,而相对于 PeBADH1,PeBADH2是一个热力学稳定性较低的酶。

图6 PeBADH1和PeBADH2蛋白的热力学稳定性Fig. 6 Thermal stability of recombinant PeBADH1 and PeBADH2.
3 讨论
植物在干旱、盐碱等逆境条件下可积累大量的甜菜碱[14],而甜菜碱的积累对改善渗透调节、保护细胞膜具有重要作用。植物体内甜菜碱的合成途径相对简单,主要包含两步氧化反应完成,甜菜碱一旦被合成后就不能被进一步代谢,积累在细胞中作为永久性或半永久性渗透调节剂[4]。因此,甜菜碱合成途径中的限速酶基因BADH被认为是最有希望的胁迫抗性基因之一,BADH的分子生物学与生物化学研究引起广泛的关注。在本研究中,我们从胡杨中克隆到2个BADH基因,对该基因的基因结构、表达模式、所编码蛋白的结构与生化功能进行了比较详细的研究。
在水稻、大麦和甜菜中,Northern blotting分析发现 BADH在正常生长和盐胁迫条件下均表达,但在盐胁迫条件下表达量升高,进一步研究发现这些基因能够提高植物细胞的渗透调节能力,降低因渗透失水造成对细胞膜、酶及蛋白质结构与功能的伤害[8-9,15]。在本研究中,在正常生长条件下的根中没有检测到胡杨PeBADH1基因的表达,但当受到盐胁迫时表达,这说明PeBADH1基因表达的积累与盐胁迫存在紧密关联,这个基因可能在胡杨细胞免受盐胁迫压力的损害中发挥重要作用。而PeBADH2基因在正常生长条件和盐胁迫下的根中微弱表达,说明PeBADH2可能在胡杨抗盐反应中没有发挥至关重要的作用,由于PeBADH2基因在正常生长条件下,在根、茎、叶和顶芽中均表达,说明这个基因在植物的生长发育中可能发挥着重要作用。

图7 PeBADH1和PeBADH2蛋白的辅酶NAD+结合位点分析Fig. 7 The NAD+-binding sites of PeBADH1 and PeBADH2 proteins. Blue dashed lines represent hydrogen bond interaction.
BADH蛋白一般都具有辅酶NAD+结合结构域[16],结构模拟发现PeBADH1和PeBADH2蛋白中有6个氨基酸残基能与NAD+形成氢键 (图7),说明这6个氨基酸是和辅酶NAD+结合的关键活性位点氨基酸。在这6个氨基酸中,除了第159位的Thr和Trp外,其他5个氨基酸在其他物种的BADH蛋白中高度保守,而第159位的Thr在菠菜、滨藜和碱蓬BADH蛋白中被Ser替代 (图 1)。PeBADH1和 PeBADH2蛋白的第231~480位氨基酸是催化结构域 (图3),这个结构域能特异结合不同的反应底物,菠菜 BADH蛋白的晶体结构显示在催化结构域有1个Cys残基是结合底物的关键活性位点氨基酸[16],序列比较分析显示这个氨基酸是 PeBADH1和PeBADH2蛋白的第294位Cys残基,这个氨基酸在所有的BADH蛋白中绝对保守 (图1中的第297位氨基酸位点)。
PeBADH1和PeBADH2蛋白的序列相似性高达92%,虽然蛋白序列相似性很高,两蛋白的酶学活性却有较大差异,PeBADH2的酶活性是PeBADH1的 1.5倍,而热力学稳定性分析发现PeBADH2的热力学稳定性低于PeBADH1,这说明两蛋白的生化性质有较大的差异。另外,基因的表达模式结果也显示PeBADH1与PeBADH2表达模式上有较大的变异。因此,本研究结果预示着胡杨PeBADH1与PeBADH2基因可能存在功能上的分化。
REFERENCES
[1] Rhodes D, Hanson AD. Quaternary ammonium and tertiary sulfonium compounds in higher plants. Annu Rev Plant Physiol Plant Mol Biol, 1993, 44: 357−384.
[2] Hanson AD, Rhodes DC.14CTracer evidence for synthesis of choline and betaine via phosphoryl base intermediates in salinized sugarbeet leaves. Plant Physiol, 1983, 71(3): 692−700.
[3] Rathinasabapathi B, McCue KF, Gage DA, et al. Metabolic engineering of glycine betaine synthesis: plant betaine aldehyde dehydrogenases lacking typical transit peptides are targeted to tobacco chloroplasts where they confer betaine aldehyde resistance. Planta, 1994, 193(2): 155−162.
[4] Kumar S, Dhingra A, Daniell H. Plastid-expressed betaine aldehyde dehydrogenase gene in carrot cultured cells, roots, and leaves confers enhanced salt tolerance. Plant Physiol, 2004, 136(1): 2843−2854.
[5] Kishitani S, Takanami T, Suzuki M, et al. Compatibility of glycinebetaine in rice plants: evaluation using transgenic rice plants with a gene for peroxisomal betaine aldehyde dehydrogenase from barley. Plant Cell Environ, 2000, 23(1): 107−114.
[6] Luo XL, Xiao JL, Wang ZA, et al. Overexpression of Spinacia oleracea betaine aldehyde dehydrogenase (SoBADH) gene confers the salt and cold tolerant in Gossypium hirsutum L.. Chin J Biotech, 2008, 24(8): 1464−1469.罗晓丽, 肖娟丽, 王志安, 等. 菠菜甜菜碱醛脱氢酶基因在棉花中的过量表达和抗冻耐逆性分析. 生物工程学报, 2008, 24(8): 1464−1469.
[7] Kirch HH, Bartels D, Wei YL, et al. The ALDH gene superfamily of Arabidopsis. Trends Plant Sci, 2004, 9(8): 371−377.
[8] Nakamura T, Yokota S, Muramoto Y, et al. Expression of a betaine aldehyde dehydrogenase gene in rice, a glycinebetaine nonaccumulator, and possible localization of its protein in peroxisomes. Plant J, 1997, 11(5): 1115−1120.
[9] McCue KF, Hanson AD. Salt-inducible betaine aldehyde dehydrogenase from sugar beet: cDNA cloning and expression. Plant Mol Biol, 1992, 18(1): 1−11.
[10] Weretilnyk EA, Hanson AD. Betaine aldehyde dehydrogenase from spinach leaves: purification, in vitro translation of the mRNA, and regulation by salinity. Arch Biochem Biophys, 1989, 271(1): 56−63.
[11] Zeng F, Yan H, Arndt SK. Leaf and whole tree adaptations to mild salinity in field grown Populus euphratica. Tree Physiol, 2009, 29(10): 1237−1246.
[12] Chen SL, Li JK, Bi WF, et al. Genotypic variation in accumulation of salt ions, betaine and sugars in Poplar under conditions of salt stress. Chin Bull Bot, 2001, 18(5): 587−596.陈少良, 李金克, 毕望富, 等. 盐胁迫条件下杨树盐分与甜菜碱及糖类物质变化. 植物学通报, 2000, 18(5): 587−596.
[13] Black S. Yeast aldehyde dehydrogenase. Arch Biochem Biophys, 1951, 34(1): 86−97.
[14] Hanson AD, May AM, Grumet R, et al. Betaine synthesis in chenopods: localization in chloroplasts. Proc Natl Acad Sci USA, 1985, 82(11): 3678−3682.
[15] Ishitani M, Nakamura T, Han SY, Takabe T. Expression of the betaine aldehyde dehydrogenase gene in barley in response to osmotic stress and abscisic acid. Plant Mol Biol, 1995, 27(2): 307−315.
[16] Johansson K, El-Ahmad M, Ramaswamy S, et al. Structure of betaine aldehyde dehydrogenase at 2.1 A resolution. Protein Sci, 1998, 7(10): 2106−2117.

