琼脂糖凝胶中DNA检测方法的研究进展
尤伟静,陈茂,马卫德,丛维涛,金利泰
作为研究生命遗传物质载体——核酸的工具,琼脂糖凝胶电泳已成为高效分离和分析核酸分子的必备手段,而在电泳后对 DNA 的检测是关键环节,因为它直接关系到实验结果显现与否。目前,常用的琼脂糖凝胶上 DNA 检测方法有荧光染色法、染料染色法、金属离子染色法、负染法等。近几十年来,许多研究者对染色方法进行了改进,使灵敏度和线性动态范围都有较大程度提高。本文主要对琼脂糖凝胶电泳后的 DNA 检测技术方面的研究历程进行总结,并展望其未来的发展方向。
1 各种 DNA 染色方法
1.1 荧光染色法
荧光染色法是检测 DNA 的一种常用分析技术。目前,用于琼脂糖凝胶上 DNA 研究和测定的荧光染色方法有Hoechst 33258、Pico Green[1]、YOYO、TOTO[2]、EB 染色法[3]、生物素染料染色法[4]、DAPI[5-6]、SYBR Green I 染色法[7-10]、SYBR Gold 荧光染色法[11]等。
LePecq 和 Paoletti[12]观察到 EB 染料与 DNA 结合时其荧光性增强的现象,此后在 1973 年,Sharp 等[13]首次将EB 法引入电泳凝胶上 DNA 的检测,并获得较好的效果,自此该检测方法就被广泛地应用于 DNA 的研究[14]。EB 染色法作为琼脂糖凝胶上 DNA 检测最经典的染色方法,具有操作快速、简便,对 DNA 专属性高,同时可满足大部分的实验要求。但它却具有两个比较严重的缺点:①EB 能嵌入 DNA 分子双螺旋结构中破坏 DNA 样品,从而具有较强的致畸、致突变作用,对实验操作者具有潜在危险;②EB 检测的凝胶需要在紫外灯下进行检识,而紫外射线常常导致核酸杂交、聚合[15-16],对其结构产生影响。Naimski 等将前人的 DAPI 染色法进行改进,通过优化操作步骤与条件来增加其灵敏度,他们最终优化的结果可使其灵敏度超过EB 荧光染色法[5-6]。
离子对技术应用于 DNA 检测是其检测方法学上的一大理论突破。通常应用于 DNA 检测的染料都带有铵根离子和芳香环,染料的铵根离子可与 DNA 的磷酸根离子通过静电作用相互结合,从而实现对 DNA 的染色。此方法不足之处在于铵根离子同时还会与琼脂糖中的硫酸根离子以及碳酸根离子结合,使得背景颜色较深,从而导致染色结果对比度较差[8,17]。而离子对理论的提出有望改善这一情况,提高灵敏度并降低凝胶背景。离子对检测技术的原理是通过采用两种相反电荷的染料,正、负离子染料在溶液中可形成疏水性离子对,使得染色液中游离存在的染料浓度降低,从而提高染料对 DNA 的选择性,增加 DNA 条带与背景的对比度,最终提高检测的灵敏度。同时,离子对染色法不需要脱色步骤,可使检测时间相对缩减。Choi 等[8,17]将离子对技术运用于 DNA 荧光染色,在增加灵敏度的同时还可避免 EB 的致畸作用。他们将小檗碱(BB)/媒染黄3R(MY3R)离子对染料运用于琼脂糖上 λDNA/HindIII 的检测,其灵敏度为 10 ng 左右,虽比 EB 染色方法低两倍,但比常用的亚甲基蓝染色方法高两倍[18],两者染料结构见图 1、图 2。

图 1 Berberine 的化学结构
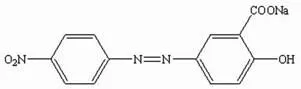
图 2 Mordant Yellow 的化学结构
近来人们将花青素类染料大量地应用于各研究领域,如:作为细胞、微团、组织[19-22]的荧光标记和探针、蛋白检测[23-24]、DNA 测序[25-26]、毛细管和凝胶电泳上核酸的定量分析[27-29]等。Hila 和 Taylor[4]对 23 种花青素染料进行筛选,最终发现了 5 种花青素染料可用于 DNA 检测,并比EB 染色法具有更高的灵敏度。
在对 DNA 染料进行筛选时,灵敏度是其中一个重要的指标,Schneeberger等[10]采用 SYBR Green I 对 PCR 产物进行检测,发现其灵敏度与 EB 相比具有较大的提高。同时,Vitzthum 等[9]用 SYBR Green I 对琼脂糖凝胶上的DNA 进行检测,结果显示其灵敏度可达到 20 pg,表明SYBR Green I 在 DNA 定量检测方面也是一种优良的检测试剂。另外,用 SYBR Green I 对琼脂糖凝胶上的 dsDNA进行检测,通过对体系条件(染料浓度、染色时间、染料与样品的比例等)的优化,得到最佳荧光强度条件,结果显示其灵敏度比 EB 高 6 ~ 8 倍,同时证明了 SYBR Green I染料对 dsDNA 有较强的专属性[9-10]。
SYBR Gold 荧光染料对琼脂糖凝胶上的 DNA 检测,可通过三种不同检测方式进行,包括电泳前染色、电泳与染色同时进行和电泳后染色。Suenaga 和Nakamura[11]将这三种染色方式进行比较,结果显示电泳前染色所得到的效果比其余两种方式要好,并且其灵敏度比 EB、SYBR Green I 都要高,但该染料存在着价格昂贵的缺点,并且其染色效果受较多外界因素(例如:染料-DNA 浓度比例、染料-DNA 配对比例等)的影响。
同时,Kirsanov 等[30]研究发现 SYBR Gold 和 SYBR Green II 在 Ames 试验中不具有变异性,该结果可为未来染料的安全性检测提供参考。近几年,SYBR Gold 荧光染料在实时 PCR 的研究中也有较快发展[31]。
2010 年,Huang 等[32]研发的 GelRed 无论是在预制凝胶染色还是在凝胶电泳后染色,都表现出了极高的灵敏度。在凝胶电泳后染色中优于或者至少相当于 SYBR Gold,但与 SYBR Gold 不同,GelRed 在预制凝胶中使用时仍然有极高的灵敏度。
1.2 染料染色法
荧光染色方法虽然具有较高的灵敏度,但其染色后的凝胶需要紫外透射仪进行数据读取,使得 DNA 染料染色法成为常规实验选用的检测技术。常用的 DNA 染料染色法主要有亚甲基蓝[33]、亮甲酚蓝[34]、乙基紫[35]和尼罗蓝[36]等,但是这些染料染色方法或需要较长的脱色时间(约 1 h),或灵敏度较低,或兼具两个缺点。因此,Yong 等[37]将尼罗蓝染色法进行改进,他们通过优化染色的顺序(先凝胶电泳,然后再染色),使其灵敏度有很大的提高。虽然该方法与 EB相比,其灵敏度仍较低[34,37],但因尼罗蓝染色方法的低毒性使得它在常规的实验应用方面具有较好的实用性。
Yang 等[38]将结晶紫(CV)/甲基橙(MO)离子对染料应用于琼脂糖上的 DNA 检测:(0.0025% CV,0.0005%MO,pH 6 ~ 7),在 30 min 内即可完成对 3 kb DNA 的检测,其灵敏度为 8 ng。两者染料结构见图 3、图 4。Hwang等[7]采用吲哚因蓝(IB)/甲基橙(MO)离子对染料对琼脂糖上的 DNA 进行检测:(0.008% IB、0.002% MO、10% 乙醇、0.2 mol/L 醋酸钠、pH 4.7),可在 60 min 内完成对λDNA/HindIII 的检测,其灵敏度可达 5 ng。两者染料结构见图 4、图 5。
1.3 银染法
银染法是迄今为止灵敏度最高的一种 DNA 染色法,该染色法广泛应用于聚丙烯酰胺凝胶上 DNA 的检测,其基本原理是银离子与 DNA 分子上的磷酸基团结合,然后结合的银离子被还原为金属银,可使 DNA 呈现深棕色至黑色。银染后的凝胶还可干燥、保存,但经银染后的 DNA不能回收,因此这种染色方法只能用于进行 DNA 分析,而不能用于 DNA 的制备与分离[39-40]。
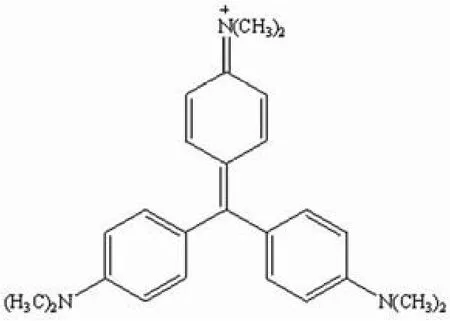
图 3 结晶紫的化学结构

图 4 甲基橙的化学结构

图 5 吲哚因蓝的化学结构
近年来,也不断有人尝试将银染法引入到琼脂糖凝胶上DNA 的检测,但是实验结果显示染色背景太深,较难观察到 DNA 条带[18,41]。Gottlieb 和 Chavko[40]将前人的银染法进行改进,对琼脂糖凝胶上的真核 DNA 进行检测,根据不同的样品采用不同的缓冲液和电泳条件,结果表明该方法检测真核生物 DNA 的灵敏度比 EB 高,可达 2.5 ng,并且对 5 ~ 30 ng 的单链和双链 DNA 均具有很好的线性相关性。
尽管琼脂糖上 DNA 银染法具有较高灵敏度,但是其缺点也是非常显著的,如:①可能造成很高的检测背景,特别在水不够纯的情况下;②费时费力,操作步骤繁琐;③费用较高;④需接触有毒的甲醛[41]等。因此,很多研究人员正不断寻求安全、灵敏的染色方法来代替银染法。
1.4 负染法
DNA 分子经过大量化学试剂处理之后,其生物活性往往受到较大影响,为了尽可能地保留核酸的生物活性,Hardy等[16]研发了一种新的检测方法——锌咪唑负染法,其灵敏度为 5 ~ 60 ng。该染色法的原理主要是锌离子与咪唑结合沉淀在胶面上,而在锌-DNA-咪唑区域不生成沉淀,因此染色后 DNA 条带区域是透明的。
2 结语及展望

图 6 近 37 年时间里重要的琼脂糖凝胶染色方法的时间进程表

表 1 常用的几种 DNA 检测方法优缺点的比较
琼脂糖凝胶上 DNA 染色是核酸分析研究中的一个重要领域,目前其问题主要集中在如何提高染色灵敏度,如何使操作简捷,如何降低试剂毒性和费用等方面。前人虽经过不懈努力取得一定进展(图 6),但仍然存在许多有待改进之处(表 1)。例如:①染料染色法虽操作简单,但其灵敏度还有待提高;②银染法是目前公认灵敏度较高的染色方法,但它的背景较深,操作步骤繁多,接触的试剂毒性较大,虽进行了不少改进,但还不尽如人意;③虽然荧光染色法可以使灵敏度接近银染,但荧光染色往往会改变凝胶中 DNA的性质,给进一步分析造成障碍,而且荧光染色的结果需要紫外透射仪才能够观察到;④负染法虽在一定程度上可以克服上述的某些缺点,但负染后胶条与背景的对比度往往不高,且负染后的条带是透明的,不易进行数据采集。
有效克服以上这些方面的缺陷已成为将来琼脂糖凝胶上 DNA 检测的发展方向。例如:如何在保持银染法高灵敏度的同时,又能够有效地降低染色背景,同时用无毒的化学物质去代替有毒的物质;针对荧光染色可能造成 DNA的化学性质改变的缺点进行改进;如何在保持负染法不破坏DNA 结构优势的同时,增加其条带与背景的对比度等。我们还可以尝试在染色同时使用几种染色方法,取长补短,达到最佳检测效果。上述方面如果能够得到改善,相信会对DNA 检测方法的改进带来极大的影响。
[1] Singer VL, Jones LJ, Yue ST, et al. Characterization of PicoGreen reagent and development of a fluorescence-based solution assay for double-stranded DNA quantitation. Anal Biochem, 1997, 249(2):228-238.
[2] Rye HS, Yue S, Wemmer DE, et al. Stable fluorescent complexes of double-stranded DNA with bis-intercalating asymmetric cyanine dyes:properties and applications. Nucleic Acids Res, 1992, 20(11):2803-2812.
[3] Maniatis T, Fritsch EF, Sambrook J. Molecular cloning: a laboratory manual. 2nd Ed. New York: Cold Spring Harbor, 1989.
[4] Hilal H, Taylor JA. Cyanine dyes for the detection of double stranded DNA. J Biochem Biophys Methods, 2008, 70(6):1104-1108.
[5] Naimski P, Bierzynski A, Fikus M. Quantitative fluorescent analysis of different conformational forms of DNA bound to the Dye,4',6-diamidine-2-phenylindole, and separated by gel electrophoresis.Anal Biochem, 1980, 106(2):471-475.
[6] Nairn RS, Dodson ML, Humphrey RM. Comparison of ethidium bromide and 4',6-diamidino-2-phenylindole as quantitative fluorescent stains for DNA in agarose gels. J Biochem Biophys Methods, 1982,6(2):95-103.
[7] Hwang SY, Jin LT, Yoo GS, et al. Counterion-dye staining for DNA in electrophoresed gels using indoine blue and methyl orange.Electrophoresis, 2006, 27(9):1739-1743.
[8] Jung DW, Yoo GS, Choi JK. Detection of DNA in agarose gels using berberine and mordant yellow 3R. Anal Biochem, 1999,272(2):254-256.
[9] Vitzthum F, Geiger G, Bisswanger H, et al. A quantitative fluorescence-based microplate assay for the determination of double-stranded DNA using SYBR Green I and a standard ultraviolet transilluminator gel imaging system. Anal Biochem, 1999, 276(1):59-64.
[10] Schneeberger C, Speiser P, Kury F, et al. Quantitative detection of reverse transcriptase-PCR products by means of a novel and sensitive DNA stain. PCR Methods Appl, 1995, 4(4):234-238.
[11] Suenaga E, Nakamura H. Prestaining method as a useful tool for the agarose gel electrophoretic detection of polymerase chain reaction products with a fluorescent dye SYBR gold nucleic acid gel stain.Anal Sci, 2005, 21(6):619-623.
[12] LePecq JB, Paoletti C. A fluorescent complex between ethidium bromide and nucleic acids. J Mol Biol, 1967, 27(1):87-106.
[13] Sharp PA, Sugden B, Sambrook J. Detection of two restriction endonuclease activities in Haemophilus parainfluenzae using analytical agarose--ethidium bromide electrophoresis. Biochemistry,1973, 12(16):3055-3063.
[14] Seville M. A whole new way of looking at things: the use of Dark Reader technology to detect fluorophores. Electrophoresis, 2001,22(5):814-828.
[15] Brunk CF, Simpson L. Comparison of various ultraviolet sources for fluorescent detection of ethidium bromide-DNA complexes in polyacrylamide gels. Anal Biochem, 1977, 82(2):455-462.
[16] Hardy E, Pupo E, Casalvilla R, et al. Negative staining with zinc-imidazole of gel electrophoresis-separated nucleic acids.Electrophoresis, 1996, 17(10):1537-1541.
[17] Jin LT, Choi JK. Usefulness of visible dyes for the staining of protein or DNA in electrophoresis. Electrophoresis, 2004, 25(15):2429-2438.
[18] Lasne F, Benzerara O, Lasne Y. A fast silver staining method for protein detection after isoelectric focusing in agarose gels. Anal Biohem, 1983, 132(2):338-341.
[19] Kovalska VB, Kyvorotenko DV, Balanda AO, et al. Fluorescent homodimer styrylcyanines: synthesis and spectral-luminescent studies in nucleic acids and protein complexes. Dyes Pigm, 2005, 67(1):47-54.
[20] Sims PJ, Waggoner AS, Wang CH, et al. Studies on the mechanism by which cyanine dyes measure membrane potential in red blood cells and phosphatidylchline vesicles. Biochemistry, 1974, 13(16):3315-3330.
[21] Moreda W, Forrester AR. Novel heterocyclic dyes as DNA markers.Part II. Structure and biological activity. Tetrahedron, 1997, 53(37):12605-12614.
[22] Moreda W, Forrester AR. Novel heterocyclic dyes as DNA markers.Part I. Synthesis and characterization. Tetrahedron, 1997, 53(37):12595-12604.
[23] Yarmoluk SM, Kryvorotenko DV, Balanda AO, et al. Proteins and cyanine dyes. Part III. Synthesis and spectroscopic studies of benzothiazolo-4-[1,2,6-trimethylpyridinium] monmethine cyanine dyes for fluorescent detection of bovine serum albumin in solutions.Dyes Pigm, 2001, 51(1):41-49.
[24] Patonay G, Salon J, Sowell J, et al. Noncovalent labeling of biomolecules with red and near-infrared dyes. Molecules, 2004, 9(3):40-49.
[25] Selvin P. Science innovation 92: the San Francisco sequel. Science,1992, 257(5072):885-886.
[26] Ju J, Glazer AN, Mathies RA. Energy transfer primer: a new fluorescent labeling paradigm for DNA sequencing and analysis.Nature Med, 1996, 2(2):246-249.
[27] Karlsson HJ, Eriksson M, Perzon E, et al. Groove-binding unsymmetrical cyanine dyes for staining of DNA: syntheses and characterization of the DNA-binding. Nucleic Acids Res, 2003, 31(21):6227-6234.
[28] Rye HS, Quesada MA, Peck K, et al. High-sensitivity two-color detection of double-stranded DNA with a confocal fluorescence gel scanner using ethidium homodimer and thiazole orange. Nucleic Acids Res, 1991, 19(2):327-333.
[29] Matselyukh BP, Yarmoluk SM, Mtselyukh AB, et al. Interaction of cyanine dyes with nucleic acids: XXXI. Using of polymethine cyanine dyes for the visualisation of DNA in agarose gels. J Biochem Biophys Methods, 2003, 57(1):35-43.
[30] Kirsanov KI, Lesovaya EA, Yakubovskaya MG, et al. SYBR Gold and SYBR Green II are not mutagenic in the Ames test. Mutat Res, 2010,699(1):1-4.
[31] Jor E, Myrmel M, Jonassen CM. SYBR Green based real-time RT-PCR assay for detection and genotype prediction of bovine noroviruses and assessment of clinical significance in Norway. J Virol Methods, 2010, 169 (1):1-7.
[32] Huang Q, Baum L, Fu WL. Simple and practical staining of DNA with GelRed in agarose gel electrophoresis. Clin Lab, 2010, 56(3-4):149-152.
[33] Yung-Sharp D, Kumar R. Protocols for the visualization of DNA in electrophoretic gels by a safe and inexpensive alternative to ethidium bromide. Technique, 1989, 1(3):183-187.
[34] Santillán-Torres JL, Ponce-Noyola P. A novel stain for DNA in agarose gels. Trends Genet, 1993, 9 (2):40.
[35] Cong WT, Zhu ZX, He HZ, et al. A visible dye-based staining method for DNA in polyacrylamide gels by ethyl violet. Anal Biochem, 2010,402(1):99-101.
[36] Adkins S, Burmeister M. Visualization of DNA in agarose gels as migrating colored bands: applications for preparative gels and educational demonstrations. Anal Biochem, 1996, 240(1):17-23.
[37] Yong YI, Hong HY, Lee IS, et al. Detection of DNA using a visible dye, Nile blue, in electrophoresed gels. Anal Biochem, 2000, 280(2):322-324.
[38] Yang Y, Jung DW, Bai DG, et al. Counterion-dye staining method for DNA in agarose gels using crystal violet and methyl orange.Electrophoresis, 2001, 22(5):855-859.
[39] Peats S. Quantitation of protein and DNA in silver-stained agarose gels. Anal Biochem, 1984, 140(1):178-l82.
[40] Gottlieb M, Chavko M. Silver staining of native and denatured eucatyotic DNA in agarose gels. Anal Biochem, 1987, 165(1):33-37.
[41] Willough EW, Lambert A. A sensitive silver stain for proteins in agarose gels. Anal Biochem, 1983, 130(2):353-358.

