长牡蛎基因组微卫星引物的开发和特性描述
韩斐斐,亓海刚,李莉,张国范,闫喜武,李慧娟,杨霏,王琳楠
(1. 大连海洋大学,辽宁 大连 116023;2. 中国科学院海洋研究所,山东 青岛 266071)
长牡蛎基因组微卫星引物的开发和特性描述
韩斐斐1,2,亓海刚2,李莉2,张国范2,闫喜武1,李慧娟1,2,杨霏1,王琳楠1
(1. 大连海洋大学,辽宁 大连 116023;2. 中国科学院海洋研究所,山东 青岛 266071)
长牡蛎于20世纪80年代从日本引进中国。在我国,长牡蛎已经成为重要的贝类养殖产业之一。本实验从全基因组上筛查微卫星序列,在微卫星筛查的范围、数目和类型上是传统的富集文库法开发微卫星所无法比拟的。利用基因组微卫星序列总共设计了104对引物,54对引物能扩增出目的片段,其中有34对引物显示多态性扩增,占32.7%,20对引物显示单态性扩增,占19.2%。在自然群体48个个体样本中分析了这些位点的多态性,结果表明:等位基因数目在2~8之间,观测杂合度和期望杂合度分别在0.065 2~0.795 5和0.063 8 ~0.853 4之间。34对微卫星分子标记中有7对符合哈迪-温伯格平衡,27对或多或少的偏离平衡。微卫星分子标记可以用作分子遗传育种、遗传连锁图谱的构建、种群遗传结构的分析、亲缘关系分析等方面。
长牡蛎;微卫星;标记开发;基因组
长牡蛎(Crassostrea gigas)又称太平洋牡蛎,属于软体动物门,瓣鳃纲,翼形亚纲,珍珠贝目,牡蛎科,具有个体大、生长速度快、适应性强、成活率高等特点[1]。20世纪80年代初从日本引入我国,80年代末在我国北部沿海大面积养殖,是我国海产贝类养殖中规模大、产量高的养殖品种之一[2],也是世界上产量最高的养殖贝类品种之一[3]。我国牡蛎养殖产量在近 10年一直呈上升趋势,长牡蛎的产量约占牡蛎总产量的1/3[4]。
微卫星(microsatellite)DNA,又称短串联重复序列(short tandem repeats,STRs)或简单序列重复(simple sequence repeats,SSRs),是指由1~6 个核苷酸组成的简单串联重复DNA 序列,它是一种DNA长度多态性标记,并且具有多态性强,呈共显性遗传等特点。微卫星作为一种分子标记,已成为种群研究和进化生物学最常用的分子标记之一,广泛地应用于生物杂交育种、遗传连锁图谱、种群遗传多样性、系统发生等研究领域[5]。微卫星的特点主要体现在共显性标记、多态信息含量高、结果稳定可靠。
李琪[6]等采用磁珠杂交选择和PCR筛选法,从长牡蛎DNA选择片段文库中,分离含有微卫星序列的阳性克隆56个,其中41个(20.5%)有随机侧翼区,可以进行引物设计,还获得两个小卫星克隆。于红[7]等从公共数据库中4 201条长牡蛎EST开发10个微卫星标记,9个位点可以在C. plicatula和C. ariakensis中成功扩增。李莉[8]等研究了在一个回交家系中 15个微卫星的分离情况。Sekino[9]等通过长牡蛎微卫星富集文库发现了123个包含引物设计区域的重复序列,设计了 9条分型引物,8个位点显示多态性,并发现6、4、5个位点分别可以在C. sikamea, C. ariakensis, C. nippona中扩增获得预期大小的等位基因。Huvet[10]等应用微卫星标记对长牡蛎和与之亲缘关系很近的葡萄牙牡蛎 C.angulata进行了分析,为这2种牡蛎在自然群体中存在杂交现象提供了证据。Sauvage[11]等通过扫描9 272个长牡蛎EST-contigs设计22组微卫星分型引物,显示 18个多态性位点。虽然长牡蛎中开发并应用微卫星引物有一系列相关报道,但在大规模开发微卫星标记上存在缺陷。
而大规模开发微卫星分子标记是推动长牡蛎分子遗传学研究深入开展并走向应用的必要条件。本文所开发的微卫星标记来自 FOSMID大片段文库的末端测序,在微卫星筛查的范围、数目和类型上是传统的富集文库法开发微卫星所无法比拟的。
1 材料和方法
1.1 实验材料
Silvia[12]等在进行遗传学分析时,发现群体的样本容量要大于30。本实验选用自然群体48个长牡蛎个体。
1.2 基因组DNA的提取
取长牡蛎肌肉,参照 Sambrook[13]等采用常规的酚/氯仿抽提的方法提取DNA。
1.3 微卫星序列的来源
实验所用的序列来源为基因组测序时构建的大片段fosmid文库末端测序,该文库的基因组序列覆盖度达到10倍,共完成451.164条fosmid-end测序,双向成功reads数为225.582对,测序深度覆盖整个基因组的0.17×,充分保证了在全基因组的层面上进行大规模的微卫星筛查。
1.4 引物的设计与合成
利用软件RIMER PREMIER 5.0设计引物,引物长度在18~22 bp左右,GC含量在40%~60%之间,扩增目的片段长度在100~250 bp之间,在序列保守区内设计引物。总共设计了104对长牡蛎微卫星引物,由上海生工生物技术公司合成。
1.5 PCR扩增
PCR反应体系(15μl):20 ng的基因组DNA、10×buffer缓冲液(20 mM Mg2+)、0.15 μl Tag酶、dNTP各0.2 mmol/L、引物各0.3 μmol/L。PCR反应条件:95℃变性5 min;94℃ 30 s,退火30 s,72℃ 30 s,30个循环;72℃延伸10 min,4℃保存。各对引物的退火温度见表1。用12%非变性聚丙烯酰胺(丙烯酰胺和甲叉双丙烯酰胺体积比为29︰1)凝胶电泳分离PCR反应产物,电泳液为1×TBE缓冲液,电压300 V,电泳1~2 h(北京六一仪器厂DYY-Ⅱ型电泳仪,DYCZ-30型电泳槽),Gel-red染色,利用UVP凝胶成像仪成像。
1.6 引物的筛选和数据处理
104对微卫星引物均先用长牡蛎自然群体的 6个DNA样本扩增,能扩增出条带的引物即被初选,然后初选的引物用自然群体的 48个个体扩增、跑跤。每对引物的电泳条带按有条带的记为 1,无条带的记为 0,不同大小的条带总数记为总等位基因数。
根据每个个体的条带确定基因型,利用POPGENEGENE(VERSION 1.31)软件统计微卫星基因座的等位基因数(Observed number of alleles,Na)、有效等位基因数(Effective number of alleles,Ne)、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)、遗传相似系数(Genetic similarity index,I),群体间遗传距离(Genetic Distance,Ds),并进行Hardy-Weinberg平衡检验。
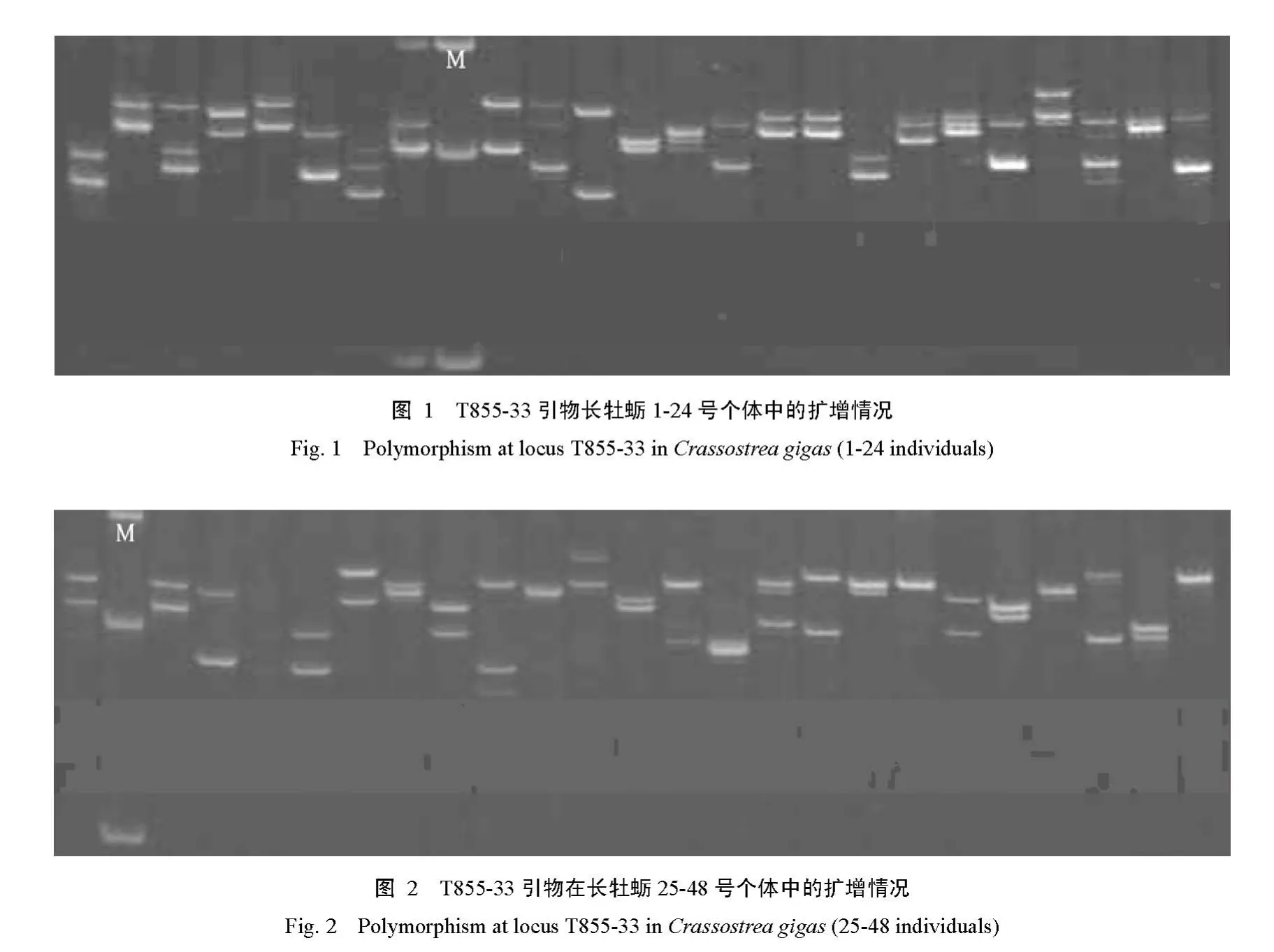
参照Botstein 等[14]的方法计算多态性信息含量(Polymorphism Information Content,PIC):
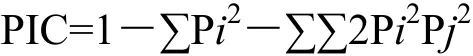
公式中,Pi、Pj分别为群体中第i和第j个等位基因频率,n为等位基因数。
2 结 果
2.1 PCR扩增反应结果
本实验总共设计了104对引物,其中有54对引物能在长牡蛎的6个样本中扩增出目的片段。用长牡蛎自然群体的48个个体进行分析,34对引物显示多态性扩增,占32.7%,20对引物显示单态性扩增,占19.2%。
2.2 遗传多态性分析
在长牡蛎自然群体的 48个个体样本中分析了这些位点的多态性(图1,图2是T855-33引物分别在1~24和25~48个DNA样本上的凝胶电泳图)以T855-33引物为例,有等位基因7个,平均等位基因5.030 6,观测杂合度0.520 8,期望杂合度0.809 6,并未偏离Hardy-Weinberg 平衡。
各位点分析如表1所示,结果表明:每个位点的等位基因数目在 2~8之间,平均等位基因数是5.0,观测杂合度在0.065 2~0.795 5之间,期望杂合度在0.063 8~0.853 4之间。34对微卫星分子标记中有7对符合哈迪-温伯格平衡,有27对或多或少的偏离平衡。
3 讨 论
微卫星分子标记在分子遗传育种、遗传连锁图谱的构建、种群遗传结构的分析、亲缘关系分析等方面得到了广泛的应用。目前开发微卫星分子标记的主要途径有3种:一是整个基因组序列测序,检索到微卫星序列,根据位点两端序列设计引物;二是检索基因组数据库中已发表的序列,设计引物;三是利用SSR分子标记的可转移性和通用性,在相近物种中筛选可用的引物。虽然传统的富集文库法开发微卫星分子标记的方法比较繁琐,消耗大,但在未经过基因组测序的物种中,也常利用此方法开发微卫星分子标记。检索基因组数据库中已发表的序列开发微卫星标记,已有相关报道[7]。研究证明,利用微卫星引物的通用性,可以在相近物种中开发SSR分子标记,如李琪[15]等通过长牡蛎基因和EST序列开发 15个微卫星标记,发现这些微卫星至少可以在C. plicatula, C. hongkongensis, C. ariakensis,C. nippona和C. sikamea中的一种中扩增。对序列完全未知的物种可以利用微卫星标记的通用性来开发相近物种的SSR标记,可以提高研究效率。
实验通过对整个基因组序列进行测序,采用新方法在全基因组上筛查微卫星序列,为大规模开发微卫星分子标记,从而推动长牡蛎分子遗传学研究深入开展并走向应用提供必要条件。
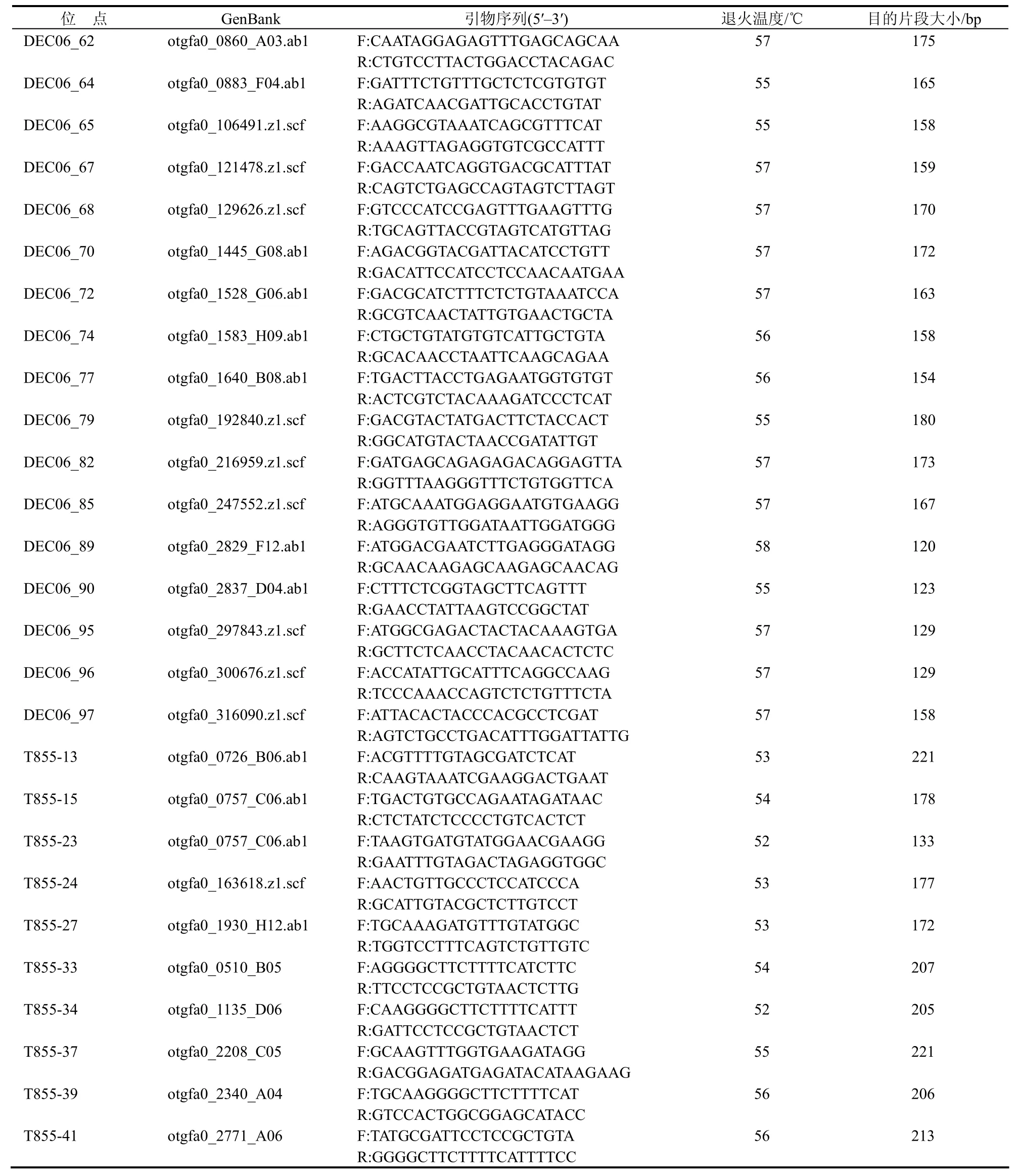
表 1 34对多态性微卫星引物序列Tab. 1 Sequences of 34 novel polymorphic microsatellite loci for the Pacific Oyster Crassostrea gigas

续表1
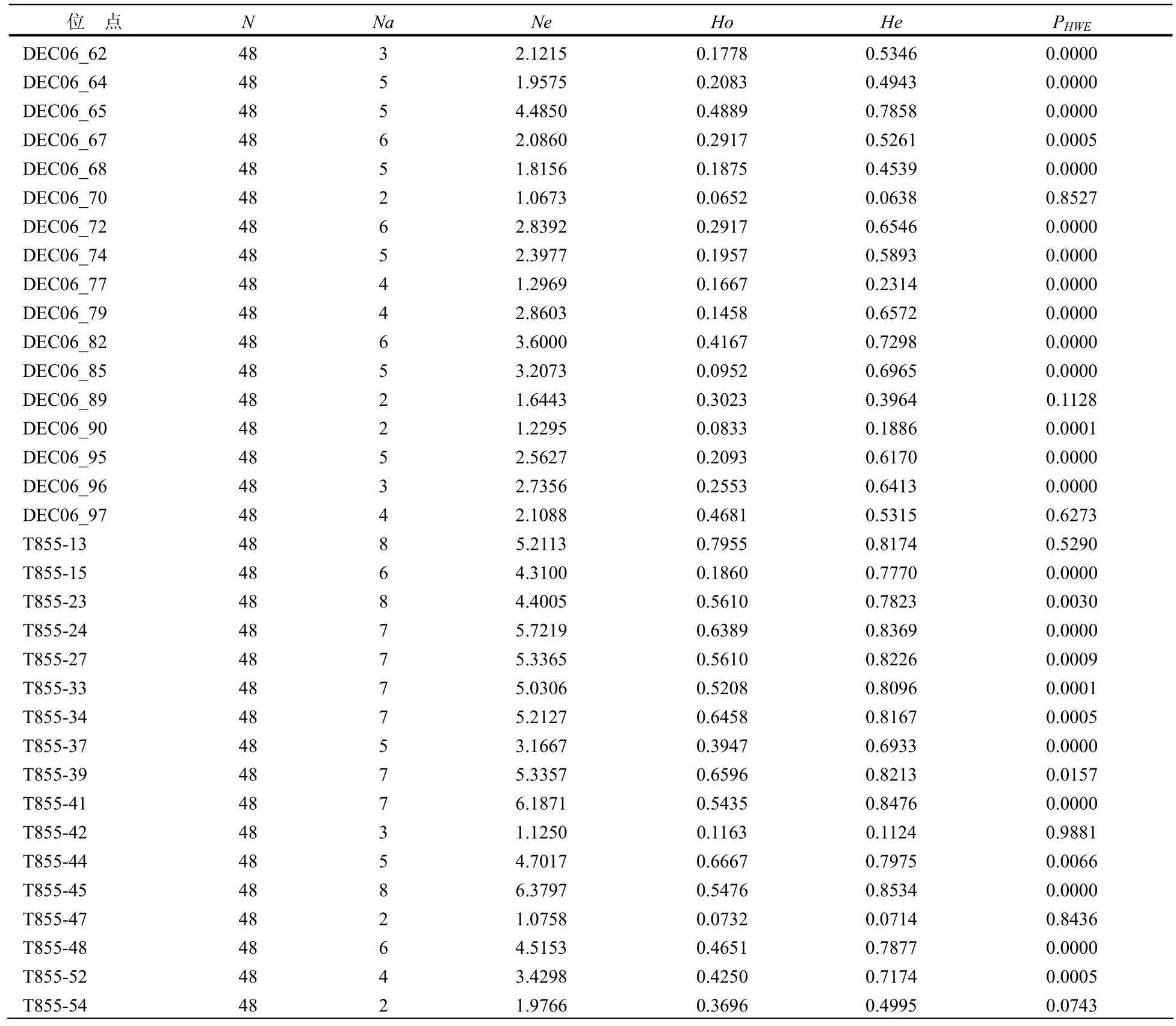
表 2 34对多态性微卫星引物特性Tab. 2 Characterization of 34 novel polymorphic microsatellite loci for the Pacific Oyster Crassostrea gigas
致谢:中国科学院海洋研究所李娟、王家丰、王威对实验的帮助,在此表示感谢。
[1] 李华琳. 长牡蛎苗种培育 [J]. 齐鲁渔业, 2005, 22(10): 4-5.
[2] 毛玉泽, 周毅, 杨红生, 等. 长牡蛎代谢率的季节变化及其夏季死亡关系的探讨 [J]. 海洋与湖沼, 2005, 36(5): 445-451.
[3] FAO. 2008年世界渔业和水产养殖状况 [R]. 罗马: FAO, 2009.
[4] 于瑞海, 李琪, 王照萍, 等. 我国北方太平洋牡蛎育苗及养殖现状 [J]. 科学养鱼, 2008, (6): 3-5.
[5] Goldstein D B, Pollock D D. Launching microsatellites: a review of mutation processes and methods of phylogenetic interference [J].Hered, 1997, 88: 335-342.
[6] 李琪, 木岛明博. 长牡蛎(Crassostrea gigas)微卫星克隆快速分离及特性分析 [J]. 海洋与湖沼, 2004, 35: 364-370.
[7] 于红, 李琪. 太平洋牡蛎养殖与野生群体遗传变异的微卫星研究[J]. 遗传学报, 2007, 34(12): 1114-1122.
[8] 李莉, 郭希明, 张国范. Inheritance of 15 microsatellites in the Pacific oyster Crassostrea gigas: segregation and null allele identification for linkage analysis [J]. 中国海洋与湖沼学报: 英文版, 2009, 27(1): 74-79.
[9] Sekino M, Hamaguchi M, Aranishi F, et al. Development of novel microsatellite DNA markers from the pacific oyster Crassostrea gigas [J]. Marine Biotechnology, 2003, 5(3): 227-233.
[10] Huvet A, Fabioux C, McCombie H, et al. Natural hybridization in genetically differentiated populations of Crassostrea gigas and C.angulata highlighted by sequence variation in flanking regions of a microsatellite locus [J]. Marine Ecology Progress Series, 2004, 272:141-152.
[11] Sauvage C, Boudry P, Lapegue S. Identification and characterization of 18 novel polymorphic microsatellite makers derived from expressed sequence tags in the Pacific oyster Crassostrea gigas [J].Molecular Ecology Resources, 2009, 9(3): 853-855.
[12] Silvia E P, Russo C A M. Techniques and statistical data analysis in molecular population genetics [J]. Hydrobiologia, 2000, 420(1):119-135.
[13] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: A Laboratory Manual, 2nd edn. Cold Spring Harbor [R]. New York:Cold Spring Harbor Laboratory Press, 1989.
[14] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. American Journal of Human Genetics, 1980,32(3): 314-331.
[15] Li Q, Liu S K, Kong L F. Microsatellites within genes and ESTs of the Pacific oyster Crassostrea gigas and their transferability in five other Crassostrea species [J]. Electronic Journal of Biotechnology,2009, 12(3): 1-6.
[16] 贾智英, 梁利群, 孙效文. 方正银鲫微卫星序列的筛选及PCR扩增产物的初步检测 [J]. 中国水产科学, 2006, 13(6): 990-994.
Development and characterization of SSR in Genome for the Pacific Oyster Crassostrea gigas
HAN Fei-fei1,2, QI Hai-gang2, LI Li2, ZHANG Guo-fan2, YAN Xi-wu1, LI Hui-juan1,2,YANG Fei1, WANG Lin-nan1
(1. Dalian Ocean University, Dalian 116023, China; 2. Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China)
The Pacific Oyster (Crassostrea gigas) was introduced into China from Japan in 1980s. Since then, the Pacific Oyster culture has been one of the important mollusks in China. Microsatellite (Simple Sequence Repeats,SSRs) sequences were screened from the whole genome, which has advantage over the traditional microsatellitescreening method including the scope, number and type of microsatellites. Primers were designed for one hundred and four SSR loci derived from genome microsatellite sequences. Fifty-four primer pairs amplified the target DNA fragments and 34 primer pairs were polymorphic in the Pacific Oyster, accounting for 51.9﹪ and 32.7﹪ of the total designed SSR loci. The polymorphisms of these loci were analyzed in 48 individuals from a natural population. The number of alleles per locus ranged from 2 to 8 with average of 5.0. The observed heterozygosity ranged from 0.0652 to 0.7955, and the expected heterozygosity ranged from 0.063 8 to 0.853 4. Seven of 34 microsatellite markers accord with Hardy-Weinberg equilibrium (HWE), and 27 markers show more or less bias from HWE. These SSRs can be used in inheritance cross-breeding, linkage genetic mapping, and other domains in the species.
Crassostrea gigas; Microsatellite; Marker development; Genome
Q751
A
1001-6932(2011)05-0566-06
2011-03-11;
2011-05-11
国家重点基础研究发展计划(973计划项目,2010CB126402);国家自然基金(40730845);国家公益性行业(农业)科研专项(3-53);中国博士后基金(20090461270)。
韩斐斐(1984-),女,硕士研究生,从事水产养殖分子生物学研究。电子邮箱:hanfeifei0325310406@126.com。
闫喜武,教授。电子邮箱:yanxiwu2002@163.com。

