HIV相关性口腔白色念珠菌rDNA基因型研究
张云操,刘 奇,武有聪,郭利军,申元英,白 丽
(大理学院基础医学院,云南大理 671000)
HIV相关性口腔白色念珠菌rDNA基因型研究
张云操,刘 奇,武有聪,郭利军,申元英,白 丽*
(大理学院基础医学院,云南大理 671000)
目的:了解HIV感染人群和健康人群口腔白色念珠菌基因型的分布情况。方法:运用CA-INT特异性引物聚合酶链反应方法扩增白色念珠菌包含1类内含子的编码rRNA的25S rDNA区,根据扩增条带大小进行基因分型,并应用χ2检验对54株HIV感染人群和54株健康人群口腔白色念珠菌基因型别进行分析。结果:CA-INT特异性引物PCR法可将108株口腔白色念珠菌分为A、B和C型,以A型最多见,两组人群口腔白色念珠菌基因型分布基本相同,但HIV感染人群口腔白色念珠菌A型所占构成比高于健康人群,差别有统计学意义。结论:HIV感染人群和健康人群口腔白色念珠菌均具有基因多态性,CA-INT特异性引物PCR法是较好的口腔白色念珠菌种内菌株间鉴定的方法。
白色念珠菌;HIV;聚合酶链反应;rDNA基因型
口腔念珠菌病(oral candidiasis,OC)是一组由念珠菌属感染所引起的口腔粘膜疾病,然而在口腔念珠菌感染中又以白色念珠菌(Candida albicans,CA)为最主要的条件致病菌。OC是HIV感染人群最常见的并发症之一,机体一旦出现OC预示疾病即将从HIV感染期进入至AIDS发病阶段,故其是早期发现和诊断AIDS的重要线索〔1〕。从基因水平对口腔白色念珠菌菌株间差异进行基因分型研究,能克服表型研究出现的缺点,无疑是能更好认识到OC流行病学的意义。本文采用特异性引物PCR分型方法,以分离来自HIV感染人群口腔白色念珠菌为研究对象,健康人群口腔白色念珠菌为对照,扩增白色念珠菌包含1类内含子的编码rRNA的25S rDNA区,以探讨2组人群口腔白色念珠菌基因型分布情况,并为HIV感染人群的OC分子流行病学研究提供方法。
1 材料与方法
1.1 材料 分离自云南大理地区HIV感染人群和健康人群的口腔念珠菌,经形态染色、培养特性、芽管形成实验、假菌丝及厚膜孢子形成实验、YBC酵母鉴定卡及CHROMagar培养基培养综合鉴定为白色念珠菌,其中HIV感染人群分离到54株口腔白色念珠菌,健康人群分离到54株口腔白色念珠菌,质控菌株为ATCC10231(本教研室提供)。
1.2 方法
1.2.1 菌株DNA提取 参照文献〔2〕修改后进行,实验菌株在沙氏琼脂培养基激活2次后,取单个菌落(直径2 mm)加入含0.5 mL无菌去离子水的离心管中,然后室温离心后,弃去上清;加入0.5 mL 0.25M Tris(pH8.0)-1.5%SDS裂解液,恒温混匀仪上加热100℃30 min,后摇匀2 min;加入等体积的酚∶氯仿∶异戊醇(25∶24∶1)(pH>7.8)混匀,室温离心后取上清;加入1/10体积的3 mol/L乙酸钠(pH5.2)和2.2倍体积的无水乙醇,混匀;室温离心后,弃去上清,加入1 mL冰预冷的70%乙醇洗涤DNA后,室温离心,弃去上清;室温干燥后用30 μl 1×Tris-EDTA(pH7.6)重悬DNA,然后-20℃保存备用;用紫外分光光度计测定OD260/OD280,估计DNA纯度和浓度,比值控制在1.8~2.1范围较理想,-20℃保存备用。
1.2.2 PCR扩增 针对白色念珠菌25S rDNA转座内含子保守序列分型,特异性引物参考文献〔3〕设计。特异性引物:CA-INT-R(5′-CCTTGGCTGTGGTTTCGCTAGATAGTAGAT-3′)和 CA-INT-L(5′-ATAAGGGAAGTCGGCAAAATAGAT-CCGTAA-3′)(上海生工生物工程公司合成)。PCR反应条件参照2×Taq PCR MasterMix扩增试剂盒(购自北京天根生化科技有限公司)进行,反应总体积为25 μL,1 μL模板(<1 μg),1 μl CA-INT-R(10 μM),1 μl CA-INT-L(10 μM),12.5 μL 2×MasterMix,ddH2O补至25μL。PCR反应循环的设置:94℃预变性3min,94℃30 s,55℃30 s,72℃1 min,共30个循环,72℃延伸5 min。扩增产物用1%琼脂糖凝胶电泳,溴化乙锭染色,凝胶成像系统照相分析,根据扩增产物中450 bp和840 bp的有无将白色念珠菌分为3种基因型,用SPSS17.0统计学软件对2组人群口腔白色念珠菌基因型别进行χ2检验。
2 结果
2.1 PCR扩增结果 经CA-INT特异性引物PCR方法扩增后,2组人群的口腔白色念珠菌均可扩增出450 bp、840 bp或二者皆有的片段,根据PCR扩增产物中450 bp和840 bp的有无将两组人群的口腔白色念珠菌基因型分为3型:A型(450 bp),B型(840 bp),C型(450 bp和840 bp)。见图1。2组人群口腔白色念珠菌CA-INT引物PCR分型结果,见表1。2组人群均以A型白色念珠菌最为多见,标准株ATCC10231也属于A型,另外B型次之以及C型较少。
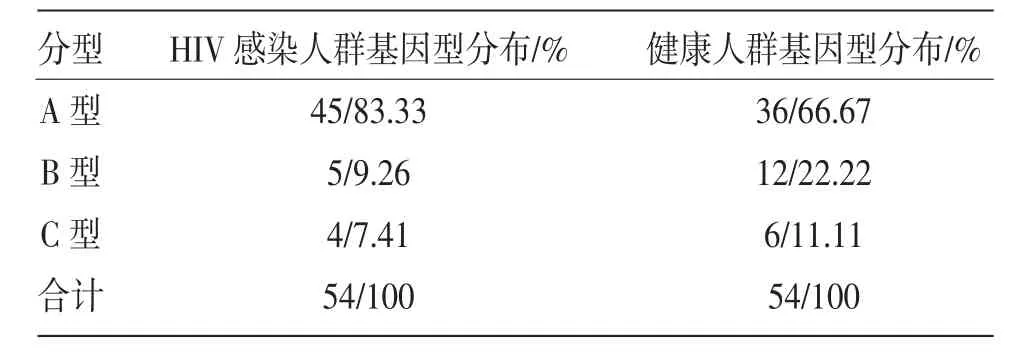
表1 两组人群口腔白色念珠菌CA-INT引物PCR分型结果

图1 白色念珠菌25Sr DNA-PCR的部分图谱
2.2 统计分析 HIV感染人群口腔白色念珠菌中A型所占构成比大于健康人群口腔白色念珠菌中A型所占构成比(表1),经χ2检验,P<0.05,差别有统计学意义;而HIV感染人群口腔白色念珠菌中B型所占构成比小于健康人群口腔白色念珠菌B型所占构成比,经χ2检验,P>0.05,差别无统计学意义;HIV感染人群口腔白色念珠菌中C型所占构成比小于健康人群口腔白色念珠菌C型所占构成比,经χ2检验,P>0.05,差别无统计学意义。HIV感染人群和健康人群的口腔白色念珠菌基因型别分布经SPSS17.0 χ2检验结果显示:χ2=4.282,P=0.118,按P=0.05水准,两组间A、B和C基因型别分布差异无统计学意义。
3 讨论
随着白色念珠菌感染在临床病例中不断增加,耐药现象逐渐突出,对白色念珠菌病流行病学及合理选择抗真菌药物的研究显得十分重要。近年来,PCR分型方法已被广泛的应用于对念珠菌间的DNA核苷酸序列的差异进行基因分析研究,用于临床分离株快速分型,追踪念珠菌感染的传染源,明确传播途径,防止爆发流行,确定同一患者反复感染是源于复发或再感染等研究,并表现出简易、快速、可靠,重复性好等优点〔4〕。运用CA-INT引物PCR法是进行白色念珠菌基因分型的方法之一,CA-INT引物是针对白色念珠菌的可换位基因内区(CA25SRRN)设计的特异引物,在白色念珠菌25S rDNA的CA25SRRN插入一个大小为379 bp的基因,即可扩增出840 bp片段(B组),没有插入该基因则扩增450 bp片段(A组),两种片段都有则扩增840 bp片段和450 bp片段(C组),后者可能是真菌有性生殖的结果,此方法设计科学合理、简便快速、结果稳定、准确可靠,适合临床推广应用〔3,5〕。本文采用CA-INT PCR基因分型方法,对54株HIV感染人群和54株健康人群口腔白色念珠菌进行基因型分析,发现2组人群口腔白色念珠菌均可扩增出清晰而稳定的条带,均具有A、B和C型,均以A型最多见,B型次之,C型最少,3种基因型的分布情况与文献〔6-7〕报道一致。本研究结果显示HIV感染人群口腔白色念珠菌A型所占构成比要高于健康人群,其原因可能由于HIV感染人群口腔与健康人群口腔局部微生态环境不同,影响了白色念珠菌的寄生环境而致寄生菌株发生差异,确切的原因有待进一步研究。武有聪〔2〕等采用随机引物P2-RAPD方法对同一组HIV感染人群的口腔白色念珠菌进行基因型研究,将其分为11个基因型,而本研究采用特异性INT引物法将这些白色念珠菌分成3个基因型,可见随机引物RAPD法分辨率高于特异性INT引物法,但特异性INT引物分型法所扩增的白色念珠菌基因条带清晰而稳定,检测成功率为100%,故认为CA-INT引物PCR法结果稳定、准确可靠、直观。因此,特异性CA-INT引物PCR法是较好的白色念珠菌种内菌株间鉴定的方法,可用于白色念珠菌基因型与表型关系研究,对确定口腔念珠菌病的复发和再感染及其分子流行病学研究均具有重要意义。
〔1〕Fidel PL Jr.Candida-Host interactions in HIV disease:implications for oropharyngeal candidiasis〔J〕.Adv Dent Res,2011,23(1):45-49.
〔2〕武有聪,白丽,袁有华,等.HIV相关性口腔念珠菌RAPD多态性分析〔J〕.微生物学通报,2009,36(5):728-734.
〔3〕Nawrot U,Pajaczkowska M,Wtodarczyk K,et al.rDNA-based genotyping of clinical isolates of Candida albicans〔J〕.Pol J Microbiol,2010,59(3):213-216.
〔4〕JewtuchowiczVM,MujicaMT,BruscaMI,etal.Phenotypic and genotypic identification of Candida dubliniensis from subgingival sites in immunocompetent subjects in Argentina〔J〕.Oral Microbiol Immunol,2008,23(6):505-509.
〔5〕Hattori H,Iwata T,Nakagawa Y,et al.Genotype analysis of Candida albicans isolates obtained from differentbody locations of patients with superficial candidiasis using PCRs targeting 25S rDNA and ALT repeat sequences of the RPS〔J〕.J Dermatol Sci,2006,42(1):31-46.
〔6〕Nawrot U,Skala J,Wlodarczyk K,et al.Proteolytic activity of clinical Candida albicans isolates in relation to genotype and strain source〔J〕.Pol J Microbiol,2008,57(1):27-33.
〔7〕Jayaguru P and M.Raghunathan.Group I intron renders differential susceptibility of Candida albicans to bleomycin〔J〕.Mol Biol Rep,2007,34(1):11-17.
rDNA Genotyping of Oral Candida albicans Isolated from HIV-infected Population
ZHANG Yuncao,LIU Qi,WU Youcong,GUO Lijun,SHEN Yuanying,BAI Li*
(School of Basic Medicine,Dali University,Dali,Yunnan 671000,China)
Objective:To investigate the distribution of genotypes of oral Candida albicans isolated from HIV-related group and healthy carriers group.Methods:PCR with CA-INT primers was designed to amplify group 1 intron-containing region in 25S rDNA of oral Candida albicans,the different strains of oral Candida albicans were classified into genotypes on the basis of bands from amplicons.Then the genotypic distribution results of all oral Candida albicans were analyzed with SPSS 17.0 by chi-square test. Results:One hundred and eight strains of oral cavities Candida albicans were classified into genotype A,B and C.Genotype A was the most predominant one.Identical genotypes distribution appeared in both groups.But the constituent ratio of genotype A of oral Candida albicans strains in HIV-positive group was statistically higher compared to the healthy people group.Conclusion:Genotypes of oral Candida albicans in both groups were highly polymorphic.The method in text is suitable to apply in genotyping research of oral Candida albicans.
Candida albicans;HIV;PCR;rDNA genotyping
R378.99[文献标志码]A[文章编号]1672-2345(2011)08-0024-03
国家自然科学基金资助项目(C30260005);大理学院高层次人才基金资助项目(KY430440)
2011-06-09
张云操,硕士研究生,主要从事病原生物学研究.
*通信作者:白丽,教授.
(责任编辑 董 杰)

