利用正交设计优化番茄SRAP-PCR反应体系
郭彩杰 侯丽霞 崔 娜 韩明利
(1山东省农业科学院蔬菜研究所,山东省设施蔬菜生物学重点实验室,国家蔬菜改良中心山东分中心,山东济南 250100;2沈阳农业大学生物科学技术学院,辽宁沈阳 110161)
利用正交设计优化番茄SRAP-PCR反应体系
郭彩杰1,2侯丽霞1*崔 娜2*韩明利2
(1山东省农业科学院蔬菜研究所,山东省设施蔬菜生物学重点实验室,国家蔬菜改良中心山东分中心,山东济南 250100;2沈阳农业大学生物科学技术学院,辽宁沈阳 110161)
以番茄耐低温材料抗寒0号和不耐低温番青的 F2为材料,利用正交试验设计对 SRAP-PCR反应体系中的5因素(模板DNA、引物浓度、Mg2+浓度、dNTPs浓度、Taq DNA聚合酶)在4个水平上进行正交优化试验。结果表明:各因素水平变化对反应体系影响的大小依次为:引物>Taq DNA聚合酶>dNTPs>模板DNA>Mg2+。建立番茄耐低温SRAP-PCR的20 μL最佳反应体系为:模板DNA为15 ng、引物浓度0.75 μmol·L-1、Mg2+浓度2.0 mmol·L-1、dNTPs浓度0.125 mmol·L-1、Taq DNA 聚合酶1.0 U。
番茄;正交设计;优化;SRAP
相关序列扩增多态性(squence-related amplified polymorphism,SRAP)是由美国加州大学蔬菜作物系Li和Quirost(2001)提出的一种基于PCR技术的新的DNA分子标记技术。SRAP具有简单、高效、高共显性、重复性好、易测序等优点,尤其可以检测基因的可译读码框(ORFs)区域,从而提高了扩增结果与表现型的相关性。目前SRAP标记已开始在植物种质资源鉴定评价、种缘进化关系、遗传图谱构建、基因定位、重要性状标记以及比较基因组学方面得到成功应用。
番茄(Lycopersion esculentumMill.)是一种喜温蔬菜,最适宜的生长温度是15~30 ℃。低温严重影响番茄的品质和产量。番茄的耐低温性是由多个基因控制的非常复杂的数量性状,也受环境因素的影响(Kharti et al.,1994)。近年来,对于番茄抗冷性状的选择正由传统的表型选择向DNA分子水平的基因直接选择发展。本试验采用正交试验设计的方法,建立番茄耐低温SRAPPCR的最优反应体系,为番茄耐低温分子标记及分子育种提供可靠的理论依据。
1 材料与方法
1.1 材料
据2007年露地栽培耐低温试验,抗寒0号和番青同时播种、定植,抗寒0号生长正常,番青全部死亡;据低温发芽试验,抗寒0号发芽正常,番青不发芽,表明抗寒0号耐低温,番青不耐低温。抗寒0号(♀)与番青(♂)由山东省农业科学院蔬菜研究所自主选育,将二者的F2于2009年9月播种在山东省农业科学院蔬菜研究所试验基地日光温室中,幼苗生长4~5片叶时取样,提取叶片基因组DNA。Mg2+、dNTPs、TaqDNA聚合酶均购自宝生物工程(大连)有限公司,引物由上海生工生物工程技术服务有限公司合成。P1:5′TGAGTCCAAACCGGATA3′和P2:5′GACTGCGT ACGAA TTTGC3′为本试验正交试验设计方案中的固定引物。
1.2 方法
1.2.1 DNA的提取 快捷型植物基因组DNA提取系统购自天根生化科技(北京)有限公司,紫外分光光度计及1.5 %琼脂糖凝胶检测其纯度和浓度,并稀释到15 ng·L-1备用。
1.2.2 正交试验设计方案 采用正交设计L16(45)进行试验,设计模板DNA、引物、Mg2+、dNTPs、TaqDNA聚合酶等5因素4水平(表1),3次重复。PCR反应体系20 μL,程序为:95 ℃5 min,94 ℃1 min,35 ℃1 min,72 ℃1 min,5个循环;94 ℃1 min,50 ℃1 min,72 ℃1 min,40个循环,72 ℃10 min,-4 ℃保存。产物用1.5 %琼脂糖凝胶电泳检测,拍照,Bandscan软件分析图像,根据条带数目和背景依次给16个反应打分,条带最多的记为16,最少的记为1,3次重复分别计分(谢云海 等,2005)(表2),将得分的平均值输入DPS V7.05软件进行统计分析,分别得出5因素对PCR试验影响大小的值。
1.2.3 SRAP-PCR优化体系的验证 采用SRAP-PCR已优化的反应体系,用14对引物(表4)对耐低温材料抗寒0号、不耐低温材料番青DNA进行扩增,以验证该体系的效果。
2 结果与分析
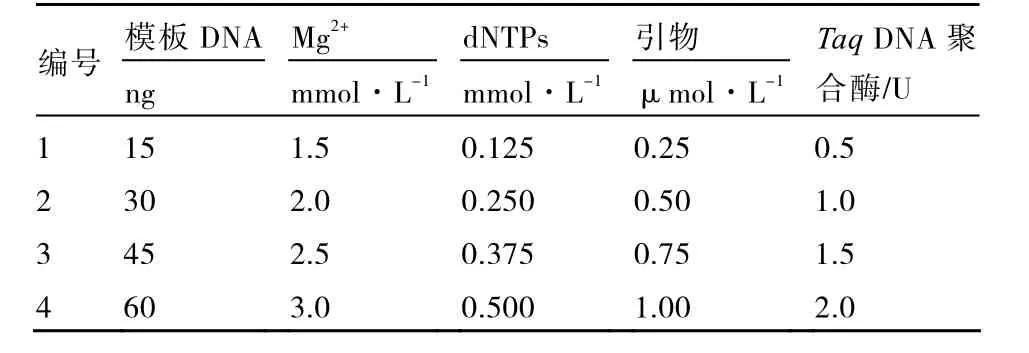
表1 PCR体系的因素水平
2.1 电泳结果评分
按照表2设计的16个反应进行PCR试验,电泳的检测结果如图1所示,得分如表2所示,取3次重复得分平均值在假设不存在交互作用下进行直观分析。求各列各水平得分平均值的和(T1、T2、T3和 T4),各水平的平均值(、、和),用各列最大平均值减去最小平均值得极差R(表3),极差越大说明该因素对反应结果影响越大。分析极差R可知,引物对反应结果影响最大,Mg2+对反应结果影响最小,各因素对反应结果的影响大小依次:引物>TaqDNA聚合酶>dNTPs>模板DNA>Mg2+。
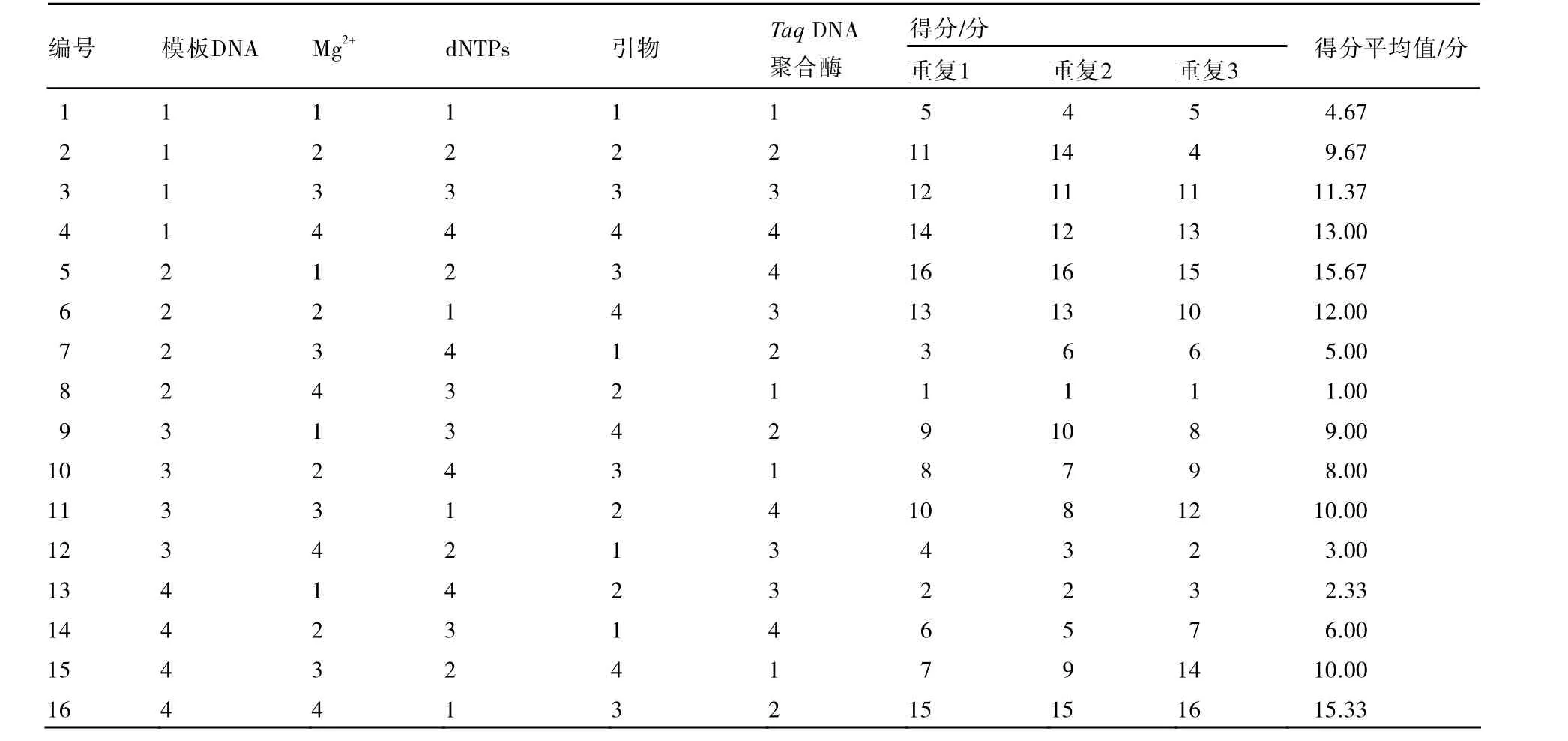
表2 PCR正交试验设计表L16(45)
2.2 各因素对PCR结果的影响

2.2.1 引物浓度对PCR结果的影响 PCR反应中引物浓度影响PCR扩增特异性效果(表3),PCR引物浓度在0.25 μmol·L-1和0.50 μmol·L-1时对PCR结果的影响差异不大,PCR反应得分平均值均较低(、),0.75 μmol·L-1水平与各水平差异较大。表明0.75 μmol·L-1为体系引物浓度的最佳条件。
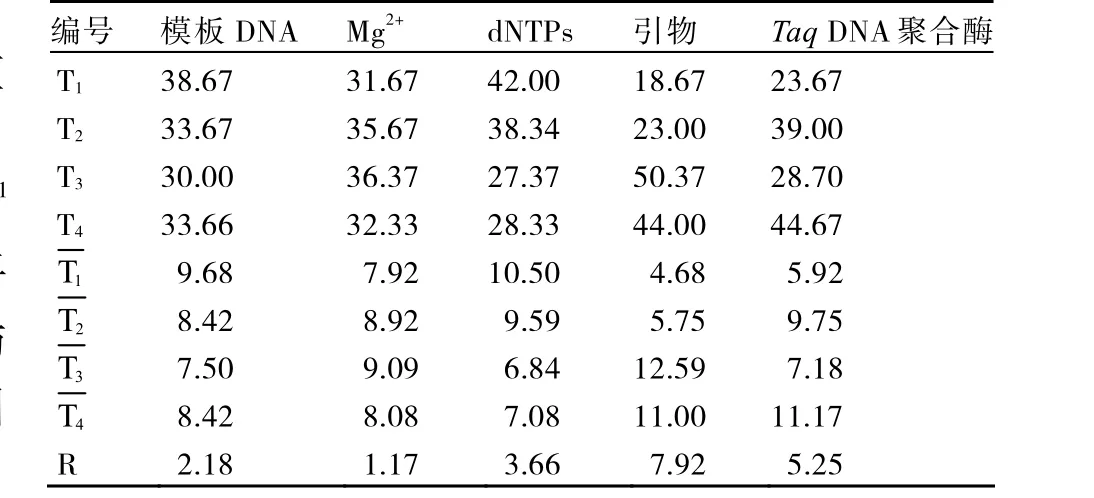
表3 PCR正交试验得分统计分析结果

表4 SRAP引物组合序列
2.2.2TaqDNA聚合酶对PCR结果的影响TaqDNA聚合酶量是影响PCR反应的一个重要因素(表3)。本试验体系中TaqDNA聚合酶量为0.5 U时,PCR反应得分平均值偏低;TaqDNA聚合酶量为1.0 U和2.0 U时,PCR反应得分平均值(和)相差不大。TaqDNA聚合酶价格较高,在确保稳定的情况下,选择TaqDNA聚合酶量1.0 U为最佳反应条件。
2.2.3 dNTPs对PCR结果的影响 dNTPs是PCR的反应原料,一般浓度范围为0.05~0.20 mmol·L-1。本试验中随着dNTPs浓度的增高,PCR反应得分平均值呈下降趋势,dNTPs最佳浓度为0.125 mmol·L-1(表3)。

图1 PCR产物电泳图
2.2.4 模板DNA对PCR结果的影响 模板DNA的用量和纯度对PCR的结果有着重要的作用,它对产物的产量和特异性都有影响。本试验中,随着模板DNA浓度的升高,PCR反应得分平均值呈下降趋势(表3)。在保证扩增条带清楚的条件下,应尽量减少模板DNA的用量。所以本试验中模板DNA用量15 ng应为最佳反应量。
2.2.5 Mg2+浓度对PCR结果的影响 Mg2+浓度对PCR扩增的特异性和产量有明显的影响。随着Mg2+浓度增大,PCR反应得分平均值逐渐升高,当Mg2+浓度超过2.5 mmol·L-1时,得分平均值开始下降,Mg2+浓度在2.0、2.5 mmol·L-1时,PCR反应得分平均值相差不大(表3),从节约的角度选择2.0 mmol·L-1Mg2+作为体系的最佳浓度。
2.3 SRAP-PCR优化反应体系的验证结果
采用SRAP-PCR已优化的20 μL反应体系(模板DNA为15 ng、引物浓度0.75 μmol·L-1、Mg2+浓度2.0 mmol·L-1、dNTPs浓度0.125 mmol·L-1、TaqDNA聚合酶1.0 U),以14对引物对番茄不耐低温、耐低温材料用2.0 mmol·L-1的DNA进行扩增,以验证该体系的效果。从图2中可以看出,利用SRAP-PCR优化体系进行 PCR扩增,采用聚丙烯酰胺凝胶电泳对扩增产物进行检测,结果条带清晰、多态性较丰富。

图2 SRAP-PCR产物电泳验证图
3 结论与讨论
刘立军等(2006)认为不同植物的SRAP-PCR反应体系是不同的,建立一个特定试验条件下适合该植物的最佳SRAP-PCR反应体系可为SRAP分子标记提供良好的基础。本试验获得的番茄耐低温SRAP-PCR反应体系中,20 μL的最佳反应体系:模板DNA15 ng、引物浓度0.75 μmol·L-1、Mg2+浓度2.0 mmol·L-1、dNTPs浓度0.125 mmol·L-1、Taq DNA聚合酶1.0 U。此最佳体系与正交设计表中得分最高的编号为5和16的反应大部分相似。王燕等(2007)对番茄基因组DNA的SRAPPCR反应体系进行优化,得到的最佳反应体系为:Mg2+浓度为1.5~3.0 mmol·L-1,模板DNA为10~20 ng(每10 μL体系)、引物浓度为0.25 μmol·L-1、dNTPs浓度为0.05~0.20 mmol·L-1。李晓慧等(2008)对西瓜SRAP-PCR反应体系的优化结果为:20 μL体系中,模板DNA为100 ng、引物浓度60 ng、Mg2+浓度2.0 mmol·L-1、dNTPs浓度0.3 mmol·L-1、Taq DNA聚合酶0.75 U。本试验通过正交试验设计确立了番茄耐低温的SRAP-PCR反应体系中各因素的最佳具体浓度,同西瓜SRAPPCR反应体系相比有一定的差异,表明不同植物间的SRAP-PCR反应体系存在明显差异;同王燕等(2007)对番茄基因组DNA的SRAP-PCR反应体系相比有细微的差异,表明同一作物在不同品种间也会存在不同。番茄耐低温的SRAP-PCR反应体系的确立是在标准SRAP-PCR的基础上增加5个循环,在琼脂糖凝胶电泳上进行的,最佳反应体系存在丰富的扩增条带,此方法简单快捷,为SRAP分子标记的聚丙烯酰胺凝胶电泳提供了依据,利用本优化体系进行PCR扩增,采用聚丙烯酰胺凝胶电泳对扩增产物进行检测,结果是条带清晰、多态性较丰富。此方法也为番茄耐低温育种提供了重要的辅助方法。
谢云海,夏德安,姜静,林萍.2005.利用正交设计优化水曲柳ISSR-PCR反应体系.分子植物育种,3(3):445-450.
王燕,龚义勤,赵统敏,刘广,郁樊敏,叶海龙,柳李旺.2007.番茄SRAP-PCR体系优化与品种分子鉴定.南京农业大学学报,30(1):23-29.
李晓慧,王从彦,徐小利,常高正,张四普.2008.西瓜SRAP-PCR反应程序的建立与体系优化.华北农学报,23(3):38-41.
刘立军,蒙祖庆,邢秀龙,彭定祥.2006 .苎麻基因组SRAP扩增体系的优化研究.分子植物育种,4(5):726-730.
Li G,Quiros C F.2001.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica.Theor and Appl Genet,103(2):455-461.
Kharti V G,Polesskaya L M,Zhakoteh A G.1994.Comparative evaluation of genetic organization of quantitative cold-resistance traits of tomato(Lycopersicon esculentum L. hirsutum)during germination and the early vegetative stage.Genetika,30(4):502-508.
Optimization of SRAP-PCR System for Tomato Based on Orthogonal Design
GUO Cai-jie1,2, HOU Li-xia1*, CUI Na2*, HAN Ming-li2
(1Institute of Vegetables, Shandong Academy of Agricultural Sciences, Key Lab for Biology of Greenhouse Vegetable of Shandong Province, National Center for Vegetable Improvement(Shandong), Jinan250100, Shandong, China;2Biological Science and Technology College, Shenyang Agricultural University, Shenyang110161, Liaoning, China)
The orthogonal design was used to optimize SRAP-PCR amplification system at4 levels of5 factors in tomato(Lycopersion esculentum Mill.)(DNA template, primer, Mg2+, dNTPs and Taq DNA polymerase). The results showed that the order of each factor in different levels affected the result of PCR was: primer>Taq DNA polymerase>dNTPs>DNA template>Mg2+. The most suitable SRAP-PCR reaction system for tomato was total20 μL containing15 ng DNA template,0.75 μmol·L-1primer,2.0 mmol·L-1Mg2+,0.125 mmol·L-1dNTPs and1.0 U Taq DNA polymerase.
Tomato; Orthogonal design; Optimization; SRAP
S641.2
A
1000-6346(2011)02-0048-05
2010-09-06;接受日期:2010-10-20
国家“863”计划项目(2006AA100108-3-1),辽宁省教育厅科学技术研究项目(2008623)
郭彩杰,女,硕士研究生,主要从事植物发育分子生物学的研究,E-mail:guocaijie@163.com
*通讯作者(Corresponding authors):侯丽霞,博士,副研究员,主要从事番茄育种与分子生物学研究,E-mail:houlx2006@126.com
崔娜,博士,副教授,硕士生导师,主要从事植物发育分子生物学研究,E-mail:syaua@163.com

