湖南甘薯品种遗传多样性的SSR分析
张超凡,黄艳岚,周 虹,易九红
(湖南省作物研究所,湖南 长沙 410125)
简单重复序列(simple sequence repeat,SSR),又称微卫星DNA和短串联重复,可依据微卫星序列两端互补序列设计引物,通过PCR反应扩增微卫星片段,将扩增产物进行凝胶电泳,再根据分离片段的大小决定基因型并计算等位基因频率。SSR标记缺点是引物开发难度大,但由于是共显性标记,具有用量少,数量丰富、重复性好等优点[1]。目前,SSR 标记已广泛应用于水稻[2-4]、小麦[5-6]、玉米[7]、大豆[8]、油菜[9-10]等的遗传多样性分析和图谱构建。本研究利用SSR分子标记对来自湖南31份甘薯品种进行遗传多样性分析及亲缘关系鉴定,目的在于探讨湖南甘薯地方品种与育成品种之间的遗传多样性,为进一步分析湖南甘薯遗传多样性提供可靠的标记手段。
1 材料与方法
1.1 材料
1.1.1 供试材料 本研究所用甘薯材料取自湖南省作物研究所,包括11份育成品种和20份地方品种(表1),均来自于湖南省。
1.1.2 试 剂 SSR引物如表2所示,由北京奥科生物技术有限责任公司合成。Taq DNA聚合酶(2.5 U/mL)和DNA Marker购自天根生化有限公司,其余试剂均为国产分析纯。
1.2 方法
1.2.1 基因组的提取 DNA取顶部展开新鲜叶片3~4片,置已编号保鲜袋中,采用CTAB[11]法对甘薯幼嫩叶片进行基因组DNA提取。
1.2.2 SSR分析 SSR的PCR扩增体系:2μL的10×PCR Buffer,2.5 μL 的 2.0 mM dNTPs,50 ng 甘薯DNA,1U Taq酶,2μL的5 mM引物(对),加ddH2O至20μL。SSR扩增条件为:94℃预变性4min;然后进行以下35个循环:94℃变性30 s,58℃复性30s,72℃延伸1min,最后72℃充分延伸7min。PCR产物加上样缓冲液(98%甲酰胺,10 mmol/L EDTA pH 8.0,0.25%溴酚蓝,0.25%二甲苯腈)变性后自然冷却,用5%变性聚丙烯酰胺凝胶电泳分离,硝酸银染色,并拍照保存。
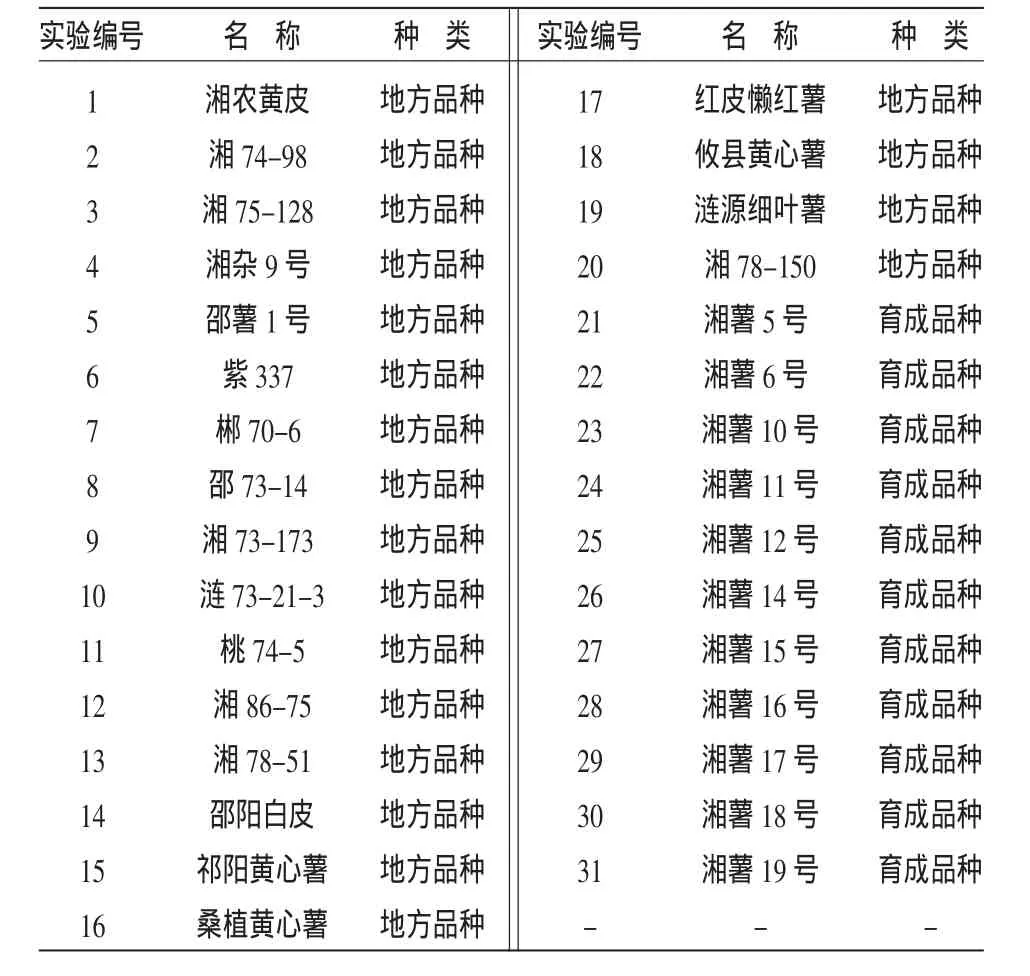
表1 本研究所用的品种名称
1.2.3 统计方法 胶板晾干后在灯箱上读带。统计方法参照文献[12]。
2 结果与分析
2.1 SSR分子标记的多态性分析
对SSR分子标记分析的12对SSR引物进行PCR扩增,引物对SSR11电泳结果如图1所示,共检测出74等位基因位点,每对引物检测出的等位基因位点3~10个,平均为6个。其中引物SSR7扩增的等位基因位点最多,为10个,引物SSR12扩增的等位基因位点最少,为3个。

表2 SSR引物组合及扩增条带
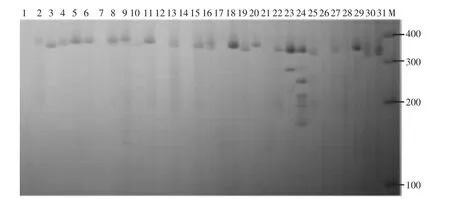
图1 SSR引物组合SSR11对31个甘薯品种的扩增结果
2.2 甘薯品种间的遗传差异
用SSR标记对31份甘薯品种进行了聚类分析,供试材料的遗传距离为0.272 7~0.626 9,平均0.520 4,表明供试材料之间遗传多样性丰富,遗传差异大。根据甘薯SSR标记分析,对供试材料进行UPGMA聚类分析(图2)。从聚类图可以看出,湘78-150与湘薯12号的遗传距离最近。
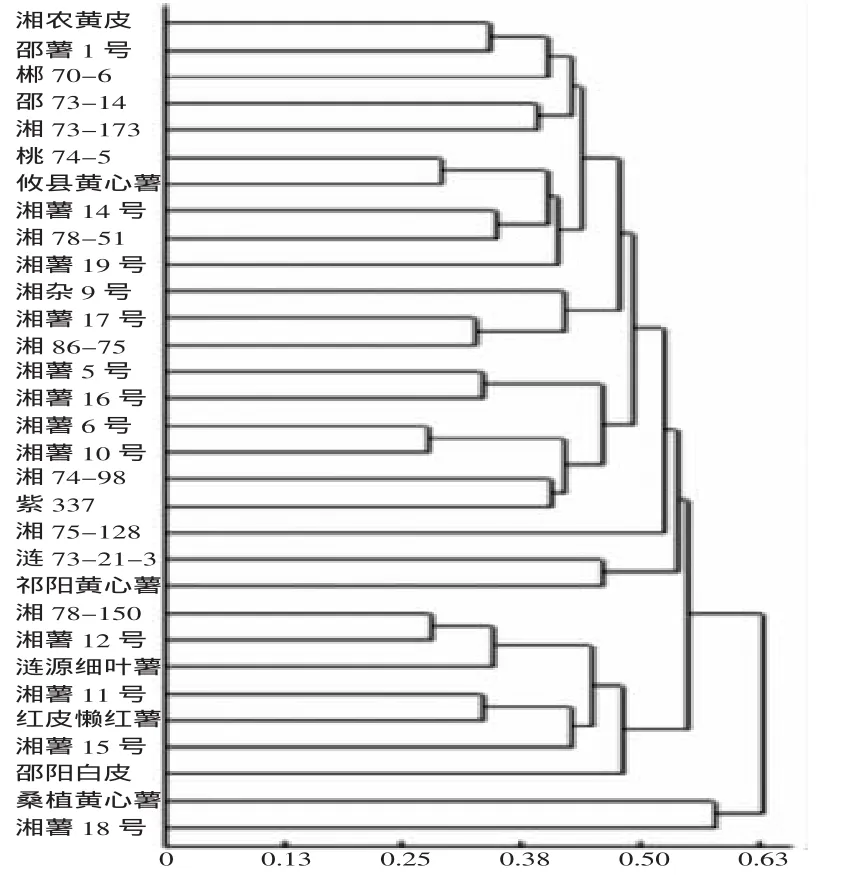
图2 基于SSR标记得到的31份甘薯品种的聚类图
2.3 甘薯地方品种与育成品种遗传差异分析
经过遗传距离计算,20份湖南地方品种遗传距离为 0.288 1~0.814 8,平均 0.524 0;11 份育成品种遗传距离为0.272 7~0.894 7,平均0.511 5。两类材料平均遗传距离大小为GDMC>GDL。湖南甘薯地方品种与湖南育成品种的遗传距离0.288 1~0.7037,平均 0.516 7。
3 讨论
分子标记技术可以不受环境、发育时期、不同器官等的限制,从基因组水平上揭示遗传变异程度。利用SSR标记进行研究时,需要知道研究材料两端的序列信息,开发有一定困难,费用也较高,使得其在甘薯遗传多样性研究中受到限制。研究利用SSR方法分析31份湖南甘薯地方品种与育成品种的遗传多样性,扩增的等位基因位点丰富。聚类图上可以看出湘薯10号与湘薯6号的亲缘关系较近;湘薯17号与湘薯86-75亲缘关系较近,这与它们的系谱图相吻合,说明SSR标记适用于检测甘薯的遗传多样性。
[1] 周延清.DNA分子标记技术在植物研究中的应用 [M].北京:化学工业出版社,2005.
[2] 徐大勇,钟 环,周 峰,等.中粳水稻品种资源的遗传多样性II.黄淮稻区近期育成品种的SSR多样性比较[J].江苏农业学报,2010,26(1):15-21.
[3] 张 彦,郭士伟,何 冰,等.利用SSR标记建立杂交水稻分子指纹图谱数据库[J].江苏农业学报,2006,22(2):181-183.
[4] Navinder S,Neelu J,Sunita J,et al.Assessment of genetic diversity within and among Basmati and non-Basmati rice varieties using AFLP,ISSR and SSR markers[J].Euphytica,2004,140:133-146.
[5] Gupta P K,Rustgi S,Sharma S,et al.Transferable EST-SSR markers for the study of polymorphism and genetic diversity in bread wheat[J].Mol Gen Genomics,2003,270:315-323.
[6] Medini M,Hamza S,Rebai A,et al.Analysis of genetic diversity in Tunisian durum wheat cultivars and related wild species by SSR and AFLPmarkers.Genet.Res[J].Crop Evol,2005,52:21-31.
[7] 孟庆长,陈艳萍,杨兴华,等.利用SSR技术鉴定苏玉20的纯度[J].江苏农业学报,2009,25(3):508-512.
[8] Tae Y H,Takashi S,Masakazu T,et al.High-density integrated linkage map based on SSR markers in soybean[J].DNA Research,2009,16(4):213-225.
[9] 谭祖猛,李云昌,胡 琼,等.SSR和SRAP标记研究油菜杂交骨干亲本的遗传多样性 [J].农业生物技术学报,2009,17(5):882-890.
[10] 张洁夫,戚存和,浦惠明,等.甘蓝型油菜SSR检测体系的优化[J].江苏农业学报,2004,20(4):225-229.
[11] 黄艳岚,张超凡.甘薯叶片基因组DNA提取方法[J].湖南农业科学,2011,(1):4-6.
[12] 张超凡,黄艳岚,周 虹,等.湖南甘薯品种AFLP标记的遗传差异分析[J].江苏农业学报,2010,26(4):706-710.

