盐芥TOR基因的克隆及对盐芥生长发育的影响*
王臻吴春霞扈玉婷张慧赵彦修
(山东师范大学山东省逆境植物重点实验室,山东济南250014)
雷帕霉素靶蛋白(TOR)是存在于真核生物中的一类非常保守的脯氨酸调控的丝氨酸/苏氨酸(Ser/Thr)蛋白激酶家族,属于磷酸肌醇相关激酶(PIKKs)家族成员[1-2].20世纪90年代,科学家首先在酵母菌中发现两种雷帕霉素的靶蛋白TOR1和TOR2[3].1994年,Brown等在哺乳动物细胞内发现了TOR1和TOR2的同源基因,命名为mTOR[4].酵母TOR激酶分子和mTOR分子均由由5个功能区域构成,从氨基端开始依次为:约20个串联的HEAT重复序列、中心区的FAT域、靠近羧基端的FRB域、激酶域和FATC域[5-7].
TOR作为一种重要的调节基因通过调节细胞周期、蛋白质合成、细胞能量代谢等多种途径发挥重要的生理功能,在细胞的增殖、生长、分化过程中起着中心调控点的作用[1-2].在酵母及哺乳动物细胞中,TOR的调节作用主要通过TORC1和TORC2两组TOR复合物完成.其中,TORC1主要调控由生长因子、能量、营养、逆境等引起的细胞生长方面的变化,如RNA翻译、蛋白质合成等;TORC2主要对形成细胞骨架的肌动蛋白起调控作用.
植物的生长和发育具有很强的可塑性,其形态及器官会因环境的变化发生很大的变化.因此,TOR在植物生长发育中所起的作用也越来越受到关注.2002年,法国科学家Menand等[8]在拟南芥中找到了TOR的同源基因,命名为AtTOR(At1g 50030).AtTOR由2 481个氨基酸组成,相对分子质量为279000,也含有mTOR和酵母TOR所包含的几个结构域,其中FRB位于1930-2022,激酶域位于2092-2340,FATC位于2451-2481[8].
拟南芥TOR突变体纯合子无法完成生活史,在胚胎时期就已致死[8].在AtTOR完全被敲除掉的纯合子中,虽然胚细胞也可进行细胞分裂,但却不正常,胚无法形成极性的胚轴.不仅如此,纯合子在胚乳的发育上也有缺陷[8].由此可见,植物的TOR对其生长发育起着举足轻重的作用.Deprost等[9]发现,当拟南芥体内的AtTOR表达下调时,多聚核糖体的积累将大大减少.从这个角度来说,AtTOR在调控蛋白质的合成上,起了一定的作用.
作为一种新型的模式植物,盐芥具有许多优点:个体小,生长周期较短,自花授粉,种子数目多,基因组也不大[10-12],仅为拟南芥的两倍.除此之外,盐芥对各种非生物胁迫的耐受能力远远强于拟南芥.盐芥cDNA和蛋白的表达与拟南芥的同源性很高,分别为90%和95%[10,13].本实验利用分子生物学方法克隆得到盐芥TOR基因(ThTOR),并进行了初步研究.
1 材料与方法
1.1 材料
1.1.1 植物材料
盐芥(Thellungiella halophila)种子采自山东东营,植株为山东师范大学逆境植物重点实验室自行种植.
1.1.2 菌种与质粒
大肠杆菌(Escherichia coli)DH5α、农杆菌(Agrobacterium tumefaciens)GV3101菌株为山东师范大学逆境植物重点实验室保存.
pMD18-T载体为TaKaRa公司产品;pBluescript KS和pCAMBIA3301质粒为山东师范大学逆境植物重点实验室保存,pRT101-INT4为山东师范大学逆境植物重点实验室孙伟老师改造,用于构建RNA干扰(RNAi)框架.
1.1.3 主要培养基与培养条件
LB液体培养基(含相应抗生素)用于培养各种重组菌及DH5α、GV3101等菌株[14].DH5α的摇床培养条件为37℃、220 r/min;GV3101的摇床培养条件为28℃、220 r/min.
LB固体培养基(含相应抗生素)用于培养重组大肠杆菌和农杆菌[14].DH5α的培养条件为37℃恒温培养箱过夜培养,GV3101的培养条件为28℃恒温培养箱培养48h.
1.1.4 主要酶及试剂
TaqDNA聚合酶、限制性内切酶、反转录试剂盒及RACE试剂盒购自宝生物工程(大连)有限公司及INVITROGEN公司,其它试剂和药品为市售分析纯.
1.2 方法
1.2.1 盐芥总RNA的提取及反转录
取盐芥的新鲜叶片,用Trizol法抽提RNA,分别以3'端引物和Oligo dT18为引物按宝生物工程有限公司反转录试剂盒说明书进行互补DNA(cDNA)第1条链的合成.
1.2.2 盐芥TOR基因cDNA序列的获得

表1 克隆ThTOR基因cDNA保守区所用的兼并引物序列Table 1 Degenerate primer sequences used in the cloning of ThTOR cDNA conservative regions

表2 其它克隆ThTOR基因cDNA所用的引物序列Table 2 Other primer sequences used in the cloning of ThTOR cDNA
依据Genbank数据库中已知的植物TOR家族的蛋白保守区域,尤其是盐芥近缘物种拟南芥的基因序列设计扩增兼并引物(见表1),以反转录获得的cDNA为模板,进行聚合酶链式反应(PCR),扩增盐芥TOR的保守片段.经克隆测序后以保守区序列为模板设计引物(见表2),扩增盐芥TOR的非保守区.通过cDNA末端快速扩增技术(RACE)获得盐芥TOR的两端序列,3'RACE按照宝生物工程公司试剂盒要求进行,5'RACE按照INVITROGEN公司5'RACE试剂盒进行.两次RACE的扩增产物测序后,与其它序列拼接后获得ThTORcDNA的全长序列.
1.2.3 盐芥TOR基因全长序列的获得
采用改良的十六烷基三甲基溴化铵(CTAB)法提取盐芥新鲜叶片的基因组DNA,根据已获得的ThTORcDNA的开放阅读框(ORF)设计引物(见表3).使用宝生物工程公司的DNA聚合酶(Pyrobest®DNA Polymerase),具体方法参照宝生物工程公司说明书.PCR产物经凝胶回收纯化后与pBluescript KS载体连接,转化DH5α感受态细胞后进行测序.将所有测序所得序列拼接后获得盐芥TOR基因组(ThTOR)的全长序列.
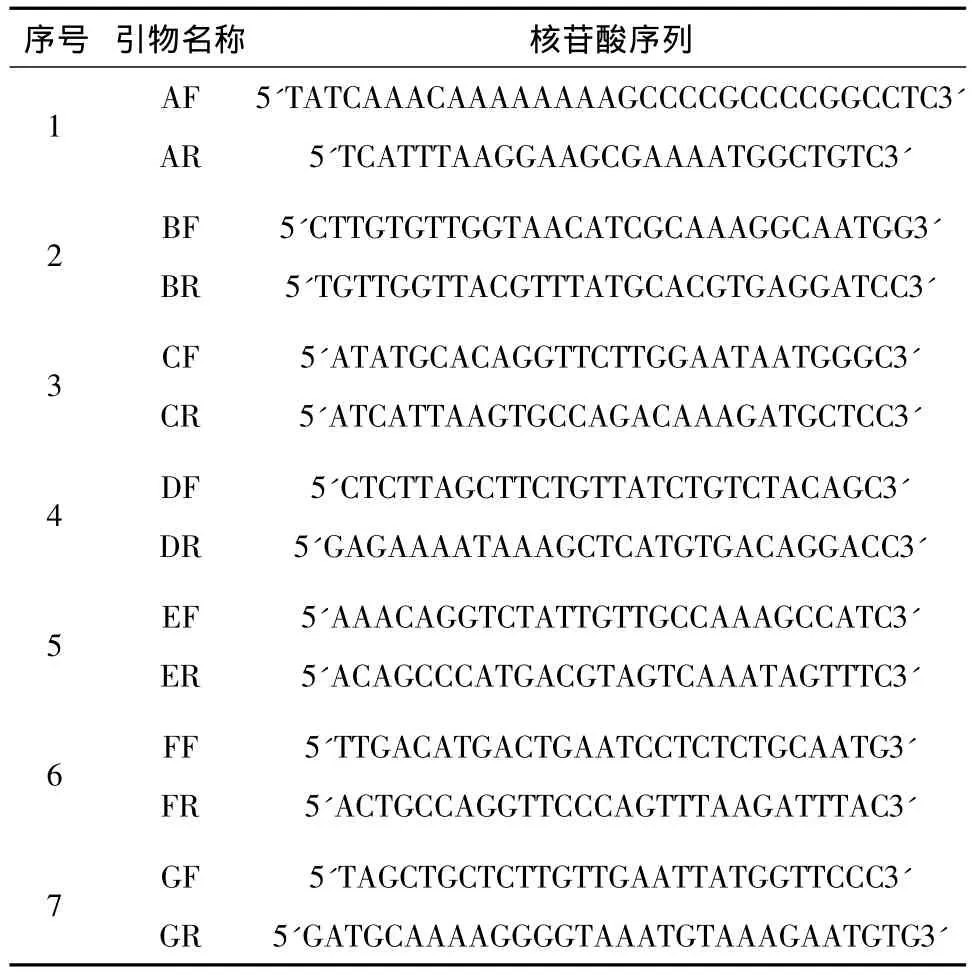
表3 克隆ThTOR基因组所用引物序列Tabal 3 Primer sequences used in the cloning of ThTOR genomic
1.2.4 盐芥TOR转基因沉默株系的获得
选择ThTOR基因FAT域约450 bp的特异性强的序列设计引物,用于构建盐芥TORRNA干扰沉默载体(ThTOR-RNAi).在引物序列中引入合适的酶切位点,正向引物为5'TGAATTCTTTACCGTTTGATCCAGAAGTATCAC3',反向引物为5'ATATCCCGGGACTGCAGAAACAACG3'.以pRT101-INT4载体为中间载体构建RNAi表达框架(见图1),成功后,再用合适的酶克隆到植物表达载体pCAMBIA3301上.构建成功的ThTOR-RNAi植物表达载体转化农杆菌GV3101,挑取阳性克隆,利用花浸染法转化盐芥.
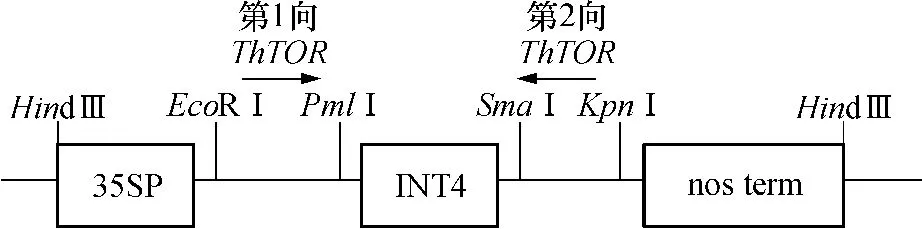
图1 ThTOR-RNAi框架Fig.1 Frame of the ThTOR-RNAi
T0幼苗出苗2~3周后,用0.1%(体积分数)的Basta喷洒筛选,转基因植株由于具有除草剂抗性得以存活.用改良CTAB法提取抗性植株的叶片总DNA,以ThTOR基因沉默表达载体DNA为阳性对照,野生型植株和无菌双蒸水为阴性对照,对基因沉默的目的片段进行PCR检测.PCR检测阳性的抗性植株为转基因株系.
1.2.5 盐芥TOR转基因沉默株系的性状分析
将转基因株系用实时定量PCR的方法进一步确定ThTOR在沉默株系中的表达量.根据ThTOR的cDNA全长序列设计特异正向引物5'TCAATGAAGTCCCTCAATTAGCCTTGCTTG3'和反向引物5'CCAAGCATATTCACAGCCTGAAGAATTTCC3'.分析中选用表达相对稳定的盐芥肌动蛋白7的基因(actin7)作为内标基因.根据基因的保守序列(从山东师范大学逆境植物重点实验室盐芥cDNA文库获得)设计正向引物5'AAATACAGTGTCTGGATCGGAGGATCTATC3'和反向引物5'TAACAAACTCACCACCACGAACAAGAGAAG3'.
本实验采用的实时定量PCR方法是相对定量方法,是使用内源性的对照来校正样品的加样量,通过与对照样品相比较计算未知样品的量.PCR反应中,在其它参数恒定的反应条件下,产物开始进入指数期增长的循环数(CT)只与反应体系中原始模板的量有关,通过2-ΔΔCT计算将统计数据转化成线性形式.其中ΔCT表示目标基因和内标基因CT值的差异,ΔΔCT表示纠正过加样误差后不同处理株系与对照株系目标基因CT值的差异,2-ΔΔCT则表示不同处理株系与对照株系目标基因表达的相对变化.根据定义,对照株系中目标基因的表达量为1,而相对于对照株系,处理株系中目标基因表达的倍数为2-ΔΔCT.
根据检测结果,选用ThTOR基因表达量下调的株系.将野生型盐芥种子与转基因盐芥种子同时用水浸泡于4℃2~3周,取出均匀播种于培养土(育苗介质、蛭石、珍珠岩体积比为4∶3∶1)中,温度为18~22℃,16h光照,8h黑暗.植株生长至有4-6片真叶时,将其移入装满培养土直径约7 cm的小盆中.
2 结果与分析
2.1 ThTOR cDNA全长的克隆与分析
以盐芥cDNA为模板,分别用保守区兼并引物及非保守区引物(如表1、2所示)进行PCR反应,将PCR产物回收克隆后进行测序,通过序列拼接获得含有一个完整编码区的基因序列,命名为ThTOR,Genbank登录号为HQ420260.所获得的ThTOR基因的cDNA全长7836bp,ORF为7437 bp,5'非编码区为196bp,3'非编码区为203 bp,与拟南芥TORcDNA的同源性高于95%.全序列的第197-199个碱基编码起始密码子M,7634-7636处的TGA为终止密码子,编码2 479个氨基酸,相对分子质量为279000.
2.2 ThTOR基因组全长的克隆与分析
以盐芥基因组DNA为模板,用表3中的引物进行PCR扩增,得到的特异条带经测序后拼接得到ThTOR基因组序列全长.ThTOR基因组DNA长度为19162bp,其中有55段内含子和56段外显子,Genbank登录号为HQ420259.拟南芥AtTOR基因组DNA长度为17555 bp,比盐芥少了约2 kbp,但其外显子片段个数与盐芥相同.盐芥与拟南芥外显子在DNA序列中的位置比较如表4所示.由表4可以看出,内含子及外显子的分布在拟南芥及盐芥中都较为均匀,没有较大差异.
2.3 ThTOR推导的蛋白序列的分析
ThTOR蛋白序列由2 479个氨基酸组成,经过分析发现,ThTOR与AtTOR的蛋白序列仅有63个氨基酸发生变化,同源性高达97.5%.表5示出了ThTOR与AtTOR中差异氨基酸的位置及其突变类型.ThTOR具有TOR基本的结构域,FAT域位于第1309-1887个氨基酸;FRB域位于第1920-2023个氨基酸;激酶域位于第2 090-2 340个氨基酸;FATC位于第2447-2479个氨基酸.
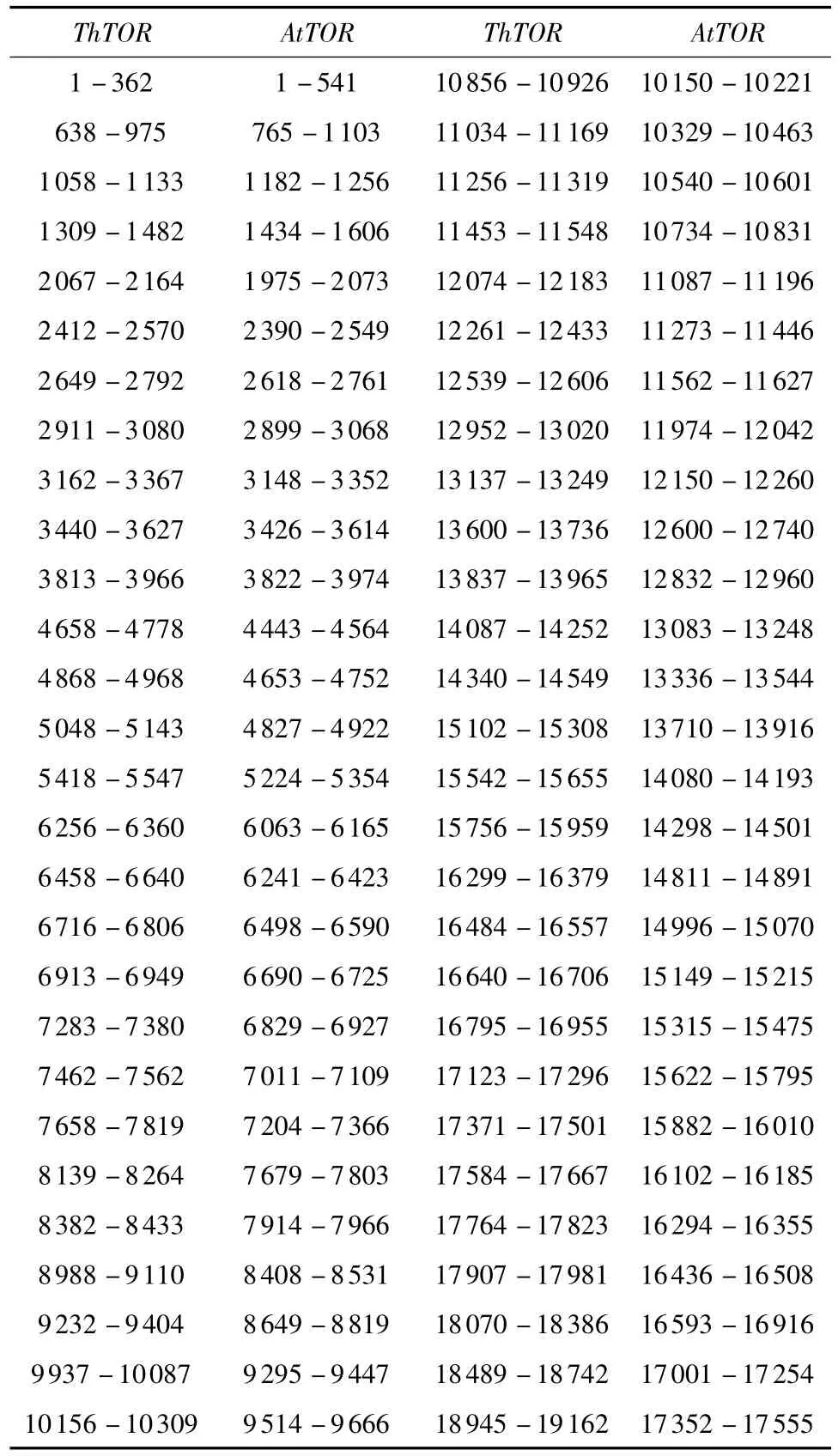
表4 盐芥ThTOR与拟南芥AtTOR外显子位置的比较Table 4 Exon location comparison between ThTOR and AtTOR
2.4 ThTOR-RNAi转基因株系的获得与鉴定
2.4.1 盐芥ThTOR-RNAi植物表达载体的构建
由PCR获得用于RNAi的ThTOR基因片段(约450bp),测序后与已获得的ThTORcDNA进行比对,结果完全一致.
将构建好的ThTOR-RNAi表达框架克隆入植物表达载体,重组质粒命名为3301-THCM.
2.4.2 盐芥ThTOR-RNAi转基因株系的鉴定
提取Basta初步筛选的抗性植株的DNA,用PCR方法对ThTOR基因的RNAi片段进行检测,检测结果为阳性即证明抗性植株中已成功转入RNAi表达框架,PCR检测结果见图2.水阴性对照及野生型盐芥中均未扩增出ThTOR-RNAi片段,而转基因植株中均扩增出与阳性对照大小一致的片段.
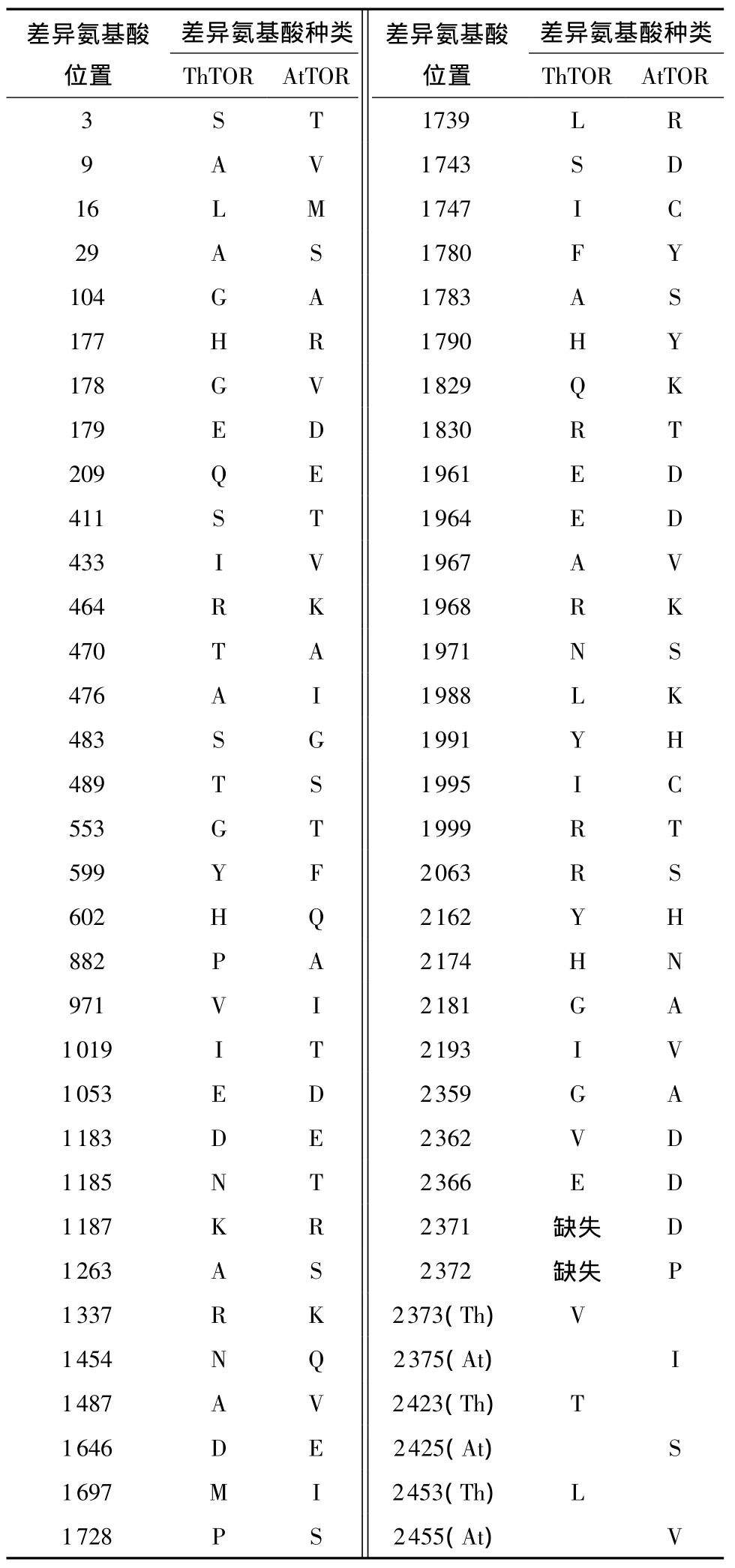
表5 ThTOR与AtTOR差异氨基酸的比较Fig.5 Comparison of differentamino acids between ThTOR and AtTOR
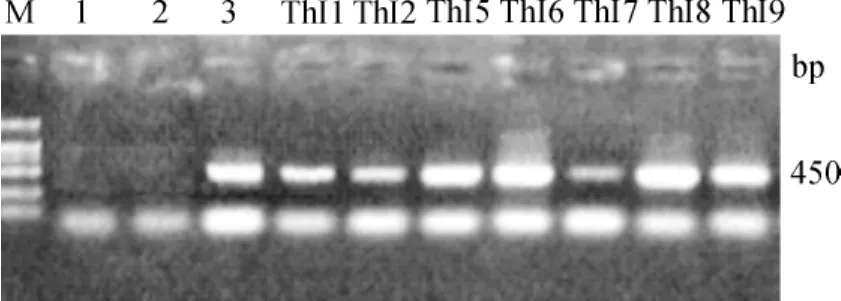
图2 部分转基因株系的PCR鉴定Fig.2 PCR identification of some transgenic plants
2.5 盐芥ThTOR-RNAi转基因株系ThTOR基因的表达及表型分析

图3 ThTOR-RNAi转基因植株与野生型植株的生长情况Fig.3 Growth of wild type and the ThTOR-RNAi transgenic plants

图4 ThTOR-RNAi转基因植株与野生型植株果荚的生长情况Fig.4 Infruit development of wild type and the ThTOR-RNAi transgenic plants
2.5.1 盐芥ThTOR-RNAi转基因株系中ThTOR基因的表达分析
本实验把野生型盐芥(wt)作为对照样品(本研究中所有检测及照片涉及到的野生型均为同一株系).通过实时定量PCR,共对3个转基因沉默株系ThI1、ThI5、ThI6的mRNA表达水平进行了分析.野生型和转基因株系ThI1、ThI5、ThI6的2-ΔΔCT值分别为1、0.8060、0.5873、0.7457.可见,3个转基因沉默株系的ThTOR表达水平均有不同程度的降低.其中,ThI5和ThI6降低更为显著.
2.5.2ThTOR对盐芥的生长及果荚发育的影响
根据实时定量PCR的分析结果,选取ThTOR下调明显的ThI5和ThI6与生长发育时间一致的同一株野生型盐芥相比,见图3、4.由图3可知,ThTOR基因沉默株系的生长明显缓慢,野生型盐芥种子已处于成熟状态,而转基因沉默株系仅处于抽苔前期.由图4可知,ThI5和ThI6这两个株系的盐芥植株的果荚有明显败育现象.跟野生型相比,ThTOR基因沉默盐芥春化开花后,无法正常形成果荚,或形成的果荚发育不全,不饱满,种子数量也远远小于野生型.
3 结论
AtTOR和ThTOR的蛋白序列同源性很高,达97.5%,cDNA序列的同源性也高于95%.结果表明,尽管ThTOR的基因组序列比AtTOR的基因组序列多了近2 kbp,但两个物种的内含子及外显子分布都较为均匀,没有明显的差异.ThTOR基因沉默株系具有明显的生长迟缓和败育的现象.说明ThTOR
对盐芥的生长发育,尤其是盐芥的生殖器官,起重要作用.这一现象与拟南芥的研究结果相一致.
[1]Harris T E,Lawrence JC.Tor signaling[J].Science Signaling:Signal Transduction Knowledge Environment,2003,2003(212):1-17.
[2]Jacinto E,Hall M N.Tor signalling in bugs,brain and brawn[J].Nature Review Molecular Cell Biology,2003,4(2):117-126.
[3]Kunz J,Henriquez R,Schneider U,et al.Target of rapamycin in yeast,TOR2,is an essential phosphatidylinositol kinase homolog required for G1 progression[J].Cell,1993,73(3):585-596.
[4]Brown E J,Albers M W,Shin T B,et al.A mammalian protein targeted by G1-arresting rapamycin-receptor complex[J].Nature,1994,369(6483):756-758.
[5]Schmelzle T,Hall M N.TOR,a central controller of cell growth[J].Cell,2000,103(2):253-262.
[6]Asnaghi L,Bruno P,Priulla M,et al.mTOR:a protein kinase switching between life and death[J].Pharmacological Research,2004,50(6):545-549.
[7]Sarbassov D D,Guertin D A,Ali SM,et al.Phosphorylation and regulation of Akt/PKB by the rictor-mTOR complex[J].Science,2005,307(5712):1098-1101.
[8]Menand B,Desnos T,Nussaume L,et al.Expression and disruption of theArabidopsisTOR(target of rapamycin)gene[J].Proceedings of the National Academy of Sciences,2002,99(9):6422-6427.
[9]Deprost D,Yao L,Sormani R,et al.TheArabidopsisTOR kinase links plant growth,yield,stress resistance and mRNA translation[J].European Molecular Biology Organization Reports,2007,8(9):864-870.
[10]Bressan R A,Zhang C,Zhang H.etal.Learning from the arabidopsis experience.the next gene search paradigm[J].Plant Physiology,2001,127(4):1354-1360.
[11]Xiong L,Zhu JK.Molecular and genetic aspects of plant responses to osmotic stress[J].Plant Cell and Environment,2002,25(2):131-139.
[12]Amtmann A,Bohnert H J,Bressan R A.Abiotic stress and plant genome evolution.search for new models[J].Plant Physiology,2005,138(1):127-130.
[13]Zhu JK.Plant salt tolerance[J].Trends in Plant Science,2001,6(2):66-71.
[14]Sambrook J,Russell DW.分子克隆实验指南[M].3版.黄培堂,译.北京:科学出版社,2002:1595.

