申克孢子丝菌菌丝相和酵母相cDNA消减文库的构建及差异表达基因的筛选*
周 汛,杨致邦,肖异珠
申克孢子丝菌为一种双相型真菌,25℃表现为菌丝相,37℃为酵母相,其酵母相可在感染者体内组织增殖,形成小的芽生孢子而致病。申克孢子丝菌的双相性表明该菌不同菌相存在差异表达基因,在不同的环境条件下,通过菌体信号转导的调控,不同的基因表达,形成酵母相或菌丝相。目前申克孢子丝菌双相转换的相关研究仅涉及少数几个基因[1-4],其双相转换的分子机制尚未完全明了,研究申克孢子丝菌双相转换机制对揭示其发病机理有重要意义。
本研究在应用抑制性消减杂交(suppression subtractive hybridization,SSH)技术成功构建了高特异性的申克孢子丝菌菌丝相(mycelium,M)和酵母相(yeast,Y)的正反cDNA消减文库的基础上,对差异表达基因进行生物信息学分析,以筛选出与双相转换相关的差异表达基因。
1 材料与方法
1.1 材料
1.1.1 菌株 申克孢子丝菌标准株CMCC(F)D1a,购于中国医学科学院南京皮肤病研究所。
1.1.2 试剂 植物总RNA提取试剂盒(Autolab,China);PCR-SelectTMcDNA Subtraction Kit(Clontech,American);SMART cDNA Amplification Kit(Clontech,American);PCR产物纯化试剂盒(Autolab,China);T-载体连接试剂盒(Promega,American);质粒提取试剂盒(Autolab,China);
1.1.3 引物和接头序列(Clonetech PCR-Select cDNA Subtraction Kit提供)

接头1:

接头2R:

1.2 方法
1.2.1 菌丝相和酵母相细胞总RNA的提取 分别取孢子丝菌菌丝相(标记M)和酵母相(标记 Y)菌体约200mg,按植物总RNA提取试剂盒按说明书操作提取总RNA,1.2%琼脂糖凝胶电泳分析其完整性。
1.2.2 cDNA的合成 分别取3μL M和 Y的总RNA为模板,按 SMART PCR cDNA Synthesis试剂盒说明书操作合成cDNA,主要过程为合成M、Y的cDNA第一链及第二链;去除cDNA短片段;RsaI酶切处理双链cDNA。
1.2.3 抑制性消减杂交 按照PCR-Select cDNA Subt raction Kit说明书操作。分别以RsaⅠ酶切后的M、Y的cDNA为检测子和驱动子,连接接头1和和接头2R,将检测子与过量的驱动子混合后进行两次杂交,杂交后的“M+Y”、“Y+M”再行两轮抑制性PCR扩增;所得的二次 PCR产物利用 PCR purification kit进行纯化。
1.2.4 cDNA消减文库的构建 按照Diatchenko L[5]参考文献方法操作。
1.2.5 转化重组子的筛选及单克隆检测 挑取有插入片段的白色单克隆菌落,接种于含100mg/L Amp的LB培养基中,37℃静止培养5h;取1μL于96孔PCR反应板内,利用消减杂交试剂盒中提供的巢式引物 PrimerT7和 PrimerSP6进行 PCR扩增,扩增产物电泳观察插入的差异表达片断的大小及分布范围。
1.2.6 “M+Y”、“Y+M”文库测序 在紫外透射仪上挑取插入片断大于250bp、带型单一的阳性克隆,以 T7或SP6为正向引物,用 ABI 9600测序仪进行单向测序,将所测序列,即表达序列标签(expressed sequence tag,ESTs)进行生物信息学分析。
1.2.7 ESTs数据的生物信息学分析 包括 ESTs数据预处理、EST数据的拼接和聚类分析、Unigene开放性读码框的预测、利用 NCBI Nt,NCBI Nr,SwissProt,KEGG,COG,InterPro及 GO数据库对筛选的差异表达基因进行注释和功能分类。
2 结 果
2.1 申克孢子丝菌菌丝相和酵母相正反cDNA消减文库的构建
2.1.1 申克孢子丝菌丝相(M)和酵母相(Y)总RNA的的鉴定 M和 Y RNA样品经1.2%琼脂糖凝胶电泳后,在紫外灯下观察,均可见28S、18S、5.8S(5S)3条清晰的rRNA条带,见图1。

图1 菌丝相及酵母相细胞总RN AFig.1 The total RNA isolated from yeast and Mycelium cells M:Mycelium Y:Yeast
2.1.2 申克孢子丝菌丝相(M)和酵母相(Y)cDNA合成:M、Y cDNA合成效果好,cDNA主要分布在0.5~2 kb,平均长度1 kb,呈长瀑布条带,见图2,图3。


2.1.3 cDNA经RsaI酶切效果检测RsaI酶切前,M、Y样品cDNA主要分布在0.5~2 kb,平均长度1 kb;酶切后片段明显减小,平均500bp左右,见图4。
2.1.4 接头连接效率检测 (Marker 1kb plus:10kb,8kb,6kb,5kb,4kb,3kb,2kb,1.5kb,1kb,0.8kb,0.5kb,0.3kb)PCR结果示条带弥散,与酶切完cDNA类似,提示接头已连上,见图5。

图4 cDNA及 RsaI酶切电泳图谱Fig.4 cDNA and cDNA ofRsaI digestedM:Marker 1kb plus;1,3:M,Y cDNA;2,4:M,Y cDNA ofRsaI digested
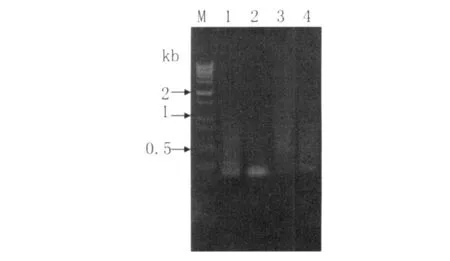
图5 接头连接PCR扩增图谱Fig.5 Adaptor ligation amplified by PCRM:Marker 1kb plus;1:M+1;2:M+2;3:Y+1;4:Y+2
2.1.5 SSH产物的电泳鉴定 经SSH消减后的cDNA片段较消减前分布区域明显缩小,两轮PCR扩增后平均片段长度为500-600bp左右,表明 cDNA得到了良好的均衡和消减,见图6。

图6 两轮PCR电泳图谱M:Marker 1kb plus;1:Y+M第一轮PCR产物;2:M+Y第一轮PCR产物;3:+M第二轮PCR产物;4:M+Y第二轮PCR产物Fig.6 PCR production after twice subtraction
2.1.6 第二轮PCR产物纯化后浓度及纯度 纯化后的M+Y及 Y+M PCR产物浓度分别为:0.101 μg/μL,0.075μg/μL;OD260/280 分别为:1.78,1.70。提示cDNA纯度较高,无蛋白质、核酸及盐类等污染。
2.1.7 消减文库单克隆的PCR扩增 随机挑取白色菌落进行 PCR扩增,鉴定阳性克隆插入片断大小,插入片断主要集中于400~1 000bp,见图7。
2.1.8 “M+Y”、“Y+M”文库测序及分析 M+Y文库获得751条ESTs,其中高质量718条,低质量28条,载体 ESTs5条。ESTs序列长度主要分布在600~1 000bp,平均690.95bp,带载体序列的 ESTs长度主要分布在600~900bp,平均694.1bp;不带载体序列的 ESTs长度主要分布在300~500 bp,平均为410.5bp。Y+M文库获得875条ESTs,其中高质量809条,低质量55条,载体 ESTs11条。ESTs序列长度主要分布在400~900bp,平均长度为575.9bp,带载体序列的 ESTs长度主要分布在400~800bp,平均长度为 582.3bp,不带载体序列的ESTs长度主要分布在300~600bp,平均长度为411.4bp。

图7 消减文库部分阳性克隆的PCR扩增结果Fig.7 Some samples amplified by PCRM:marker DL2000 Plus:250bp,500bp,800bp,1kb,2kb,3kb,4.5kb
2.2 ESTs数据的生物信息学分析
2.2.1 ESTs数据预处理结果 M+Y文库的718条高质量 ESTs经过拼接组装后,获得52条序列重叠群(Contigs),49条单一序列(Singlets),共101条非冗余序列簇(Unigenes),占14.06%;Y+M文库的809条高质量ESTs经过拼接组装后,获得109条 Contigs,140 条 Singlets,共 249 条 Unigenes,占30.77%(见表3)。M+Y文库的101条 Unigenes平均长度为530.39bp,最大的由70个 ESTs组成,有 ORF的 Unigenes63条,占 62.4%,无 ORF的有38条,占37.6%;最长ORF的平均长度为186.19 bp。Y+M文库的249条 Unigenes平均长度为499.28bp,最大的由 62个 ESTs组成,有 ORF的Unigenes194条 ,占 77.9%,没有 ORF 的 55 条 ,占22.1%。最长ORF的平均长度为193.59bp。
2.2.2 Unigenes数据库比对结果 Blastn NCBI Nt:M+Y文库的101条Unigenes有注解的共93条,占92.1%,其中有多个重复序列,去除重复序列后得到35条同源基因序列;Y+M文库的249条Unigenes有注解的共232条,占93.21%,去除重复序列后得到91条同源基因序列。Blastx NCBI Nr:M+Y文库有注解的共47条,占46.5%;Y+M文库有注解的共 213条,占 85.5%。Blastx SWISSPROT:M+Y文库有注解的共14条,占13.9%;Y+M文库有注解的共61条,占24.5%。Blastx KEGG:M+Y文库有注解的共34条,占33.7%;Y+M文库有注解的共149条,占59.8%。COG分类:M+Y文库有注解的共6条,占5.9%,其中,2条为转录后修饰,蛋白质转换和伴侣蛋白;氨基酸运输和代谢的、能源生产和转换、一般功能预测及功能未知的各1条;Y+M文库有注解的共27条,占18.5%,其中,8条为翻译,核糖体结构和生物起源;6条为能源生产和转换;转录:3条;碳水化合物运输和代谢:3条;氨基酸运输和代谢、脂质运输和代谢、辅酶的运输和代谢、转录后修饰,蛋白质转换和伴侣蛋白、复制,重组和修复、一般功能预测及细胞骨架各1条;Interpro:M+Y文库有注解的共10条,占9.9%;Y+M文库有注解的共 42条,占16.9%。数据库比对分析结果:M+Y文库有注解的共9条,占8.9%;Y+M文库有注解的共32条,占12.9%。
3 讨 论
抑制性消减杂交(SSH)技术[6]是一种以抑制性PCR反应为基础,将标准化测试cDNA单链步骤和消减杂交步骤合为一体的技术。该技术将待测的cDNA分为检测子(tester)和驱动子(driver),标准化步骤均等了检测子中的cDNA单链丰度,消减杂交步骤去除了检测子和驱动子之间的共同序列,使差异表达序列得到扩增。SSH技术的本质是除去共同的cDNA序列,有效富集差异表达基因的序列,故敏感性较高,在克隆差异表达基因方面得到广泛使用[7-9]。该方法也应用于部分致病性真菌的致病性和耐药性的研究当中,如曲霉、巴西副球孢子菌、粗球孢子菌,白念珠菌等[10-12]。刘红芳、席丽艳等应用SSH技术构建了马内菲青霉酵母相和菌丝相的消减cDNA文库,并进行了差异表达基因的初步筛选[13]。
生物信息学(bioinformatics)[14]是生物学与计算机科学以及应用数学等学科相互交叉而形成的一门新兴学科。它通过对生物信息的获取、加工、存储、检索与分析,综合运用数学、计算机科学和生物学的各种工具,进而达到揭示数据所蕴含的生物学意义的目的。从已建好的cDNA库中随机取出一个,从5’末端或3’末端对插入的cDNA片段进行一轮单向自动测序,所获得的一段短的cDNA部分序列,即为表达序列标签(EST)[15]。EST作为表达基因所在区域的分子标签因编码DNA序列高度保守而具有自身的特殊性质,能穿越家系与种的限制,容易在EST库中搜寻到新的基因。
本研究是在成功构建申克孢子丝菌标准株酵母相和菌丝相差异表达cDNA文库的基础上进行生物信息学分析,对所有数据进行去载、拼接、聚类和Blastn分析及数据整理,M+Y文库获得101条Unigenes;Y+M文库获得249条Unigenes。这些拼接后得到的Unigenes序列通过与多个数据库的比对分析,发现M+Y,Y+M两个消减文库中都出现有多个重复的基因序列,在两轮PCR中也出现了几条特异条带,这几个基因都多次重复,从M+Y/Y+M两者的比对结果来看,这些多次重复基因没有在 Y+M/M+Y中出现,表明这不是假阳性产生的,而是正常被消减出来的结果,理论上讲重复序列的出现可能是样本间表达差异大,差异丰度高的基因。申克孢子丝菌酵母相菌丝相的转换伴随着不同菌相细胞差异基因的高表达,这些高表达的差异基因可分为四类:(1)结构基因类:如18S、25sRNA基因,某些结构基因在申克孢子丝菌酵母相菌丝相状态下的独特表达,可能是菌相转换的结果;(2)代谢酶类:参与碳水化合物、氨基酸、脂质和辅酶运输和代谢,环境的改变势将影响细胞代谢的增强或代谢途径的改变。(3)细胞表面分子类,这些分子参与细胞内的信号转导,推测其在菌相转换中起介导细胞基因表达的调控或细胞行为的改变;(4)功能不明的细胞分子。它们在申克孢子丝菌双相转换机制中的作用也有待进一步验证,在已知这些基因全部或部分序列后对其功能及其与菌相转换的关系进行更深入的研究显得尤为重要。
另外,我们的试验结果未能验证已报道的与申克孢子丝菌双相转换相关的基因。如:编码钙/钙调蛋白激酶家族中的 SSCM K1基因sscmk1,其在GENEBAN K的编号是A Y823267.1;编码胞浆磷脂酶A2的基因 ssg-2,其编号是A Y078408;细胞周期素依赖激酶的编码基因 PhoSs,其编号是AF116453;PKC的编码基因 pkcSs-1,其编号是AF12492.1。与M+Y及 Y+M cDNA差异基因文库中的所有基因序列进行比对,没有找到相同的基因。分析可能的原因为:1.虽然SSH技术的本质是除去共同的cDNA序列,有效富集差异表达基因的序列,敏感性较高,但对差异丰度不显著的基因可能不会全部被检出;2.虽然在筛选出具有插入片段的克隆后进行了大规模的测序,仍然可能漏掉部分差异基因片段;3.对 ESTs进行拼接组装后得到的unigene的序列可能不是完全准确。所以,成功构建申克孢子丝菌标准株酵母相和菌丝相差异表达cDNA文库及差异表达基因的生物信息学分析只是为进一步筛选与申克孢子丝菌双相转换相关基因奠定了基础,还需要针对筛选出的有意义的差异表达基因进行下游验证试验。
[1]Aquino-Piñero EE,Rodríguez del Valle N.Different protein kinase C iso-forms are prent in the yeast and mycelium forms of Sporothrix schenckii[J].Med Vet Mycol,1997,138(3):109-115.
[2]de Jesús-Berríos M,Rodríguez-del Valle N.Expression of a Pho85 cyclin-dependent kinase is repressed during the dimorphic transition inS porothrix schenckii[J].Fungal Genet Biol,2002,37(1):39-48.
[3]Valle-Aviles L,Valentin-Berrios S,Gonzalez-Mendez R R,et al.Functional,genetic and bioinformatic characterization of a calcium/calmodulin kinase gene inS porothrix schenckii[J].BMC Microbiol.2007(7):107-117.
[4]Valentín-Berríos S,González-Velázquez W,Pérez-Sánchez L,et al.Cytosolic phospholipase A2:a member of the signalling pathway of a new G proteinαsubunit inS porothrix schenckii[J].BMC Microbiol,2009(9):100-124.
[5]Diatchenko L,Lau YF,Campbell AP,et al.Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries[J].Proc Natl Acad Sci U S A,1996 Jun 11;93(12):6025-30.
[6]Diatchenko L,Lau YF,Campbell AP,et al.Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries[J].Proc Natl Acad Sci U S A,1996,93(12):6025-30.
[7]Dios S,Poisa-Beiro L,Figueras A,et al.Suppression subtraction hybridization(SSH)and macroarray techniques reveal differential gene expression profiles in brain of sea bream infected with nodavirus[J].Mol Immunol,2007,44(9):2195-204.
[8]Pan YS,Lee YS,Lee YL,et al.Differentially profiling the lowexpression transcriptomes of human hepatoma using a novel SSH/microarray approach[J].BMC Genomics,2006,31(7):131.
[9]Bui LC,Léandri RD,Renard J P,et al.SSH adequacy to preimplantation mammalian development:scarce specific transcripts cloning despite irregular normalization[J].BMC Genomics,2005,8(6):155.
[10]R te Biesebeke,A Levin C Sagt,J Bartels,et al.Identification of growth phenotype-related genes in As pergillus oryzae by heterologous macroarray and suppression subtractive hybridization[J].Mol Gen Genomics,2005,273:33-42.
[11]Marques E R,Ferreira M E S,Drummond R D.Identification of genes referentially expressed in the pathogenic yeast phase of Paracoccidioides brasiliensis,using suppression subtraction hybridization and differential macroarray analysis[J].Mol Gen Genomics,2004,271:667-677.
[12]Delgado N,Hung C Y,Tarcha E,et al.Profiling gene expression in Coccidioides posadasii[J].Med Mycol,2004,42(1):59-71.
[13]刘红芳,席丽艳,李彦清,等.马尔尼菲青霉酵母相消减cDNA文库的构建及初步筛选[J].中国人兽共患病学报,2007,23(3):218-226.
[14]Alba R,Fei Z,Payton P,Liu Y,et al.ESTs,cDNA microarrays,and gene expression profiling:tools for dissecting plant physiology and development[J].Plant J,2004,39(5):697-714.
[15]Teufel A,Krupp M,Weinmann A,et al.Current bioinformatics tools in genomic biomedical research(Review)[J].Int J Mol Med,2006,17(6):967-73.

