β-1,3-1,4-葡聚糖酶基因的克隆与序列分析
孙军涛, 王洪新, 吕文平*, 马朝阳, 戴易兴, 姚红
(1.食品科学与技术国家重点实验室,江南大学,江苏无锡 214122;2.江南大学食品学院,江苏无锡 214122)
β-1,3-1,4-葡聚糖酶基因的克隆与序列分析
孙军涛1,2, 王洪新1,2, 吕文平*1,2, 马朝阳1,2, 戴易兴1,2, 姚红1,2
(1.食品科学与技术国家重点实验室,江南大学,江苏无锡 214122;2.江南大学食品学院,江苏无锡 214122)
依据GenBank中收录的解淀粉芽孢杆菌(Bacillus amyloliquef aciens)β-1,3-1,4-葡聚糖酶基因序列,设计特异性引物,以提取的解淀粉芽孢杆菌基因组DNA为模板,利用聚合酶链式反应(PCR)扩增获得758 bp的β-1,3-1,4-葡聚糖酶基因(bgl),将此目的基因克隆至p TG19-T Easy载体中,经PCR、限制性内切酶鉴定和克隆片段的序列测定、比较,结果表明该克隆片段扩增准确、可靠。序列比较发现,此片段与解淀粉芽孢杆菌(B.am y loliquefacines,M 15674)、枯草芽孢杆菌(B.subtilis,D00518)和地衣芽孢杆菌(B.licheniform is,A Y365256)分别有99%、95%和94%的同源性。
解淀粉芽孢杆菌;β-1,3-1,4-葡聚糖酶;克隆;序列分析
β-1,3-1,4-葡聚糖是禾本科高等植物(谷物类)细胞壁的多糖组分,是由1 200个以上的β-D-葡萄糖残基通过β-1,3和β-1,4糖苷键连接而成的线性分子,在大麦、燕麦、高粱、大米和小麦等谷物胚乳细胞壁中的含量尤为丰富,不同谷物β-葡聚糖中两种糖苷键键型的比例和寡糖单元的长度有所不同,如大麦,90%以上的水溶性β-葡聚糖是由单一的β-1,3糖苷键连接纤维三糖和纤维四糖所组成[1]。
β-1,3-1,4-葡聚糖酶 (1,3-1,4-β-D-glucan 4-glucanohydrolase, 1, 3-1, 4-glucanase,E.C.3.2.1.73,简称β-葡聚糖酶或地衣多糖酶),它是只降解β-葡聚糖中与β-1,3糖苷键相邻的β-1,4糖苷键的酶,其降解产物主要是纤维三糖和纤维四糖[2]。它是一种有经济价值的工业用酶,在啤酒酿造中常用于糖化或发酵以提高啤酒质量和稳定性[3],并且作为饲料添加剂也能够有效的提高饲料转化率[4-5]。但是现有的β-1,3-1,4-葡聚糖酶的耐热性较差、酸性条件下活性较低,在工业应用中,较高的加工温度或酸性环境,就会使β-1,3-1,4-葡聚糖酶的活性降低甚至失活,限制了β-1,3-1,4-葡聚糖酶的应用范围,增加生产成本。因此,开发出在高温、强酸条件下具有高活性的β-1,3-1,4-葡聚糖酶将具有很好的社会经济价值。
许多不同来源的β-1,3-1,4-葡聚糖酶基因已经克隆得到,如芽孢杆菌属[6-10]、球菌[11]和真菌[12]等,并在大肠杆菌(Escherichia coli)、芽孢杆菌(Bacillus strains)、啤酒酵母(Saccharomyces cerevisiae)以及其他转基因植物大麦和烟草等不同宿主菌中进行了表达。通过单一菌株的基因的克隆表达、定点诱变以及基因缺失等手段得到的β-1,3-1,4-葡聚糖酶在耐热、耐酸和活性方面不够理想。随着分子生物学技术的发展,基因重叠延伸PCR技术在基因工程中有很好的应用,通过此技术采用具有互补末端的引物,使PCR产物形成重叠链,从而在随后的扩增反应中通过重叠链的延伸,将不同来源的扩增片段重叠拼接起来[13]。为了获得工业生产条件下具有较高热稳定性的酶,将不同来源的β-1,3-1,4-葡聚糖酶基因通过基因重叠延伸PCR技术进行杂合,构建杂合基因工程菌,表达出适合工业生产需要的杂合酶将是一种较好的手段。
作者以解淀粉芽孢杆菌(Bacillus amyloliquefaciens)为研究对象,克隆了β-1,3-1,4-葡聚糖酶的基因,并对基因序列进行了分析,为进一步构建耐热、耐酸高活性的β-1,3-1,4-葡聚糖酶奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒 解淀粉芽孢杆菌(Bacillus amyloliquef aciens)、DH5a由作者所在实验室保存。
1.1.2 培养基与试剂 LB培养基(g/L):蛋白胨10,酵母膏5,氯化钠10。氨苄青霉素的使用浓度为100μg/m L,固体培养基按2%的量加入琼脂。
限制性内切酶BamH I、XhoI、T4 DNA 连接酶、TaqDNA 聚合酶、p TG19-T Easy Vector、DL 2000 M arker、小量质粒快速提取试剂盒均购于上海捷瑞生物工程公司;X-gal和IPTG购于Sigma公司;其他主要试剂均为国产AR级产品。
1.2 仪器与设备
PCR仪 PTC-2000:美国MJ公司产品;凝胶成像透射仪chemi system:美国UVP公司产品;One Drop Spectrophotometer OD-1000:上海采邑生物科技有限公司产品;冷冻气浴恒温振荡器BS-1E:金坛市瑞华仪器有限公司产品。
1.3 方法
1.3.1 解淀粉芽孢杆菌基因组DNA提取 活化的解淀粉芽孢杆菌接种到LB培养基中,37℃下200 r/mim振荡培养过夜,收集菌液按照小量质粒快速提取试剂盒提供的方法提取解淀粉芽孢杆菌基因组DNA,用1.5 g/dL的琼脂糖凝胶电泳对提取的DNA进行检测。同时用OD-1000紫外、可见光分光光度计检测DNA浓度和纯度。

1.3.3 β-1,3-1,4-葡聚糖酶的基因的PCR扩增以提取的解淀粉芽孢杆菌基因组DNA为模板,利用PCR扩增β-1,3-1,4-葡聚糖酶的基因,反应体系(50μL):10 ×PCR buffer 5μL,M gCl2(25 mmol/L)2.5μL,dN TP(10 mmol/L)1μL,引物各2μL,模板3μL,Taq酶(5 U/μL)0.5μL,重蒸水 34μL。PCR反应条件:94℃2 min;94℃1 min;42℃1 min,每循环增加0.1℃,72℃1 min,重复30次;72℃10 min;4℃保存。
1.3.4 β-1,3-1,4-葡聚糖酶基因的克隆 PCR产物经1.5 g/dL的琼脂糖凝胶电泳,割胶回收试剂盒回收目的基因片段,回收产物与p TG19-T vector 4℃连接过夜,连接产物加入感受态DH5a中,冰浴30 min,42℃下热激45 S,立即放入冰浴中5 min,加入800μL的LB培养基,37℃下200 r/mim振荡培养1 h。将转化好的菌液接种在含有氨苄青霉素的LB平板上(已经涂过X-gal和IPTG)37℃过夜培养,挑取白色菌落,接种到含有氨苄青霉素的LB液体培养基中,37℃过夜培养。提取质粒备用。
1.3.5 阳性克隆的鉴定
1)PCR鉴定 以提取的重组克隆质粒进行PCR扩增,反应体系和条件同基因扩增,扩增产物用1.5 g/dL的琼脂糖凝胶电泳检测。
2)BamH I和XhoI双酶切鉴定 酶切反应体系为:buffer 2μL,BamH I 1μL,Xho I1μL,克隆质粒8μL,无菌水8μL;混匀,37 ℃酶切4 h后,65℃灭活15 min,酶切产物经1.5 g/dL琼脂糖凝胶电泳检测。
1.3.6 测序及序列分析 经PCR和酶切鉴定为阳性的克隆质粒送上海生物工程公司进行测序。应用DNA Tools软件对β-1,3-1,4-葡聚糖酶基因的核苷酸序列进行分析,将推导的氨基酸序列与 Gen-Bank上发表的已知序列做同源性比较与分析。
2 结果与分析
2.1 解淀粉芽孢杆菌基因的提取
通过琼脂糖凝胶电泳对所提取的DNA进行检查可以清晰地看到特征性条带,证明得到了解淀粉芽孢杆菌的基因。用OD-1000紫外、可见光分光光度计检测DNA的质量浓度为215.33 ng/μL,A260和A280之比为1.954,符合1.8~2.0的要求,说明DNA的纯度较高,可用于PCR的扩增。
2.2 β-1,3-1,4-葡聚糖酶的基因的 PCR扩增
以解淀粉芽孢杆菌基因组DNA为模板,进行PCR扩增出的目的片段在750 bp左右,其条带单一,特异性强,表现与目的基因预期大小一致的条带,琼脂糖电泳图如图1。
2.3 阳性克隆的鉴定
2.3.1 PCR鉴定 在LB平板上筛选白斑,得到一些阳性克隆子。挑取得到的白斑于LB液体培养基中培养,提取质粒,经PCR扩增后,琼脂糖凝胶电泳结果如图2,克隆得到的基因片段与目的基因片段大小一致,说明目的基因片段 bgl已经插入到p TG19-T vector中。

图1 β-1,3-1,4-葡聚糖酶的基因的 PCR扩增Fig.1 Gel electrophoresis analysis of PCR amplification product ofβ-1,3-1,4-glucanase gene
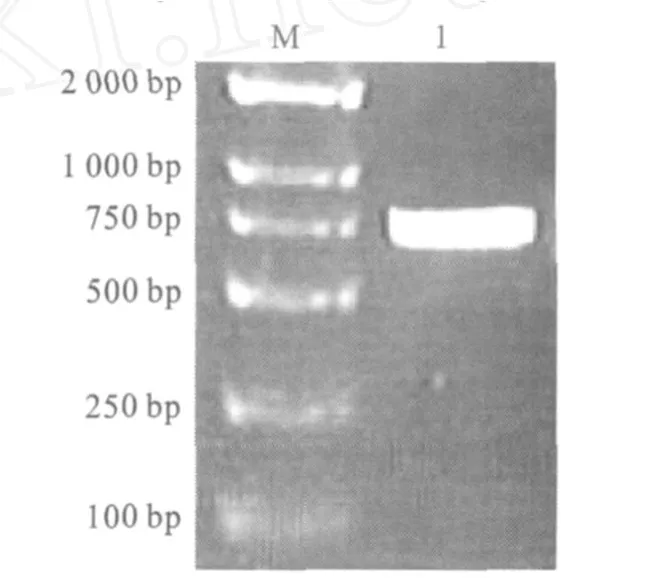
图2 阳性克隆子PCR鉴定电泳图Fig.2 Gel electrophoresis analysis of PCR amplification product of positive cloning plasm id
2.3.2BamH I和XhoI双酶切鉴定 克隆载体经BamH I和XhoI双酶切后,琼脂糖凝胶电泳结果如图3,与未经酶切的克隆质粒对比发现,在750 bp左右出现明显的条带,进一步证实β-1,3-1,4-葡聚糖酶目的基因已经插入到p TG19-T vector中,成功获得阳性克隆子p TG19-T-bgl。
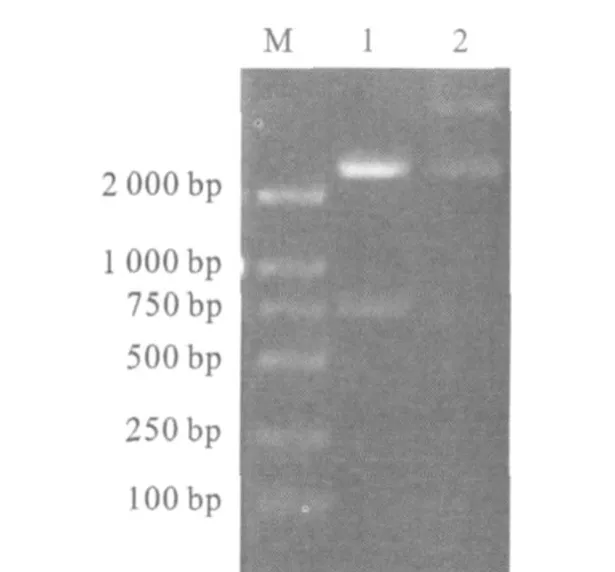
图3 阳性克隆子双酶切电泳图Fig.3 Restriction enzyme cutting of positive clon ing plasm id
2.4 测序结果及序列分析
序列测定结果表明,克隆得到的β-1,3-1,4-葡聚糖酶扩增片段全长为758 bp,已被 GenBank收录(Accession A Y205562)。如图4,与预期长度相同。经Blast同源性分析结果表明,克隆得到的β-1,3-1,4-葡聚糖酶基因与 GenBank登录的解淀粉芽孢杆菌(B.amyloliquef acines,M 15674)、枯草芽孢杆菌(B.subtilis,D00518)和地衣芽孢杆菌(B.licheniform is,A Y365256)分别有 99%、95%和94%的同源性。用DNA Tools分析表明,该基因ORF为717 bp,编码239个氨基酸,计算相对分子质量为26 807,等电点为6.51,其中疏水性氨基酸占36.8%,亲水性氨基酸占44.8%,酸性氨基酸占8.8%,碱性氨基酸占9.6%。
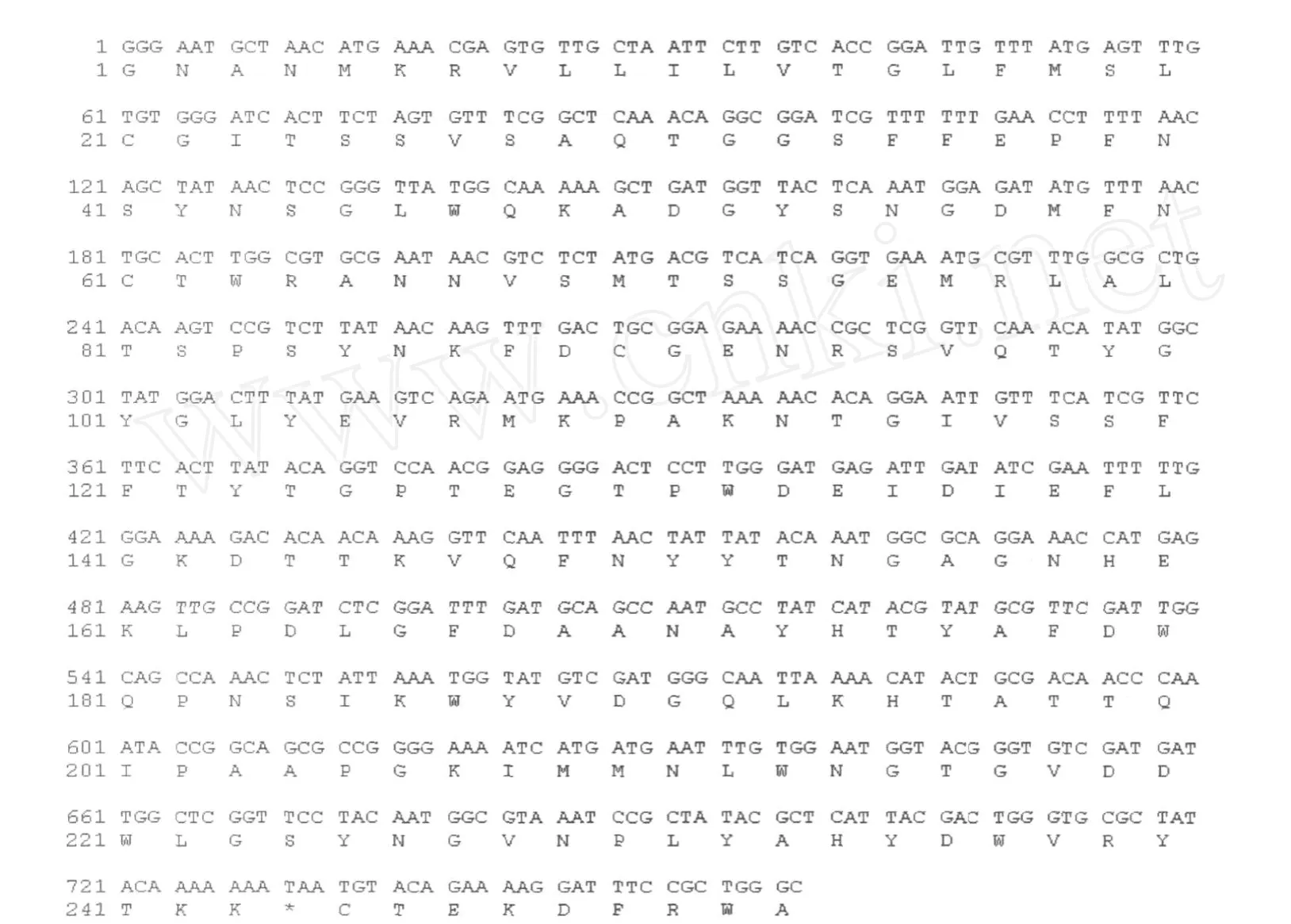
图4 解淀粉芽孢杆菌β-1,3-1,4-葡聚糖酶核苷酸序列及其推译的氨基酸序列Fig.4 The nucleotides sequence and its deduced amino acid sequence ofβ-1,3-1,4-glucanade from Bacillusamyloliquefaciens
3 结 语
β-1,3-1,4-葡聚糖酶是一种重要的工业用酶,广泛应用于酿酒和饲料工业。β-葡聚糖在啤酒生产和饲料加工过程中会产生一些问题,如降低麦芽汁和啤酒过滤速度、影响啤酒风味、在啤酒储存过程中易引起沉淀;过多的β-葡聚糖增加饲料的粘性和不可消化性,降低饲料的营养价值。在啤酒生产和饲料加工过程中,由于高温、低p H的条件,往往使谷物内源性β-1,3-1,4-葡聚糖酶失活,因此添加适量的耐热、耐酸、活性高的β-1,3-1,4-葡聚糖酶能够解决工业生产中β-葡聚糖引起的负面影响。但现有的β-1,3-1,4-葡聚糖酶的热稳定性、耐酸性较差,在酿酒和饲料工业应用时增加了生产成本。通过分子生物学技术构建适应工业需要的β-1,3-1,4-葡聚糖酶,降低其在酿酒和饲料工业应用时的生产成本,已经成为近年来的热点。
作者根据 GenBank提供的β-1,3-1,4-葡聚糖酶基因已知序列,应用Primer5软件设计特异性引物,运用PCR及T-A克隆技术成功获得了包含758 bp的β-1,3-1,4-葡聚糖酶基因片段,并与p TG19-T Easy Vector载体连接,经 PCR、酶切和测序鉴定,结果与 GenBank报告的序列完全一致,成功构建出p TG19-T-bgl真核表达载体,为下一步构建耐热、耐酸、活性高的杂合β-1,3-1,4-葡聚糖酶基因奠定基础。
(References):
[1]H rmova M,Banik M,Harvey A J,et al.Polysaccharide hydrolases in germinated barley and their role in the depolymerization of plant and fungal cell walls[J].International journal of biological macromolecules,1997,21(1):67-72.
[2]Anderson M A,Stone B A.A new substrate for investigating the specificity of beta-glucan hydrolases[J].FEBSLett,1975,52(2):202-207.
[3]Schimming S,Schwarz W H,Staudenbauer W L,Properties of a thermoactive beta-1,3-1,4-Glucanase(lichenase)from Clostridium thermocellum exp ressed inEscherichia coli[J].Biochemical and Biophysical Research Communications,1991,177:447-452.
[4]Gohl B,Alden S,Elwinger K,et al.Influence ofβ-glucanase on feeding value of barley for poultry and moisture content of excreta[J].British Poultry Science,1978,19:41-47.
[5]Mathlouthi N,Serge M S,Luc S,et al.Effects of xylanase and β-glucanase addition on perfo rmance,nutrient digestibility,and physico-chemical conditions in the small intestine contents and fecal microflora of broiler chickens fed a wheat and barley-based diet[J].Animal Research,2000,51:395-406.
[6]Akita M,Kayatama K,Hatada Y,et al.A novel beta-glucanase gene from Bacillus halodurans C-125[J].FEMS Microbiolgy Letters,2005,248:9-15.
[7]Kim JY.Overproduction and secretion of Bacillus circulans endo-β-1,3-1,4-glucanase gene(bglBC1)inB.subtilisandB.megaterium[J].Biotechnology Letters,2003,25:1445-1449.
[8]Qiao J Y,Dong B,Li Y H,et al.Cloning of aβ-1,3-1,4-d-glucanase gene fromBacillussubtilisMA 139 and its functional expression inEscherichia coli[J].Applied Biochemistry and Biotechnology,2009,152:334-342.
[9]Teng D,Wang J H,Fang Y,et al.Cloning ofβ-1,3-1,4-d-glucanase gene fromBacillus licheniform isEGW 03(CGMCC0635)and its exp ression inEscherichia coliBL21(DE3)[J].Applied Microbiology Biotechnology,2006,72:705-712.
[10]李永仙,郑飞云,李岐,等.重组大肠杆菌BL 21(DE3)-p ET28a(+)-bgl诱导表达β-葡聚糖酶的条件优化[J].食品与生物技术学报,2009,28(2):250-255.
LI Yong-xian,ZHENG Fei-yun,LI Qi,et al.Optimization of fermentation conditions of recombinant E.coliBL21(DE3)-p ET28a(+)-bgl producingβ-glucanase[J].Journal of Food Science and Biotechnology,2009,28(2):250-255.(in Chinese)
[11]Ekincl M S,Flint H J.Expression of bifunctional genes encoding xylanase andβ(1,3-1,4)-glucanase in gram-positive bacteria[J].Turkish Journal of Veterinary&Animal Science,2001,25:771-775.
[12]Yang SQ,Yan Q J,Jiang ZQ,et al.Biochemical characterization of a novel thermostableβ-1,3-1,4-d-glucanase(Lichenase)fromPaecilomyces thermophila[J].Journal of agricultural and food chemistry,2008,56:5345-5351.
[13]Ho SN,Hunt H D,Horton R M,et al.Site-directed mutagenesis by overlap extension using the polymerase chain reaction[J].Gene,1989,77(1):51-59.
Cloning and Sequence Analysis ofβ-1,3-1,4-Glucanase Gene
SUN Jun-tao1,2, WANG Hong-xin1,2, LV Wen-ping*1,2,M A Chao-yang1,2, DA I Yi-xing1,2, YAO Hong1,2
(1.State Key Laboratory of Food Science and Technology,Jiangnan University,Wuxi 214122,China;2.School of Food Science and Technology,Jiangnan University,Wuxi 214122,China)
Based on the sequence alignment ofBacillus amyloliquef aciens,two pairs of primers were designed and synthesized in this study.Using the total DNA ofBacillus amyloliquef aciensas temp late,the gene ofβ-1,3-1,4-glucanase(bgl)was amplified by PCR,which has about 750 bp in length.The gene was cloned into pTG19-T Easy vector,and the recombinant plasm id was identified by PCR,restriction enzyme analysis and sequencing.The homology analysis revealed that the nucleotide sequences of the clonedβ-1,3-1,4-glucanase gene sim ilar to the B.amyloliquef acines(M 15674),B.subtilis(D00518)andB.licheniform is(A Y365256)were 99%,95%and 94%,respectively.
Bacillus amyloliquef aciens,β-1,3-1,4-glucanase,cloning,sequence analysis
*通信作者:吕文平(1968-),男,山西寿阳人,工学博士,副教授,主要从事生物技术研究。Email:lwpkxy@163.com
Q 556.2
A
1673-1689(2011)04-0618-05
book=622,ebook=312
2010-08-11
国家自然科学基金项目(20090964);无锡市科技创业计划(CIE00920)。
孙军涛(1982-),男,河南漯河人,食品科学与工程博士研究生,主要从事食品营养与安全研究。Email:jtsfly@163.com

