丛粒藻形态多样性与遗传多样性研究*
王朋云,茅云翔,孔凡娜,马 梅,马 飞
(中国海洋大学海洋生物遗传学与基因资源利用教育部重点实验室,山东青岛266003)
丛粒藻(Botryococcus braunii)[1],又名布朗葡萄藻,属绿藻门(Chlorophyta)、共球藻纲(Trebouxiophyceae)、丛粒藻科(Botryococcaceae)、丛粒藻属(Botryococcus),是1种营聚落生活的单细胞绿藻[2]。研究发现,丛粒藻在生长过程中能够大量合成并储存多种烃类物质,其总烃含量一般超过细胞干重的30%,最高可达86%[3-4]。研究还证明,丛粒藻是构成石油生烃母质的主要成分[5-7],所产生的烃类物质热值较高[8]。因此,该藻被认为是一种潜在的可再生生物燃料来源。
丛粒藻广泛分布于世界热带、亚热带和温带的淡水和微咸水湖等水体中[9]。不同地理株系间具有形态多样性,细胞形状大小、胶质鞘包埋程度、细胞排列方式、聚落细胞数目和连接方式等均存在差异,这些形态指标是丛粒藻分属、定种的主要指标[10]。同时,研究还证明不同来源丛粒藻合成的烃类物质也存在显著差异[11-13]。丛粒藻形态多样性与生化多样性的基础在于不同来源藻株的遗传差异。
本研究以3株不同来源的丛粒藻为实验材料,一方面通过观察显微和亚显微结构,克隆并分析18S~28S rDNA序列特征,为丛粒藻的形态分类和分子鉴定提供系统的数据;另一方面研究丛粒藻不同株系间形态多样性-遗传多样性的关系,结合生理生化数据,将为优良藻株的遗传选育提供资料。
1 材料和方法
1.1 材料
1.1.1 实验藻株 实验中所用3株丛粒藻AGBBb01、AGB-Bb02和A GB-Bb03均来自于中国科学院水生生物研究所淡水藻种库,其中AGB-Bb01采集自英国剑桥(原始藻株编号U TEX 572),AGB-Bb02采集自中国云南(原始藻株编号FACHB-759),AGB-Bb03采集自中国湖北(原始藻株编号FACHB-775)。藻株均培养于Chu10培养基中,培养温度(23±1)℃,光暗周期比12 h∶12 h,光照强度25μmol·m-2·s-1。
1.1.2 菌株和克隆载体 基因克隆载体为pMD18-T,宿主菌株为E.coli DH5α,均购自TaKaRa公司。
1.1.3 试剂盒、工具酶和试剂 PCR产物凝胶回收试剂盒(Gel Extraction Kit)购自OM EGA公司,Taq酶等PCR试剂购自MBI公司,试剂为分析纯。
1.2 方法
1.2.1 光学显微镜样品处理 取对数生长期藻液滴于载玻片上制成临时装片,直接在Olympus显微镜下观察并显微拍照,测量指标包括细胞长度、细胞宽度、聚落长度、聚落宽度、聚落细胞数目,每株微藻观察聚落数为30个。
1.2.2 扫描电镜样品处理 取1 m L对数生长期藻液于圆底离心管,800 r/min离心10 min收集藻细胞,加入1 m L 1%戊二醛固定(4℃过夜),1 000 r/min离心10 min,用0.1 mol/L磷酸缓冲液漂洗3次,然后依次用30%,50%,70%,90%,100%乙醇进行逐级脱水,用吸管将含有样品的乙醇溶液滴在有明胶膜的盖玻片上,可防止细胞被冲走,采用临界点干燥法干燥,离子溅射镀膜法镀膜[14]。
1.2.3 基因克隆 取对数生长期藻液,4℃下8 000 r/min离心10 min收集藻细胞,无菌水洗涤2次。将藻细胞于液氮中充分研磨后转入1.5 mL离心管,用酚氯仿法提取微藻基因组DNA[15]。
根据NCBIGenBank数据库中已公布的丛粒藻序列设计引物,PCR扩增产物包含大部分18S rDNA、ITS区和小部分28S rDNA序列片段(见图1),所用引物由上海英俊(Invitrogen)生物技术有限公司合成。正向引物序列S1F:5’-TGCCAGTAGTCA TA TGCTT GTC-3’;反向引物序列S2R:5’-TAAGTTCAGCGGGTGCTCTTAC-3’。

图1 丛粒藻18S~28S rDNA序列结构和引物结合位点Fig.1 The arrangment of the 18S~28S rDNA sequences and binding site of primers in B.braunii
PCR扩增条件:94℃预变性5 min;94℃变性1 min,55℃退火1 min,72℃延伸2 min,30个循环;72℃延伸5 min。
PCR扩增产物用OM EGA凝胶回收试剂盒(Gel Extraction Kit)回收后用T4 DNA连接酶将目标片段连接到质粒载体pMD18-T上,转入E.coli DH5α,由北京六合华大基因科技股份有限公司测序。
1.2.4 核酸序列分析 核酸序列同源性分析采用NCBI blastn在线程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)。多序列对位分析采用Bio Edit(version 7.0.5.2)包含的Clustal Wpackage(参数设定为gap open penalty=15,gap extend penalty=6.66);系统进化树构建采用M EGA 3.1 Unweighted Pair-Group Method Using(NJ),Kimura 2-parameter计算遗传距离值(重复1000次计算bootstrap值)。
2 结果
2.1 丛粒藻细胞形态特征及不同藻株间的差异
显微观察发现,3株丛粒藻藻细胞均呈黄绿色,细胞侧面观卵形或宽卵形,顶面观圆形。藻细胞色素体单个片状侧生,位于细胞底部至中部;胞内有数个至数十个大小不等的球状小颗粒,内为淀粉或油滴。细胞基部埋藏于藻落的透明胶质中,符合丛粒藻的形态学特征(见图2)。藻细胞合成的烃类物质大部分运输到细胞外,胞外烃呈颗粒状储存在细胞壁中。测量结果显示,3株藻在细胞大小(见表1)、聚落大小和聚落细胞数目方面存在较明显的差异(见表2)。其中,藻株AGB-Bb03细胞最大,而AGB-Bb02细胞最小;藻株A GB-Bb03聚落最大,聚落细胞数目最多,而AGBBb02聚落最小,AGB-Bb01聚落细胞数目最少。

图2 3株丛粒藻细胞形态显微观察Fig.2 Mo rphological characters of different strains of B.braunii observed under lightmicroscope
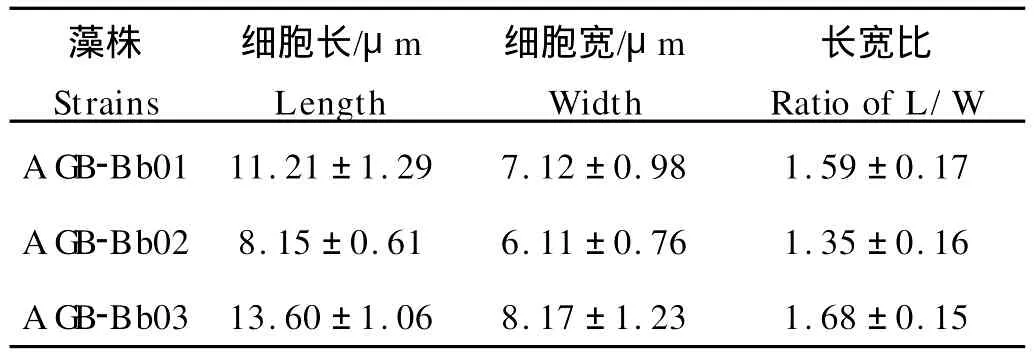
表1 不同株系丛粒藻细胞大小Table 1 Cell size of different strainsof B.braunii
扫描电镜下,可见藻细胞基部至中上部被母细胞壁残余加厚形成的杯状结构包被,顶部裸露不具加厚的细胞壁。3株微藻杯状鞘的厚度及细胞的包埋程度略有差别:AGB-Bb02的细胞壁较厚,AGB-Bb03次之,AGB-Bb01最薄;AGB-Bb02细胞大部分被杯状细胞壁包埋,仅顶部小部分未被杯状鞘包被,AGB-Bb01和AGB-Bb03的细胞包埋程度不及前者,杯状鞘约至细胞中上部。聚落通常是由2、4、8或更多的细胞通过胶丝连接成葡萄串状的集结体或复合集结体,胶丝长短各异且形状不规则,细胞在聚落表面略呈辐射排列(见图3)。
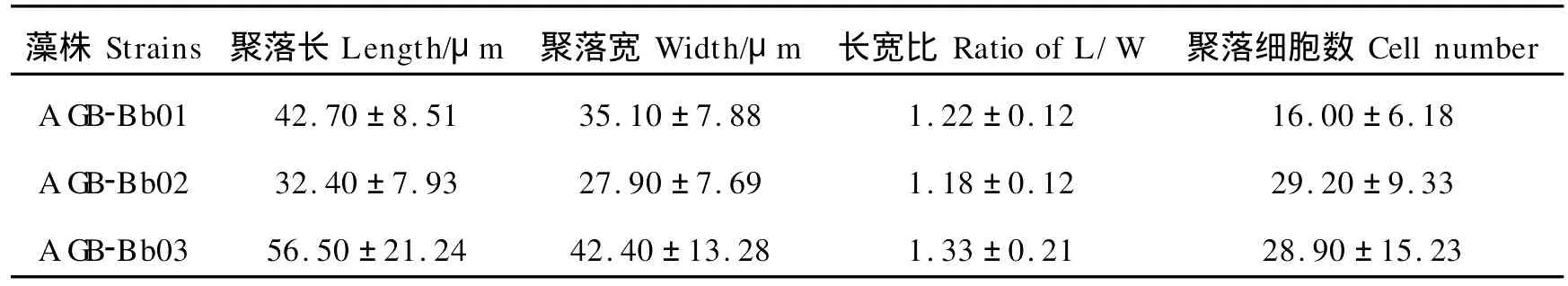
表2 丛粒藻不同株系聚落大小和聚落细胞数目Table 2 Colony size and cell numbersof the different strains of B.braunii

图3 3株丛粒藻细胞扫描电子显微镜观察Fig.3 Morphological and structural characters of three different strains of B.braunii observed by a scanning electron microscope
2.2 丛粒藻遗传多样性
PCR扩增产物分子量约为2 500 bps,包含18S rDNA、ITS区和28S rDNA序列片段。采用NCB I blastn在线程序进行核酸序列同源性分析,结果显示3株实验藻株的18S-28S rDNA序列与GenBank公共核酸数据库中丛粒藻的相关序列高度相似,其中AGBBb01与B.braunii Titicaca序列相似性为91.02%,A GB-Bb02与B.braunii Songkla Nakarin序列相似性为94.42%,AGB-Bb03与B.braunii Ayame序列相似性为99.60%。结果表明3株实验藻株均为丛粒藻,但不同藻株间存在遗传差异。将3株实验藻株的序列向GenBank公共核酸数据库提交,获得登录号为GU 951518、GU 951519、GU 951520。
2.2.1 基于18S rDNA的遗传多样性分析 去除引物序列后,18S rDNA序列长度分别为A GB-Bb01 1755bp、A GB-Bb02 1753bp,A GB-Bb03 1754bp,将本文克隆的18S rDNA序列分别与公共核酸数据库(GenBank)中已登录的丛粒藻相应序列,可以发现不同藻株相应序列之间存在碱基数目的差别(见表3)。
遗传距离和序列相似性分析结果显示:8株丛粒藻中遗传亲缘关系最近的是采自湖北武汉的藻株AGBBb03与源于非洲科特迪瓦的藻株Ayame,其遗传距离和序列相似性分别为0.001 1和0.998 3;遗传亲缘关系最远的是采自云南抚仙湖的藻株A GB-Bb02与源于美国明尼苏达艾塔斯卡湖的藻株Tow 9/21 P-16w,遗传距离和相似性分别为0.040 0和0.957 9。8株丛粒藻之间的平均遗传距离为0.025 2,平均序列相似性为0.972 7;而与同科不同属的Botryosphaerella sudetica U TEX 2629亲缘关系最近的是藻株AGB-Bb01,二者之间的遗传距离值为0.102 0,序列相似性为0.902 7(见表4)。
以与丛粒藻同科但不同属的南向球葡萄藻Botryosphaerella sudetica U TEX 2629相关序列为外群采用邻接法构建系统发育树,8株藻可划分为2个簇群(见图4)。在本文实验研究的3株微藻中,AGB-Bb02与AGB-Bb03亲缘关系更近,属于同一簇群;其中AGB-Bb03首先与源自科特迪瓦的Ayame株聚群(支持率100%,遗传距离值0.001 1,序列相似性0.998 3),AGB-Bb02则先与源自泰国的Songkla Nakarin株聚群(支持率99%,遗传距离值0.007 5,序列相似性0.992 6)。而AGB-Bb01则属于另一簇群,与源于玻利维亚的Titicaca株和美国明尼苏达的Tow 9/21 P-16w亲缘关系更近(支持率为99,遗传距离值0.009 8,序列相似性0.988 0)。藻株Ayame和AGB-Bb03的序列相似性最大(0.998 3),AGB-Bb02和Tow 9/21 P-16w的序列相似性最小(0.957 9),8株丛粒藻的序列相似性均值为0.972 7。
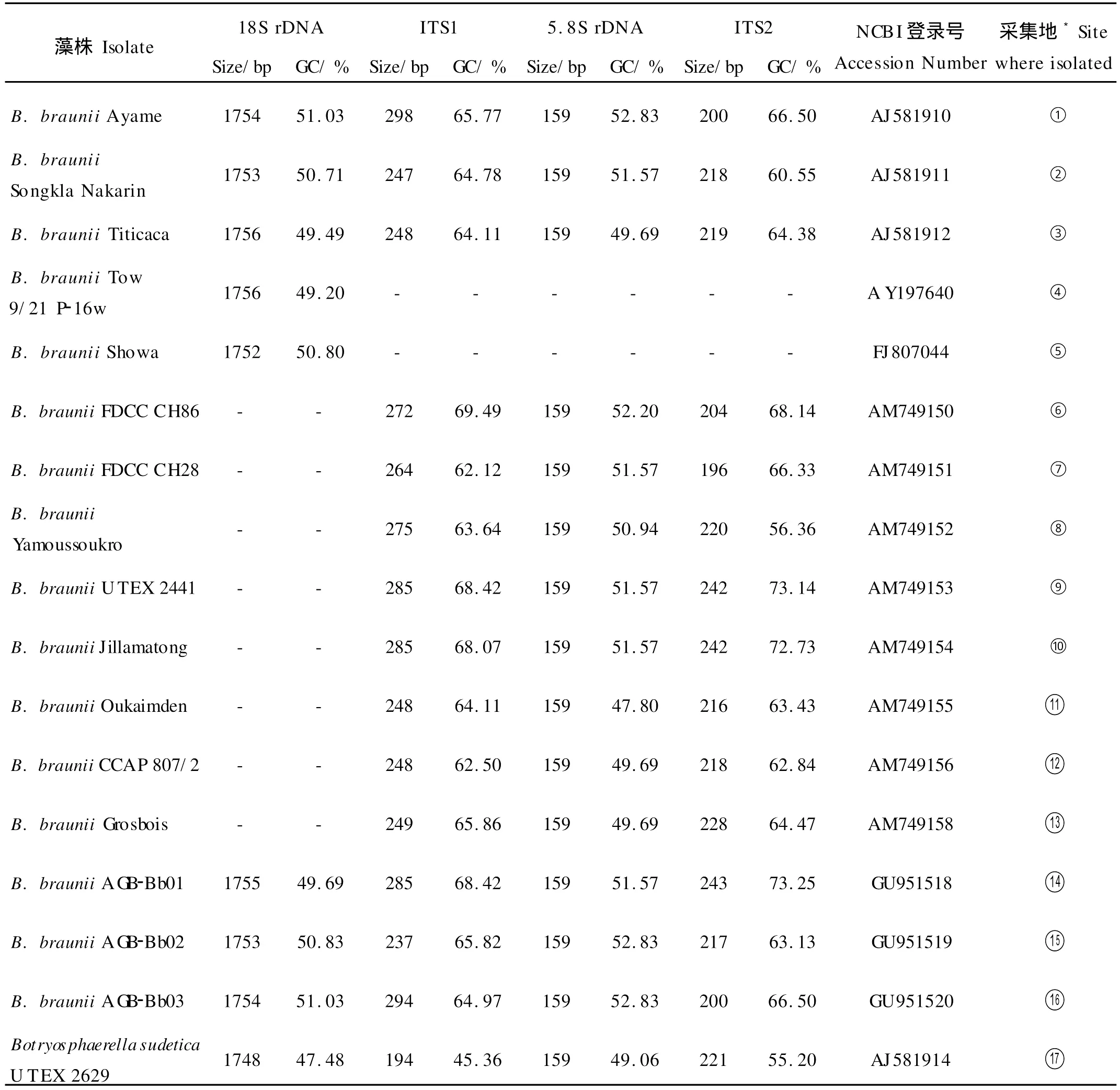
表3 用于系统树构建的丛粒藻18S rDNA和ITS区域的基本信息Table 3 Basic characters of 18S rDNA and ITS region of Botryococcus fo r phylogenetic tree construction
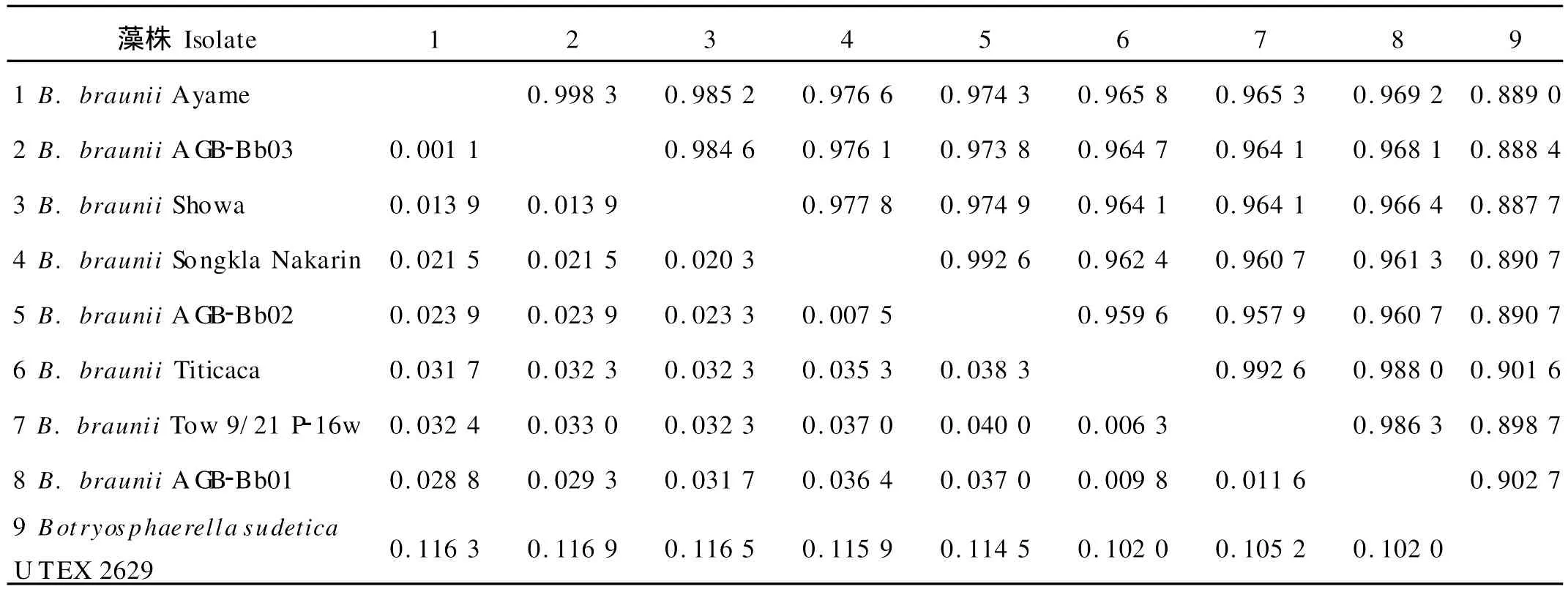
表4 基于18S rDNA序列的不同丛粒藻藻株的遗传距离和序列相似性Table 4 Genetic distances and sequence similarities of different Bot.strains based on 18S rDNA sequences
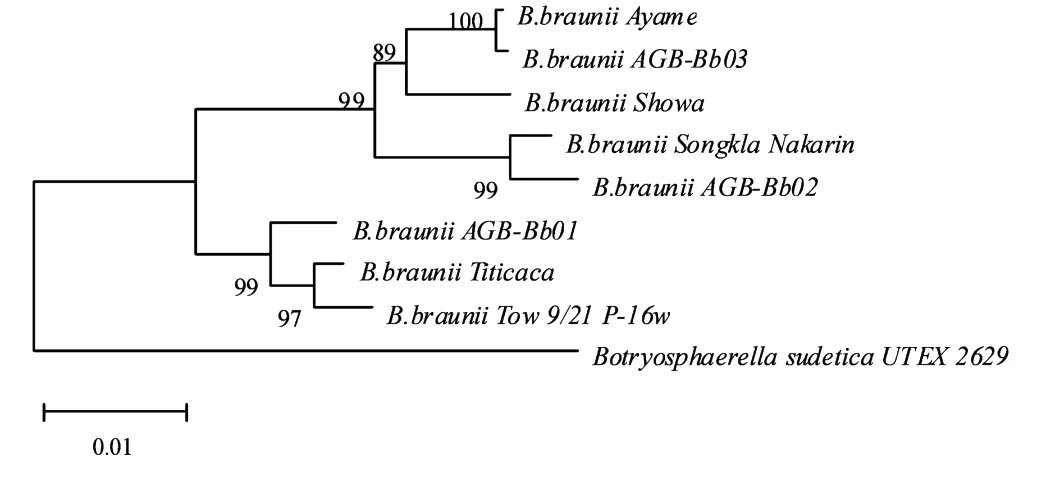
图4 基于18S rDNA序列的丛粒藻系统发育树Fig.4 Phylogenetic tree of genus Bot.based on 18S rDNA sequences using Neighbor-Joining method
2.2.2 基于ITS区序列的遗传多样性分析 本文克隆的3株丛粒藻ITS区序列长度分别是AGB-Bb01 687bp、AGB-Bb02 613bp和AGB-Bb03 653bp。比较全部14株相关序列已知的丛粒藻,发现5.8S rDNA核苷酸长度一致,均为159bp,显示出遗传上的稳定性;但5.8S rDNA的GC含量、ITS1和ITS2长度及碱基使用偏好变化很大,又显示出藻株间的遗传多样性(见表3)。
遗传分析显示:在14株丛粒藻中,源于英国剑桥的藻株AGB-Bb01与源于澳大利亚新南威尔士的藻株Jillamatong具有最近的亲缘关系,遗传距离和序列相似性分别为0.002 3和0.988 4;亲缘关系最远的是源自英国坎布里亚的CCAP807/2与科特迪瓦藻株Yamoussoukro,遗传距离和序列相似性分别为0.494 8和0.577 4。14株丛粒藻之间的平均遗传距离为0.327 5,平均序列相似性为0.663 9;而与近缘属物种Botryosphaerella sudetica U TEX 2629的平均遗传距离为0.678 0,平均序列相似性为0.463 6(见表5)。数据分析表明丛粒藻藻株的5.8S rDNA-ITS位点具有较高的遗传多态性,可用于不同藻株基因分型研究。同时数据分析也表明,尽管种内不同藻株之间遗传距离较大,序列相似性也较低,但与近缘物种相关数据比较仍有显著差异,因此该位点也可用于鉴定种属间的差异。
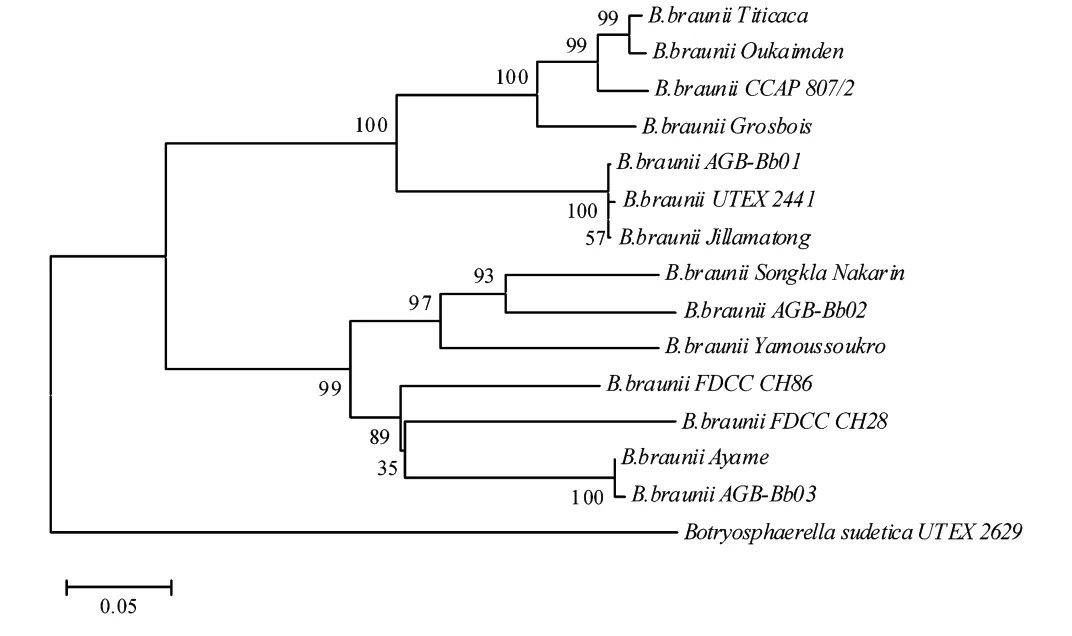
图5 用邻接法重建的基于丛粒藻ITS区序列的系统发育树Fig.5 Neighbor-joining tree of Bot.based on ITS region sequences
以B.sudetica U TEX 2629的ITS区序列为外群重建的系统发育树显示,所有丛粒藻藻株可以分为2大簇群(见图6),完全支持了18S rDNA序列系统发生树对丛粒藻亲缘关系的划分。本实验研究的3株丛粒藻显示出遗传上的差异,AGB-Bb02和AGB-Bb03属于同一个簇群,而AGB-Bb01属于另一个簇群。AGBBb01与来自秘鲁库斯科的U TEX 2441及澳大利亚新南威尔士的Jillamatong聚为1个分支(支持率100,遗传距离值0.002 3,序列相似性0.988 4);源自云南抚仙湖的AGB-Bb02先与源自泰国的Songkla Nakarin聚群(支持率93,遗传距离值0.153 9,序列相似性0.804 4),再与来自科特迪瓦的Yamoussoukro聚在一起;源自湖北武汉的A GB-Bb03则在与A yame聚群后(支持率为100,遗传距离值0.004 5,序列相似性0.990 9),再依次与2株来自美国明尼苏达的FDCC CH28、CH86聚为1个分支。另外,系统发生树还显示丛粒藻2大簇群各包含7个藻株,而每个簇群又可进一步细分为不同的亚簇,表明不同藻株之间遗传上的多样性。

表5 基于ITS区序列的不同丛粒藻藻株的遗传距离和序列相似性Table 5 Genetic distances and sequence similarities of different strains based on ITS region sequences
3 讨论
3.1 丛粒藻的分类鉴定技术
微藻藻种鉴定工作要求研究者具有丰富的经验,所依据的形态特征需要借助光学显微镜来观察,某些细节特征还需进一步借助电子显微镜。但由于微藻细胞形态的可塑性,在不同的环境下往往出现明显的差异,因此依据形态的鉴定结果常常出现同种异名或同名异种的情况。利用基因序列分析判断藻株的亲缘关系是形态观察的辅助手段,特别是形态上难以区别的藻株,二者结合能够对微藻分类提供更多的依据,结果的准确性也更有保证。
丛粒藻的鉴定方法主要有形态观察、基因序列分析及化学成分检测3种。根据不同地理株形态上的差别,Komarek和M arvan[16]将丛粒藻属分为13个种。本研究也表明不同地理来源的丛粒藻藻株在细胞大小、聚落大小和聚落细胞数目方面存在较明显的差异,电镜观察不同藻株的亚显微结构也显示其杯状鞘厚度及细胞包埋程度存在差异。但是否可依据这些特征将其分为不同的种,仍需开展深入的研究。
丛粒藻在代谢过程中能够合成不同的烃类,研究发现不同的藻株所合成烃的种类具有特异性,因此Plain等人提出[17]将产烃种类相同的种归为同一化学族(Race)。根据所合成烃类的类型,目前将丛粒藻分为A、B、L和Gb 4个化学族[18-20]。
本研究表明,丛粒藻18S rDNA位点具有一定的多态性,而ITS区位点多态性极高,因此将二者结合起来可用于从属、种至地理株系等不同分类阶元的分子鉴定。本研究依据核苷酸序列信息所有藻株可分为2大簇群和4个亚群,但分子鉴定的划分方式与依据形态特征区分的种、亚种或地理种的对应关系尚待进一步的研究。Senousy等人[2]分析了不同化学族藻株的18S rDNA序列,发现利用核苷酸序列区分的类群与化学分类方法具有一致性。因此,利用分子遗传学方法鉴定藻株的基因型,可迅速了解其化学组成特性,提高了研究效率。
3.2 丛粒藻遗传多样性
核基因组中的18S rRNA、ITS区序列和叶绿体基因组中的rbcL基因、matK基因、trnL内含子和trnLF基因间隔区、ndhF基因、atpB基因以及线粒体基因组中的cox I、atpA基因是目前植物分子系统学研究中的常用片段[21]。本文共分析了16株丛粒藻藻株的18S rDNA序列和ITS区序列,结果未见序列完全相同的藻株,结合形态学观察显示丛粒藻遗传多样性较高,也表明分布于世界不同地点的丛粒藻存在一定程度的地理隔离。
丛粒藻作为1种具有潜在应用价值的能源微藻,制约其应用的最大的障碍在于其生长速度很慢,但丛粒藻丰富的遗传多样性为分离筛选快速生长藻株,或通过遗传育种技术培育速生藻株提供了丰富的遗传材料。
[1] 曾呈奎,毕列爵,高亚辉,等.藻类名词及名称[M].2版,北京:科学出版社,2005,77.
[2] Senousy H H,Beakes G W,Hack E.Phylogenetic Placement of Botryococcus braunii(Trebouxiophyceae)and Botryococcus sudeticus isolate U TEX 2629(Chlorophyceae)[J].Journal of Phycology,2004,40(2):412-423.
[3] Wake L V,Hillen L W.Nature and hydrocarbon content of bloomsof the alga Botryococcus braunii occurring in australian freshwater lakes[J].Journal of Marine and Freshwater Research,1981,32(3):353-367.
[4] Brow n A C,Knights B A,Conway E.Hydrocarbon content and its relationship to physiological state in the green alga Botryococcus braunii[J].Phytochemistry,1969,8(3):543-547.
[5] 周凤英,彭德华,边立增,等.柴达木盆地未熟—低熟石油的生烃母质研究新进展[J].地质学报,2002,76(1):107-113.
[6] 贺振建,贾凤华,蒋光秀,等.新疆和田杜瓦地区葡萄藻化石的发现及其意义[J].新疆石油地质,2004,25(4):400-402.
[7] Ji L M,Yan K,M eng F W,et al.The oleaginous Botryococcus from the triassic yanchang formation in ordos basin,Northwestern China:morphology and its paleoenvir on mental significance[J].Journal of Asian Earth Sciences,2010,38(5):175-185.
[8] Held W,Peter M,Buhs C.Production of Hydrocarbon from Biomass[J].Energy Biomass,1985,25(3):744-748.
[9] Banerjee A,Sharma R,Chisti Y,et al.Botryococcus braunii:a renew able source of hydrocarbons and other chemicals[J].Critical Review s in Biotechnology,2002,22(3):245-279.
[10] 毕列爵,胡征宇.中国淡水藻志第八卷绿藻门绿球藻目(上)[M].北京:科学出版社,2004:111-115.
[11] Gelpi E,Schneider H,Mann J,et al.Hydrocarbons of geochemical significance in microscopic algae[J].Phytoche mistry,1970,9(3):603-612.
[12] Metzger P,Berkaloff C,Casadevall E,et al.Alkadiene-and botryococcene-producing races of wild strainsof Botryococcusbraunii[J].Phytochemistry,1985,24(10):2305-2312.
[13] Metzger P,Allard B,Casadevall E,et al.Structure and chemistry of Anew chemical race of Botryococcusbraunii(Chlorophyceae)that produces lycopadiene,Atetraterpenoid hydrocarbon[J].Journal of Phycology,1990,26(2):258-266.
[14] 张菊平,张兴志.植物游离细胞的扫描电镜样品的制备法[J].生物学通报,2008,43(8):56.
[15] Marmur J.A Procedure for the isolation of the deoxyribonucleic acid from micro-organisms[J].Journal of Molecular Biology,1961,3(2):208-218.
[16] Komarek J,Marvan P.Morphological differences in natural populations of the genus Botryococcus(Chlorophyceae)[J].A rchivfur Protistenkunde,1992,141(1-2):65-100.
[17] Plain N,Largeau C,Derenne S,et al.Morphological variability of Botryococcus braunii(Chlorococcales,Chlorophyta):correlations with grow th conditions and lipid content[J].Phycologia,1993,32(4):259-265.
[18] Inoue S,Dote Y,Sawayama S,et al.Analysisof oil derived from liquefaction of Botryococcusbraunii[J].Biomass and Bioenergy,1994,6(4):269-274.
[19] Templier J,Largeau C,Casadevall E.Biosynthesis of n-alkatrienes in Botryococcusbraunii[J].Phytochemistry,1991,30(7):2209-2215.
[20] Simpson A J,Zhang X,Kramer R,et al.New Insights on the Structure of Algaenan from Botryococcus brauniirace A and its hexane insoluble botryals based on multidimensional NMR spectroscopy and electrosp ray-mass spectrometry techniques[J].Phytochemistry,2003,62(5):783-796.
[21] 张峰.用于植物分子系统学研究的基因片段[J].山东科学,2004,17(1):55-58.

