真菌甲壳素脱乙酰酶(CDA)研究进展
朱利平,黄惠莉
(华侨大学化工学院,福建厦门361021)
真菌甲壳素脱乙酰酶(CDA)研究进展
朱利平,黄惠莉*
(华侨大学化工学院,福建厦门361021)
甲壳素脱乙酰酶(Chitin deacetylase,CDA)可以脱去甲壳素上N-乙酰-D-葡糖胺的乙酰氨基,使之转化成壳聚糖。壳聚糖是一种具有特殊性质的生物多聚物,在食品、医药、化工等领域具有广泛的应用前景,它也是很好的食物纤维,对人体生理活动起着多方面的调节作用,在食品工业和医药方面有重要应用价值。本文概述了真菌中CDA的研究进展,包括真菌CDA的来源、纯化和酶学性质,在生物体内的功能、基因、作用方式和结构机制,并探讨了今后研究的方向。
甲壳素脱乙酰酶,甲壳素,壳聚糖
甲壳素是2-乙酰氨基-2-脱氧-β-D-葡萄糖(GlcNAc)通过(1→4)糖苷键连接形成的线性同聚物,是自然界中含量最丰富、最易获得、可再生的聚合物之一,仅次于纤维素。但是甲壳素不溶于水和有机溶剂,所以其没有太大的商业价值,在工业上没有重要的应用,而且甲壳素的堆积会造成蚊蝇滋生、河流污染等环境问题。但是甲壳素N端脱乙酰基后的衍生物—壳聚糖具有广泛的用途[1-3],而且随着社会的发展和人们对健康的认识,对壳聚糖的品质和产量的需求也与日俱增,生产壳聚糖及其寡聚物具有相当大的商业前景。但是目前为止,工业上生产壳聚糖一般是通过热化学方法脱去甲壳素上的乙酰基而实现的。这个过程耗能大,需要大量的强碱,排放的废水对环境有很大的危害,并且整个脱乙酰过程不易控制,多糖在热碱中易降解,会导致产出的壳聚糖分子量和脱乙酰度不均一。相对于化学法,利用生物酶法生产壳聚糖已经引起了广泛的关注,这种方法具有新颖、环境友好、无降解以及可以形成特定位点脱乙酰的产物等优点,其中甲壳素脱乙酰酶起重要作用。甲壳素脱乙酰酶,简称 CDA(EC3.5.1.41),它可以对甲壳素上的GlcNAc进行脱乙酰作用,催化甲壳素转化为壳聚糖(图1),在CAZY数据库中,CDA属于糖脂酶家族4(CE-4s)成员[4],这种酶是在Mucor rouxii提取物中首次发现的,随后在其他真菌[5,6-9]和昆虫[8]中都有关于CDA的报道。本文主要综述了真菌CDA的酶学性质,在生物体内作用、基因水平以及作用方式和结构机制,并对CDA的应用前景和发展方向做了简单的展望。
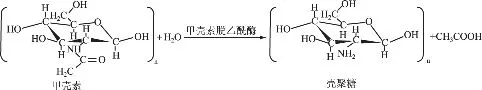
图1 甲壳素脱乙酰酶催化方式[1]
1 真菌CDA纯化与酶学性质
不同真菌CDA的分子量为27~150kDa,最适温度为37~60℃,最适pH为4.5~8.5,具有激活剂和抑制剂不同(表1)。
大多数的真菌CDA都具有一定的热稳定性[12-14,17],并且是糖蛋白[14-15,17-18],但是也报道称 CDA存在不含糖的活性形式,E.coli中克隆表达的Saccharomyces cerevisiae CDA2基因时[16],发现纯化的重组酶是单体形式,分子量是35kDa,与S.cerevisiae的天然Cda2p去糖基化后的分子量一致,缺少CoCl2的重组酶就会失活,而CoCl2对S.cerevisiae的天然酶并不是必需的,只是起激活作用[15],并且S.cerevisiae的天然Cda2p脱糖基化后完全失活,加入CoCl2后酶活才恢复;利用质谱分析重组 P.pastoris中表达的C.lindemuthianum UPS9 CDA[19],发现其不含糖类,而且不需要糖基化和 Co2+就可以表现出活性,但是Co2+有显著的激活作用。
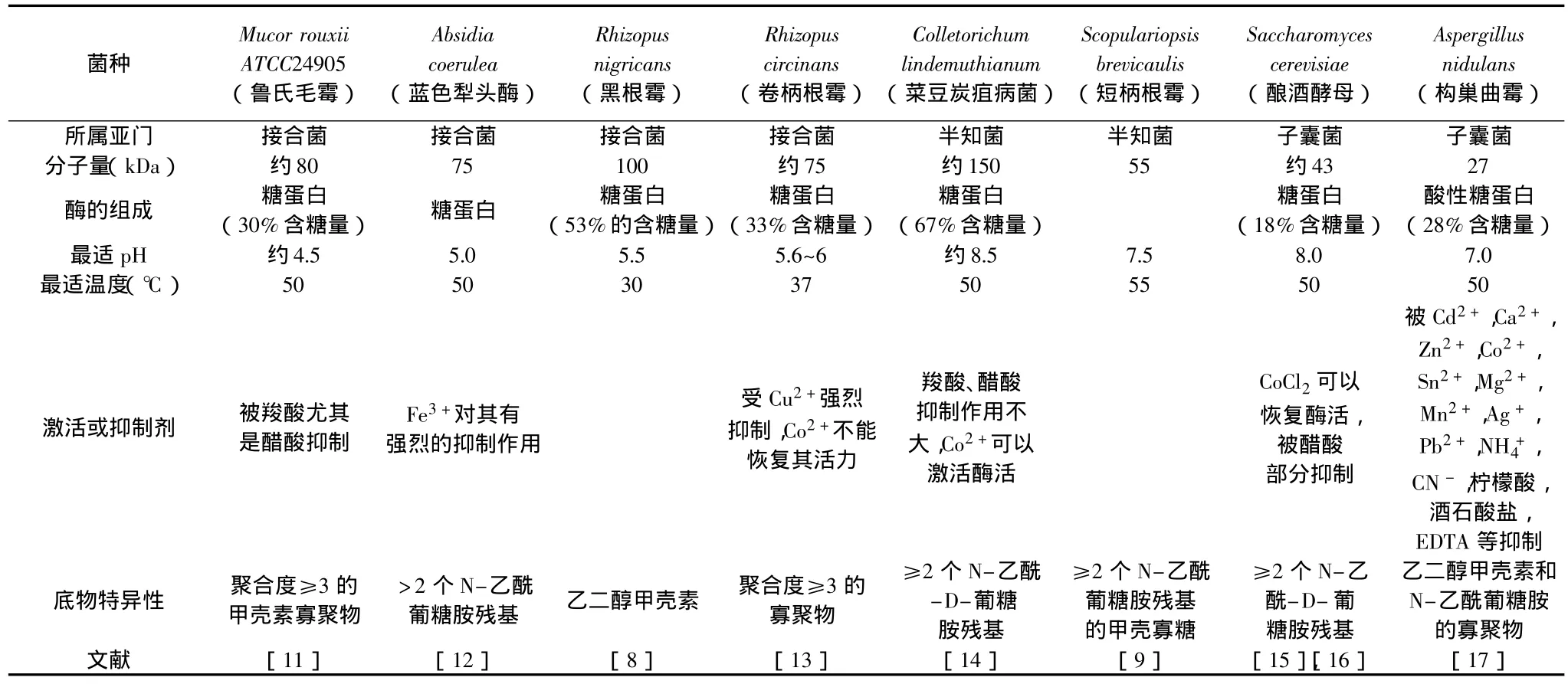
表1 不同菌株的甲壳素脱乙酰酶性质
在一些体外实验中,A.coerulea CDA在体外合成可以对壳聚糖的转化率达到90%,这与A.coerulea体内壳聚糖脱乙酰作用的程度(95%)极为接近,说明此CDA在体外具有很好的转化活性。另外 C. lindemuthianum CDA[20]具有一个很有趣的性质,此CDA在3.0mol/L醋酸钠存在时会乙酰化自由的氨基糖残基,使之转化为N-乙酰化形式,即通过逆水解反应将GlcN2合成GlcNAcGlcN。这为研究酶N-乙酰化氨基糖提供了一个新的方法,还可以合成一个不常见的N-乙酰化的产物(GlcNAcGlcN),同时这也是对CDA能力多样化的一个重要发现。
2 真菌CDA在生物体内的功能
关于真菌CDA在生物体内的功能研究集中于C.lindemuthianum 和 S.cerevisiae比较多。在 C. lindemuthianum中研究发现,CDA主要参与病原性入侵的过程,而在S.cerevisiae中CDA主要是参与正确的合成孢子壁中的壳聚糖成分,这说明在不同亚门的真菌中CDA的生物学功能不同,所以有必要进一步的研究CDA在其他亚门中的作用,对其生物学功能多样性做进一步的研究。
Iason Tsigos等[14]提出了C.lindemuthianum中的CDA可能在此植物病原菌入侵植物的机制中起作用。甲壳素寡聚物可以激活植物防御机制,但是这些寡聚物的脱乙酰形式不具有激活植物防御功能的作用,CDA的作用就是对这些寡聚物进行脱乙酰作用,所以C.lindemuthianum的CDA可能是在胞外将寡聚物转化成其脱乙酰的形式,这样就可以避免被植物的几丁质酶降解,从而保证真菌顺利地侵入植物。此外,与CDA作用的底物只有第二个GlcNAc糖被脱乙酰化[21],作用后的产物既不能被几丁质酶水解也不能被壳聚糖酶水解。因此 CDA可能是C.lindemuthianum的一个重要毒性因子,当 C. lindemuthianum侵入并在宿主组织中繁殖时,CDA可以伪装它以免被宿主的多糖酶水解掉。
CDA对形成正确的S.cerevisiae子囊孢子壁具有重要作用[22],CDA突变的孢子对水解酶、乙醚和热相当的敏感。S.cerevisiae中有两个CDA,即Cda1p和Cda2p,它们只在S.cerevisiae孢子形成期专一性表达,Cda2p在孢子细胞壁的脱乙酰作用中起主要作用,而Cda1p可能对过程起精细调控作用。Yasuhiro Matsuo等[23]在Schizosaccharomyces pombe中也报道了类似现象,破坏编码CDA的Cda1+基因之后,菌株不能正确的形成孢子,说明S.pombe正确形成孢子需要CDA,这与CDA在酿酒酵母的作用相一致。
3 真菌CDA的基因
关于CDA基因研究在Saccharomyces cerevisiae[16]、Gongronella butleri[6]、Rhizopus nigricans[8]、Rhizopus circinans[13]、 Colletotrichum lindemuthianum[6,24]、Schizosaccharomyces pombe[23]、甚 至 于 Tribolium castaneum[10]中都有报道。很多研究都是在E.coli[16,24]和Pichia pastoris[13,19]中克隆表达重组CDA,并且多个菌株CDA的cDNA文库已经构建成功[6,8,13]。
在S.cerevisiae中有两个CDA基因[16]:CDA1和CDA2,它们在孢子形成期的不同时间限制性表达。敲除这两个基因的细胞在形成孢子时没有CDA活力,这说明酵母中没有其他的基因编码CDA。但同时有另外两个基因DIT1和DIT2也在子囊孢子壁形成时特异性表达,与CDA基因相似。这说明产孢子特异性基因的关键调节因子(包括DIT1和DIT2,IME1和IME2)应该在CDA基因的表达中扮演重要角色。野生型细胞和一个△cda被破坏的细胞在荧光、蜗牛酶敏感性、热激、乙醚等方面没有区别,这意味着一个CDA基因对足以正确的形成孢子,但是残余的CDA活力近似原来的一半。如果两个CDA基因都被敲除后,菌株会对上述反应敏感,并且经酶水解后会失去自然荧光。根据与M.rouxii的CDA序列相似性对比,逐步的去除N或C端的保守区域,发现去除N端12个或更多氨基酸会导致酶活完全丧失,这可能是由于成熟酶N端氨基酸涉及蛋白的正确折叠;去掉C端7个氨基酸没有明显的酶活丧失,去除27个或更多个C端氨基酸会导致酶失活。
C.lindemuthianum CDA的基因开放阅读框是由一个N端27个氨基酸的前序列和成熟CDA组成,成熟酶的氨基酸序列与鲁氏毛霉中的CDA有26%是一致的,46%是相似的,由氨基酸序列数据推测的蛋白质分子大小为24.3kDa。将此基因克隆至E.coli中[17],利用S.lividans几丁质酶的信号肽,蛋白质成功的定位于胞外,并且在预计的位点被大肠杆菌信号肽酶切除。生产出活性形式的CDA,并且与原来的酶Km和kcat值相似。尽管Sl-CDA的N端或C端的修饰作用会导致活力下降,但是没有观察到失活或聚合的现象。
对G.butleri CDA基因的cDNA文库整个基因测序[6],发现其包含一个1290个核苷酸的开放阅读框,可编码430个氨基酸残基,在其中间区域含有一个编码多聚糖脱乙酰酶的核苷酸序列,占整个基因序列的34%。根据初级结构推测在氨基酸序列中有9个可能的N端糖基化位点,其中6个(Asn75,Asn95,Asn157,Asn296,Asn312,Asn341)与Mucor rouxii的CDA相应位点一致。为了测定CDA之间的进化关系,对八种产CDA菌进行了同源进化树分析,发现CDA形成的簇刚好与真菌的分类相吻合。这说明在不同产CDA的真菌中,各酶虽具有相似的功能但其进化关系不同。测序R.nigricans中CDA基因的cDNA文库[8],发现整个基因包含了一个1341个核苷酸的开放阅读框,可以编码447个氨基酸残基。核苷酸序列中间有一个编码保守多聚糖脱乙酰酶的区域,占总序列的34%,有八个可能的N端连接的糖基化位点。
从一个编码CDA的R.circinans cDNA文库[13]中分离出三个CDA的cDNA(RC,D2和I3/2)并对其测序。将其分别克隆进P.pastoris的表达系统中,虽然利用蛋白质印迹免疫检测发现所有的重组克隆都分泌重组蛋白,但是只在含有RC片段的培养基上清液中检测出酶活。多聚组蛋白标记的RC CDA通过一步可再生性过程被纯化至电泳纯,尽管在重组蛋白的N端含有多聚组氨酸标记,纯化的蛋白仍具有高的活力。这是首次在同一接合菌中分离多个可能的CDA,而且发现R.circinans中可能含有更多的CDA,这也是首次提出真菌CDA可能是由一个多基因家族编码。
4 真菌CDA的作用方式和结构机制
关于真菌CDA的作用方式的研究和推测在比较早的时候就有报道,利用高效液相色谱和1H核磁共振光谱对M.rouxii CDA脱乙酰后产物的结构进行了研究[25],此M.rouxii CDA对聚合度小于3的甲壳素寡聚物不能有效地脱乙酰化,只有(GlcNAc)4和(GlcNAc)5完全脱乙酰化,而(GlcNAc)3、(GlcNAc)6和(GlcNAc)7的还原端残基仍然是完整的。推测酶是先去除实验所用甲壳素寡聚物非还原端残基上的一个乙酰基,然后进一步按顺序水解下一个乙酰基,之后它与底物分离并与另一条甲壳素寡聚物形成活性复合体。脱乙酰作用的程度依赖于底物的长度,而对于(GlcNAc)4和(GlcNAc)5,酶-底物复合物的几何构型可能更适合CDA对底物完全脱乙酰化,或长或短的寡聚物还原端残基都不能被脱乙酰化。
C.lindemuthianum cDAH[18]含有四个次级位点-2,-1,0,+1,其中0位点是催化性位点,次级位点-2对GlcNAc单位上N-乙酰基识别作用强烈。利用连续的分光光度计法实验和ESI-MS相结合,直接分析cDAH作用(GlcNAc)2~6后的产物,发现这些次级位点与GlcNAc残基的作用是从底物的非还原端到还原端进行的。其中C.lindemuthianum UPS9 CDA有一个单核金属酶的区域[21],它含有一个结合锌的保守组氨酸-组氨酸-天冬氨酸三联体,此三联体与保守的催化碱(Asp)和酸(His)紧密结合完成酸碱的催化。数据表明C.lindemuthianum具有一个高度保守的底物结合沟,具有微妙的选择性,此选择性可以影响底物的特异性和次级位点亲和性,并且发现此酶至少要被(GlcNAc)2占据0和+1次级位点才具有活性。
5 真菌CDA的应用展望
关于甲壳素脱乙酰酶的研究国内的一些研究人员[26-28]也做出了积极的贡献。真菌体内,CDA在不溶性甲壳素转化成可溶性壳聚糖过程中起主要作用,CDA的存在可以保证病原性真菌成功地入侵植物,CDA在细胞增殖过程中也起到重要的作用。同时,由于CDA作用过后形成的壳聚糖具有很大的商业价值,使得CDA的工业化应用引起了广泛的关注。
关于真菌CDA的研究可以为工业化应用提供理论依据,结合现代生物学技术可以产生大量的、不同性质的CDA,可以制备新型的壳聚糖聚合物和寡聚物,这对于生物医学、食品添加剂、医药、天然杀虫剂、抗菌剂、金属胶合生物聚合物、化妆品[1]等方面的发展都有巨大促进作用。在食品工业中,壳聚糖及其衍生物具有很强的抑菌[29-30]、保鲜作用[31-34],而且对人体无任何不良作用,是一种理想的保鲜和防腐剂,已广泛应用于鲜鱼、水果、蔬菜等生鲜食品和豆制品以及酱油、醋等调味品的防霉保鲜,并可用作食醋的防沉淀剂,原料糖汁的纯化剂,面包和饼干等食品的添加剂。在牙膏、漱口水及口香糖中添加壳聚糖,有预防牙周炎、除去或减轻口臭的作用。
但是由于此酶的底物范围较窄[36],对于一般不溶性甲壳素的脱乙酰化程度并不高,现阶段的研究还处于实验室阶段,需要尝试一些新的方法促进CDA的应用,例如寻找底物范围更广的产酶菌、固定化[36]、和其他真菌杀虫剂联用[7]、寻找其他更适合工业化应用的菌株(如细菌)等。现在关于CDA的研究主要集中在陆生性真菌和一些病原性真菌中,对于一些微生物丰富的地方(如沿海地区)应该积极地探索新的产CDA的菌源(如海洋微生物),同时这些区域的甲壳素类物质较多可直接进行商业化生产和应用,减少成本,获得良好的社会效益和经济效益。
[1]TsigosI,Martinou A,KafetzopoulosD,etal.Chitin deacetylases:New,versatile tools in biotechnology[J].Trends Biotechnol,2000,18(7):305-312.
[2]乔德亮.低聚壳聚糖制备及其在功能食品中应用[J].食品工业科技,2007,28(4):228-231.
[3]王瑾,李默馨,李红玲,等.壳聚糖/海藻酸钠固定化β-葡萄糖苷酶的研究[J].食品工业科技,2009,30(3):164-167.
[4]Coutinho P M,Henrissart B.Carbohydrate-active enzymes:an integrated database approach[M].Edited by:Gilbert H J,Davies G,Henrissart B,Svensson B.Recent Advances in Carbohydrate Bioengineering.Cambridge:The Royal Society of Chemistry,1999:3-12.
[5]Kauss H,Jeblick W,Young D H.Chitin deacetylase from the plant pathogen Colletotrichum Lindemuthianum[J].Plant Sci Lett,1983,28(2):231-236.
[6]Maw T,Tan T K,Khor E,et al.Complete cDNA Sequence of Chitin Deacetylase from Gongronella butleri and its Phylogenetic Analysis Revealed Clusters Corresponding to Taxonomic Classification of Fungi[J].J Biosci Bioeng,2002,93(4):376-381.
[7]Nahar P,Ghormade V,Deshpande M V.The extracelluar constitutive production of chitin deacetylasein Metarhizium anisopliae:possible edge to entomopathogenic fungiin the biological control of insect pests[J].J Invertebr Pathol,2004,85(2):80-88.
[8]Jeraj N,KunicˇB,Lenasi H,et al.Purification and molecular characterization of chitin deacetylase from Rhizopus nigricans[J]. Enzyme Microb Tech,2006,39(6):1294-1299.
[9]Cai J,Yang J,Du Y,et al.Purification and characterization of chitin deacetylase from Scopulariopsis brevicaulis[J].Carbohyd Polym,2006,65(2):211-217.
[10]Dixit R,Arakane Y,Specht C A,et al.Domain organization and phylogenetic analysis of proteins from the chitin deacetylase gene family of Tribolium castaneum and three other species of insects[J].Insect Biochem Molec,2008,38(4):440-451.
[11]Tsigos I,Zydowicz M,Domard A,et al.Mode of action of chitin deacetylase from Mucor rouxii on N -acetylchitooligosaccharides[J].Eur J Biochem,1999,261(3):698-705.
[12]Gao X D,Katsumoto T,Onodera K.Purification and Characterization of Chitin Deacetylase from Absidia coerulea[J].J Biochem,1995,117(2):257-263.
[13]Gauthier C,Clerisse F,Dommes J,et al.Characterization and cloning of chitin deactylases from Rhizopus circinans[J].Protein Expres Purif,2008,59(1):127-137.
[14]Tsigos I,Bouriotis V.Purification and Characterization of Chitin Deacetylase from Colletorichum lindemuthianum[J].J Biol Chem,1995,270(44):26286-26291.
[15]Martinou A,KoutsioulisD,BouriotisV.Expression,Purification and Characterization of a Cobalt-Activated Chitin Deacetylase(Cda2p)from Saccharomyces cerevisiae[J].Protein Expres Purif,2002,24(1):111-116.
[16]Martinou A,Koutsioulis D,Bouriotis V.Cloning and expression of a chitin deacetylase gene( CDA2)from Saccharomyces cerevisiae in Escherichia coli Purification and characterization of the cobalt-dependent recombinant enzyme[J].Enzyme Microb Tech,2003,32(6):757-763.
[17]Alfonso C,Nuero O M,Santamaría F,et al.Purification of a Heat-Stable Chitin Deacetylase from Aspergillua nidulans and Its Role in Cell Wall Degradation[J].Curr Microbiol,1995,30(1):49-54.
[18]Tokuyasu K,Mitsutomi M,Yamaguchi I,et al.Recognition of Chitooligosaccharides and Their N-acetyl Groups by Putative Subsites of Chitin Deacetylase from a Deuteromycete,Colletotrichum lindemuthianum[J].Biochemistry,2000,39(30):8837-8843.
[19]Shrestha B,Blondeau K,Stevens W F,et al.Expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris:purification and characterization[J].Protein Expres Purif,2004,38(2):196-204.
[20]Tokuyasu K,Ono H,Hayashi K,et al.Reverse hydrolysis reaction of chitin deacetylase and enzymatic synthesis of β-DGlcNAc-(1→4)-GlcN from chitobiose[J].Carbohyd Res,1999,322(1-2):23-31.
[21]Blair D E,Hekmat O,Schüttelkopf A W,et al.Structure and Mechanism of Chitin Deacetylase from the Fungal Pathogen Colletotrichum lindemuthianum[J].Biochemistry,2006,45(31):9416-9426.
[22]Christodoulidou A,Briza P,Ellinger A,et al.Yeast ascospore wall assembly requires two chitin deacetylase isozymes[J].Febs Lett,1999,460(2):275-279.
[23]Matsuo Y,Tanaka K,Matsuda H,et al.cdal+,encoding chitin deacetylase is required for proper spore formation in Schizosaccharomyces pombe[J].Febs Lett,2005,579(12):2737-2743.
[24]Tokuyasu K,Kaneko S,Hayashi K,et al.Production of a recombinantchitin deacetylase in the culture medium of Escherichia coli cell[J].Febs Lett,1999,458(1):23-26.
[25]Tsigos I,Zydowicz M,Domard A,et al.Mode of action of chitin deacetylase from Mucor rouxii on N -acetylchitooligosaccharides[J].Eur J Biochem,1999,261(3):698-705.
[26]蔡俊,杜予民,杨建红,等.甲壳素脱乙酰酶产生菌的筛选及产酶条件[J].武汉大学学报:理学版,2005,51(4):485-488.
[27]蒋霞云,周培根,李燕.几种霉菌产甲壳素脱乙酰酶活力比较及部分酶学性质[J].上海水产大学学报,2006,15(2):211-215.
[28]蒋霞云,邹曙明,周培根.总状毛霉(Mucor racemosus)甲壳素脱乙酰酶全长cDNA的克隆及序列分析[J].农业生物技术学报,2007,15(6):981-985.
[29]屠洁,刘冠卉,程曦.壳聚糖的抑菌效果研究及其与苯甲酸钠的比较[J].食品工业科技,2008,29(5):83-85.
[30]吴清平,陈威,张菊梅,等.壳聚糖衍生物抗菌活性研究进展[J].食品科学,2009,30(5):269-272.
[31]王娟慧,谭兴和,熊兴耀,等.壳聚糖涂膜对鲜切马铃薯保鲜效果的影响[J].食品工业科技:2007,28(8):215-218.
[32]纪淑娟,高倩.羧甲基壳聚糖的制备及其在黄瓜保鲜中的应用研究[J].食品工业科技,2008,29(2):201-203.
[33]万丽,申琳,赵丹莹,等.壳聚糖复合涂膜对鲜切冬枣安全性与贮藏品质的影响[J].食品科学,2009,30(4):277-281.
[34]王兰菊,屠琼芳,刘颖,等.壳聚糖涂膜对鲜切山药品质的影响[J].食品工业科技,2009,30(4):309-311.
[35]Win N N,Stevens W F.Shrimp chitin as substrate for fungal chitin deacetylase[J].Appl Microbiol Biot,2001,57(3):334-341.
[36]Jaworsha M M,Bryjak J,Liesiene J.A search of an optimal carrier for immobization of chitin deacetylase[J].Cellulose,2008,16(2):261-270.
Progress of research on fungi chitin deacetylase
ZHU Li-ping,HUANG Hui-li*
(College of Chemical Engineering,Huaqiao University,Xiamen 361021,China)
Chitin deacetylase(CDA)is an enzyme that catalyzes the hydrolysis of acetamine groups of N-acetyl-D -glucosamine in chitin,converting it to chitosan.Chitosan is a biopolymer of unique properties which has bright prospects in industries such as food,pharmaceuticals,chemical and so on.lt’s also a good food fiber which regulates certain activities in human body,the application value of chitosan in food and pharmaceutical industry should be given enough attention.The latest advances of CDAs of fungi,including sources,purification and characterization,biological role,genes,mode of action were presented.And the direction of future research was also discussed.
chitin deacetylase;chitin;chitosan
TS201.2+5
A
1002-0306(2010)11-0394-05
2009-09-28 *通讯联系人
朱利平(1986-),男,硕士研究生,研究方向:海洋微生物资源在食品方面的应用。
福建省自然科学重点项目基金(2007T010)。

