PCR检测牛乳中大肠杆菌基因组DNA的提取方法
宋宏新,邹联柱,马 冬,薛海燕
(1.陕西科技大学生命科学与工程学院,陕西 西安 710021;2.华北煤炭医学院,河北 唐山 063000)
PCR检测牛乳中大肠杆菌基因组DNA的提取方法
宋宏新1,邹联柱1,马 冬2,薛海燕1
(1.陕西科技大学生命科学与工程学院,陕西 西安 710021;2.华北煤炭医学院,河北 唐山 063000)
为获得一种适于牛乳样品大肠杆菌PCR检测的基因组DNA提取方法,对饱和酚法、CTAB法、试剂盒法及溶剂裂解法等4种提取方法加以比较,通过考察DNA的纯度、定量分析以及PCR分析,确定一套有效、快速、适合牛乳中提取大肠杆菌的基因组DNA方法——溶剂热裂解法。结果显示该方法提取的DNA模板,适于PCR扩增大肠杆菌DNA,可以用于牛乳中的大肠杆菌检测。
乳品;大肠杆菌;基因组总DNA;聚合酶链式反应
伴随着近几年人们对食品中微生物污染与危害的公共意识提高,快速、灵敏和可靠的检测食源性病原微生物方法发展迅速,基于核酸分析的PCR方法成为确定和检测食品中微生物,甚至死菌的可靠方法[1-3]。但是,直接检测乳品样品中细菌通常容易受到来自富集培养基、DNA分离剂、食品基质本身和其他非目标微生物等PCR抑制因子的影响[4-6]。因此研究从乳品中高效提取DNA的方法,消除 PCR反应抑制因子,对利用PCR直接检测食品中的致病菌[7-9]具有重要意义。本研究拟比较4种常用大肠杆菌基因组DNA提取方法在乳品PCR检测中的应用,为PCR直接检测乳中大肠杆菌及其他病原菌提供参考。
1 材料与方法
1.1 材料、试剂与仪器
非致病性大肠杆菌ATCC 25922菌株 中国医学科学院菌种保藏中心;液态牛乳样品 市购。
蛋白酶K、DNA Marker DL2000 北京天根生物有限公司;PCR buffer、dNTPs、Taq酶 大连宝生物科技有限公司;L B培养基试剂、酚、氯仿、异丙醇、无水乙醇、石油醚和氨水均为国产分析纯。
PCR仪 美国MJ公司;水平电泳槽 北京六一仪器厂;JD-801凝胶成像系统 南京捷达科技公司。
1.2 方法
1.2.1 菌种活化及牛乳样品制备
将大肠杆菌ATCC 25922菌种经二次斜面活化再挑取单菌落,在LB液体培养基摇床过夜培养(37℃、100r/min),经平板菌落计数后4℃冰箱保藏;牛乳样品经巴氏灭菌后按国标法检测证实无大肠杆菌,每份取1mL灭菌乳,接种10μL LB培养基(平板法测得大肠杆菌数为108CFU),分别采用4种方法提取大肠杆菌DNA。
1.2.2 提取方法
将人工污染乳样室温下1200×g离心10min,弃上清液,沉淀4℃保存。
1.2.2.1 酚/氯仿抽提法[5]
①取1mL人工污染乳样于Eppedorf管中,4000r/min室温离心10min,去上清液;②取500μL STE溶液悬浮细胞,加入20% SDS 100μL,37℃条件下轻摇过夜,使细胞裂解;③加入等体积的饱和酚混合均匀,放置5min后3500r/min离心10min;④去上相,加入等体积的酚/氯仿,如步骤③离心;⑤取上相,加入等体积的氯仿,如步骤④;⑥取上相转入干净的Eppendorf管中,加入2倍体积的冷乙醇,-20℃放置10min,4℃、12000r/min离心10min,弃上清液;⑦用70%乙醇洗一次,4℃、12000r/min离心10min,弃乙醇,室温倒置10min,用50μL水或TE溶液溶解DNA。
1.2.2.2 CTAB法[6]
①加入567μL的TE缓冲液悬浮,加入30μL质量浓度100g/L SDS和3μL 20mg/mL蛋白酶K,混匀,37℃、1h;②加入100μL 5mol/L NaCl,充分混匀,再加入80μL CTAB/NaCl(质量浓度50g/L CTAB,0.5mol/L NaCl)混匀,65℃温浴10min;③加入等体积的酚-氯仿-异戊醇(质量比为25:24:1)抽提;④加入0.6倍体积的异戊醇混匀,沉淀DNA,离心沉淀用70%(V/V)乙醇洗2次,再用TE溶解沉淀,4℃备用或-20℃保存。1.2.2.3 UNIQ-10株式细菌基因组DNA抽提试剂盒
①取1mL人工污染乳10000×g室温离心1min,弃上清液;②沉淀加入含溶菌酶的TE 200μL, Digestion Buffer 400μL,蛋白酶K 3μL,混匀55℃保温5min;③加入260μL无水乙醇混匀,然后将样品全部转移到套放于2mL收集管的UNIQ-10柱中,室温8000×g离心1min;④弃去收集管中废液,向柱中加入500μL Wash Solution,室温10000×g离心30s,重复步骤④一次,弃去收集管中的全部废液,10000×g室温离心1min,去除残留Wash Solution;⑤将柱放入洁净Eppedorf管中,加入50μL Elution Buffer,室温2min,10000×g离心1min,离心管中的液体即为基因组DNA,根据用途样品可以4℃或-20℃保存。
1.2.2.4 溶剂提取热裂解法[7]
①1mL人工污染样品加入0.2mL无水乙醇、0.2mL氨水和0.2mL石油醚混匀后12000×g离心10min;②沉淀用300μL 10mmol/L TE(pH7.8)溶解后,加入5μL 10mg/mL溶菌酶,37℃水浴振荡1h。然后加入10% SDS 50μL煮沸5min;③加入等体积的氯仿充分振荡,13000×g离心10min,上清液用0.1倍体积 2.5mol/L乙酸铵(pH5.4)和2.5倍体积预冷无水乙醇沉淀DNA,13000×g离心20min,DNA沉淀干燥后用100μL灭菌超纯水溶解,4℃贮藏备用。
1.2.3 PCR反应
用以上不同方法所提取的DNA作为模板,进行PCR反应,用以验证上述方法是否可有效从乳品中提取大肠杆菌DNA,PCR反应使用的引物、反应条件及应用检测参见文献[8]。
PCR采用25μL体系(2.5μL 10×PCR buffer、2.5μL dNTPs、0.2μL 2.5U/μL Taq酶,引物 alr-1和引物alr-2各2μL 4umol/L,5μL模板DNA),94℃变性2min后进入PCR循环:94℃、30s,66℃、1min,72℃、1min,25个循环,72℃充分延伸补足5min。PCR以LB培养基中大肠杆菌ATCC 25922为阳性对照,以牛乳样作为阴性空白对照,PCR产物5μL在1.5%琼脂糖凝胶上进行电泳,采用凝胶成像系统观察结果,进行比较,实验电泳结果仅显示了阳性及阴性对照的溶剂裂解法样品。
2 结果与分析
2.1 基因组DNA定性和定量分析

图1 4种方法从牛乳中提取基因组DNA电泳结果Fig.1 Electrophoresis of genomic DNA in milk extracted by four methods
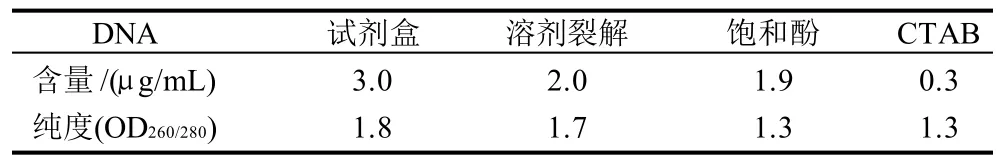
表1 不同方法得到的DNA含量和纯度Table 1 Contents and purities of genomic DNA extracted by four methods
图1为基因组DNA琼脂糖电泳图谱,表1为基因组DNA含量和纯度。从图1明显可以看出,在起始菌量相同的情况下,不同方法提取基因组DNA的效果存在明显差异,1、2、3带的亮度比4强,但是3带却在点样槽,说明在提取的过程中没有充分除掉蛋白,以致其与DNA结合从而影响在凝胶中的泳动,而且亮度与含量成比例关系,所以可以认为溶剂提取热裂解法的提取效果与试剂盒法最接近。
从表1可以看出,无论从DNA提取的量还是纯度(OD260/280阈值在1.8~2.0之间),试剂盒法和溶剂裂解法均可提供足量可靠的PCR模板。
2.2 PCR产物电泳结果
将提取的DNA进行丙氨酸消旋酶基因扩增,结果如图2所示。

图2 不同提取方法扩增结果Fig.2 PCR amplification results using genomic DNA extracted by different methods
电泳结果表明,1、2条带最亮,说明利用溶剂裂解法和试剂盒方法均能有效提取大肠杆菌基因组DNA,适合作检测乳品中大肠杆菌基因扩增的模板。
3 讨 论
影响PCR产量主要发生在3个操作阶段:分离和浓缩目标菌;模板DNA的提取效率;PCR反应条件[9]。本实验主要通过对乳样简单的离心浓缩大肠杆菌,重点研究采用不同方法提取大肠杆菌基因组DNA,有效除去PCR抑制因子及杂蛋白,最后进行PCR扩增反应及分析。
通过比较,酚/氯仿抽提法和CTAB法,需要过夜消化,全过程接近一整天,严重影响实验进度和效率,加入溶菌酶及蛋白酶K会增加检测试验成本,且需要多次酚、氯仿抽提转相,除去牛乳中杂蛋白,实验时间长,造成NDA提取率降低,可以造成DNA降解现象。试剂盒的应用在价格上相对昂贵,对于大量的实验来说大大增加了实验整体成本。因此,本实验选取上述的几种方法并从其中挑选出一套适合于在牛乳样品中提取DNA的方法——溶剂裂解法,其主要优点在于:提取的全过程大约在2h左右,节省时间;提取的基因组DNA纯度高、数量多,成本远比试剂盒少;同时此方法不使用CATB、溶菌酶、蛋白酶K等生物试剂,降低了实验成本,有利于实验的推广。通过溶剂裂解法提取的大肠杆菌基因组DNA,在应用PCR检测乳品中大肠杆菌中提供足量、可信的模板,从而完成E.coli在牛乳卫生安全上的检测研究。
[1] SCHEU P M, BERGHOF K, STAHL U. Detection of pathogenic and spoilage microorganisms in food with the polymerase chain reaction[J].Food Microbiology, 1998, 15: 13-31.
[2] JOSEPHSON K L,GERBA C P, EPPER I L. Polymerase chain reaction detection of nonviable bacterial pathogens[J]. Applied and Environmental Microbiology, 1993, 59: 3513-3515.
[3] 关晶岩, 赵新准. 原料乳样品中嗜冷菌快速PCR计数方法研究[J]. 食品工业, 2009(5): 63-66.
[3] ROSSEN L, NORSKOV P, HOLMSTROM K, et al. Inhibition of PCR by components of food samples, microbial diagnostic assays and DNA-extraction solutions[J]. Food Microbiology, 1992, 17:37-45.
[4] WILSON I G. Inhibition and facilitation of nucleic acid amplification[J].Applied and Environmental Microbiology, 1997, 63: 3741-3751.
[6] 李英军, 刘景武, 袁耀武, 等. 肉中金黄色葡萄球菌PCR检测模板的制备方法[J]. 食品与发酵工业, 2007(11): 107-110.
[7] MENG R C, BUCKLEY S A, KUBENA L F. Bactericidal effect of sodium chlorate on Escherichia coli O157:H7 and Salmonella typhimurium DT104 in rumen contents in vitro[J]. Food Prot, 2000, 63(8): 1038-1042.
[8] FITZMAURICE J, GLENNON M, DUFFY G, et al. Application of real-time PCR and RT-PCR assays for the detection and quantitation of VT1 and VT2 toxin genes in E.coli O157:H7[J]. Molecular and Cellular Probes, 2004, 18: 123-132.
[9] AL-HOLY M A, LIN M, CAVINATO A G, et al. The use of Fourier transforminfrared spectroscopy to differentiate Escherichia coli O157: H7 from other bacteria inoculated into apple juice[J]. Food Microbiology,2006, 23: 162-168.
[10] 李任强. 生物技术试验精选[M]. 广州: 暨南大学出版社, 2006.
[11] 王关林, 方宏筠. 植物工程原理与技术[M]. 北京: 科学出版社, 1998.
[12] HERMAN L M F, de BLOCK J H G E, MOERMANS R J B. Direct detection of Listeria monocytogenes in 25 milliliters of raw milk by a two-step PCR with nested primers[J]. Applied and Environmental Microbiology, 1995, 61: 817-819.
[13] 马冬, 宋宏新, 李明亮, 等. PCR检测乳品中大肠杆菌的研究[J]. 食品科学, 2009, 30(4): 260-263.
[14] CAO Anli, MUTHARIA L, RAYMOND M, et al. Improved template DNA preparation procedure for detection of Mycobacterium avium subsp.paratuberculosis in milk by PCR[J]. Microbiological Methods, 2007,69: 417-420.
Extraction Methods of PCR-detectable Escherichia coli Genomic DNA in Milk
SONG Hong-xin1,ZOU Lian-zhu1,MA Dong2,XUE Hai-yan1
(1. College of Life Science and Engineering, Shaanxi University of Science and Technology, Xi’an 710021, China ;2. North China Coal Medical University, Tangshan 063000, China)
In order to obtain an appropriate sample for PCR detection, four extraction methods including saturated phenol method, CTAB method, kit method and solvent pyrolysis method for E. coli genomic DNA in milk samples were compared.Through evaluating the purity, quantitative analysis and PCR analysis of genomic DNA extracted by four methods, an effective and fast solvent-thermal pyrolysis method was suitable for the extraction of E. coli genomic DNA in milk. The genomic DNA extracted by this method used as the template is suitable for PCR amplification of E. coli DNA, which can be used for the detection of E. coli in milk.
dairy products;Escherichia coli;total genomic DNA;PCR
TS252.1
A
1002-6630(2010)24-0308-03
2010-07-23
陕西省科学技术厅农业攻关项目(2009K02-09);陕西省教育厅产业化培育项目(09JC19)
宋宏新(1959—),男,教授,主要从事生物化学与分子生物学、食品科学研究。E-mail:hongxinsong@163.com

