通过体外分子进化技术提高淀粉液化芽胞杆菌BS5582 β-1,3-1,4-葡聚糖酶热稳定性
秦久福,高威威,李崎,李永仙,郑飞云,刘春风,顾国贤
1 江南大学 工业生物技术教育部重点实验室,无锡 214122 2 江南大学生物工程学院 酿酒科学与工程研究室,无锡 214122
通过体外分子进化技术提高淀粉液化芽胞杆菌BS5582 β-1,3-1,4-葡聚糖酶热稳定性
秦久福1,2,高威威1,2,李崎1,2,李永仙1,2,郑飞云1,2,刘春风1,2,顾国贤2
1 江南大学 工业生物技术教育部重点实验室,无锡 214122 2 江南大学生物工程学院 酿酒科学与工程研究室,无锡 214122
应用基于易错PCR随机突变的体外分子进化技术,来提高淀粉液化芽胞杆菌β-1,3-1,4-葡聚糖酶的热稳定性。利用建立的基于96微孔板高通量筛选模型,经过两轮定向进化与高通量筛选,共筛选得到3株热稳定性明显提高的突变体2-JF-01、2-JF-02和2-JF-03。将野生型β-葡聚糖酶基因和热稳定性提高的突变基因的高效表达产物经镍亲和层析柱纯化后,酶学性质测定表明突变酶2-JF-01、2-JF-02和2-JF-03的T50值分别比野生酶(53℃)提高2.2℃、5.5℃和3.5℃。突变酶2-JF-01、2-JF-02和2-JF-03在60℃下的半衰期t1/2,60℃(min)分别比野生酶(18 min)提高4 min、13 min和17 min。突变酶 2-JF-01、2-JF-02 和 2-JF-03 的Vmax值为 286 µmol/(mg·min)、304 µmol/(mg·min)和 279 µmol/(mg·min),分别比野生型下降8.3%、2.6%和10.6%。突变酶2-JF-01、2-JF-02和2-JF-03的Km值分别为6.76 mg/mL、6.19 µmg/mL和6.84 mg/mL,与野生型(6.29 mg/mL)基本相同。序列分析表明,3个突变体共发生7个氨基酸替代:2-JF-01(N36S,G213R)、2-JF-02(C86R,S115I,N150G)和2-JF-03(E156V,K105R)。同源建模表明,7个氨基酸替代中5个位于蛋白质表面或表面洞穴中,42.8%的替代氨基酸是精氨酸,也表明精氨酸在提高β-1,3-1,4-葡聚糖酶热稳定性中起重要的作用。
β-1,3-1,4-葡聚糖酶,体外分子进化,易错PCR,热稳定性
Abstract:In vitroevolution methods are often used to modify protein with improved characteristics.We developed a directed evolution protocol to enhance the thermostability of the β-1,3-1,4-glucanase.The thermostability of the enzyme was significantly improved after two rounds of directed evolution.Three variants with higher thermostability were obtained.The mutant enzymes werefurther analyzed by their melting temperature, halftime and kinetic parameters.Comparing to intact enzyme, the T50of mutant enzymes 2-JF-01, 2-JF-02 and 2-JF-03 were increased by 2.2°C, 5.5°C and 3.5°C, respectively, the halftime(t1/2,60°C)of mutant enzymes 2-JF-01, 2-JF-02 and 2-JF-03 were shortened by 4,13 and 17 min, respectively, theVmaxof mutant enzymes were decreased by 8.3%, 2.6% and 10.6%, respectively, whileKmof mutant enzymes were nearly unchanged.Sequence analysis revealed seven single amino acid mutant happened among three mutant enzymes, such as 2-JF-01(N36S, G213R), 2-JF-02(C86R, S115I, N150G)and 2-JF-03(E156V, K105R).Homology-modeling showed that five of seven substituted amino acids were located on the surface of or in hole of protein.42.8% of substituted amino acids were arginine, which indicated that arginine may play a role in the improvement of the thermostability of the β-1,3-1,4-glucanase.This study provide some intresting results of the structural basis of the thermostability of β-1,3-1,4-glucanase,and provide some new point of view in modifying enzyme for future industrial use.
Keywords:β-1,3-1,4-glucanase,vitroevolution, error-prone PCR, thermostability
β-1,3-1,4-葡聚糖属植物细胞壁中的结构性非淀粉多糖,是以混合(1→3)和(1→4)β-糖苷键连接形成的D 型葡萄糖聚合物,它在大麦整粒和胚乳中的含量高达4.0%~8.0%[1-2]。β-1,3-1,4-葡聚糖酶葡聚糖酶(EC 3.2.1.73)是一种能够有效水解β-1,3-1,4-葡聚糖的酶,它能水解其中的β-1,3邻接的β-1,4键,其水解的主要产物为三糖(3-O-β-D-纤维二糖-D-葡萄糖)和四糖(3-O-β-D-纤维三糖-D-葡萄糖)。葡聚糖酶在工农业中的用途广泛,在啤酒工业中,β-葡聚糖酶可降低麦汁粘度,提高麦汁滤速及得率,改善啤酒风味,保持成品酒稳定性[3]。β-葡聚糖酶在饲料工业中也有较广泛的应用,β-葡聚糖也被称为谷物的重要抗营养因子,主要是因为单胃动物由于自身不具备合成β-葡聚糖酶的微生物及分解β-葡聚糖的酶系,使食糜在肠道中具有较高的粘度,而阻止蛋白质和脂肪营养的吸收,降低饲料的转化率,因此 β-葡聚糖也被称为谷物的重要抗营养因子[4],在饲料中添加β-葡聚糖酶,可以大大降低β-葡聚糖粘度,从而改善麦类营养价值,同时减少排泄物从而减轻对环境的污染。
然而,葡聚糖酶较差的热稳定性限制了它在工业生产中的使用,如在啤酒糖化和饲料造粒过程中易失去大部分酶活性[5]。因此提高 β-葡聚糖酶的热稳定性具有一定实践意义,不仅可稳定 β-葡聚糖酶的工业应用效果,而且可以此为基础研究 β-葡聚糖酶结构与功能之间的关系。
定向进化技术是由美国Trost等于上世纪90年代初期开发出来的一项蛋白质工程技术[6]。它是基因工程、蛋白质结构和计算机技术互补发展和渗透的结果,标志着人类可以按照自己的意愿和需要改造蛋白质,甚至设计出自然界中原本不存在的全新蛋白质。分子定向进化技术[7],简单地说就是在实验室中对达尔文自然进化原理进行模拟,从而对目标蛋白进行改造。随着分子生物学技术的不断发展,特别是PCR和基因重组技术的应用,定向进化已从在体内转为在体外进行,实验方法也变得简便、快速和高效[8-9]。体外展示技术等高通量筛选方法的发展更进一步拓展了体外定向进化的应用范围,使原本操作起来耗时耗力或难以进行直观筛选的蛋白质分子也能够得到改造[10]。
近年来国内外学者,大量运用体外分子进化技术,对碱性脂肪酶、木聚糖酶、β-糖苷酶以及 L-天门东酰胺酶[11]等多种工业用酶的性能进行了改造,取得较好的效果。对于 β-葡聚糖酶的研究,目前国内外研究主要集中在酶的规模化制备方面[12-14],而对于其性能的分子改造较少,尤其是在 β-葡聚糖酶的热稳定性定向进化研究方面,尚未见相关报道。
本研究基于易错PCR随机突变技术对来自淀粉液化芽胞杆菌β-1,3-1,4-葡聚糖酶基因bgl进行了分子改造,通过建立的基于 96微孔板高通量筛选模型,经过两轮定向进化与高通量筛选,共筛选得到3株热稳定性明显得到提高的葡聚糖酶突变体,2-JF-01、2-JF-02和 2-JF-03,并初步分析了热稳定性提高的机理。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
文中使用的菌株和质粒见表1。

表1 菌株和质粒Table 1 Strains and plasmids
1.1.2 主要试剂
TaqDNA聚合酶、dNTPs购自上海博彩生物科技有限公司;溶菌酶、蛋白酶K购自北京博大泰克有限公司;限制性内切酶BamH I、XhoI、T4 DNA连接酶、λDNA、蛋白质 Marker、硫酸卡那霉素、异丙基硫代-β-D-半乳糖苷(IPTG)购自上海生工生物工程公司,PCR产物纯化试剂盒、胶回收试剂盒、质粒提取试剂盒,丙烯酰胺、亚甲双丙烯酰胺为加拿大BBI公司产品;β-葡聚糖底物为美国Sigma公司产品;酵母提取物和胰蛋白胨为英国 Oxoid公司产品;其他未特别注明的试剂均为国产分析纯,国药集团化学试剂有限公司产品。
1.1.3 培养基
LB培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,琼脂粉15(固体培养基),固体和液体培养基在需要时加入硫酸卡那霉素至 30 μg/mL。TB培养基(g/L):胰蛋白胨 12,酵母提取物24,甘油6,NaCl 10,KH2PO42.4,K2HPO4·3H2O 12.5。培养基接种前加入卡那霉素溶液,终浓度为0.03 g/L。
1.2 方法
1.2.1 易错PCR扩增
使用高浓度Mg2+和不均衡dNTPs浓度来进行易错PCR,针对β-1,3-1,4-葡聚糖酶全基因,构建随机突变库。根据淀粉液化芽胞杆菌bgl序列(GenBank Accession:M15674)设计合成PCR所需的引物。
设计引物如下:PAG1-F1:(5′-ACATCGGATCC ATGAAACGAGTGTTGCTAATTCTTG-3′,BamH I)和 PAG1-R:(5′-GTAGTCATCTCGAGTTATTTTTT TGTATAGCGCACCCAG-3′,XhoI)。以含bgl基因的质粒pET28a(+)-bgl为模板,进行易错PCR 扩增。每100 µL反应体系为:1×TaqDNA聚合酶缓冲液、0.2 mmol/L dATP和 dGTP、1.0 mmol/L dCTP和dTTP、7 mmol/L Mg2+、0.25 mmol/L Mn2+、上下游引物各 50 pmol、Template DNA 1 μg、TaqDNA 聚合酶5 U。PCR扩增条件为:94℃预变性3 min;94℃变性50 s,56℃退火45 s,72℃延伸1 min,30个循环;4℃保温。
1.2.2 突变体文库的构建
易错PCR产物经1%(W/V)琼脂糖凝胶电泳检测后利用胶回收试剂盒回收。用限制性内切酶BamH I和XhoI分别对载体pET28a(+)和易错PCR产物进行消化,将bgl基因与线性化载体片段连接,连接产物转化感受态细胞E.coliBL21(DE3),涂布硫酸卡那霉素(50 mg/L)抗性平板构建突变体库。
1.2.3 突变文库的高通量筛选
从LB平板上挑选单个克隆子菌落接种于含有1 000 μL LB培养基(内含30 μg/mL Kan)的96深孔板中,每孔对应一特定转化子。每块深孔板同时接种野生型克隆,作为阳性对照。37℃、200 r/min振摇培养 12 h。在无菌条件下,从 96深孔板中取出20 μL菌液,对应接入含1 000 μL新鲜LB培养基的另一96深孔板中,并进行37℃、200 r/min振摇培养。4℃临时冷藏种子96深孔板。37℃、200 r/min振摇培养重组菌4 h后,每孔加入2 g/L IPTG 40 μL以及180 g/L乳糖100 μL,混匀后于24℃、200 r/min振摇培养6 h。在酶标仪上测定OD600并保存数据后于4℃、3 000 r/min离心20 min。以每孔一一对应为原则,利用排枪快速移取 20 μL上清粗酶液至 2块96 PCR反应板中。将其中一块含有粗酶液的96 PCR反应板经过80℃金属浴处理0.5 h,另一对应板4℃冷藏。将2块96 PCR反应板40℃预热10 min后(利用酶标仪加热模块),每孔中加入40℃蓝色葡聚糖底物(使用前和pH 6.5的20 mmol/L的磷酸缓冲液1∶19混合)80 μL,混匀后40℃反应10 min。每孔中加入300 μL沉淀液,利用Eppendorf低温离心机,于4℃、3 000 r/min离心20 min,利用排枪移上清至2块96反应板中并测定OD590吸光值。利用酶标仪软件计算每个克隆(OD590,未钝化−OD590,热钝化)/OD600的数值(残余活性),以阳性对照为参考,将结果高于阳性对照的克隆挑出并进行斜面保藏。
1.2.4 高通量筛选方法的统计学分析
从野生型E.coliBL21(DE3)-pET28a(+)-bgl平板上挑取200个单菌落,在96深孔培养板上进行培养、诱导表达后离心制得粗酶液,随后进行β-1,3-1,4-葡聚糖酶活高通量测定。基于数值符合正态分布的原理,利用公式R=1−F((X0−µ)/σ)对筛选方法的精确性进行统计学分析,其中X0为人为设定的预期值,用来表示由实验误差导致假阳性的活力数值;µ为野生型β-1,3-1,4-葡聚糖酶活力的平均值;R为所求假阳性出现的概率;σ为野生型β-1,3-1,4-葡聚糖酶活的标准方差。当确定单一基因库标准方差(σ)和活力平均值(µ)后,预期值(X0)在96孔板中假阳性概率就可以被计算,以此作为衡量筛选方法精确性的依据。
1.2.5 β-1,3-1,4-葡聚糖酶的分离纯化
纯化过程全部在4℃下进行,离心收集的重组菌溶于pH 8.0的Tris-HCl缓冲液中,超声破碎后,离心取上清液,即为粗酶液。粗酶液按美国通用公司的 HisTrap HP affinity column亲和层析方法进行纯化,纯酶液超滤浓缩后用于后续实验。
1.2.6 β-1,3-1,4-葡聚糖酶活力的测定
采用 3,5-二硝基水杨酸(DNS)法及改良 AZO测定方法相结合进行测定。蓝色大麦葡聚糖底物法测定 β-1,3-1,4-葡聚糖酶的原理见图1。酶活定义:在40℃和 pH值为 6.5条件下,每分钟水解β-葡聚糖生成相当于1 μmol的葡萄糖还原物质的量为1个酶活力单位。带蓝色基团的大麦β-葡聚糖,在β-葡聚糖酶的作用下,由于 β-葡聚糖大分子中的β-1,3-1,4键的断裂,将蓝色基团释放到反应体系中,加入特定的沉淀液后,经过离心,清液在590 nm处具有吸光值,且吸光值的大小直接与酶活力的高低成正比。具体测定步骤见Ekinci MS[15]等介绍方法。
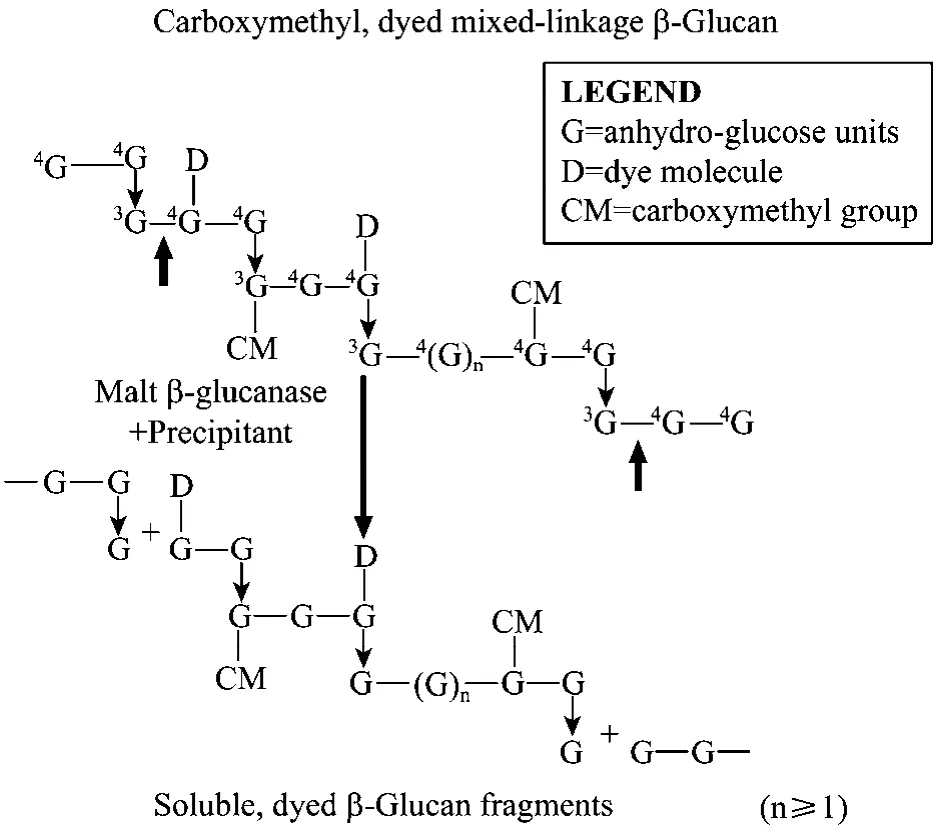
图1 蓝色大麦葡聚糖底物法测定 β-1,3-1,4-葡聚糖酶的原理示意图Fig.1 Theoretical basis of the β-1,3-1,4-glucanase assay employing Azo-Barley Glucan.
1.2.7 野生酶和热稳定性提高突变体的Michaelis-Menten动力学分析
底物为 0~10 mg/mL的大麦 β-葡聚糖,溶解于0.02 mol/L的pH 6.5的磷酸缓冲液。在相同酶浓度下,于40℃测定β-1,3-1,4-葡聚糖酶活力。以Lineweaver-Burk作图法计算 Michaelis-Menten常数。从Lineweaver-Burk双倒数图中可得到动力学参数Km以及Vmax。
1.2.8 野生酶和热稳定性提高突变体的热稳定性测定
纯化得到的酶液分别在 25℃~80℃的水浴中保温10 min,立即冰浴20 min,按照1.2.5描述方法测定酶活力。记酶溶液不经过钝化处理时的酶活力为100%,以不同温度对相应的残余酶活力百分比作图,求出酶活力丧失50%时的温度,即该酶的半失活温度 T50。定义 β-1,3-1,4-葡聚糖酶特定温度下酶活丢失一半的时间为其半衰期,记为t1/2,X℃。
1.2.9 淀粉液化芽胞杆菌BS5582 β-1,3-1,4-葡聚糖酶的三维结构同源模拟与突变位点分析
在 SWISS-MODEL数据库(http://swissmodel.expasy.org/workspace)中输入 β-1,3-1,4-葡聚糖酶野生型以及突变酶的的氨基酸序列并选择模板,生成同源模拟图。借助 PYMOL蛋白质三维结构分析软件并根据预测的结构信息,分析其位点突变效应。
2 结果
2.1 易错PCR反应条件的优化
易错PCR随机突变方法主要基于降低DNA聚合酶在扩增过程中的保真度而向靶基因中引入错误脱氧核糖核苷酸[6,16]。反应体系中的 Mn2+浓度高低与DNA聚合酶保真性好坏直接相关。通过调节易错PCR反应体系Mn2+浓度可以实现对靶基因突变频率进行精细调节,使其突变频率稳定在一定范围内。本文对不同Mn2+浓度下bgl的突变频率进行了研究,从表2中可以得知,易错PCR反应系统具有0.03%的净突变,主要原因是TaqDNA聚合酶没有3′→5′核酸外切酶活性进而影响其对 PCR反应的校对功能[17]。靶基因的突变频率随着Mn2+浓度的增加而提高,如果突变频率太高,产生的绝大多数酶将失去活性,如果突变频率太低,野生型的背景太高,样品的多样性则较少。0.25 mmol/L至0.35 mmol/L的Mn2+浓度对我们所建立的随机突变体系来说是比较合适的,可以使β-葡聚糖酶分子产生了2~3个氨基酸的替代。后期的进化与筛选实验的结果也进一步表明,0.25~0.35 mmol/L的Mn2+浓度非常适合于bgl(717 bp)基因的定向进化。本研究亦对含不同浓度的易错 PCR突变产物进行琼脂糖凝胶电泳分析,由图2可以看出,当Mn2+浓度大于0.25 mmol/L时,非突变产物的量明显地高于突变产物的量,Mn2+氧化生成的微量Mn3+抑制了部分DNA聚合酶活性,可能是产生此结果的原因之一[18]。综合考虑,本研究选择0.25 mmol/L作为易错PCR反应体系Mn2+浓度。
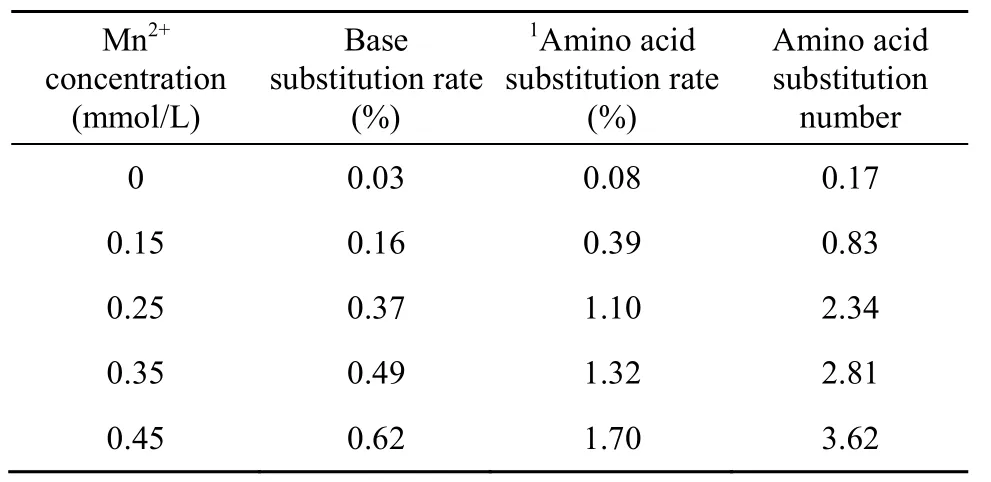
表2 Mn2+浓度对bgl突变频率的影响Table 2 The mutation rate of ep-PCR at different Mn2+concentration
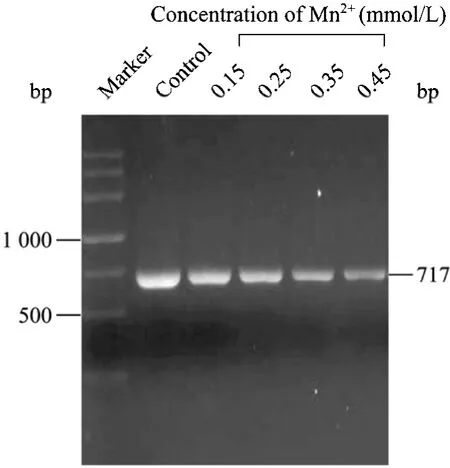
图2 Mn2+浓度对易错PCR反应体系产物的影响Fig.2 Effect of Mn2+concentration on the products of ep-PCR marker: DNA marker; control: standard PCR control;0.15: ep-PCR with 0.15 mmol/L of Mn2+; 0.25: ep-PCR with 0.25 mmol/L of Mn2+; 0.35: ep-PCR with 0.35 mmol/L of Mn2+;0.45: ep-PCR with 0.45 mmol/L of Mn2+.
2.2 高通量筛选方法的统计学分析
欲使酶的定向进化获得成功,灵敏准确的高通量筛选方法(图3)是其必要条件。为了检验96酶标板测定β-1,3-1,4-葡聚糖酶酶活的精确性,本研究从野生型E.coliBL21(DE3)-pET28a(+)-bgl平板上挑取200个单菌落,在96深孔培养板上进行培养及诱导表达后离心制得粗酶液,随后进行β-1,3-1,4-葡聚糖酶活高通量测定。结果表明,代表β-1,3-1,4-葡聚糖酶活OD590均数 µ=0.63,其数据组标准差为±0.088(0.14 µ),基于数值符合正态分布的原理,利用公式R=1−F((X0−µ)/σ)对筛选方法的精确性进行统计学分析,表观为 2倍均数的假阳性出现概率为0.75×10−6当活力预期值为1.15倍于野生型总体均数,假阳性克隆的概率为1.7×10−4。研究结果表明,利用96酶标板进行β-1,3-1,4-葡聚糖酶活测定假阳性背景很低,可以有效地在定向进化中加以应用。
2.3 突变体文库的构建及突变株的筛选和鉴定
在优化确定的易错PCR反应体系下,进行PCR扩增,产物经1%(W/V)琼脂糖凝胶电泳检测后利用胶回收试剂盒回收。用限制性内切酶BamH I和XhoI分别对载体pET28a(+)和易错PCR产物进行消化,将易错bgl基因产物与线性化载体片段连接,连接产物转化感受态细胞E.coliBL21(DE3),涂布硫酸卡那霉素(50 mg/L)抗性平板构建突变体库,质粒构建流程见图3。按照上述文库构建及筛选方法对β-1,3-1,4-葡聚糖酶进行了两轮进化,共完成对约4 500株克隆的初筛,得到活性比值提高突变体87株。随后从相应母 96培养板中挑取这些克隆在96孔板以及试管中进行多次复筛,定量比较这些突变株和野生型β-葡聚糖酶在80℃、30 min后的剩余活性。最终获得热稳定性得到提高的3个突变体:2-JF-01、2-JF-02和2-JF-03。
序列分析表明,3个突变体共发生 7个氨基酸替代(图4):2-JF-01(N36S,G213R)、2-JF-02(C86R,S115I,N150G)和 2-JF-03(E156V,K105R)。基于SWISS−MODEL 数 据 (http://swissmodel.expasy.org/workspace),对野生型及热稳定性提高突变体进行了蛋白三维结构同源模拟。选择的 PDB模板[19]为同源性与淀粉液化芽胞杆菌 BS5582 β-1,3-1,4-葡聚糖酶达到97%的1gbgA(1.80 Å),同源建模的结果由 PROCHECK验证。借助 PYMOL蛋白质三维结构分析软件并根据预测的结构信息,分析了 β-葡聚糖酶蛋白位点突变效应。图5三维结构显示,7个氨基酸替代中5个位于蛋白质表面或洞穴中。
2.4 野生酶和热稳定性提高突变体的 Michaelis-Menten动力学分析
野生型及突变体β-1,3-1,4-葡聚糖酶按美国通用公司的HisTrap HP affinity column亲和层析方法进行纯化并经超滤浓缩后用于动力学研究。突变酶2-JF-01、2-JF-02 和 2-JF-03 的Vmax值为 286 µmol/(mg·min)、304 µmol/(mg·min)和 279 µmol/(mg·min),分别比野生型下降8.3%、2.6%和10.6%。突变酶2-JF-01、2-JF-02和2-JF-03的Km值为6.76 mg/mL、6.19 µmg/mL和6.84 mg/mL,与野生型6.29 mg/mL基本相同(表3)。
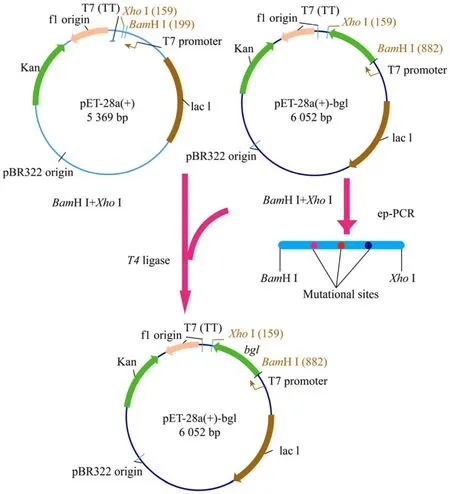
图3 易错PCR介导的β-1,3-1,4-葡聚糖酶定向进化质粒构建流程Fig.3 Mutagenesis procedure of the β-1,3-1,4-glucanase structural gene bgl.

图4 野生型及热稳定性提高突变体β-1,3-1,4-葡聚糖酶氨基酸序列比对Fig.4 Amino acid sequence alignment for variants 2-JF-01, 2-JF-02 and 2-JF-03, comparing to wide type β-1,3-1,4-glucanase.The alignment was performed by using the DNAMEN alignment programmer.Amino acid substitutions that differ from wide type are indicated in white.
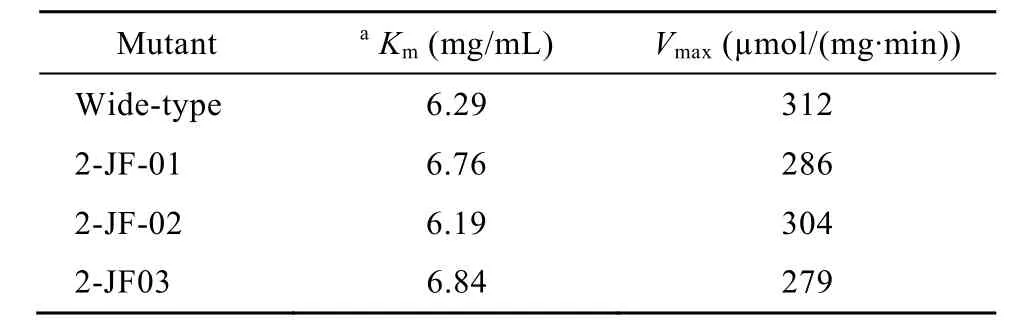
表 3 野生酶和热稳定性提高突变体 β-1,3-1,4-葡聚糖酶动力学参数Table 3 Kinetic parameters of wide-type β-1,3-1,4-glucanase and its mutants
2.5 野生酶和热稳定性提高的突变酶的热稳定性测定
纯化得到的酶液分别在 25℃~80℃的水浴中保温10 min,立即冰浴20 min,按照1.2.5描述方法测定酶活力。记酶溶液不经过钝化处理时的酶活力为100%,以不同温度对相应的残余酶活力百分比作图,求出酶活力丧失50%时的温度,即该酶的半失活温度T50。β-1,3-1,4-葡聚糖酶特定温度下酶活丢失一半的时间为其半衰期 t1/2,X℃。实验结果表明(图6)突变酶2-JF-01、2-JF-02和2-JF-03的T50值分别比野生酶 53℃提高 2.2℃、5.5℃和 3.5℃,达到55.2℃、58.5℃和 56.5℃。突变酶 2-JF-01、2-JF-02和2-JF-03在60℃下的半衰期t1/2,60℃(min)分别比野生酶18 min提高4 min、13 min和17 min,达到22 min、31 min和 35 min。
3 讨论
本研究对来源于淀粉液化芽胞杆菌 BS5582 β-1,3-1,4-葡聚糖酶基因bgl进行了定向进化,获得3个热稳定性得到提高的突变体。序列分析表明,3个突变体共发生7个氨基酸替代。
酶学性质及动力学性质分析表明,热稳定性提高突变体β-1,3-1,4-葡聚糖酶对底物的亲和力基本不变,但突变体相对于葡聚糖底物的催化活力下降明显。虽然有研究报道,通过定向进化,酶的热稳定性和催化活性同时得到提高[8],但以活性的损失来换取热稳定性的提高在定向进化中非常普遍。因为酶的活性是建立在其空间构象的柔性基础上的,而热稳定性正好相反,需要其构象具有刚性,以抵御外力(高温钝化作用)的破坏[20]。因此很多情况下,一个酶的活性和其热稳定性是一种此消彼长的关系,本研究的结果也与之符合。
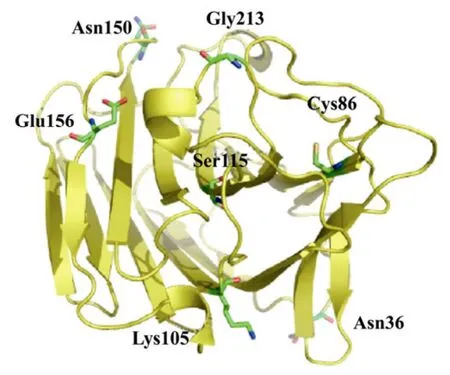
图5 β-1,3-1,4-葡聚糖酶热稳定性提高突变体氨基酸替代三维结构定位Fig.5 Crystal structure of the wide-type β-1,3-1,4-glucanase(yellow cartoon)with the seven mutations(labeled and indicated by sticks)of the three variants selected in the directed evolution.The figure was made using PYMOL.
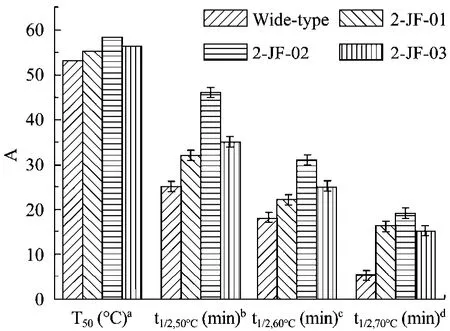
图6 野生酶和热稳定性提高的突变酶 β-1,3-1,4-葡聚糖酶热稳定性参数Fig.6 Thermostability parameters of wide-type β-1,3-1,4-glucanase and its mutants.aTemperature at which β-1,3-1,4-glucanase loses 50% activity upon incubation for 20 min.bHalf-life of 50°C inactivation.cHalf-life of 60°C inactivation.dHalf-life of 70°C inactivation A: the absolute value of T50and half-life.
最近有研究表明水解酶类的蛋白质表面环形区域(loop)或 310螺旋的氨基酸残基组成对于蛋白质的稳定性非常重要[20-22],如在热稳定性较好的栖热菌麦芽糖淀粉酶共有9个氨基酸替代,其中6个位于蛋白质表面环形区域(loop)或 310螺旋中。本研究同源建模表明,7个氨基酸替代中的 5个位点位于蛋白质表面环形区域(loop)、310螺旋或蛋白表面洞穴中,这有利于葡聚糖酶突变酶的热稳定性提高。
通过突变位点分析,显示7个氨基酸替代中有3个突变成精氨酸(G213R、C86R和K105R),占整个突变的比例为 42.8%。蛋白表面精氨酸突变热点现象已见相关报道[11,23],精氨酸的电荷分布、分子结构及形成多重氢键的能力,使得它能与带有负电荷的分子结合。因此,精氨酸在蛋白质的外围,能在带电荷的环境下产生相互作用,从而维系蛋白质更加稳定的结构。图7显示,86位的半胱氨酸突变为精氨酸后,与其邻近的85位的天冬氨酸及88位的谷氨酸形成盐桥,在蛋白质表面形成亲水性离子簇从而加强了酶蛋白分子的稳定性。
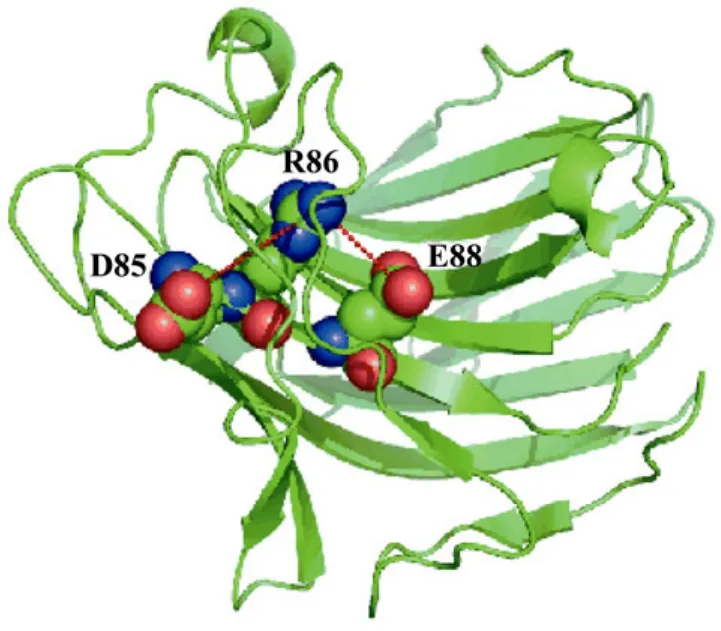
图7 β-葡聚糖酶蛋白三维结构中86位氨基酸的定位Fig.7 Location of mutations in the crystal structure of β-1,3-1,4-glucanase site 86Cys86 is surrounded by Asp85 and Glu88, resulting in a hydrophilic cluster.Substitution of the Cys increases the electrostatic interaction by forming salt bridge with Asp85 and Glu88.The figure was made using PYMOL.
本文所研究的葡聚糖酶蛋白分子中 115位的丝氨酸突变成异亮氨酸以及 156位的谷氨酸突变成缬氨酸,使突变酶蛋白更加稳定,其原因可能是被替代的疏水氨基酸增加蛋白质内部的疏水核包装密度,从而增加了蛋白质的稳定性[24]。而第36位的天冬酰胺突变成丝氨酸后,与分子邻近的38位的酪氨酸形成氢键,新产生的氢键也可能是突变酶蛋白稳定性提高的原因之一。
本研究通过非理性方法(易错 PCR)对 β-1,3-1,4-葡聚糖酶热稳定性定向进化,一方面解释了酶蛋白结构与功能的信息,另一方面,结构与功能的信息也为下一步的理性改造(定点突变)打下了坚实的基础。
REFERENCES
[1]Cantwell B.An analysis ofBacillus subtilisendo-beta-1,3-1,4-glucanase gene expression inEscherichia coli.Gene, 1983, 23(2): 211–219.
[2]Cantwell B, Brazil G, Hurley J,et al.Expression of the gene for the endo-beta-1,3-1,4-glucanase fromBacillus subtilisinSaccharomycescerevisia.J Inst Brew, 1985,91(3): 129–129.
[3]Gormley EP, Cantwell BA, Barker PJ,et al.Secretion and processing ofBacillussubtilisendo-beta-1,3-1,4-glucanaseinEscherichia coli.Mol Microbiol, 1988, 2(6):813–819.
[4]Asan M, Ozcan N.Expression of the beta-(1,3-1,4)-glucanase gene inStreptococcus salivariussubsp.thermophilus.Turk J Vet Anim Sci, 2007, 31(5): 319–324.
[5]Fukamizo T, Hayashi K, Tamoi M,et al.Enzymatic hydrolysis of 1,3-1,4-beta-glucosyl oligosaccharides by 1,3-1,4-beta-glucanase fromSynechocystisPCC6803: a comparison with assays using polymer and chromophoric oligosaccharide substrates.Arch Biochem Biophys, 2008,478(2): 187–194.
[6]Trost BM, Dumas J, Villa M.New strategies for the synthesis of vitamin-D metabolites via PD-catalyzed reactions.J Am Chem Soc, 1992, 114(25): 9836–9845.
[7]Giver L, Gershenson A, Freskgard PO,et al.Directed evolution of a thermostable esterase.Proc Natl Acad Sci USA, 1998, 95(22): 12809–12813.
[8]Kim MS, Lei XG.Enhancing thermostability ofEscherichia coliphytase AppA2 by error-prone PCR.Appl Microbiol Biot, 2008, 79(1): 69–75.
[9]Leemhuis H, Nightingale KP, Hollfelder F.Directed evolution of a histone acetyltransferase-enhancing thermostability, whilst maintaining catalytic activity and substrate specificity.FEBS J, 2008, 275(22): 5635–5647.
[10]Emond S, Potocki-Veronese G, Mondon P,et al.Optimized and automated protocols for high-throughput screening of amylosucrase libraries.J Biomol Screen, 2007, 12(5):715–723.
[11]Kotzia GA, Labrou NE.Engineering thermal stability of L-asparaginase byin vitrodirected evolution.FEBS J,2009, 276(6): 1750–1761.
[12]Jung YJ, Yoo JS, Lee YS,et al.Purification and characterization of thermostable beta-1,3-1,4 glucanase fromBacillussp.a8-8.Biotechnol Bioproc E, 2007, 12(3):265–270.
[13]Hong MR, Kim YS, Joo AR,et al.Purification and characterization of a thermostable beta-1,3-1,4-glucanase fromLaetiporus sulphureusvar.miniatus.J Microbiol Biotechnol, 2009, 19(8): 818–822.
[14]Qiao JY, Dong B, Li YH,et al.Cloning of a beta-1,3-1,4-glucanase gene fromBacillus subtilisMA139 and its functional expression inEscherichia coli.Appl Biochem Biotech, 2009, 152(2): 334–342.
[15]Ekinci MS, McCrae SI, Flint HJ.Isolation and overexpression of a gene encoding an extracellular beta-(1,3-1,4)-glucanase fromStreptococcus bovisJB1.Appl Environ Microb, 1997, 63(10): 3752–3756.
[16]Matsumura I, Ellington AD.In vitroevolution of thermostable p53 variants.Protein Sci, 1999, 8(4):731–740.
[17]Zhao HM, Arnold FH.Directed evolution converts subtilisin E into a functional equivalent of thermitase.Protein Eng, 1999, 12(1): 47–53.
[18]Ghadessy FJ, Ong JL, Holliger P.Directed evolution of polymerase function by compartmentalized self-replication.Proc Natl Acad Sci USA, 2001, 98(8): 4552–4557.
[19]Arnold K, Bordoli L, Kopp J,et al.The Swiss-model workspace: a web-based environment for protein structure homology modelling.Bioinformatics, 2006, 22(2):195–201.
[20]Nakazawa H, Okada K, Onodera T,et al.Directed evolution of endo-glucanase III(Cel12A)fromTrichoderma reesei.Appl Microbiol Biot, 2009, 83(4):649–657.
[21]Liu D, Schmid RD, Rusnak M.Functional expression ofCandida antarcticalipase B in theEscherichia colicytoplasm-a screening system for a frequently used biocatalyst.Appl Microbiol Biot, 2006, 72(5): 1024–1032.
[22]Asako H, Shimizu M, Itoh N.Engineering of NADPH-dependent aldo-keto reductase fromPenicillium citrinumby directed evolution to improve thermostability and enantioselectivity.Appl Microbiol Biot, 2008, 80(5):805–812.
[23]Fang YW, Lu YJ, Lv FX,et al.Improvement of alkaline lipase from Proteus vulgaris T6 by directed evolution.Enzyme Micrb Tech, 2009, 44(2): 84–88.
[24]Mordukhova EA, Lee HS, Pan JG.Improved thermostability and acetic acid tolerance ofEscherichia colivia directed evolution of homoserine o-succinyltransferase.Appl Environ Microb, 2008, 74(24): 7660–7668.
Improvement of thermostability of β-1,3-1,4-glucanase from Bacillus amyloliquefaciens BS5582 through in vitro evolution
Jiufu Qin1,2, Weiwei Gao1,2, Qi Li1,2, Yongxian Li1,2, Feiyun Zheng1,2, Chunfeng Liu1,2,and Guoxian Gu2
1 The Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, China 2 Laboratory of Brewing Science and Tecnology, School of Biotechnology, Jiangnan University, Wuxi 214122, China
Received:February 8, 2010;Accepted:April 22, 2010
Supported by:Key Projects in the National Science and Technology Pillar Program during the Eleventh Five-Year Plan Period(Nos.2008BAI63B06,2007BAK36B01), National High Technology Research and Development Program of China(863 Program)(No.2006AA020204), Program for Changjiang Scholars and Innovative Research Team in University(No.IRT0532).
Corresponding author:Qi Li.Tel: +86-592-2185869; Fax: +86-592-2184822; E-mail: liqi@jiangnan.edu.cn“十一五”国家科技支撑计划(Nos.2008BAI63B06, 2007BAK36B01),国家高技术研究发展计划(863计划)(No.2006AA),长江学者和创新团队发展计划项目(No.IRT0532)资助。

