中国明对虾(Fenneropenaeus chinensis)肠道微生物区系组成的分子分析
刘淮德 刘 梅 王宝杰 蒋克勇 张国范 王 雷
中国明对虾主要分布在我国黄渤海和朝鲜西部沿海,南海部分海域也有少量存在,是我国重要的海水养殖对虾之一。中国明对虾是中国的特产,也是重要的出口水产品,无论是品质还是市场价格都远远高于其他养殖对虾品种,其最高养殖年产量曾达到近20万吨。而随其养殖规模的扩大,对虾疾病的发生越来越频繁,1993年爆发的病毒性流行病,使中国明对虾的养殖遭受毁灭性打击,此后始终未能全面恢复。近年来,随着生态养殖和健康养殖的发展,一些微生态制剂开始应用于水产养殖,以改善养殖环境,减少病害和提高免疫力。目前有关微生物制剂的研究与开发在国内外均得到很大的重视,市场上已有大量用于水产养殖的专用产品销售。水产微生态制剂是以后水产养殖病害防治的发展方向,但其被水产动物口服后的作用机理并不十分清楚。了解对虾肠道微生物区系微生物的组成、结构及其生理功能如何,在此基础上可以进一步研究外源有益微生物制剂对其影响,将有助于微生态制剂在水产养殖中的有效应用与推广。
随着水产养殖业的发展和分子生物学技术的成熟,利用分子生物学手段研究肠道微生物组成日益成为热点。水产动物的肠道内存在着大量的微生物,肠道微生物区系的动态平衡是动物保持健康和正常生长所必需的条件,肠道内有益微生物群对宿主起着屏障、营养和免疫等作用。本文采用不依赖培养的分子生物学手段16S rRNA基因克隆文库法,分析中国明对虾肠道群落组成,以期为水产微生态制剂的使用提供参考依据。
1 材料与方法
1.1 材料
1.1.1 样品的采集与处理
海捕中国明对虾,于2008年夏天购自青岛小港水产品市场,为健康对虾成体,雄性,平均体长为12 cm,购回的活体蓄养空腹后于超净台内用70%乙醇进行体表消毒后,无菌条件下解剖取出肠道。
1.1.2 主要试剂和仪器
PCR 仪(PTC-100),GelDoc凝胶成像系统(Bio-Rad)。
DNA纯化试剂盒购自上海生工,质粒pMD18-T,限制性内切酶(MspI和HhaI)购自大连宝生物工程有限公司。
1.2 肠道微生物总DNA的提取
取200 mg肠道样品,液氮研磨,加入l ml PBS(pH 值 7.4,0.1 mol/l磷酸钠缓冲液) 和 20 μl 20%PVPP(聚乙烯聚吡咯烷酮),充分均质化后200×g离心6 min,取上清;沉淀中再加人1 ml PBS,离心,取上清;合并两次上清,再次300×g离心6 min,取上清;再以12 000×g离心6 min收集菌体,并用PBS洗涤。在得到的菌体中加入300 μl裂解液I(150 mM NaCl,100 mM EDTA·Na2,pH 值 8.0)、10%溶菌酶 100 μl和1%RNaseA 20 μl,混匀后37℃保温 30 min。加入300 μl裂解液 II(100 mM NaC1,500 mM Tris-HCl,pH 值 8.0)及50 μl 20%SDS(十二烷基磺酸钠)和 50 μl 20%PVPP,混匀后冰浴5 min。加入等体积Tris饱和酚:氯仿:异戊醇为25:24:1溶液,13 000×g离心8 min,取上清;再加入等体积氯仿:异戊醇(24:1),13 000×g离心 8 min,取上清(重复操作 1 次)。加入1/10体积3 M醋酸钠及两倍体积乙醇,-20℃沉淀2 h以上。15 000×g离心15 min,70%乙醇洗涤1次,超净台下干燥沉淀。沉淀用40 μl TE(100 mmol/l Tris-HCl,1 mmol/l EDTA,pH值8.0)溶解,-20℃冷冻保存备用。
1.3 16S rRNA基因的PCR扩增
PCR 引物:27F(5'-AGAGTTTGATCMTGGCTC-3';M=A 或 C);1387R(5'-GGGCGGWGTGTACAAGGC-3';W=A或T)。
PCR 体系:无菌双蒸水 34.5 μl,10×PCR 缓冲液5 μl,2.5 mmol/l dNTP 4 μl,2.5 μmol/l PCR 引物各 2 μl,Taq 酶 0.5 μl,模板 DNA 2 μl。反应条件:94 ℃ 预变性 4 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 2 min,30个循环;72℃10 min。PCR产物用1.0%的琼脂糖凝胶进行电泳,切下条带,用上海生工胶回收试剂盒回收。
1.4 16S rDNA克隆转化和文库构建
按照pMD18-T载体试剂盒说明,将纯化后的产物与载体pMD18-T连接后,转化到大肠杆菌E.coli Top10中。
1.5 阳性克隆子的RFLP分析
用菌落PCR方法验证16S rRNA基因文库中的阳性克隆。选择通用引物T7和SP6扩增pMD18-T载体多克隆位点之间的序列,扩增条件同上。扩增产物用4碱基限制性内切酶MspI和HhaI进行酶切消化(37℃过夜),酶切产物用3%的琼脂糖凝胶进行电泳分析。
1.6 DNA测序和分析
挑取代表性克隆,送上海生物工程有限公司进行序列测定,根据测序结果,计算文库的覆盖率。测定的序列在NCBI数据库中用BLAST程序进行相似性搜索,从中选择相近的典型菌株16S rRNA序列,用Clustal X软件进行多重序列比对,构建系统发育进化树。
2 结果和分析
2.1 总DNA的抽提
肠道微生物基因组总DNA提取电泳图见图1。总DNA经0.8%的琼脂糖凝胶电泳,在大于10 kb处出现条带。

2.2 16S rDNA片段的扩增
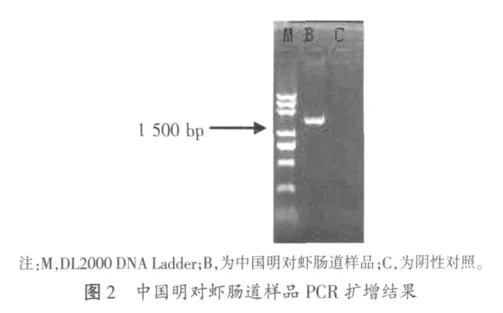
总DNA的PCR产物用1.0%琼脂糖凝胶电泳检测,发现均获得了特异性扩增片段。从图2中可以看出,片段大小在1 500 bp左右,且没有非特异性扩增现象,得到的PCR扩增产物适合下一步16S rDNA克隆文库的构建。
2.3 16S rDNA基因克隆文库分析
从文库中挑选60个白色菌落,菌落PCR检测到阳性克隆为55个。根据样品酶切片段大小和数量区别电泳条带谱型,结果表明,在中国明对虾肠道16S rDNA文库55个阳性克隆共有13种细菌RFLP谱型。
以覆盖率(C)来评估所构建的文库对环境微生物多样性的体现,计算公式为:
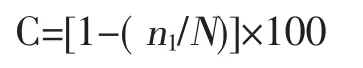
式中,N代表所分析的克隆数,n1代表具有不重复序列的克隆数 (16S rDNA序列>97%为重复序列)。计算结果表明,所构建的16S rDNA克隆文库覆盖率达到76.36%,即包含了对虾肠道中76.36%的细菌多样性状况。因此,从覆盖率分析可以说明所构建的16S rDNA克隆文库能够较完整地反映所属环境中的细菌多样性状况。
2.4 序列比对和系统发育分析
挑选具有代表性的克隆子测序,去除载体序列部分,将所得序列在GenBank中进行BLAST分析,搜索相关序列,比对结果如表1所示。55个阳性克隆子与GenBank数据库中已知16S rDNA序列相似性最高为99%,最低为87%;其中31个克隆子与已知序列相似性高于98%,其余克隆子(24个,占43.6%)与已知序列同源性低于94%。测序55克隆,弧菌22克隆,占40.0%,柔膜细菌11克隆,占20.0%,光合细菌3克隆,占54.5%,不可培养细菌19克隆,占34.5%。结果显示中国明对虾肠道优势菌为弧菌,其次为不可培养细菌和柔膜细菌。
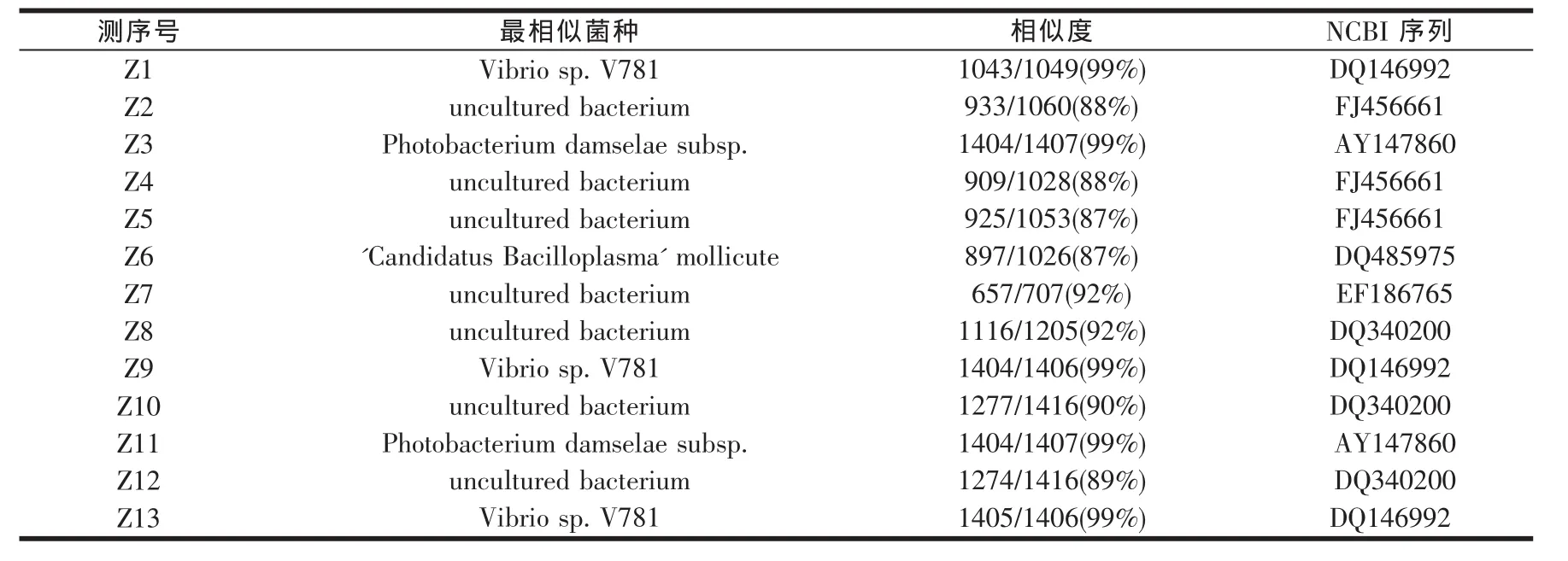
表1 部分中国明对虾肠道微生物DNA文库比对结果
3 讨论
对虾肠道微生物的早期研究主要是传统的培养法,而近年来分子方法开始被大多数人采用。普遍认为水生无脊椎动物肠道微生物的组成为弧菌属、假单胞菌属、黄杆菌属、微球菌属和气单胞菌属。Yasuda等(1980)发现幼虾中,弧菌属占优势,而成年虾中,假单胞菌属占优势;Dempsey等(1989)发现,弧菌属、产碱菌属、气单胞菌属、发光杆菌属和假单胞菌属为虾肠道优势种群;王祥红等(2000)发现,弧菌属和发光杆菌属在野生中国明对虾肠道中为优势菌属。Moss等(2000)发现,对虾肠道优势菌为弧菌属、气单胞菌属和假单胞菌属。Oxley等(2002)研究发现,野生和养殖对虾有相似的肠道微生物组成,包括气单胞菌属、发光杆菌属、假单胞菌属和弧菌属。Esiobu发现,健康的桃红对虾肠道中优势菌为弧菌属。这些研究都是采用传统的培养方法,然后鉴定得到优势种群。李志勇等(2005)发现,不同对虾及同一种对虾的鳃部与肠道内的细菌组成差异性非常大;而罗鹏等(2006)经比较研究发现,凡纳滨对虾养殖系统内不同环境的优势种群十分明显。两者采用的都是PCR-DGGE方法,但仅仅比较了不同对虾之间的肠道微生物组成差异,并没有比较分析对虾肠道微生物中各种群之间的关系。本研究采用16S rDNA基因文库法分析中国明对虾肠道微生物区系组成,结果与前人研究结果基本一致,采用分子生物学方法得到一些不可培养细菌的信息,更贴近真实情况,但是由于所选阳性克隆数较少,文库的覆盖率为76.36%,如果能再增加阳性克隆数目,就能更加完整地反映肠道微生物区系的真实组成。
16S rRNA基因克隆文库技术现在已经广泛地用于各种生态系统微生物组成的多样性研究中,例如人们在对废水处理系统、土壤、海洋、人体胃肠道系统等生态系统中的微生物进行研究时都用到该技术。目前,基于16S rRNA基因克隆文库的分析方法是用于调查一个环境样品中微生物组成情况的常用方法。
在水产养殖中,应用克隆文库技术可建立一种不依传统分离培养技术而能原位揭示对虾体内细菌种群组成的分子诊断技术和病原菌分子诊断技术。通过检测水产动物肠道正常微生物区系组成,建立基于机体内优势菌群结构的健康水产动物评价技术。还可检测水产动物肠道微生物的动态变化和病害后微生物区系组成的差异,以及使用微生态制剂前后微生物区系组成的差异,为水产动物健康养殖提供理论依据。通过对虾肠道内细菌群落结构的揭示将为新的水产微生物制剂菌株的开发和应用提供候选菌株和理论基础。
4 结论
本文采用不依赖于培养的分子生物学手段16S rRNA克隆文库法分析中国明对虾肠道微生物群落组成。从中国明对虾肠道中提取微生物基因组DNA,以细菌16S rRNA基因通用引物27F/1387R扩增16S rRNA基因序列,构建16S rRNA基因文库。结果表明,中国明对虾肠道微生物主要由弧菌、光合细菌、不可培养细菌组成,弧菌为优势菌。16S rRNA克隆文库法能够反映对虾肠道微生物中种群的组成关系,是研究水产动物肠道微生物区系组成的可行方法。
17篇,刊略,需者可函索)

