底泥水体中适用于PCR的不同形态DNA的提取方法
冯凌,罗义
南开大学环境科学与工程学院 环境污染过程与基准教育部重点实验室,天津300071
底泥水体中适用于PCR的不同形态DNA的提取方法
冯凌,罗义*
南开大学环境科学与工程学院 环境污染过程与基准教育部重点实验室,天津300071
提出直接从环境河流底泥和表层水体中同时提取不同形态DNA(胞内、胞外、染色体和质粒DNA)的有效方法.利用该方法提取了海河典型区域底泥的胞外DNA和胞内DNA及水体中细菌染色体DNA和质粒DNA.结果表明,NaH2PO4提取的胞外DNA分子量大于1kb,提取率40%~72%,没有共提取胞内DNA.胞内DNA提取的分子量均大于23.1kb,细菌质粒DNA与染色体DNA同时分离.16S rDNA与sul2扩增验证提取方法可靠性.16S rDNA扩增结果显示所有胞外DNA呈阴性,胞内DNA呈阳性.质粒DNA和染色体DNA的16S rDNA扩增显示,染色体DNA结果显阳性,质粒DNA呈阴性,sul2的结果相反.
胞外DNA;胞内DNA;染色体DNA;质粒DNA;提取方法
Received 13 December 2009 accepted 10 January 2010
Abstract:In this paper,effective methods were presented to extract different gene types(extracellular DNA,intracellular DNA,genome DNA and plasmid DNA)from the sediments and water in environment.The methods were used for extracted extracellular DNA and intracellular DNA in sediments while genome DNA and plasmid DNA of bacteria in water in typical regions of Haihe River.Results showed that extracellular DNA was more than 1kb in size;its recovery ratio was 46%~72%without containing intracellular DNA by using NaH2PO4.All intracellular DNA were more than 23.1kb in size.Genome DNA and plasmid DNA were separated simultaneously.PCR amplification of 16S rDNA and sul2 were applied for assurance of the methods.Results showed that products of 16S rDNA of all the extracellular were negative while intracellular DNA positive.The genome DNA amplified of 16S rDNA turned out to be positive while of sul2 was negative,the results of plasmid DNA were the opposite.
Keywords:extracellular DNA;intracellular DNA;genome DNA;plasmid DNA;extraction
1 引言(Introduction)
近年来,抗生素的滥用导致在动物肠道细菌诱导产生抗生素抗性基因(Antibiotic resistance genes, ARGs),抗性基因从动物肠道细菌向环境土著细菌传播扩散,使其成为一类新型环境污染物,其在不同环境介质中的传播扩散目前已引起广泛关注(Knapp et al.,2008;Krumperman,1983;Nielsen et al.,1998;2007;Pei et al.,2006;Pruden et al., 2006;罗义等,2008;高盼盼等,2009).现代分子生物学PCR技术已被越来越多地用于环境样本中抗性基因的检测,如何获得高质量的DNA模板是进行PCR扩增的前提.目前采用的环境样本DNA的提取方法大都建立在对细胞内 DNA的提取上(Volossiouk et al.,1995;Clesceri et al.,1995).然而,无论在陆地生态系统还是水生态系统都已检测到胞外游离DNA的存在(Zhu,2006;Gallori et al.,1994;Poté et al.,2003).许多研究表明,环境中包含持久性存在的游离态DNA.这些胞外DNA可以吸附在底泥和土壤的复杂颗粒物上,减少与脱氧核糖核酸酶接触而避免降解(Niemeyer and Gessler,2002).Romanowski等(1991)在土壤中加入质粒DNA,在60d后仍能检测到其存在.研究表明,细胞外游离态的DNA分子同样可以作为抗性基因的携带者,可通过与环境中的细菌接触发生基因水平扩散从而将抗性基因整合或转导进入其它微生物体内(Paget and Simonet,1994),使得这些菌体获得抗性.因此,在抗生素抗性基因的研究中需要重视对环境中胞外游离态DNA的研究.
本文旨在建立一种可靠的河流底泥中胞外DNA的提取方法,并与胞内DNA提取方法相结合,同时分别提取河流水体中细菌体内的染色体DNA和质粒DNA.并将此方法应用于海河底泥和表层水体不同形态DNA的提取,为进一步研究海河水体中抗性基因的主要存在形态提供可行性.
2 材料与方法(Material and methods)
2.1 采样点选取
海河是中国北方重要水系,全长73公里,从西向东流经整个天津.本研究选取海河支干流5个代表性采样点:1)大沽桥:干流,位于天津的几何中心的商业娱乐区;2)洪泥河:支流,处于农业区;3)新开河:支流,城市泄洪与排涝;4)马厂减河:农业灌溉与城市排涝;5)入海口:海河的终点,流入渤海.采集的样品对分析对比不同样点的DNA形态有较好的代表性.
具体采样布点见图1.

图1 海河采样点Fig.1 Sampling sites of the Haihe River
2.2 样品采集
采样在2008年12月进行.采集海河表层底泥(0~20cm),水样在底泥的垂直高度上采集,采集表层水样(0.5m),记录水温.采集的底泥样品转移到无菌塑料袋中,水样收集到无菌塑料瓶中,4℃条件下立即运送回实验室,24h内分析.
2.3 底泥胞外DNA和胞内DNA提取
2.3.1 胞外DNA提取
1g底泥,放入10mL灭菌塑料试管,分别加入4mL NaH2PO4(0.12mol·L-1,pH 8.0) 和 100μL sodium dodecyl sulphate(SDS,10%)混匀,250r·min-125℃振荡10min;添加0.2g polyethylene poly pyrrolidone(PVPP) 混匀,4℃、6000r·min-1离心10min,0.22μm灭菌膜(Osmonics,USA)过滤,上清液收集到另一个试管A中至于冰上.离心后的沉淀再加4mL NaH2PO4,250r·min-125℃振荡5min,离心(条件同上)再收集上清液到A中;同上再提取1次.提取得到的上清液放置冰上,下一步提纯(Corinaldesi et al.,2005;Ogram et al.,1987).
2.3.2 胞内DNA提取
上述离心后的沉淀和过滤后膜的截留物用于提取胞内DNA,采用SDS高盐法,并做适当修改.沉淀和剪碎的滤膜加入 4mL DNA提取液(100mmol·L-1Tris-HCl,100mmol·L-1Na2-EDTA, 100mmol·L-1Na3PO4,1.5mol·L-1NaCl,1%CTAB, 50mg·mL-1protein K)和 0.5g 1mm玻璃珠(酸洗),2500r·min-1振荡10min;加入1mL SDS(10%)混匀,液氮放置1min后立即60℃水浴20min,重复3次,4℃、8000r·min-1离心20min,上清液转移到试管B中冰上放置.沉淀再次加入2mL DNA提取液,2500r·min-1振荡 5min,37℃水浴 2h,加入1mL SDS,60℃水浴20min,4℃、8000r·min-1离心20min.收集上清液到试管B,放置冰上,下一步提纯.
2.3.3 胞外DNA提取率
1g底泥样品在马弗炉450℃下烘烤2h,加入15μg的E.coli BH5α DNA,用上述的胞外DNA提取方法提取.
2.3.4 方法质量控制
验证胞外 DNA提取方法是否共提取胞内DNA.将过夜培养的 2mL菌液(E.coli BH5α,OD600=0.35)离心,加入DNase I(10mg·mL-1)37℃温浴15min(去除胞外DNA影响),过0.22μm灭菌膜,NaCl(1.5mol·L-1)溶液去除DNase残留.过滤后的膜与经过预处理的底泥样品(马弗炉450℃,2h)混合.用上述胞外DNA提取方法提取.提取物经16S rDNA PCR扩增.
2.4 水体细菌中质粒DNA和染色体DNA提取
2.4.1 水体中菌体收集
1L水样过0.22μm灭菌膜(Osmonics,USA),100mL溶液(0.25mol·L-1KH2PO4,0.75mol·L-1MgCl2)(Clesceri et al.,1995)冲洗两次.膜剪切成碎片放进 15mL离心管,加入 1.8mL SET溶液(20% sucrose,50mmol·L-1Tris-HCl,pH 7.6)(Rivera et al.,2003;Sommerville et al.,1989).
2.4.2 水体中菌体总DNA提取
6000r·min-1离心10min,弃去上清液;250μL溶液Ⅰ(50mmol·L-1sucrose,25mmol·L-1Tris-HCl,10mmol·L-1EDTA,RNase 1mg·mL-1,pH 8.0)重悬菌体,混匀;加入250μL溶液Ⅱ(0.2mol·L-1NaOH和1%SDS)混匀,至于冰上2min;加入250μL KAc(5mol·L-1,pH 4.8),加入的动作要快且柔和.混合液置于冰上5min,4℃、12000r·min-1离心10min.
2.4.3 质粒DNA和染色体DNA分离
上清液含有质粒DNA,沉淀中含有染色体DNA.将上清液转移到另外的试管中.沉淀加入2mL NaCl溶液(1.5mol·L-1),重悬沉淀.
2.4.4 方法质量控制
同样方法提取过夜培养的2mL菌液(E.coli BH5α,OD600=0.35)质粒DNA和染色体DNA,作为方法质量控制.提取物经16S rDNA和sul2 PCR扩增.
2.5 纯化
提取DNA使用 DNA纯化试剂盒(3S Spin Agarose Gel DNA Purifiction Kit Shenergy Biocolor, China)纯化.所有的提取物保存在-20℃.
2.6 16S rDNA和sul2扩增
16S rDNA扩增应用于胞外DNA提取质量控制(检验是否共提取了胞内DNA),采样点底泥胞外DNA和胞内DNA(提取方法应用性),水体染色体DNA和质粒DNA(提取方法的应用性和分离验证)及E.coli BH5α(分离验证)PCR扩增.sul2扩增应用在E.coli BH5α(分离验证)水体染色体DNA和质粒DNA(分离验证).

表1 DNA扩增引物Table 1 Primers of amplification
PCR扩增引物见表1.使用 Biometra PCR(Biometra TGradient,Germany)扩增仪.反应体系溶液包含:12.5μL 2×Taq PCR预混液[(0.1U·μL-1Taq polymerase,0.5mmol·L-1dNTP(dATP,dCTP, dGTP,dTTP),3mmol·L-1MgCl2,100mmol·L-1KCl,20mmol·L-1Tris-HCl,pH 8.3)]和其他的稳定剂(Tiangen biotech Ltd.,Beijing,China),0.5μL引物(1μmol·L-1),1μg DNA模板,加dd H2O到最终体积25μL.PCR反应条件:(1)16 S rDNA:95℃预热 5min,95℃、15s 30个循环,56℃退火 30s,72℃延长1min,最后72℃ 5min;阳性对照大肠杆菌提取的基因组DNA代替模板;(2)sul2:95℃预热 5min,95℃、15s 30个循环,60.8℃退火 30s,72℃延长1min,最后72℃、5min,阳性对照是E. coli BH5α提取的质粒代替模板.阴性对照是将模板由无菌水代替,其余不变.
3 结果与分析(Results and analysis)
3.1 DNA的纯度与浓度表征
使用紫外分光光度法测定 DNA溶液在230nm、260nm、280nm的光度值.DNA纯度依据OD260/280和OD260/230比值,比值越大说明DNA样品中蛋白质和腐殖酸杂质的含量越低,DNA样品越纯,反之亦然(Steffen et al.,1988).实验提取的DNA溶液OD260/280皆在1.8左右,OD260/230在2.0左右,表明提取物DNA纯度较好,不含PCR反应的抑制剂,达到PCR扩增要求.
底泥DNA浓度:OD260×50μg·mL-1×稀释倍数× DNA溶液体积(mL)/底泥重量(g);水体DNA浓度:OD260×50μg·mL-1×稀释倍数×DNA溶液体积(mL)/水体体积(L).
电泳:PCR产物(5μL)1.2%凝胶,DNA提取物(5μL)0.7%凝胶电泳,λDNA/HindⅢ与Marker 1作为分子标准.凝胶经EB染色,凝胶成像系统分析.
3.2 DNA提取方法质量控制
底泥中胞外DNA的提取应满足:1.溶液能最大限度的溶解底泥中的DNA;2.不能提取出胞内DNA;3.提取DNA的纯度要达到后即的分子实验要求.很多其他的因素,比如DNA吸附在土壤底泥颗粒物上,共提取的酶抑制剂和DNA的降解或剪切都可能影响DNA的提取效率(Corinaldesi et al.,2005).针对胞内DNA的提取方法的评判是:1.分子量大且完整(>23kb);2.没有分子分析抑制物(OD260/280),必须适用于样品分析和后继的DNA扩增.
方法质量控制结果,胞外DNA经16S rDNA电泳未出现条带,提取溶液中未含有胞内DNA,说明建立的胞外 DNA提取方法可靠.底泥提取DNA经16S rDNA PCR扩增后,所有的胞外DNA扩增产物呈阴性,胞内DNA呈阳性(图2).提取物不经稀释即可使用.试验数据和PCR结果表明上述的提取方法适用于不同区域底泥的胞外胞内DNA提取,而且可以用于后继的分子分析,方法简易可行.

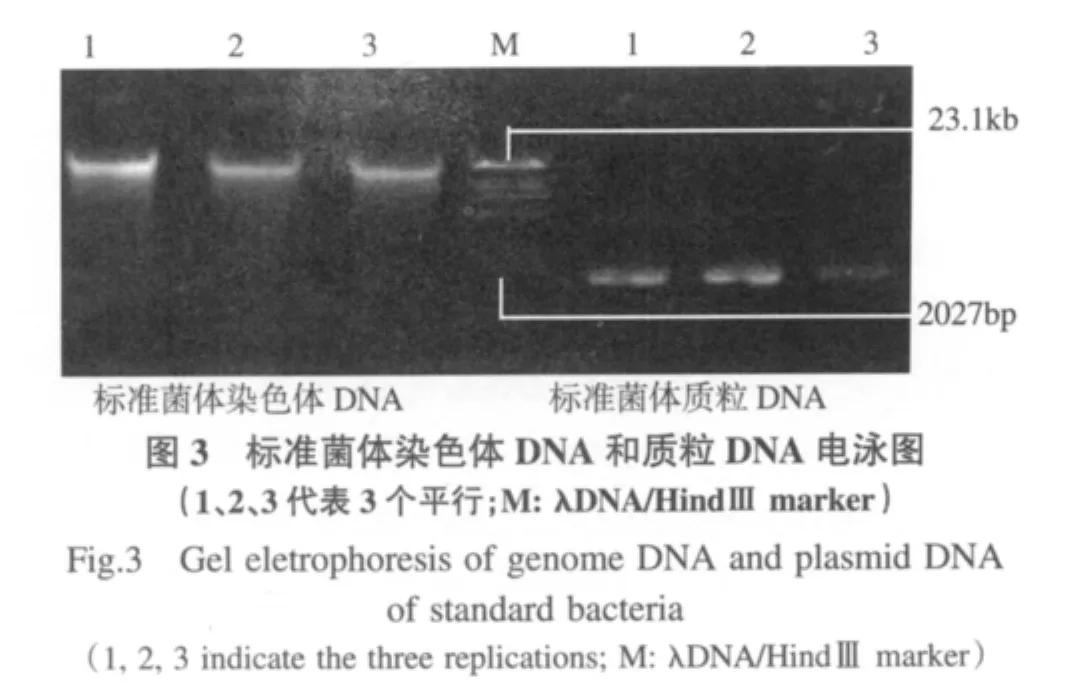
在碱性条件下大分子的线性染色体DNA彼此联结形成网状结构,与细胞膜、蛋白等形成共沉淀,共价闭合环状的质粒DNA仍然悬浮在上清液中,通过离心分离质粒DNA与染色体DNA.
验证同时提取染色体DNA和质粒DNA的方法.在方法质量控制中,利用建立的方法提取已知标准菌(构建含有 sul2抗性基因质粒的 E.coli BH5α)染色体DNA和质粒DNA.质粒DNA包括环状质粒DNA和开环线状质粒DNA.本实验提取的环状质粒DNA多于开环质粒DNA,根据图中泳带亮度可得,提取比例大约是3:1到4:1(图3).实验中发现,NaOH是实验中细胞膜裂解的关键. Frerix等(2007)表明NaOH浓度影响质粒DNA和染色体DNA的获得量.时间和动作剧烈程度对提取很重要,一旦染色体DNA在裂解中断裂,不会在中和条件下沉淀.经16S rDNA和sul2 PCR扩增发现,染色体DNA的16S rDNA结果全部阳性而sul2呈阴性,证明提取的染色体DNA没有包含质粒DNA;质粒DNA的16S rDNA结果呈阴性而sul2的 PCR结果呈阳性,证明没有参杂染色体DNA.
采样点水体中菌体提取的质粒DNA和染色体DNA全部进行16S rDNA和sul2的PCR扩增.染色体DNA的16S rDNA结果全部阳性而sul2呈阴性,证明提取的染色体DNA没有包含质粒DNA;质粒DNA的16S rDNA结果呈阴性而sul2的PCR结果呈阳性,证明没有参杂染色体DNA(图4).该方法表明同时提取染色体DNA和质粒DNA的方法可以应用在环境样品的提取过程,而且在分子分析中可以直接使用.

3.3 海河底泥中胞外DNA和胞内DNA浓度
胞外DNA提取率的分光值表明不同的底泥样品的提取率为40%~72%,与Corinaldesi等(2005)从海洋底泥中提取胞外DNA的提取率34%~60%接近,Corinaldesi等(2005)同时发现胞外 DNA的含量是胞内DNA的10~70倍.本研究结果表明,海河底泥5个采样点都存在相当浓度的游离态胞外DNA,其浓度明显高于胞内DNA(图5).不同区域的胞外DNA含量不同,大沽桥>马厂减河、洪泥河>新开河、海河入海口.本研究对5个样点底泥的统计分析表明(SPSS),海河底泥胞外DNA浓度与底泥中的黏粒含量有很好的相关性(r=0.979, p=0.01),这与底泥黏粒对DNA的吸附提供保护、减少核酸酶降解密切相关.电泳图分子量大小表明提取的胞外DNA大于1kb(图6).5个采样点底泥中胞内DNA含量不同,马厂减河、洪泥河>大沽桥>新开河>海河入海口(图5),提取物均大于23.1kb,提取过程无明显降解和剪切现象(图6).本研究表明底泥中存在的胞外DNA形态占很重要的比例,在研究中这部分形态的DNA不能忽视.

3.4 海河水体中染色体DNA和质粒DNA浓度
从5个采样点水体中菌体的染色体DNA和质粒DNA结果中发现大部分都以染色体DNA形态存在(图7).提取的水体菌体染色体DNA含量和质粒DNA含量均以洪泥河样品最高,其他各区域差异不大.

图7 海河水体中提取DNA浓度Fig.7 The concentration of DNA extraction in water of Haihe River
不同形态DNA提取的细化在研究中是非常必要的.Potè等(2003)和Agnelli等(2004)发现胞外DNA迁移性很强,可从土壤的上层迁移到底层.如果这些胞外DNA携带有致病基因并迁移到地下水中,将对人类健康造成最直接的威胁.如果携带抗性基因的胞外DNA转移到土著微生物体内,将直接影响环境中微生物群落的进化及生物种群多样性(Lorenz and Wackernagel,1994).同时质粒DNA与染色体DNA的分离,对定位环境中功能基因的研究,阐述基因分布、传播有很大的帮助.
本研究提出了从环境样本同时提取不同形态DNA的方法,是进一步细化分子生物研究有效的DNA提取方法,该方法对于进一步研究抗生素抗性基因在环境中的主要存在形态提供了有效的方法学支持.
Agnelli A,Ascher J,Corti G,Ceccherini M T,Nannipieri P, Pietramellara G.2004.Distribution of microbial communities in a forest soil profile investigated by microbial biomass, soil respiration and DGGE of total and extracellular DNA[J].Soil Biology Biochemistry,36(5):859-868
ClesceriL S,Eaton A D,Greenberg A.E.1995,Standard MethodsfortheExaminationofWaterandWastewater[M]. Washington DC:American Public Health Association
Corinaldesi C,Danovaro R,Dell’Anno A.2005.Simultaneous recovery of extracellular and intracellular DNA suitable for molecularstudies from marine sediments[J].Applied and Environmental Microbiology,71(1):46-50
Frerix A,Geilenkirchen P,Müller M,Kula M R,Hubbuch J. 2007.Separation ofgenomicDNA,RNA and open circular plasmid DNA from supercoiled plasmid DNA by combining denaturation,selective renaturation and aqueous two-phase extraction[J].Biotechnology and Bioengineering,96(1):57-66
Gallori E,Bazzicalupo M,Dal Canto L,Fani R,Nannipieri P, Vettori C,Stotzky G.1994.Transformation of Bacillus subtilis by DNA bound on clay in non-sterilesoil[J].FEMS Microbiology Ecology,15(1-2):119-126
Gao P P,Luo Y,Zhou Q X,Mao D Q.2009.Research advancement of antibiotic resistance genes(ARGs)in aquaculture environment[J].Asian Journal of Ecotoxicology,4(6):770-779(in Chinese)
Knapp C W,Engemann C A,Hanson M L,Keen P L,Hall K. J,Graham D W.2008.Indirect evidence of transposon-mediated selection of antibiotic resistance genes in aquatic systems at low leveloxytetracycline exposures[J].EnvironmentalScience & Technology,42(14):5348-5353
西部作为欠发达地区,经济社会发展水平整体落后于中东部地区,医疗技术水平也不例外。遵义医学院附属医院(以下简称“遵医附院”)院长余昌胤认为,制约西部地区医疗技术发展主要有以下四方面原因。
Krumperman P H.1983.Multiple antibiotic resistance indexing of Escherichia coli to identify high-risk sources of fecal contamination of foods[J].Applied and Environmental Microbiology,46(1):165-170
Lorenz M G,Wackernagel W.1994.Bacterial gene transfer by natural genetic transformation in the environment[J].Microbiology and Molecular Biology Reviews,58(3):563-602
Luo Y,Zhou Q X.2008.Antibiotic resistance genes(ARGs)as emerging pollutants[J].Acta Scientiae Circumstantiae,28(8):1499-1505(in Chinese)
Nielsen K M,Bones A M,Smalla K,Van Elsas J D.1998. Horizontalgene transfer from transgenic plants to terrestrial bacteri-a rare event?[J].FEMS Microbiology Reviews,22(2):79-103
Nielsen K M,Johnsen P J,Bensasson D,Daffonchio D.2007. Release and persistence of extracellular DNA in the environment[J].Environmental Biosafety Research,6(1-2):37-53
Niemeyer J,Gessler F.2002.Determination of free DNA in soils[J].Journal of Plant Nutrition and Soil Science,165(2):121-124 Ogram A,Sayler G S,Barkay T.1987.The extraction and purification of microbial DNA from sediments[J].Journal of Microbiological Methods,7(2-3):57-66
Paget E,Simonet P.1994.On the track of natural transformation in soil[J].FEMS Microbiology Ecology,15(1-2):109-118
Pei R,Kim S C,Carlson K H,Pruden A.2006.Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes(ARG)[J].Water Research, 40(12):2427-2435
Pruden A,Pei R,Storteboom H,Carlson K H.2006.Antibiotic resistance genes as emerging contaminants:Studies in Northern Colorado[J].Environmental Science& Technology,40(23):7445-7450
Rivera I N,Lipp E K,Gil A,Choopun N,Huq A,Colwell R R.2003.Method of DNA extraction and application of multiplex polymerse chain reaction to detect toxigenic Vibrio cholerae O1 and O139 from aquatic ecosystem[J].Environmental Microbiology, 5(7):599-606
Romanowski G,Lorenz M G,Wackernagel W.1991.Adsorption ofplasmid DNA to mineralsurfaces and protection against DNaseI[J].Applied and Environmental Microbiology,57(4):1057-1061
Sommerville C C,Knight I T,Straube W L,Carlson K H. 1989.Simple,rapid method for direct isolation of nucleic acids from aquatic environments[J].Applied and Environmental Microbiology,55(3):548-554
Steffan R J,Coksøyr J,Bej A K,Atlas R M.1988.Recovery of DNA from soils and sediments[J].Applied and Environmental Microbiology,54(12):2908-2915
Volossiouk T,Robb E J,Nazar R N.1995.Direct DNA extraction
for PCR-mediated assays of soil organisms[J].Applied and Environmental Microbiology,61(11):3972-3976
Weisburg W G,Barns S M,Pelletier D A,LaneD J.1991.16S Ribosomal DNA amplification forphylogeneticstudy[J]. Journal of Bacteriology,173(2):697-703
Zhu B.2006.Degradation of plasmid and plant DNA in water microcosms monitored by natural transformation and real-time polymerase chain reaction(PCR)[J].Water Research,40(17):3231-3238
中文参考文献
高盼盼,罗义,周启星,毛大庆.2009.水产养殖环境中抗生素抗性基因(ARGs)的研究及进展[J].生态毒理学报,4(6):770-779
罗义,周启星.2008.抗生素抗性基因(ARGs)——一种新型环境污染物[J].环境科学学报,28(8):1499-1505
Methods of Extraction Different Gene Types of Sediments and Water for PCR Amplification
FENG Ling,LUO Yi*
Key Laboratory of Pollution Processes and Environmental Criteria,Ministry of Education,College of Environmental Sciences and Engineering,Nankai University,Tianjin 300071
1673-5897(2010)2-280-07
X502
A
罗义(1971—),女,南开大学副教授,硕士研究生导师,主要从事污染生态化学、分子生态毒理、分子生物标记等方面的研究.
2009-12-13 录用日期:2010-01-10
国家自然科学基金(No.2077704;No.30870363);天津市自然科学基金(No.08JCYBJC02700)
冯凌(1984—),女,硕士研究生;*通讯作者(Corresponding author),E-mail:luoy@nankai.edu.cn

