耐环丙沙星大肠埃希菌对氨基糖苷类药物的耐药性探讨*
张馨琢,黄永茂,陈 庄,钟 利,陈 枫,游春芳,邓 敏
国内各地近年来报道大肠埃希菌(Escherichia coli,E.coli)对环丙沙星(ciprofloxacin,CIP)的耐药率已超过50%,并且这些耐CIP菌株对氨基糖苷类药物(aminoglycosides,AGs)的耐药形势逐渐严峻,其耐药机制最重要的是产生氨基糖苷类修饰酶,此类酶作用于药物特定的氨基或羟基,使药物钝化,使其降低或丧失对靶位核糖体的亲和力,使细菌在药物存在的情况下仍能存活。为此本文对临床分离的耐CIP大肠埃希菌对AGs药物的耐药表型和基因型进行了相关分析,为临床合理选用AGs药物提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌株来源 分离自本院 2007年 7月至2008年7月非肠道感染的住院病例大肠埃希菌75株(不含同一病人同一部位感染的重复菌株),经MicroScan WalkAway-40型全自动细菌鉴定仪鉴定。大肠埃希菌质控株ATCC 25922购自国家临床检验中心。
1.1.2 主要试剂 环丙沙星、链霉素、庆大霉素、妥布霉素、卡那霉素、阿米卡星、奈替米星纸片均购自杭州天和微生物试剂公司;M-H培养基、胰大豆蛋白胨肉汤购自英国Oxiod公司;LB液体培养基购自上海生工生物技术公司;Biospin细菌基因组DNA提取试剂盒购自杭州博日科技有限公司;2×Taq PCR MasterMix试剂盒、电泳琼脂糖购自北京天根生化科技公司。
1.2 方法
1.2.1 药物敏感试验 按NCCLS推荐的纸片扩散法(Kerby-Bauer法)测定75株大肠埃希菌对环丙沙星(CIP)的耐药率,把菌株分为耐CIP组与非耐CIP组。K-B法测定2组菌株对6种AGs(链霉素、庆大霉素、妥布霉素、卡那霉素、阿米卡星、奈替米星)的耐药性。以大肠埃希菌ATCC 25922为质控菌株。结果判读参照NCCLS2006常规标准。
1.2.2 氨基糖苷类修饰酶(AMEs)基因检测 用Biospin细菌基因组DNA提取试剂盒提取大肠埃希菌临床分离株和标准菌株ATCC 25922的基因组DNA,操作步骤按照说明书进行。参考文献〔1〕设计6对引物,由上海生工生物技术公司合成,见表1。PCR反应采用2×Taq PCR MasterMix试剂盒,按说明书进行操作。6种靶基因PCR扩增体系均为:每反应体系加入 P1、P2引物各 10μ mol/L,dNTP each 6.25μ mol/L,KCl 1 250μ mol/L,MgC12 37.5μ mol/L,Tris-HCl(pH8.3)250μ mol/L,Taq Polymerase 1.25U,总反应体积 25μL,其中模板液5μL。PCR反应循环参数为:94℃预变性 3min,94℃变性30 s,55℃退火 30s,72℃延伸30s,共 30个循环;循环结束后72℃延伸5min。PCR产物用2%琼脂糖凝胶电泳80V 50min,以DNA 100-600bp ladder Marker为分子量标记,电泳后用全自动凝胶成像系统观察PCR产物的大小并贮存结果。收集到DNA溶液(未纯化)用核酸定量仪测得浓度>390ng/μL,送至上海生工生物技术公司进行基因测序。

表1 6种氨基糖苷类修饰酶基因引物序列Table 1 The primers'sequeses of six aminoglycosides modifing enzymes genes
1.2.3 统计学方法 采用SPSS14.0进行χ2检验,比较耐CIP组与非耐CIP组之间对氨基糖苷类的耐药表型和基因型有无差别。α=0.05。
2 结 果
2.1 药物敏感试验结果 75株大肠埃希菌中,53株(70.67%)对CIP耐药。耐CIP组对庆大霉素、卡那霉素、链霉素的耐药率较高:73.58%、64.15%、62.26%,对庆大霉素、妥布霉素、卡那霉素的耐药率与非耐CIP组的差异有统计学意义(P<0.05),耐CIP组与非耐CIP组对6种AGs耐药率的比较详见表2。耐CIP组以多重耐药表型常见,且比例明显高于非耐CIP组(P=0.001),而非耐CIP组全敏感表型所占比例与耐CIP组的差异有统计学意义(P=0.002),耐CIP组与非耐CIP组对6种AGs耐药表型的比较详见表3。耐CIP组对AGs的多重耐药表型复杂:16株(30.19%)对3种AGs耐药,7株(13.21%)对5种AGs耐药;而非耐CIP组仅3株表现为多重耐药。
2.2 氨基糖苷类修饰酶基因检测结果 耐CIP组AMEs基因的总检出率为 77.36%,其中 30株(56.60%)检出aac(3)-II,22株(41.51%)检出 aac(6′)-I。22株非耐 CIP菌株 AMEs基因的总检出率为59.09%,其中 aac(3)-II、ant(3〞)-I的检出率较高。aac(6′)-I的检出率,耐CIP组明显高于非耐CIP组(P=0.006);其余5种基因检出率的差异无统计学意义(P>0.05)。耐CIP组与非耐CIP组AMEs基因检出率的比较详见表4、图1。单基因型、双基因型、三基因型的检出率在耐CIP组与非耐CIP组中的差异无统计学意义(P>0.05),见表5。耐CIP组中,对应6种AGs的耐药表型,aac(3)-II在全敏、单耐药、双耐药、多重耐药中的检出率分别为:33.33%、50%、45.45%、62.50%;aac(6′)-I在对AGs的全敏、单耐药、双耐药、多重耐药表型中的检出率分别为:0、0、27.27%、59.38%;ant(2〞)-I在对AGs的(全敏+单耐药+双耐药)表型中的检出率为9.09%,在多重耐药表型中的检出率为9.38%;ant(3〞)-I在对AGs的(全敏+单耐药+双耐药)表型中的检出率为9.09%,在多重耐药表型中的检出率为21.88%。由此可见,随着耐CIP菌株对AGs耐药种类的增加,AMEs基因的检出率也逐渐增高,以aac(3)-II、aac(6′)-I最为明显,并且对AGs多重耐药的菌株常常检出多个AMEs基因;而非耐CIP菌株耐药表型与基因型的关系未呈现此现象。各组PCR产物经基因测序后证实是E.coli的 aac(3)-II、aac(6′)-I、ant(3 〞)-I、ant(2〞)-I、aac(3)-I基因序列。
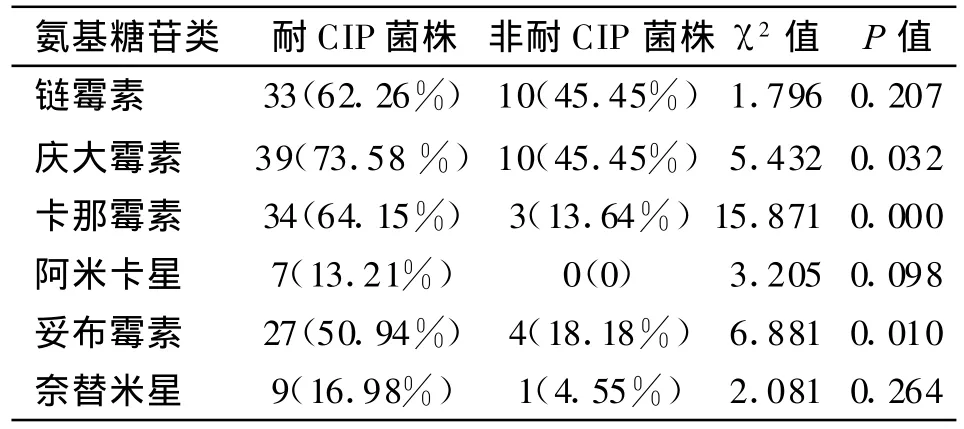
表2 耐CIP与非耐CIP菌株AGs耐药率的比较Table 2 Drug resistant differences between ciprofloxacin-resistant strains and non-ciprofloxacin-resistant strains to aminoglycoside antibiotics
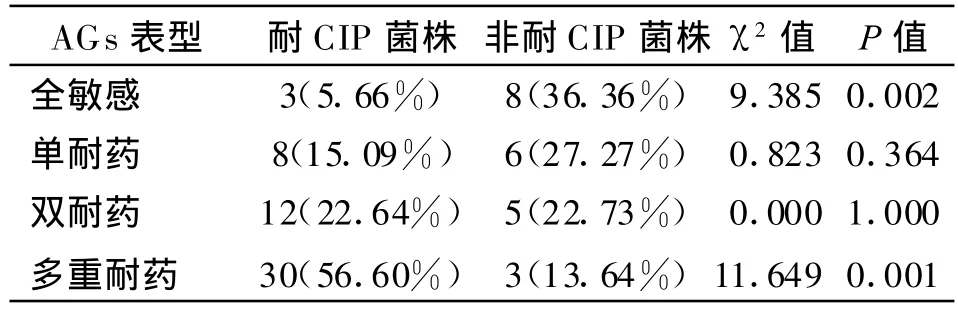
表3 耐 CIP与非耐CIP菌株AGs表型的比较Table 3 Difference of drug-resistant phenotypes between ciprofloxacin-resistant strains and non-ciprofloxacin-resistant strains to aminoglycoside antibiotics
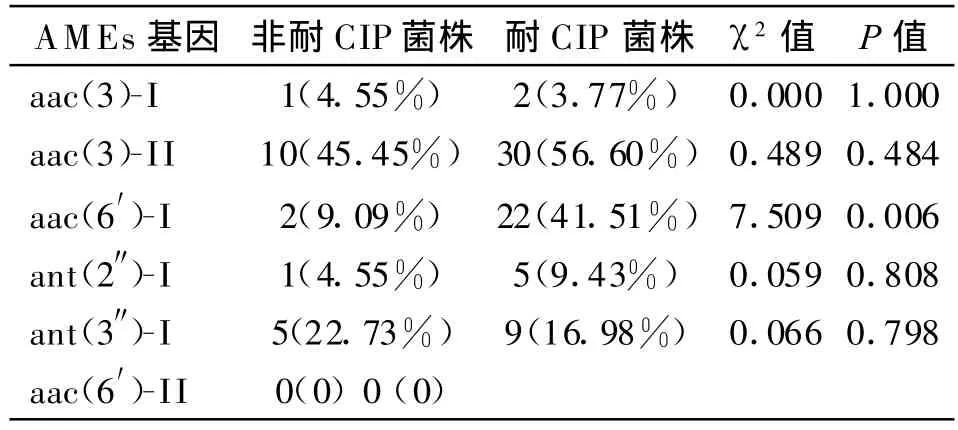
表4 耐CIP与非耐CIP菌株AMEs基因型的比较Table 4 Difference of AMEs genetypesbetween ciprofloxacin-resistant strains and non-ciprofloxacin-resistant strains
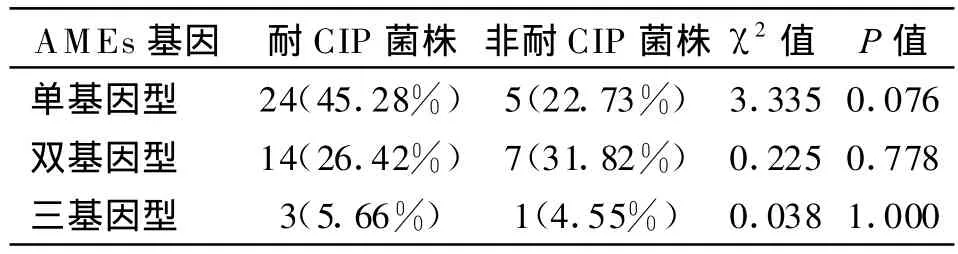
表5 耐CIP与非耐CIP菌株 AMEs基因型比较Table 5 Difference of AMEs genetypesbetween ciprofloxacin-resistant strains and non-ciprofloxacin-resistant strains
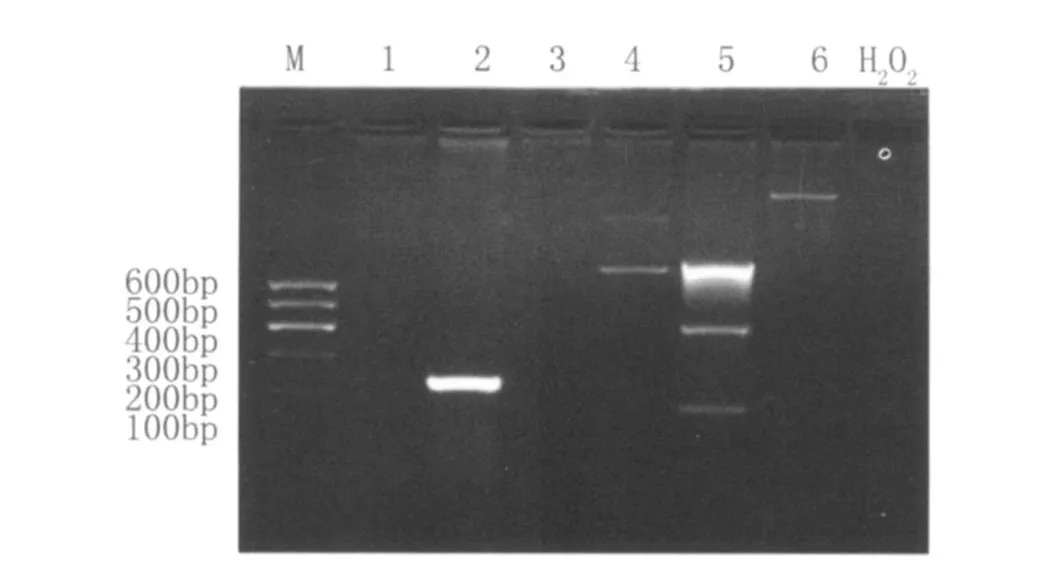
图1 PCR电泳图谱Fig.1 Electrophoregram of PCR
3 讨 论
大肠埃希菌是临床上细菌感染常见病原菌之一。随着抗菌药物的广泛应用,细菌的耐药性日趋严重,给临床抗感染治疗的抗生素选择增加了难度。本地区耐CIP的临床分离株对庆大霉素、妥布霉素、卡那霉素的耐药率比非耐CIP菌株有明显增高(P<0.05),且对AGs的耐药表型复杂,其中多重耐药表型占56.60%,明显高于非耐CIP菌株(P<0.05);而非耐CIP菌株则以全敏感、单耐药表型较为多见。耐CIP菌株对大部分AGs的耐药率增高,且容易发生对AGs的多重耐药,这可能与耐CIP菌株同时携带多种耐药基因有关〔2-5〕。
耐CIP菌株对庆大霉素(73.58%)、卡那霉素(64.15%)、链霉素(62.26%)的耐药率较高,并且对AGs药物出现了较高的交叉耐药率。其耐药机制复杂,其中最重要的机制是产AMEs。目前从美国GenBank核酸库已能检索到AMEs 30多种基因型,但在大肠埃希菌中以 aac(3)-II、aac(6′)-I、ant(3〞)-I等基因型为常见。本文AMEs基因检测结果具有相似的结果:耐CIP菌株AMEs基因的总检出率为 77.36%,其中 aac(3)-II为 56.60%,aac(6′)-I为 43.40%,耐 CIP 菌株 aac(6′)-I的检出率明显高于非耐CIP菌株(P=0.006)。本文发现一株耐CIP菌株(标本号20060)的基因扩增序列与GenBank gb|DQ303918.1|大肠埃希菌氟喹诺酮乙酰化氨基糖苷乙酰转移酶基因aac(6')-Ib-cr的核苷酸序列相匹配,与文献报道相似〔6〕。aac(6')-Ib-cr是aac(6')-Ib-cr的一种变异体,两处碱基的突变赋予了其修饰喹诺酮类的特性。AAC(6')-Ib-cr不仅可以修饰氨基糖苷类药物,对环丙沙星、诺氟沙星等喹诺酮类药物也具有乙酰化修饰作用〔7-9〕。
耐CIP菌株多表现为多基因型和多重耐药表型,且两者关系较为复杂,耐药表型和AMEs基因检测结果未能达到一一对应;而非耐CIP菌株则以单基因型和单耐药表型多见。AMEs在细菌体内的实际表达量以及细菌对药物的摄入和水解速度都可能影响分离菌最终的耐药表型〔10〕。
综上所述,本地区耐CIP大肠埃希菌对氨基糖苷类药物的交叉耐药情况较为常见,耐CIP菌株多表现为同时对多种氨基糖苷类药物耐药。耐CIP大肠埃希菌对氨基糖苷类药物的耐药与氨基糖苷类修饰酶存在一定相关性,以AAC(6′)-I最为明显。
〔1〕糜祖煌,陆亚华,黄支密,等.PCR检测氨基糖苷类修饰酶基因〔J〕.中华微生物学和免疫学杂志,2005,25(2):160.
〔2〕Li XZ.Quinolone resistance in bacteria:emphasis on plasmidmediated mechanisms〔J〕.Int J Antimicrob Agents,2005,25(6):453-463.
〔3〕Luzzaro F.Fluoroquinolones and Gram-negative bacteria:antimicrobial activity and mechanisms of resistance〔J〕.Infez Med,2008,16 Suppl 2:5-11.
〔4〕Périchon B,Courvalin P,Galimand M.Transferable resistance to aminoglycosides by methy lation of G1405 in 16S rRNA and to hydrophilic fluoroquinolones by QepA-mediated efflux in Escherichia coli〔J〕.Antimicrob Agents Chemother,2007,51(7):2464-2469.
〔5〕Périchon B,Bogaerts P,Lambert T,et al.Sequence of conjugative plasmid pIP1206 mediating resistance to aminoglycosides by 16S rRNA methylation and to hydrophilic fluoroquinolones by efflux 〔J〕.Antimicrob Agents Chemother,2008,52(7):2581-2592.
〔6〕Yang H,Chen H,Yang Q,et al.High prevalence of plasmidmediated quinolone resistance genes qnr and aac(6ˊ)-Ib-cr in clinical isolates of Enterobacteriaceae from nine teaching hospitals in China〔J〕.Antimicrob Agents Chemother,2008,52(12):4268-4273.
〔7〕Park C H,Robicsek A,Jacoby G A,et al.Prevalence in the United States of aac(6')-Ib-cr encoding a ciprofloxacin-modifying enzyme 〔J〕.Antimicrob Agents Chemother, 2006, 50:3953-3955.
〔8〕Morgan-Linnell SK,Becnel Boyd L,Steffen D,et al.Mechanisms accounting for fluoroquinolone resistance in Escherichia coli clinical isolates〔J〕.Antimicrob Agents Chemother,2009,53(1):235-241.
〔9〕Vetting MW,Park CH,Hegde SS,et al.Mechanistic and structural analysis of aminoglycoside N-acetyltransferase AAC(6')-Ib and its bifunctional,fluoroquinolone-active AAC(6')-Ib-cr variant〔J〕.Biochemistry,2008,47(37):9825-9835.
〔10〕Jakoniuk P,Wieczorek P,Sacha PT,et al.T he role of aminoglycoside-modifying enzymes in resistance of G ram-negative rods〔J〕.Med Dosw Mikrobiol,2006,58(4):363-370.

