黄瓜瓜长性状的QTL定位分析
程周超 顾兴芳 张圣平 苗 晗 张若纬 刘苗苗 杨双娟
(中国农业科学院蔬菜花卉研究所,北京 100081)
黄瓜(Cucumis sativus L.)是我国的重要蔬菜作物,培育高产、优质、多抗的黄瓜品种是黄瓜育种者的共同目标(顾兴芳 等,2005)。近年来,分子标记辅助选择(MAS)育种技术的应用,极大地加速了育种进程,然而这依赖于饱和的遗传图谱和准确的性状QTL定位(Tanksley et al.,1989)。由于黄瓜的遗传基础狭窄,自 1994年 Kennard将分子标记技术用于遗传图谱的构建到2009年第一张饱和SSR分子图谱的完成(Ren et al.,2009)期间,构建的主要黄瓜遗传图谱(Park et al.,2000;Bradeen et al.,2001;Fazio et al.,2003;Yuan et al.,2007)存在饱和度低、通用标记少、连锁群与染色体不对应等缺点,第一张饱和SSR遗传图谱(Ren et al.,2009)虽然克服了上述缺点,但是没有对黄瓜的农艺性状进行QTL分析,因此不能将其直接应用于分子标记辅助选择育种。
黄瓜果实长度是其重要的外观品质性状。由于消费习惯和用途的不同,各地对黄瓜长度的要求不一,因此,研究控制果实长度的基因,可以为黄瓜品质育种提供理论依据。前人对黄瓜瓜长的QTL进行了一些研究(Kennard & Havey,1995;Serquen et al.,1997;Fazio et al.,2003;Yuan et al.,2007),这些报道中检测到的瓜长QTL在4~6个之间,但由于所用试验材料不同,造成QTL数和位置也不同,同时缺少相同的锚定标记,且已报道的QTL也没有与相应的染色体对应,使得难于对不同图谱间的QTL进行准确的比较。
本试验以黄瓜野生变种PI 183967和新泰密刺选系931杂交获得的F2群体为作图群体,构建了一张黄瓜SSR遗传图谱,并对瓜长进行了QTL定位分析,为黄瓜MAS育种提供理论依据。
1 材料与方法
1.1 试验材料及性状调查
试验材料为黄瓜野生变种PI183967(来源于美国威斯康辛大学)、新泰密刺选系931(来源于中国农业科学院蔬菜花卉研究所)和以两者为亲本的F2、F3群体。
2008年秋分别将两个亲本和F2种植于本所温室中,F2群体共包含179个单株;2009年春将179个F2:3家系及亲本种植在中国农业科学院南口实验基地温室内,每家系种植10株,均进行常规栽培管理。当果实长度达到商品瓜大小时测量瓜长,测量标准为正常商品瓜瓜蒂至瓜顶的长度(李锡香 等,2005)。取F3各家系内瓜长的平均值用于QTL分析。
1.2 遗传分析
采用DPSv6.55数据统计软件对亲本的瓜长数据进行差异显著性分析,应用Excel 2003检验瓜长在F2群体及F2:3家系中的分布是否符合正态分布。
1.3 遗传图谱的构建
1.3.1 SSR引物的来源 本试验所用的SSR引物来源于黄瓜全基因组序列,共2112对(Ren et al.,2009)。
1.3.2 SSR分析 从亲本和 F2群体 179个单株侧枝上取叶片,冷冻处理后,用改良的 CTAB法(Clark,1998)提取基因组 DNA;SSR 反应体系:总体积 15 µL,1.5 µL 10×PCR buffer,0.2 µL dNTP(2.5 mmol·µL-1),0.15 µL Taq 酶(5 U·µL-1),2 µL DNA(10ng·µL-1),正向、反向引物(50ng·µL-1)各1 µL,其余以ddH2O补足15 µL。PCR程序为:94 ℃预变性4 min,94 ℃变性15 s,55 ℃退火15 s,72 ℃延伸30s,34个循环;72 ℃保温5 min;扩增产物用8 %非变性聚丙烯酰胺凝胶160V恒功率电泳分离,银染显色后在观察灯箱上进行数据分析。
1.3.3 标记的选择 本试验使用的 SSR标记分两部分进行选择:①将利用亲本筛选的有多态性的标记与黄瓜高密度图谱(Ren et al.,2009)上的标记进行比较分析,从中挑选出条带清晰、稳定,而且没有定位在高密度图谱上的SSR标记,全部用于分离群体的遗传连锁图谱构建;②为了保证遗传图谱上标记分布的均匀性,又从亲本筛选出的有多态性的标记中挑选出了一些标记,这些标记的选择是根据它们在黄瓜染色体上的位置,以大约每5 cM的距离进行挑选。
1.3.4 数据统计方法及图谱的构建 共显性标记的统计方法:来自母本(PI183967)的带型记为a,来自父本(931)的带型记为b,杂合的带型记为h,未扩增出的或模糊不清的记为u。显性标记的统计方法:若 P1为显性,则 F2单株中和 P1带型一致的记为 d,和 P2带型一致的记为 b;若P2为显性,则 F2单株中和 P1带型一致的记为 a,和 P2带型一致的记为 c。最后利用 JoinMap3.0(van Ooijen & Voorrips,2001),LOD阈值3.0,分析数据获得黄瓜遗传图谱。
1.4 QTL分析与命名
利用MapQTL 4.0分析软件进行QTL分析,首先以置换测验确定QTL存在的LOD阈值,然后利用区间作图法(IM)进行QTL分析,将最高LOD值所在位置的标记或与其紧密连锁的标记作为协同因子,再对IM检测到的QTL进行多座位QTL模型(Multiple-QTL model,MQM)检测,以连锁群上 LOD值最高的位置作为 QTL的位置,以 2-LOD的区间作为 95 %的置信区间(van Ooijen,1992)。QTL的命名参照Yuan等(2007)的方法。
2 结果与分析
2.1 F2遗传图谱的构建
利用两构图亲本PI183967和931进行多态性筛选,从2112对引物中,获得996对多态性引物,多态率为47.2 %;从中挑选出319对SSR引物对F2群体进行分析,排除群体分析中条带模糊不清的标记,最终得到238对可用标记,建立了包含7个连锁群,234个SSR标记的黄瓜遗传图谱(图1)。该图谱覆盖基因组长度829.7 cM,平均图距3.5 cM。每个连锁群的标记数在 20~51之间,LG3包含的标记数最多,有51个;LG7含有的标记数最少,只有20个;连锁群的长度在56.4~158.7 cM之间;图谱密度在2.7~6.5 cM·maker-1之间,LG4的平均间距最小,为2.7 cM·marker-1;LG2的平均间距最大,为6.5 cM·maker-1。应用MapInspect软件对该图谱和黄瓜高密度遗传图谱(Ren et al.,2009)进行比较,7个连锁群上共有86个标记与高密度图谱一致,本试验中的1~7个连锁群中分别有17、12、17、5、9、16、10个标记和高密度图谱的1~7条染色体对应,从而确定了本试验构建的7个连锁群和黄瓜7条染色体的对应关系(表1)。

表1 黄瓜F2SSR遗传图谱
2.2 瓜长QTL分析
2.2.1 遗传分析 差异显著性分析结果显示(表2),瓜长在两亲本间表现出极显著差异,F2群体及F3的瓜长平均值分别为12.74和10.4。标准差分别为2.75和2.24,均表现出明显的分离。并且其分布为单峰分布,F2群体的峰值为0.23,偏度为-0.26,绝对值均小于 2,是数量性状的典型特征;在F3群体中,峰值为1.25,偏度为 4.13,绝对值大于 2,说明对于标准的正态分布而言,该分布呈尖顶峰,但还是表现出明显的单峰分布,因此基本符合正态分布,可用于数量性状的定位分析(图2、3)。
2.2.2 F2群体 QTL定位 用 MapQTL 4.0复合区间作图法对 2008年秋季 F2的瓜长进行 QTL分析,共检测到2个QTL(表3),将其命名为fl1.1和fl1.2,分别位于LG1(Chr1)的SSR02734~SSR05709之间和 LG4(Chr4)的SSR19596~SSR20063之间,fl1.1和fl1.2分别和标记SSR05709和SSR20063更接近,这两点的贡献率分别为14.1 %和12.5 %;加性效应为负,表现为减效加性效应,说明这些位点上使果实长度偏短的效应主要来自于黄瓜野生变种PI183967;这两个QTL的显性势(显性效应和加性效应比值的绝对值)分别为0.21和0.23,根据Stuber等(1987)提出的QTL作用方式的判别标准,fl1.1和fl1.2均为部分显性效应。

表2 黄瓜瓜长在亲本间的显著性分析及F2群体和F2:3家系的平均值

图1 PI183967、新泰密刺931 F2连锁图谱及瓜长的QTL位点
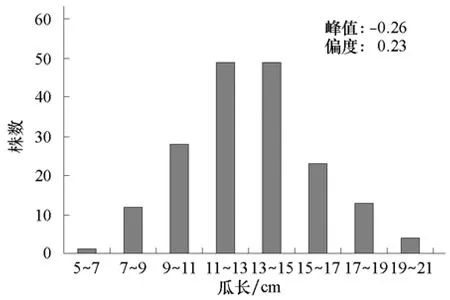
图2 瓜长在黄瓜F2群体的分布

图3 瓜长在黄瓜F2:3家系的分布
2.2.3 F2:3家系QTL定位 用同样的作图方法对2009年春季F2:3家系的瓜长进行QTL分析,共检测到3个QTL(表3),将其命名为fl1.1、fl4.2和fl6.1,分别位于LG1(Chr1)的SSR03680~SSR19914之间、LG4(Chr4)的 SSR03820~SSR12180之间和 LG6(Chr6)的 SSR15516~SSR23284之间。其中,贡献率分别为9.4 %、13.3 %和7.1 %,其中fl4.2的贡献率最高,fl6.1的贡献率最低;加性效应均为负,表现为减效加性效应,和F2检测到的QTL的效应相同,也说明了果实长度偏短的效应主要来自于黄瓜野生变种 PI183967;fl1.1、fl4.2和 fl6.1的显性势分别为0.19、0.34和0.53,依据Stuber等(1987)提出的差别标准,fl1.1为加性效应,fl4.2和fl6.1均为部分显性效应。

表3 黄瓜瓜长在F2群体及F2:3家系的QTL定位
3 讨论
基于已经构建的黄瓜高密度遗传图谱(Ren et al.,2009),本试验有目的地选择作图标记,构建了SSR遗传图谱,该图谱每个连锁群上的标记分布都很均匀,不存在较大的间隙,从而有利于进一步的QTL定位;并且除了共有标记外,在该图谱上还有208对标记没有在高密度图谱上得到定位,这也为今后的图谱整合工作奠定了基础;通过和高密度图谱的比较,确立了连锁群和染色体的对应关系,实现了数量性状位点和染色体的对应,为分子标记辅助育种提供了更直观的依据。
不同的试验群体会造成QTL数和位置的不同(Dijkuizen & Staub,2002)。本试验中F2:3家系检测到的瓜长QTL为3个,多于F2检测到的2个,并且与Owens等(1985)、Lower和Edwards(1986)根据经验估计控制瓜长的基因数为4个的结论较为接近。fl1.2和fl1.1位置相邻(≤3 cM),在两个世代间能够相互对应,推测其可能为同一个QTL,只是由于受到环境的影响而导致了不同世代间相对位置的微小变化;LG4(Chr4)上的fl4.1和fl4.2相距较远,且F2:3家系在LG6(Chr6)上检测了fl6.1,而 F2在该连锁群上未检测到瓜长 QTL,这种不同可能是由于两个世代的环境条件(温度、地域等)差异,存在基因型和环境的互作效应而产生的。
顾兴芳,张圣平,王烨.2005.我国黄瓜育种研究进展.中国蔬菜,(12):1-7.
李锡香,朱德蔚.2005.黄瓜种质资源描述规范和数据标准.北京:中国农业出版社.
张海英,葛风伟,王永健.2004.黄瓜分子遗传图谱的构建.园艺学报,31(5):617-622.
Bradeen J M,Staub J E,Wye C,Antonise R,Peleman J.2001.Towards an expanded and integrated linkage map of cucumber(Cucumis sativus L.).Genome,44:111–119.
Clark M S.1998.Experimentalmanual of plant molecular biology.Beijing:Higher Education Press:427.
Dijkuizen A,Staub J E.2002.QTL conditioning yield and fruit quality traits in cucumber (Cucumis sativus L.):effects of environment and genetic background.J New Seeds,4:1-30.
Fazio G,Staub J E.2003.Geneticmapping and QTL analysis of horticultural traits in cucumber(Cucumis sativus L.)using recombinant inbred lines.Theor Appl Genet,107:864-874
Fukino N,Ohara T,Monforte A J,Sugiyama M,Sakata Y,Kunihisa M,Matsumoto S.2008.Identification of QTLs for resistance to powdery mildew and SSR markers diagnostic for powdery mildew resistance genes in melon(Cucumis melo L.).Theor Appl Genet,118(1):165-175.
Kennard W C,Poetter K,Dijkhuizen A,Meglic V,Staub J E,and Havey M J.1994.Among RFLP,RAPD,isozyme,disease resistance and morphological markers in narrow and wide crosses of cucumber,Theor Appl Genet,89:42-48.
Kennard W C,Havey M J.1995.Quantitative trait analysis of fruit quality in cucumber,QTL detection,confirmation,and comparison with mating-design variation.Theor Appl Genet,91:53-61.
Lower R L,Edwards M D.1986.Cucumber breeding//Bassett M J ed.Breeding vegetable crops,AVI.Westport,Connecticut,USA:173-207.
Owens K W,Bliss F A,Peterson C E.1985.Genetic analysis of fruit length and weight in two cucumber populations using the inbred backcross line method.J Am Soc Hortic Sci,110:431-436.
Park Y H,Sensoy S,Wye C.2000.A geneticmap of cucumber composed of RAPDs,RFLPs,AFLPs,and loci conditioning resistance to papaya ringspot and zucchini yellow mosaic viruses.Genome,43(6):1003-1010.
Ren Yi,Zhang zhong-hua,Liu Jin-hua,Staub J E,Han Yong-hua,Cheng zhou-chao,Li Xue-feng,Lu Jing-yuan,Miao Han,Kang Hou-xiang,Xie Bing-yan,Gu Xing-fang,Wang Xiao-wu,Du Yong-chen,Jin Wei-wei,Huang San-wen.2009.An integrated genetic and cytogeneticmap of the cucumber genome.PloS ONE,4(6):e5795.
Serquen F C,Bacher J,Staub J E.1997.Mapping and QTL analysis of horticultural traits in a narrow cross in cucumber(Cucumis sativus L.)using random amplified polymorphic DNA makers.Molecular Breeding,3(4):257-268.
Stuber C W,Edwards M D,Wendel J F.1987.Molecular marker-facilitated investigations of quantitative trait loci in maize.Factors influencing yield and its component traits.Crop Science,27(3):639-648.
Tanksley S D,Young N D,Paterson A H,Bonierbale M W.1989.RFLP mapping in plant breeding:new tools for an old science.Nature Biotechnology,7:247-264.
van Ooijen J W.1992.Accuracy of mapping quantitative trait loci in autogamous species.Theor Appl Genet,84:803-811.
van Ooijen J,Voorrips R.2001.JoinMap3.0,software for calculation of genetic linkage maps.Wageningen:Plant Research International.
Yuan X J,Li X Z,Pan J S,Wang G,Jiang S,Li X H,Deng S L,He H L,Si M X,Lai L,Wu A Z,Zhu L H,Cai R.2007.Genetic linkage map construction and location of QTLs for fruit-related traits in cucumber.Plant Breeding,127:180–188.

