大黄鱼野生与养殖群体遗传结构的比较研究
陈 锦, 黎中宝, 方 秀, 雷光高, 张桂玲, 赵斌丽, 王展林
(1. 集美大学 水产学院, 福建 厦门361021; 2. 集美大学 水产生物技术研究所, 福建 厦门361021; 3. 福建闽威水产实业有限公司, 福建 福鼎 355200)
大黄鱼野生与养殖群体遗传结构的比较研究
陈 锦1,2, 黎中宝1,2, 方 秀3, 雷光高1,2, 张桂玲1,2, 赵斌丽1,2, 王展林1,2
(1. 集美大学 水产学院, 福建 厦门361021; 2. 集美大学 水产生物技术研究所, 福建 厦门361021; 3. 福建闽威水产实业有限公司, 福建 福鼎 355200)
采用垂直板型不连续聚丙烯酰胺凝胶电泳技术对福建野生与养殖大黄鱼(Pseudosciaena crocea)4个群体(湄洲野生群体、宁德野生群体、宁德养殖群体、连江养殖群体)的遗传结构进行了比较研究。结果表明, 4个大黄鱼群体的平均每位点有效等位基因数为 1.1250~1.1293, 多态位点百分数为12.50%~18.75%, 观察杂合度为 0.1250, 期望杂合度为 0.0636~0.0677。湄洲湾野生群体的遗传多样性高于养殖群体。4个大黄鱼群体间的遗传分化很低, 4个大黄鱼群体间遗传距离在0.0000~0.0001, 平均遗传距离为0.0005, 遗传分化系数(Fst= 0.0004)低, 基因流(Nm= 609.6667)大。
大黄鱼(Pseudosciaena crocea); 等位酶; 遗传多样性; 遗传分化; 遗传结构
大黄鱼(Pseudosciaena crocea), 属鲈形目(Perciformes)、石首鱼科(Sciaenidae)、黄鱼属(Pseudosciaena), 俗称黄鱼、黄花鱼等, 主要分布于中国黄海南部、东海和南海, 是中国单一品种养殖规模最大的海水经济鱼类[1,2]。大黄鱼野生资源由于过度捕捞等原因濒于枯竭, 不形成渔汛[3]。福建省大黄鱼网箱养殖自1990年突破百万尾全人工批量育苗大关后得到迅速发展, 大黄鱼养殖成为福建闽东地区渔业的支柱产业。近年来养殖大黄鱼出现性早熟、生长缓慢、病害频发等种质资源退化现象, 这些现象往往与群体遗传多样性下降有关[4]。因此大黄鱼野生资源及养殖群体遗传多样性的检测对于大黄鱼种质资源保护、繁殖和育种显得十分的必要。此前许多学者采用不同技术方法对其他地区的大黄鱼进行了相关分析[4~9]。作者采用等位酶技术对福建宁德、连江及莆田的野生及养殖共 4个大黄鱼群体的遗传多样性及分化进行了研究, 以期为大黄鱼野生资源的保护和人工养殖、育种提供遗传学资料。
1 材料与方法
大黄鱼共 4个群体, 宁德野生与养殖群体取于2007年5月, 连江养殖群体取于2007年7月, 湄洲野生群体取于2007年8月。每个群体各30尾。所有样品均加冰运送至实验室, 立即存于-80°C冰箱保存至分析。
每个群体各取30个样品, 实验时取冰冻大黄鱼背部肌肉少许, 剪碎放入研钵中, 加入2~3倍体积的Tris-HCl缓冲液(pH7.0)冰浴研磨, 4°C 离心 10 min(12 000 r/min), 取上清液按2∶1加入40%蔗糖溶液及20 μL溴酚蓝, 直接电泳或低温保存备用。
电泳采用垂直板型不连续聚丙烯酰胺凝胶(PAGE)电泳。浓缩胶和分离胶质量分数分别为2.5%和7.0%, pH值分别为8.9和6.7。实验共检测8个酶16个位点, 电泳条件参照黎中宝等[10]的方法, 等位酶的缩写参照 Shaklee 等[11]的方法, 酶谱判译参照王中仁[12]的方法, 实验共检测8个酶(表1)。
群体的等位基因频率(allelic frequency)、多态位点百分数(percentage of polymorphic loci,简称P)、平均每位点有效等位基因数(mean effective number of alleles per locus, 简称Ae)、期望杂合度(expected heterozygosity,简称He)、观察杂合度(observed heterozygosity,简称Ho)、遗传距离(genetic distance)和遗传一致度(genetic identity)及遗传分化系数(Fst)采用POPGENE 1.3.2软件进行数据的统计分析。

表1 实验所分析的等位酶、E.C.代码及位点数目Tab.1 Enzyme systems, E.C.code and Numbers of loci ofP.crocea populations
2 结果与分析
2.1 4个大黄鱼群体的遗传多样性
4个大黄鱼群体的等位基因频率见表2。在4个大黄鱼群体检测的8个酶16个位点中, 有3个多态位点(P0.99), 它们是Ldh-2、Sod-2及Est-2位点, 单态位点为13个, 它们是Ldh-1、Sod-1、Adh-1、Sdh-1、Mdh-1、Mdh-2、Me-1、Me-2、Me-3、Me-4、Aat-1、Est-1、Est-3位点。
4个大黄鱼群体平均每个位点有效等位基因数为 1.1250~1.1293, 多态位点百分数为 12.50%~18.75%, 观察杂合度为 0.1250, 期望杂合度为0.0636~0.0677(表3)。

表2 4个大黄鱼群体的等位基因频率Tab. 2 Allele frequencies in 4 P.crocea populations

表3 4个大黄鱼群体遗传多样性参数Tab. 3 Indices of genetic diversity of the 4 populations of P.crocea by allozyme
2.2 4个大黄鱼群体间的遗传分化
4个大黄鱼群体间的遗传分化很低。4个大黄鱼群体间遗传距离为0.0000~0.0001, 平均遗传距离为0.0005 (表4), 4个大黄鱼群体间的分化系数为0.0004,群体间的基因流为609.6667。
3 讨论
3.1 大黄鱼的遗传多样性
多态位点百分比、平均观察杂合度和平均每个位点有效等位基因数目是群体变异水平及等位基因丰富程度的主要指标。本研究中大黄鱼的多态位点百分比为 12.5%~18.75%, 这与梭鱼(Liza haemato-chelia)(13.1%~26.2%)[13]、牙鲆(Paralichthys olivaceus)(14.29%~23.81%)[10,14]、斜带髭鲷(Hapalogenys nitens)(17.39%)[15]相近, 高于花尾胡椒鲷(Plectorhinchus cinctus)(8.33%)[16]和状黄姑鱼(Nibea miichthioides)(4.55%~9.09%)[17], 但低于鲈鱼(Lateolabrax japonicus)(22.6%~25.8%)[18]、真鲷(Pagrus major)(25%~45%)[19]、漠斑牙鲆(Paralichthys lethostigma)(33.3%)[20]和14种鱼类平均值(30.6%)[21]。大黄鱼的平均观察杂合度(Ho)为 0.1250, 与真鲷(0.095~0.141)[19]相近, 高于牙鲆(0.0788~0.104)[10]、梭鱼(0.064~0.092)[13], 但低于漠斑牙鲆(0.1547)[20]。大黄鱼的平均每个位点有效等位基因数目为1.1250~1.1293, 这与梭鱼(1.101~1.132)[13]、状黄姑鱼(1.05~1.14)[17]相近, 高于花尾胡椒鲷(1.083)[16],但低于斜带髭鲷(1.174)[15]、牙鲆(1.19~1.24)[10]、真鲷(1.22~1.267)[19]及漠斑牙鲆(1.2196)[20]。综上可见,大黄鱼遗传多样性处于中等偏下水平。
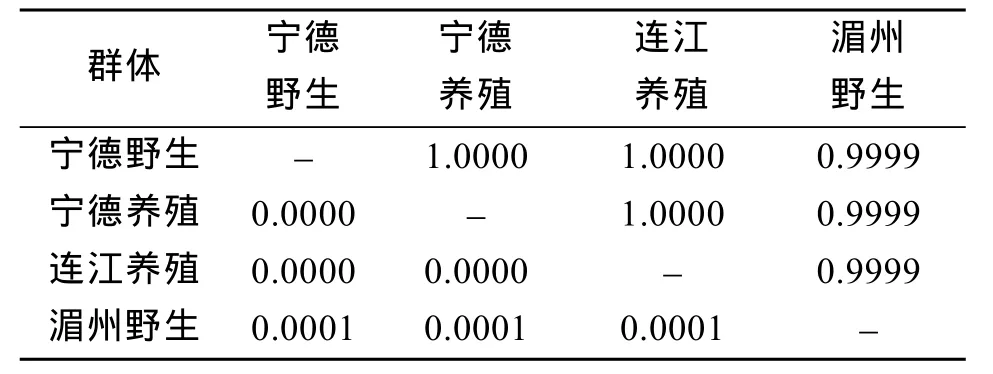
表4 4个大黄鱼群体的遗传一致度(对角线右上)和遗传距离(对角线左下)Tab. 4 Genetic distance between the 4 populations of P.crocea
宁德野生和养殖群体及连江养殖群体遗传多样性水平几乎一致, 这可能有两方面的原因。一方面,人工增殖放流影响。大黄鱼野生资源由于过度捕捞受到毁灭性的破坏, 为恢复大黄鱼野生资源量, 各地采取了人工放流大黄鱼鱼苗的措施, 仅宁波地区2003~2005年每年放流150万尾。而人工放流的大黄鱼苗多为养殖后代, 由于数量巨大, 其与野生大黄鱼的杂交, 势必对野生资源产生一定的遗传污染等影响。另一方面, 人工网箱养殖过程中, 由于台风等各种因素的影响, 一定数量逃逸的养殖大黄鱼也混入野生群体中。湄洲野生大黄鱼群体遗传多样性水平较宁德野生群体高, 推测可能由于湄洲大黄鱼养殖很少, 逃逸的大黄鱼较少的原因。本研究发现野生群体平均多态位点百分比为18.5%, 高于养殖群体的12.5%。这与其他学者在大黄鱼[6]、牙鲆[14]、 状黄姑鱼[17]、真鲷[19]等研究的结果相似。养殖群体遗传多样性的丧失主要由于亲本数量少引起的遗传瓶颈效应及其伴随发生的遗传漂变和近交衰退等作用。而养殖鱼类在遗传多样性水平上的丧失往往与其种质退化现象, 如生长缓慢、成鱼个体小、性成熟提早、抗病能力下降、肉质变差等有着很大的联系。因此在养殖过程中应注重亲鱼的数量和遗传背景, 实施科学的繁育和养殖管理措施。
3.2 大黄鱼群体间的遗传分化
大黄鱼 4个群体间的平均遗传距离为 0.0005,群体间的遗传分化较低为 0.0004, 基因流为609.6667。这可能由于人工放流和养殖逃逸的影响,致使野生群体和养殖群体间存在很大的基因交流。养殖群体逃逸对于野生资源的保护影响往往是不利的, 由于养殖群体遗传背景单一, 进入自然海区与野生种杂交, 使野生鱼类基因库损失大量的遗传变异, 从而直接影响到其遗传多样性水平。Clifford 等[22]发现一些人工孵化的大西洋鲑苗种逃逸到自然界而使得自然群体平均杂合度明显下降的现象。因此为保护大黄鱼野生种质资源, 应进行严格的封闭养殖, 或者应用不育品种于养殖。而放流人工繁殖苗种,其遗传背景也很单一, 因此在放流时应综合考虑其对野生资源的遗传污染等负面影响, 应放流遗传多样性丰富的野生群体的子一代。
[1]徐开达, 刘子. 藩东海区大黄鱼渔业资源及资源衰退原因分析[J]. 大连水产学院学报, 2007, 22(5):392-396.
[2]伏荣. 不同养殖模式大黄鱼Pseudosciaena crocea(Richardson) 品质的比较[J]. 现代渔业信息, 2008,23 (10):3-6.
[3]练兴常. 大目洋渔场大黄鱼放流现状[J]. 中国水产,2000, 1:22-23.
[4]黎中宝, 方秀, 陈锦, 等. 大黄鱼(Pseudosciaena crocea)养殖群体遗传多样性的降低[J]. 海洋与湖沼,2009, 40(4):446-450.
[5]全成干, 王军, 丁少雄, 等. 大黄鱼养殖群体遗传多样性的同工酶[J]. 厦门大学学报(自然科学版), 1999,38(4):584-587.
[6]王军, 全成干, 苏永全, 等. 官井洋野生与养殖大黄鱼同工酶的研究[J]. 海洋科学, 2001, 25(6):39-41.
[7]李明云, 张海琪, 薛良义, 等. 网箱养殖大黄鱼遗传多样性的同工酶RAPD分析[J]. 中国水产科学, 2003,6:523-525.
[8]王志勇, 王艺磊, 林利民, 等. 福建官井洋大黄鱼AFLP指纹多态性的研究[J]. 中国水产科学, 2002, 3:198-213.
[9]黄 勤, 陈 曦, 杨金先, 等. 福建养殖大黄鱼(Pseudosciaena crocea)RAPD标记及多态性调查[J].福建农业学报, 2007, 22(2):130-135.
[10]黎中宝. 牙鲆养殖群体遗传结构的研究[J]. 海洋学报, 2004, 26(3):102-108.
[11]Shaklee J B, Allendorf F W, Morizot D C,et al. Genetic nomenclature for proteincoding loci in fish [J]. Trans Amer Fish Soci, 1990, 119:2-15.
[12]王中仁. 植物等位酶分析[M]. 北京:科学出版社,1996. 77-119.
[13]杨锐, 庄志猛, 喻子牛, 等. 梭鱼养殖群体与自然群体等位基因酶的遗传变异[J]. 海洋水产研究, 2002,23(3):15-19.
[14]尤锋, 王可玲, 相建海. 山东近海褐牙鲆自然与养殖群体生化遗传结构及其遗传变异的比较分析[J]. 海洋与湖沼, 2001, 32(5):512-518.
[15]梁君荣, 王军, 苏永全. 斜带髭鲷养殖群体遗传多样性的同工酶研究[J]. 台湾海峡, 2003, 22(1):19-23.
[16]丁少雄, 王军, 苏永全, 等. 花尾胡椒鲷养殖群体遗传多样性的等位酶研究[J]. 台湾海峡, 2001, 20(2):224-227.
[17]丁少雄, 王军. 野生与养殖状黄姑鱼群体遗传多样性的同工酶比较[J]. 厦门大学学报(自然科学版),2001, 40(4):922-926.
[18]徐成, 王可玲, 张培军. 鲈鱼群体生化遗传学研究. II.种群生化遗传结构及变异[J]. 海洋与湖沼, 2001,32(3):248-254.
[19]王继伟, 孔杰, 庄志猛, 等. 真鲷野生群体和人工繁殖群体的同工酶遗传差异[J]. 生物多样性, 2000, 8(4):391-396.
[20]李鹏飞, 刘萍, 柳学周. 漠斑牙鲆引进种群同工酶的遗传多态性分析[J]. 中国水产科学, 2006, 13(1):13-19.
[21]Kirpichnilov V S. Genetic Bases of Fish Selection[M].Berlin, Heidelbarg, New York:Springer-Verlag, 1981,143-200.
[22]Clifford S L, Mcginnity P, Ferguson A. Genetic changes in an Atlantic salmon population resulting from escaped juvenile farm salmon[J]. J Fish Biology, 1998, 52:916-922.
The genetic structure of wild and cultivated populations ofPseudosciaena crocea
CHEN Jin1,2, LI Zhong-bao1,2, FANG Xiu3, LEI Guang-gao1,2, ZHANG Gui-ling1,2,ZHAO Bin-li1,2, WANG Zhan-lin1,2
(1.Fisheries College, Jimei University, Xiamen 361021, China; 2. Institute of Aquaculture Biotechnology, Jimei University, Xiamen 361021, China; 3.Fujian Minwei Aquatic Products Industrial Corporation Limited, Fuding 355200, China)
Nov. ,26, 2008
Pseudosciaena crocea;allozyme; genetic diversity; genetic differention; genetic structure
Genetic structures of wild and cultivated populations ofPseudosciaena croceawere investigated using the assay of vertical slab polyacrylamide gel electrophoresis. The results showed that the mean effective number of alleles per locus (Ae) ranged from 1.1250 to 1.1293, the percentage of polymorphic loci (P) ranged from 12.50 % to 18.75 %, the observed heterozygosity (Ho) was 0.1250, and the expected heterozygosities (He) ranged from 0.0636 to 0.0677. The genetic differentiation amongP. croceapopulations was low. Genetic distance a among populations ranged from 0.0000 to 0.0001, genetic differentiaion index (Fst)among populationswas 0.0004, and gene flow was high (Nm=609.6667).
Q953 文献标识码:A 文章编号:1000-3096(2010)02-0045-04
2008-11-26;
2009-03-18
福建省高等教育新世纪优秀人才支持计划资助项目(闽教科〔2006〕35号); 集美大学创新团队基金资助项目(2007A001); 福建省海洋与渔业厅科技项目
陈锦(1981-), 福建福安人, 硕士, 主要从事水产养殖学研究,电话:13799808167, E-mail:Cj198166@163.com; 黎中宝, 通信作者,E-mail:lizhongbao@jmu.edu.cn
谭雪静)

