紫花苜蓿NAC基因家族鉴定及在非生物胁迫下的表达模式分析












摘要:NAC是植物特有转录因子,在调控植物生长发育及响应非生物胁迫等方面发挥作用。为挖掘紫花苜蓿(Medicago sativa)响应干旱等非生物胁迫的NAC转录因子,本研究利用生物信息学方法在‘中苜4号’紫花苜蓿基因组中鉴定出143个NAC家族转录因子。研究表明,紫花苜蓿MsNAC基因在染色体上不均匀分布,MsNAC基因存在片段重复,并与大豆(Glycine max (Linn.) Merr.)、蒺藜苜蓿(Medicago truncatula)存在共线性关系,基因保守基序及基因结构分析表明MsNAC基因多数含有Motif1,Motif3保守基序及3~6个外显子。大多数MsNAC蛋白定位于细胞核中。系统进化树分析结果表明MsNACs聚类为8个亚家族。顺式作用元件分析表明MsNAC基因含有响应干旱及脱落酸、生长素等顺式作用元件。荧光定量分析结果显示部分MsNAC基因响应干旱、盐胁迫及脱落酸处理,大部分MsNAC候选基因在干旱、盐及ABA处理后上调表达。本研究结果可为紫花苜蓿抗旱、耐盐分子育种提供候选基因。
关键词:紫花苜蓿;NAC;转录因子;生物信息学;非生物胁迫;表达模式
中图分类号:S514.9""" 文献标识码:A"""" 文章编号:1007-0435(2024)08-2440-19
Identification of Alfalfa NAC Gene Family and Analysis of Their
Expression Patterns under Abiotic Stress
ZOU Wei-peng, ZHAI Jia-xing, LI Di-na, HUANG Jie-qiong, GUO Kang-jie,
CEN Hui-fang*, ZHU Hui-sen*, XU Tao
(College of Grassland Science, Shanxi Agricultural University, Taigu, Shanxi Province 030801, China)
Abstract:The NAC transcription factors are specific to plants and play a crucial role in regulating plant growth,development,and response to abiotic stresses. A total of 143 NAC family transcription factors were identified in the ‘Zhongmu No.4’ alfalfa (Medicago sativa) genome using bioinformatics methods,specifically in response to abiotic stress such as drought. The distribution of MsNAC genes on the chromosomes of alfalfa was found to be uneven,with fragment repetition and collinear relationships with soybean (Glycine max) and Medicago truncatula. Gene conserved motifs and gene structure analysis revealed that most MsNAC genes contain Motif1 and Motif3 conserved motifs and 3~6 exons,most MsNAC proteins are localized in the nucleus. The results of phylogenetic tree analysis showed that MsNACs were clustered into 8 subfamilies. The analysis of cis-acting elements showed that MsNAC genes contain cis-acting elements to drought,abscisic acid and auxin. Fluorescence quantitative PCR analysis showed that some MsNAC genes were responsive to drought,salt stress,and ABA treatment,most MsNAC candidate genes were up-regulated after drought,salt and ABA treatment. These findings provide valuable candidate genes for breeding alfalfa with improved drought and salt tolerance.
Key words:Alfalfa;NAC;Transcription factor;Bioinformatics;Abiotic stress;Expression pattern
植物在生长过程中容易受到干旱、盐碱等非生物胁迫和病虫害等生物胁迫的危害,植物通过调控相应基因的表达来调节植物的生化代谢途径及生长发育过程来抵御及适应胁迫对其产生的影响。NAC转录因子家族是植物特有的基因家族,也是植物体内最大的转录因子家族之一[1-3]。NAC蛋白具有高度保守的N端DNA结合区以及可变的C端转录调控区,N端大约由150个氨基酸残基组成且又分为高度保守的A,C,D及多变的B,E共5个亚结构域,不同亚结构域发挥不同的功能,其中A亚结构域与功能二聚体有关,B,E亚结构域与蛋白质功能的多样性有关,C,D亚结构域则与DNA结合有关[4-5]。自1996年Souer等在矮牵牛中分离到第一个属于NAC转录因子家族的NAM后,越来越多的NAC转录因子被发掘出来,并且证明其在调控植物生长发育、响应逆境胁迫、参与次生物质代谢、影响细胞发育等方面发挥着重要作用[6-8]。如在水稻(Oryza sativa)中OsNAC2基因可以结合生长素失活相关基因GH3.6/GH3.8和信号传导相关基因ARF25以及细胞分裂素氧化酶基因的启动子,从而调控水稻生长素及细胞分裂素途径,影响水稻根系生长[9];西瓜(Citrullus lanatus)ClNAC68转录因子通过抑制ClINV和ClGH3.6的表达正向调控西瓜的含糖量和种子发育[10];在小麦(Triticum aestivum)中过表达TaNACL-D1转录因子可以增强小麦对赤霉病的抗逆性[11];草莓(Fragaria×ananassa)FaRIF转录因子是草莓成熟以及色素生成和糖分积累的关键调控因子[12];在番茄(Solanum lycopersicum)中,NAC转录因子NOR-like1通过调节番茄细胞层数和细胞面积来调节果实大小[13];油菜(Brassica rapa)BraNAC87基因的过表达导致了油菜叶片叶绿素的降解及花青素积累,调控活性氧的积累和细胞死亡[14]。
NAC基因在多种植物中完成功能验证及全基因组分析,包括拟南芥(Arabidopsis thaliana)[15]、玉米(Zea mays)[16]、梭梭(Haloxylon ammodendron)[17]、月季(Rosa chinensis)[18]、山茶(Camellia sinensis)[19]等。在拟南芥中鉴定出117个NAC基因,其中AtNAC092调控由盐胁迫引发的衰老过程[20];玉米中鉴定出148个非冗余的NAC基因,ZmNAC20能提高玉米耐旱性[21];在月季中鉴定出116个NAC基因,其中RcNAC091直接靶向RcWRKY71启动子区域激活RcWRKY71表达,RcWRKY71正向调控月季脱落酸信号传导及月季耐旱性[22];在猕猴桃中鉴定出142个NAC基因,其大多调控猕猴桃果实成熟过程[23];在山茶中鉴定出104个NAC基因,过表达CsNAC28可增强山茶耐旱性[19]。
紫花苜蓿(Medicago sativa)是豆科苜蓿属多年生牧草,是我国栽培面积最大的牧草之一,具有营养品质高、产量高、适应性强、适口性好的特点,被称为“牧草之王”[24-25]。随着我国畜牧业发展,优质豆科牧草的需求量逐年升高。我国紫花苜蓿多种植于干旱半干旱地区种植区,干旱遏制了我国紫花苜蓿种植及生产[26]。因此培育耐旱紫花苜蓿新品种是发展我国畜牧业的关键。目前,多个品种紫花苜蓿的全基因组已得到解析,为进一步挖掘分析抗旱相关基因家族提供了便利。NAC基因在调控其他植物生长发育与非生物胁迫响应等方面已得到验证,本研究利用生物信息学方法对紫花苜蓿MsNAC基因家族进行鉴定和分析,并对其在干旱、盐胁迫及脱落酸(Abscisic acid,ABA)处理下的响应模式进行研究,为后续进一步探究NAC基因在紫花苜蓿中的功能提供理论基础。
1 材料与方法
1.1 NAC转录因子的鉴定
利用紫花苜蓿基因组数据(https://figshare.com/s/fb4ba8e0b871007a9e6c)和NAC转录因子保守结构域的隐马尔可夫模型文件,用hmmsearch命令鉴定紫花苜蓿NAC家族成员;并且从Phytozom(https://phytozome-next.jgi.doe.gov)和Tair(https://www.arabidopsis.org)数据库获取拟南芥、水稻及蒺藜苜蓿的基因组文件,与紫花苜蓿基因组进行Blast比对,得到紫花苜蓿NAC蛋白序列。以上获得的序列进行合并及去冗余分析,并且利用NCBI-conserved domain(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)及SMART(https://smart.embl.de/)和Pfam(http://pfam-legacy.xfam.org/)进行蛋白质保守结构域的鉴定,再根据NAM结构域氨基酸序列长度约为150 bp进行筛选[27],最终获得紫花苜蓿NAC转录因子。
1.2 蛋白理化性质、亚细胞定位分析
利用ExPasy(https://www.expasy.org)在线蛋白质分析工具ProtParam和ProtScale预测氨基酸数目、分子量等信息。利用Cell-Ploc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)及Wolf-psort(https://wolfpsort.hgc.jp)对蛋白序列进行亚细胞定位分析。
1.3 基因结构及蛋白质基序分析
利用保守基序在线预测工具MEME(https://meme-suite.org/meme)对MsNAC蛋白序列进行预测,保守基序数设置为10,其他参数设置为默认;从紫花苜蓿注释文件中获得MsNAC的基因结构信息,使用TBtools工具将MsNAC的基因结构(外显子-内含子、保守结构域)及保守基序结构进行可视化分析。
1.4 系统发育进化树分析
使用MEGA7.0软件对紫花苜蓿MsNAC基因和蒺藜苜蓿MtNAC基因的氨基酸序列的多序列比对,构建系统进化树,采用邻接法(Neighbor-Joining),泊松校正,成对删除,1000次Bootstrap重复[28],利用iTOL(https://itol.embl.de/)及Evolview(https://www.evolgenius.info/evolview/)进行系统发育树美化。
1.5 顺式作用元件分析
从紫花苜蓿注释文件中选取MsNAC基因起始密码子上游2000 bp的序列,利用PlantCARE(https://bioinformatics.psb.ugent.be/webtools/plantcare/html)工具预测并分析启动子顺式作用元件。将预测获得的数据利用TBtools工具进行可视化分析[29]。
1.6 染色体定位及基因共线性分析
从紫花苜蓿基因注释文件中获取紫花苜蓿的染色体信息及MsNAC基因的染色体定位信息,根据MsNAC基因在染色体上的位置进行编号命名。利用TBtools工具对MsNAC基因进行共线性及基因重复事件和染色体定位的可视化分析[30]。
1.7 紫花苜蓿MsNAC基因的组织表达特异性及响应不同非生物胁迫的表达模式分析
从紫花苜蓿转录组数据库(PRJNA450305)中获取不同非生物胁迫下紫花苜蓿的转录组数据。通过Hisat2建立索引,将紫花苜蓿基因组数据与RNA-seq数据进行比对,使用StringTie计数,计算每百万条阅读映射的转录本(Transcript per million,TPM)值,以log2(TPM+1)值表述紫花苜蓿MsNAC基因不同非生物胁迫下的响应模式,并利用TBtools进行可视化[31]。
植物材料为‘中苜1号’紫花苜蓿,于2023年10月种植于蛭石∶泥炭=1∶1的基质中,置于人工气候箱中,光周期为光照∶黑暗=16 h∶8 h,培养温度为25℃,湿度为60%。待幼苗生长至4周龄,取出植株于1/2 Hoagland营养液中缓苗24 h,于1/2 Hoagland营养液中分别进行干旱胁迫(15% PEG-6000)、盐胁迫(250 mmol·L-1 NaCl)和ABA处理(150 μmol·L-1)。分别取胁迫处理不同时间下(0 h,1 h,3 h,6 h,12 h,24 h)紫花苜蓿幼苗同一部位叶片提取RNA进行qRT-PCR分析。
总RNA提取采用植物RNA提取试剂盒(0416-50,北京华越洋生物科技有限公司),利用Nanodrop 2000检测RNA浓度,利用PrimeScriptRTreagen kit with gDNA eraser(Perfect Real Time)(RR047,TaKaRa)将总RNA反转录成cDNA。利用Primer 5设计引物,引物列表见表1,其中MsActin为内参基因。qRT-PCR采用Bio-Rad CFX96系统,每个反应体系含有cDNA模板1 μL、上下游引物各0.5 μL、TB Green Premix Ex Taq Ⅱ(TaKaRa)10 μL和ddH2O 7 μL。qRT-PCR反应循环条件为:95℃,30 s;95℃,10 s;60℃,30 s;72℃,10 s;40个循环。试验设置3次重复,基因相对表达量使用差异倍数(Fold change,FC)=2-ΔΔCt方法计算。
1.8 数据处理
qRT-PCR数据使用Excel 2022及SPSS 26.0软件进行整理分析。
2 结果与分析
2.1 紫花苜蓿NAC家族候选基因鉴定
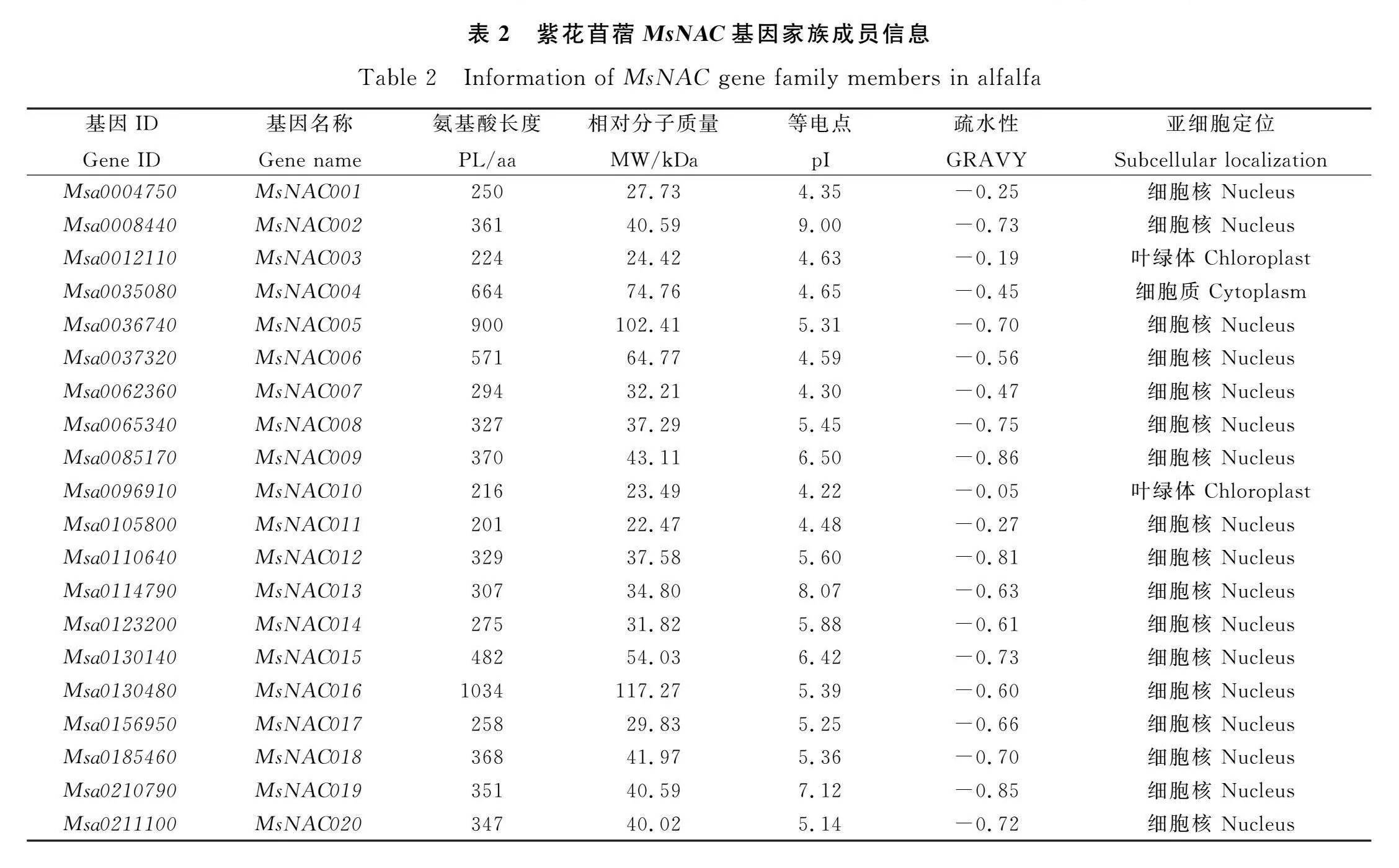
在‘中苜4号’紫花苜蓿基因组中鉴定NAC基因家族成员,通过HMM及结构域分析,共鉴定出143个非冗余MsNAC基因,按照在染色体上的分布位置依次命名为MsNAC001~MsNAC143(表2)。对紫花苜蓿MsNAC蛋白的理化性质及亚细胞定位分析结果表明,MsNAC的理化性质存在较大差异,其中分子量最大的是MsNAC016,编码1034个氨基酸,分子量为117.27 kDa,分子量最小的是MsNAC123,编码150个氨基酸,分子量为17.60 kDa。蛋白疏水性指数介于-1.02(MsNAC067)和-0.05(MsNAC010)之间,表明MsNAC蛋白均为亲水性蛋白。蛋白质理论等电点(Isoelectric point,pI)介于4.16(MsNAC056)和10.24(MsNAC041)之间,70个(48.9%)MsNAC蛋白为酸性蛋白(pIlt;6.5),55个(38.4%)MsNAC蛋白为碱性蛋白(pIgt;7.5)。对MsNAC蛋白进行亚细胞定位预测,发现MsNAC蛋白主要定位于细胞核中,共有139个MsNAC定位于细胞核中,占总数的97.2%,少数定位于细胞质、叶绿体中,如MsNAC004定位于细胞质中,MsNAC003,MsNAC010,MsNAC121定位于叶绿体中,推测大多数MsNAC转录因子在细胞核中发挥生物学功能(表2)。
2.2 紫花苜蓿MsNAC基因家族保守基序及结构域分析
利用MEME工具对紫花苜蓿保守基序(Motif)进行预测分析,依次命名为Motif1~Motif10(图1A)。如图1B所示,保守基序在不同MsNAC蛋白中存在分布差异,大多数Motif保守程度较高,少部分Motif存在特异性。其中Motif1存在于绝大多数MsNAC成员中,仅MsNAC068及MsNAC079中不存在Motif1,除MsNAC033,MsNAC052,MsNAC064,MsNA-C105及MsNAC112外,其余成员都拥有Motif3,其中Motif8,9,10在MsNAC中保守性较差,分别只存在于11,12及38个MsNAC成员中,大多数MsNAC成员都包含Motif1~Motif7。
保守结构域分析表明,MsNAC家族成员共鉴定出7种结构域,其中所有MsNAC蛋白都含有NAM结构域,而MsNAC121含有Glyco_hydro_100结构域,Glyco_hydro_100结构域主要存在于碱性蔗糖转化酶基因INV(Invertase)中,该基因能够将蔗糖水解为葡萄糖和果糖,并且参与糖分运输,从而调控植株生长发育及响应非生物胁迫[32]。MsNAC044和MsNAC050含有Zinc_peptidase结构域,而MsNAC115,MsNAC129和MsNAC056分别具有PHA02682和PHA03269结构域,MsNA-C127和MsNAC035,MsNAC043分别具有RPA_2b-aaRSs_OBF和LIM结构域(图1C),LIM结构域蛋白是一种肌动结合蛋白,在植物生长发育过程中可促进肌动蛋白束的形成[33]。这些结构域可能赋予这些MsNAC基因特殊的生物学功能。
利用TBtools对MsNAC基因家族成员进行基因结构可视化分析,结果显示MsNAC基因的外显子数目在2~17个之间,其中外显子数目在3~6个的MsNAC成员有124个,MsNAC16基因含有17个外显子,MsN-AC11,MsNAC38,MsNAC88,MsNAC97,MsNAC101,MsNAC123,MsNAC143基因仅含有2个外显子(图2)。
2.3 紫花苜蓿MsNAC家族系统进化分析
为进一步研究紫花苜蓿MsNAC基因家族的进化关系,对紫花苜蓿MsNAC蛋白与模式植物蒺藜苜蓿的NAC蛋白构建系统进化树。结果表明,紫花苜蓿的143个MsNAC蛋白被分为8个亚族。其中第Ⅴ亚族包含的NAC家族成员最多,包含34个MsNAC及35个MtNAC。第Ⅱ亚族包含的NAC家族成员最少,包含12个MsNAC及4个MtNAC。在Ⅲ,Ⅳ,Ⅵ和Ⅶ亚族中,MsNAC成员与MtNAC成员较均匀分布,而在第Ⅰ,Ⅷ亚族中MsNAC成员明显多于MtNAC成员(图3)。综上,紫花苜蓿MsNAC家族成员与蒺藜苜蓿MtNAC家族成员分布较为均匀,显示紫花苜蓿与蒺藜苜蓿亲缘关系较近且存在相似蛋白质。
2.4 紫花苜蓿MsNAC基因启动子顺式作用元件分析
为进一步探究MsNAC基因的生物学功能,对MsNAC基因起始密码子(ATG)上游2000 bp进行预测及分析,共鉴定出包括脱落酸响应元件、水杨酸响应元件、赤霉素响应元件、茉莉酸甲酯响应元件、应急防御响应元件等22种顺式作用元件,其中共有108个MsNAC基因启动子上含有脱落酸响应元件,134个MsNAC基因含有光响应元件,66个成员含有应急防御响应元件,99个成员含有茉莉酸甲酯响应元件,7个成员含有细胞周期调控元件,54个成员拥有低温响应元件,65个成员拥有MYB结合位点响应干旱胁迫的顺式作用元件(图4),这些顺式作用元件可能在MsNAC参与非生物胁迫及生长发育过程中起到关键作用。
2.5 紫花苜蓿MsNAC基因染色体定位及共线性分析
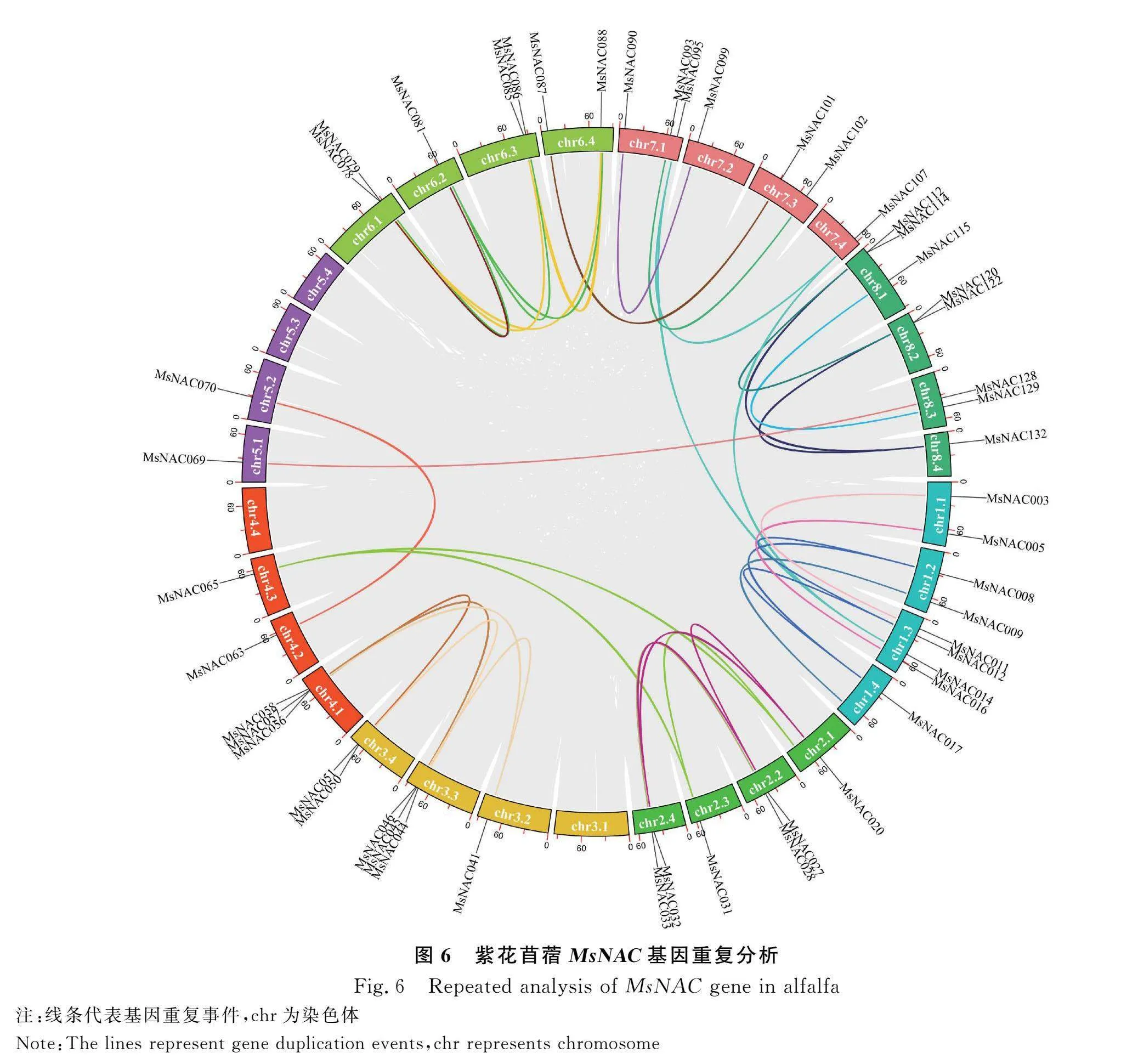
依据紫花苜蓿MsNAC基因在染色体上的位置及顺序依次命名,由染色体定位图可知(图5),133个MsNAC基因不均匀地定位到32条染色体上,chr8.1染色体上分布的MsNAC基因最多,每条染色体含有至少2个MsNAC基因,另外,有10个MsNAC基因未定位到紫花苜蓿染色体上。
利用MCScanX软件分析MsNAC家族成员的共线性关系,如图6所示,在紫花苜蓿MsNAC基因家族中共鉴定出40个基因片段重复事件,表明MsNAC基因在进化过程中发生了一定的基因复制事件,使得MsNAC基因家族迅速扩张。
在此基础上,为进一步探究NAC基因家族在不同植物进化过程中基因的亲缘关系,对紫花苜蓿以及大豆、蒺藜苜蓿、拟南芥中的NAC基因进行共线性分析。结果表明,49个MsNAC基因与拟南芥存在共线性关系,72个MsNAC基因与大豆存在共线性关系,73个MsNAC基因与蒺藜苜蓿存在共线性关系(图7)。紫花苜蓿NAC基因与蒺藜苜蓿和大豆亲缘关系更近,说明进化过程中NAC基因在豆科植物中保守演替。
2.6 紫花苜蓿MsNAC基因在不同组织及非生物胁迫处理下的表达模式分析
为探究MsNAC基因在紫花苜蓿不同组织中的表达模式,利用‘中苜1号’紫花苜蓿RNA-seq数据进行分析。如图8A所示,有12个MsNAC基因在花中有较高的表达水平,其中MsNAC040及MsNAC054的表达量明显高于其他紫花苜蓿MsNAC基因;有18个MsNAC基因在茎中具有较高的表达量,其中MsNAC057及MsNAC042在茎中的表达量最高;有17个MsNAC基因在叶片中有较高的表达水平,MsNAC031及MsNAC065在叶中拥有最高的基因表达量,而在花中的表达量最低,说明发生基因重复事件的基因具有相似的组织表达模式。另外,有6个MsNAC基因在花、茎、叶中的表达量无显著差异。大部分MsNAC基因在紫花苜蓿不同组织中均有表达但存在组织特异性,说明MsNAC基因功能存在多样性,在调控紫花苜蓿生长发育过程中起到重要作用。
为进一步分析MsNAC基因在不同非生物胁迫下的响应模式,分析了MsNAC在干旱、盐及脱落酸胁迫处理下的RNA-seq转录组数据[34]。结果显示,不同MsNAC在不同非生物胁迫处理下存在不同的响应模式。干旱胁迫下,随着胁迫时间的延长,MsNAC036,MsNAC025,MsNAC070和MsNAC-006基因的表达量逐渐升高,在24 h达到最大值,而MsNAC040,MsNAC056,MsNAC037和MsNA-C098基因的表达量降低,MsNAC057,MsNAC042和MsNAC074等基因的表达量呈现先升高后降低的趋势(图8B)。在盐胁迫下,随着胁迫时间的延长,MsNAC036,MsNAC044等基因的表达量上调,MsNAC042,MsNAC129和MsNAC019等基因的表达量下调,MsNAC015,MsNAC094等基因的表达量呈现先降低后升高的趋势(图8C)。在ABA处理下,随着处理时间的延长,MsNAC139,MsNA-C063和MsNAC031等基因的表达量上调,MsNAC088,MsNAC118等基因的表达量下调,MsNAC013,MsNAC057和MsNAC050等基因表达量呈现先升高后降低的趋势(图8D)。另外,部分基因在不同胁迫处理下表达模式相似,如MsNAC036,MsNAC025和MsNAC044等基因在干旱胁迫和盐胁迫下均随着胁迫时间的增加呈现上调表达的趋势,MsNAC040,MsNAC129等基因在干旱胁迫和盐胁迫下随着胁迫时间的增加均下调表达。表明MsNAC基因可能在紫花苜蓿响应不同非生物胁迫过程中发挥重要作用。
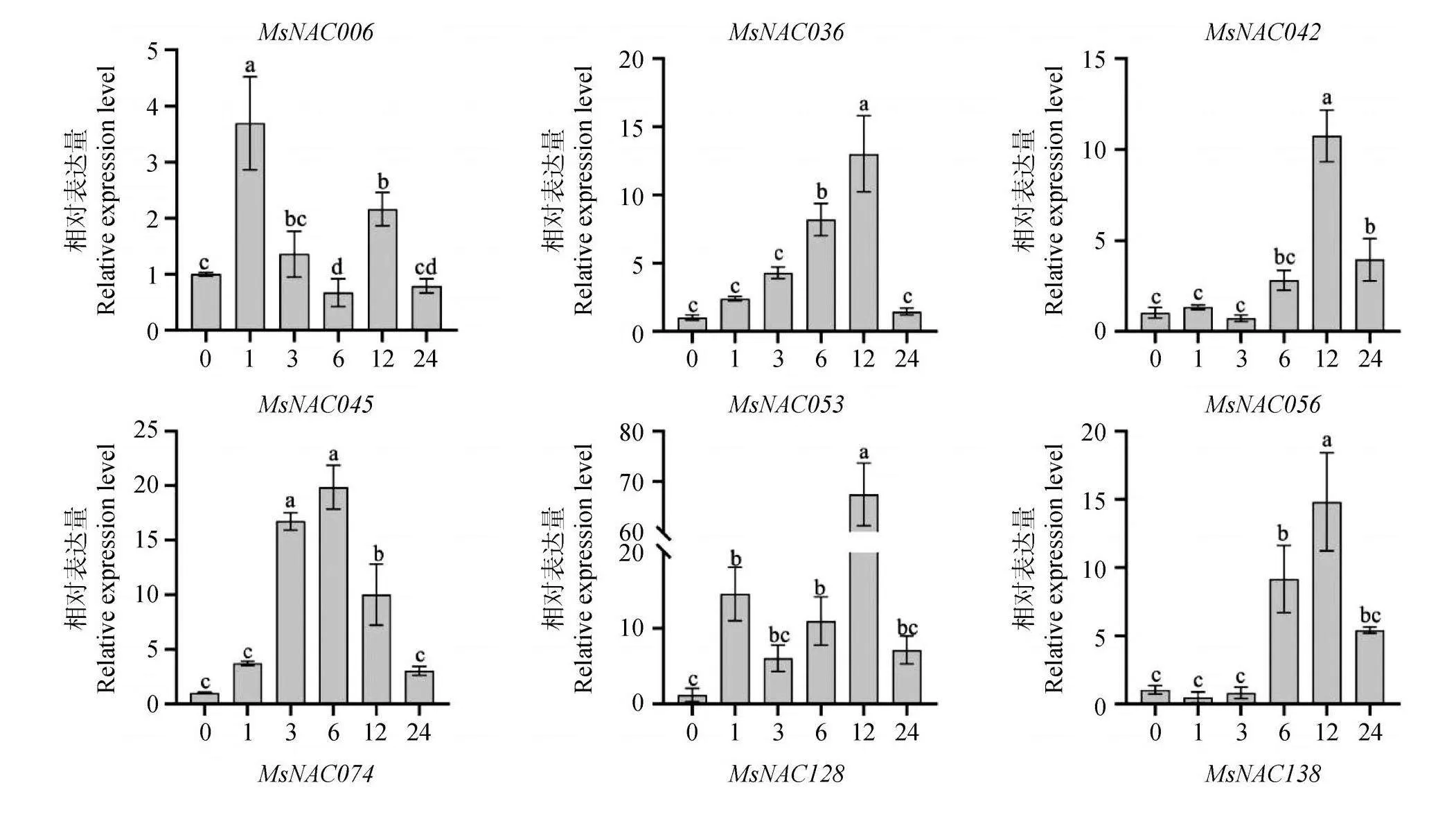
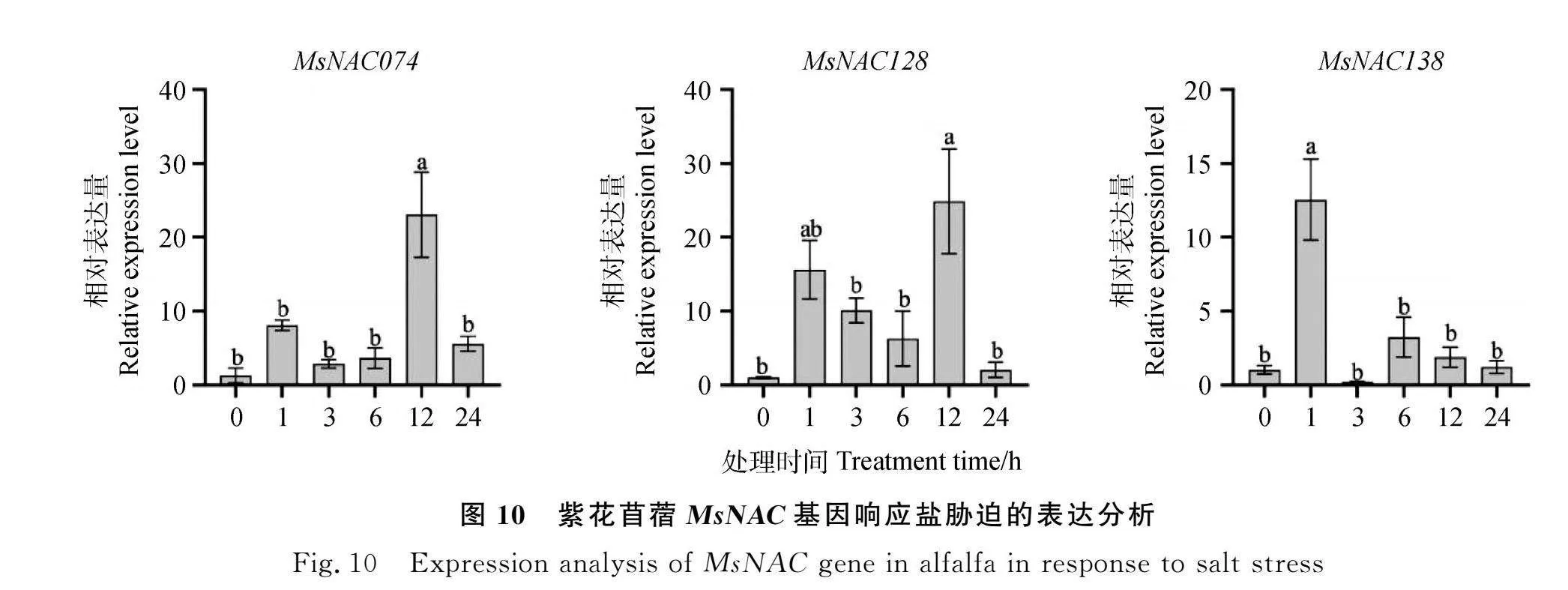
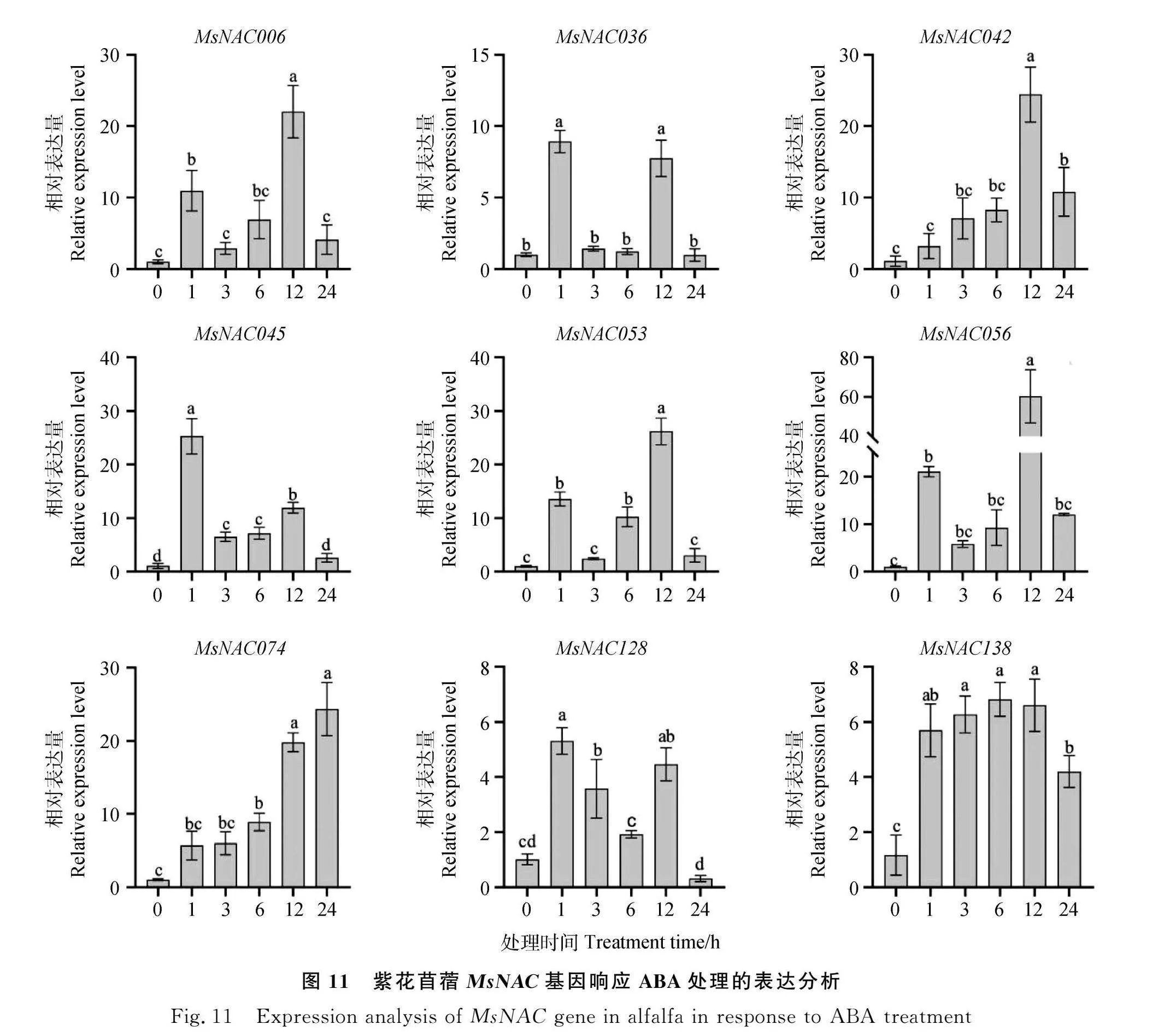
为进一步确认紫花苜蓿MsNAC基因在非生物胁迫下的表达模式,选取了9个MsNAC基因验证其对干旱、盐胁迫和ABA处理的响应情况。如图9所示,在干旱胁迫下,随着胁迫时间增加,MsNAC045,MsNAC138基因呈上调表达,MsNAC042,MsNAC053,MsNAC056和MsNA-C074基因表达量呈现先上调后下调的趋势;MsNAC006,MsNAC036和MsNAC128基因的表达量呈现先上调后下调再上调的趋势,其中MsNAC036,MsNAC045,MsNAC056,MsNAC128和MsNAC138基因的表达量在干旱胁迫下显著上调,说明其在紫花苜蓿干旱胁迫应答中可能发挥重要作用。在盐胁迫下,随着胁迫时间增加,MsNAC036,MsNAC042和MsNAC045基因先上调表达而后下调表达;MsNAC006,MsNAC053,MsNAC056,MsNAC074,MsNAC128和MsNAC-138基因表达量波动较大,但总体对盐胁迫有响应,其中MsNAC045基因的表达趋势与转录组热图分析一致(图10)。在ABA处理下,MsNAC074基因表达量随着胁迫处理时间增加呈现上调表达趋势,在24 h达到最大值;MsNAC006,MsNAC036,MsNAC045,MsNAC053,MsNAC056和MsNA-C128基因表达量随胁迫处理时间的延长波动较大,MsNAC042,MsNAC138基因表达量呈现先上调后下调的趋势(图11)。
3 讨论
NAC作为植物特有转录因子,在植物生长发育及响应胁迫过程中发挥作用,如紫花苜蓿MsNAC051能够与MsP5CS和MsPOD-P7的启动子区域相结合,直接激活其表达,增强烟草的抗旱性[35]。目前已在可可(Theobroma cacao)及蒺藜苜蓿中分别鉴定出102及97个NAC基因[36-37],本研究在紫花苜蓿‘中苜4号’基因组中鉴定出143个MsNAC基因,分布在32条染色体上,明显多于拟南芥[15]及蒺藜苜蓿中鉴定到的NAC基因数量,可能是因为紫花苜蓿为同源四倍体,拥有更多同源基因。
紫花苜蓿和蒺藜苜蓿NAC基因系统发育分析可知,MsNAC基因聚类到8个亚家族中,其中第V家族包含最多的MsNAC成员,位于同族的MsNAC成员拥有相似的Motif及基因结构,在辣椒(Capsicum annuum)[38]及梅花(Prunus mume)[39]中都有相似现象,说明MsNAC基因在进化过程中较为保守。亚细胞定位预测分析表明,大多数MsNAC蛋白定位在细胞核中,说明MsNAC蛋白主要在细胞核中发挥功能,与拟南芥AtNAC蛋白的亚细胞定位结果相似[20]。同时,基因共线性分析表明紫花苜蓿MsNAC基因与大豆及蒺藜苜蓿NAC基因在进化过程中亲缘关系更近,表明NAC基因在豆科植物进化过程中相对保守,说明了紫花苜蓿与蒺藜苜蓿等有更近的亲缘关系,在红车轴草[40](Trifolium pratense)中有类似结果。相同基因功能域预示着可能发挥类似的生物学功能。紫花苜蓿MsNAC蛋白与辣椒[38]、草莓[12]、玉米[16]的NAC蛋白都拥有典型的NAM结构域,说明MsNAC蛋白可能在紫花苜蓿响应非生物胁迫及生长发育等方面发挥作用。不同基因结构也影响基因的表达及蛋白质的功能,大多数MsNAC基因的外显子数量在3~6个,但序列长度不同,说明MsNAC的基因结构较为相似,并且与拟南芥及蒺藜苜蓿NAC基因结构相似。
基因启动子区域的顺式作用元件预示着该基因可能在不同胁迫下发挥不同的生物学功能,MsNAC基因在启动子区域含有多个与植株生长发育及逆境响应相关的顺式作用元件,可能与紫花苜蓿抵抗非生物胁迫有重要关联[36]。如MsNAC001,MsNAC50,MsNAC079和MsNAC106基因含有脱落酸及生长素响应元件,与紫花苜蓿MsNAC051研究结果相似[35]。表明它们可能在多种激素应答及生理代谢过程发挥作用,而MsNAC034及MsNAC085拥有干旱响应元件及应急防御响应元件,说明它们可能在植物抵御干旱胁迫过程中发挥重要作用。
NAC基因在其他物种中的功能也有研究,在水稻中,OsNAC3是盐胁迫及ABA信号传导的重要调控因子[41];有研究表明,GmNAC1~4是调控大豆根瘤高氮响应的中心调控因子[42];在拟南芥中,AtNAC096与ABF2及ABF4相互作用,激活拟南芥对干旱及渗透胁迫中ABA诱导基因的表达[43];香蕉(Musa acuminata)中MaNAC1随着冷胁迫时间的增加,表达量升高,说明MaNAC1是参与香蕉响应冷胁迫的重要基因[44]。
本试验通过生物信息学方法,将鉴定出的MsNAC与转录组数据库进行整合,分析出与干旱、盐、ABA处理有关的MsNAC基因家族成员,同时通过分析紫花苜蓿在干旱、盐和ABA处理后MsNAC基因的表达模式,表明MsNAC可能参与多种胁迫响应。其中,大多数MsNAC在干旱、盐及ABA处理下呈上升趋势,仅少量MsNAC呈下降趋势,如MsNAC128及MsNAC138在干旱胁迫24 h后达到最大值,而MsNAC042及MsNAC074在干旱胁迫12 h后达到最大值,与山茶CsNAC9表达模式相似[45];大多MsNAC基因在盐胁迫处理后在12 h达到最大值,表明他们拥有相似的表达模式,MsNAC138在盐胁迫下与ZmNAC59表达水平相似[45];而MsNAC128在ABA处理后在24 h表达量下调,MsNAC053及MsNAC056在ABA处理下与CsNAC9响应模式相似,说明它们可能参与脱落酸调控植物生长发育[45]。随后用qRT-PCR验证了MsNAC基因能够响应干旱、盐胁迫及ABA处理,说明MsNAC基因是紫花苜蓿抵抗非生物胁迫的重要基因,参与紫花苜蓿响应非生物胁迫的应答过程。本研究可为后续针对紫花苜蓿进行抗逆性改良提供候选基因。
4 结论
本研究从‘中苜4号’紫花苜蓿基因组中鉴定出143个拥有完整NAM结构域的MsNAC基因,被分为8个亚族,大都含有3~6个外显子并在其启动子区域含有大量响应脱落酸、生长素等激素及干旱和寒冷等胁迫的顺式作用元件。同时鉴定出40个基因重复事件,并与蒺藜苜蓿、大豆及拟南芥存在共线性关系。MsNAC基因在紫花苜蓿中存在组织特异性表达,参与调控紫花苜蓿对干旱、盐、ABA的响应过程,并在干旱、盐胁迫及ABA处理后大都上调表达,其中MsNAC036,MsNAC045及MsNAC053是响应干旱及盐胁迫的关键基因。
参考文献
[1] OLSEN A N,ERNST H A,LEGGIO L L,et al. NAC transcription factors:structurally distinct,functionally diverse[J]. Trends in Plant Science,2005,10(2):79-87
[2] BIAN Z,GAO H,WANG C. NAC transcription factors as positive or negative regulators during ongoing battle between pathogens and our food crops[J]. International Journal of Molecular Sciences,2020,22(1):81
[3] DIAO P,CHEN C,ZHANG Y,et al. The role of NAC transcription factor in plant cold response[J]. Plant Signaling amp; Behavior,2020,15(9):1785668
[4] WANG Z,DANE F. NAC (NAM/ATAF/CUC) transcription factors in different stresses and their signaling pathway[J]. Acta Physiologiae Plantarum,2013,(35):1397-1408
[5] LI W,ZENG Y,YIN F,et al. Genome-wide identification and comprehensive analysis of the NAC transcription factor family in sunflower during salt and drought stress[J]. Scientific Reports,2021,11(1):19865
[6] 宋洁琼,初若雯,赵晶莹,等. NAC转录因子在植物响应盐胁迫中的作用[J]. 生物学通报,2023,58(8):1-7
[7] NURUZZAMAN M,SHARONO A M,KIKUCHI S. Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants[J]. Frontiers in Microbiology,2013(4):248
[8] 荣梦茹,余如刚,韦英铭,等. 紫花苜蓿耐盐性相关NAC转录因子的挖掘及表达分析[J]. 草地学报,2024,32(4):1055-1067
[9] MAO C,HE J,LIU L,et al. OsNAC2 integrates auxin and cytokinin pathways to modulate rice root development[J]. Plant Biotechnology Journal,2020,18(2):429-442
[10]WANG J,WANG Y,ZHANG J,et al. The NAC transcription factor ClNAC68 positively regulates sugar content and seed development in watermelon by repressing ClINV and ClGH3.6[J]. Horticulture Research,2021,8(1):265
[11]PEROCHON A,KAHLA A,VVRNIC M,et al. A wheat NAC interacts with an orphan protein and enhances resistance to Fusarium head blight disease[J]. Plant Biotechnology Journal,2019,17(10):1892-1904
[12]MARTIN-PIZARRO C,VALLARINO J G,OSORIO S,et al. The NAC transcription factor FaRIF controls fruit ripening in strawberry[J]. Plant Cell,2021,33(5):1574-1593
[13]GAO Y,WEI,ZHAO X,et al. A NAC transcription factor,NOR-like1,is a new positive regulator of tomato fruit ripening[J]. Horticulture Research,2018,5:75
[14]YAN J,TONG T,LI X,et al. A Novel NAC-Type transcription factor,NAC87,from oilseed rape modulates reactive oxygen species accumulation and cell death[J]. Plant and Cell Physiology,2018,59(2):290-303
[15]OOKA H,SATOH K,DOI K,et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research,2003,10:239-247
[16]FAROOQI M Q U,NAWAZ G,WANI S H,et al. Recent developments in multi-omics and breeding strategies for abiotic stress tolerance in maize (Zea mays L.)[J]. Frontiers in Plant Science,2022,3:965878
[17]WANG M,ZHANG L,TONG S,et al. Chromosome-level genome assembly of a xerophytic plant,Haloxylon ammodendron[J]. DNA Research,2022,29(2):1-9
[18]GENG L,SU L,FU L,et al. Genome-wide analysis of the rose (Rosa chinensis) NAC family and characterization of RcNAC091[J]. Plant Molecular Biology,2022,108(6):605-619
[19]WANG Y X,LIU Z W,WU Z J,et al. Transcriptome-wide identification and expression analysis of the NAC gene family in tea plant [Camellia sinensis (L.) O. Kuntze] [J]. Plos One,2016,11(11):e0166727
[20]BALAZADEH S,SIDDIQUI H,ALLU A D,et al. A gene regulatory network controlled by the NAC transcription factor ANAC092/AtNAC2/ORE1 during salt-promoted senescence[J]. The Plant Journal,2010,62(2):250-264
[21]宋松波. 转录因子ZmNAC20调控玉米响应干旱胁迫的分子机制[D]. 郑州:河南农业大学,2023:39-43
[22]GENG L,YU S,ZHANG Y,et al. Transcription factor RcNAC091 enhances rose drought tolerance through the abscisic acid-dependent pathway[J]. Plant Physiology,2023,193(2):1695-1712
[23]JIA D,JIANG Z,FU H,et al. Genome-wide identification and comprehensive analysis of NAC family genes involved in fruit development in kiwifruit (Actinidia) [J]. BMC Plant Biology,2021,21(1):44
[24]LIU Z,CHEN T,MA L,et al. Global transcriptome sequencing using the Illumina platform and the development of EST-SSR markers in autotetraploid alfalfa[J]. Plos One,2013,8(12):e83549
[25]SHEN C,DU H,CHEN Z,et al. The chromosome-level genome sequence of the autotetraploid alfalfa and resequencing of core germplasms provide genomic resources for alfalfa research[J]. Molecular Plant,2020,13(9):1250-1261
[26]RAZI K,MUNEER S. Drought stress-induced physiological mechanisms,signaling pathways and molecular response of chloroplasts in common vegetable crops[J]. Critical Reviews in Biotechnology,2021,41(5):669-691
[27]MOHANTA TK,YADAV D,KHAN A,et al. Genomics,molecular and evolutionary perspective of NAC transcription factors[J]. Plos One,2020,15(4):e0231425
[28]唐芳,梅亭,高佳荷,等. 紫花苜蓿GPAT基因家族鉴定及在盐碱胁迫下的表达模式分析[J]. 草地学报,2023,31(9):2608-2620
[29]LESCOT M,DEHAIS P,THIJS G,et al. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327
[30]任明辉,张雨蓬,许涛,等. 紫花苜蓿R2R3-MYB亚家族鉴定与干旱胁迫下的表达分析[J]. 草地学报,2023,31(4):972-983
[31]CHEN C,WU Y,LI J,et al. TBtools-II:A “one for all,all for one” bioinformatics platform for biological big-data mining[J]. Molecular Plant,2023,16(11):1733-1742
[32]WANG L,ZHENG Y,DING S,et al. Molecular cloning,structure,phylogeny and expression analysis of the invertase gene family in sugarcane[J]. BMC Plant Biology,2017,17(1):109
[33]LI L,LI Y,WANG N N,et al. Cotton LIM domain-containing protein GhPLIM1 is specifically expressed in anthers and participates in modulating F-actin[J]. Plant Biology,2015,17(2):528-534
[34]JIANG X,YU A,ZHANG F,et al. Identification of QTL and candidate genes associated with biomass yield and feed quality in response to water deficit in alfalfa (Medicago sativa L.) using linkage mapping and RNA-Seq[J]. Frontiers in Plant Science,2022,13:996672
[35]ZHOU L,SHI K,CUI X,et al. Overexpression of MsNAC51 from alfalfa confers drought tolerance in tobacco[J]. Environmental and Experimental Botany,2023,205:105143
[36]SHEN S,ZHANG Q,SHI Y,et al. Genome-wide analysis of the NAC domain transcription factor gene family in theobroma cacao[J]. Genes,2019,11(1):35
[37]LING L,SONG L,WANG Y,et al. Genome-wide analysis and expression patterns of the NAC transcription factor family in Medicago truncatula[J]. Physiology and Molecular Biology of Plants,2017,23(2):343-356
[38]DIAO W,SNYDER J C,WANG S,et al. Genome-wide analyses of the NAC transcription factor gene family in pepper (Capsicum annuum L.):chromosome location,phylogeny,structure,expression patterns,cis-elements in the promoter,and interaction network[J]. International journal of molecular sciences,2018,19(4):1028
[39]ZHUO X,ZHENG T,ZHANG Z,et al. Genome-wide analysis of the NAC transcription factor gene family reveals differential expression patterns and cold-stress responses in the woody plant Prunus mume[J]. Genes,2018,9(10):494
[40]WANG Z,CHEN Z,WU Y,et al. Genome-wide identification and characterization of NAC transcription factor family members in Trifolium pratense and expression analysis under lead stress[J]. BMC Genomics,2024,25(1):128
[41]ZHANG X,LONG Y,CHEN X,et al. A NAC transcription factor OsNAC3 positively regulates ABA response and salt tolerance in rice[J]. BMC Plant Biology,2021,21(1):546
[42]WANG X,QIU Z,ZHU W,et al. The NAC transcription factors SNAP1/2/3/4 are central regulators mediating high nitrogen responses in mature nodules of soybean[J]. Nature Communications,2023,14(1):4711
[43]XU Z Y,KIM S Y,HYEON DO Y,et al. The Arabidopsis NAC transcription factor ANAC096 cooperates with bZIP-type transcription factors in dehydration and osmotic stress responses[J]. Plant Cell,2013,25(11):4708-4724
[44]YIN Q,QIN W,ZHOU Z,et al. Banana MaNAC1 activates secondary cell wall cellulose biosynthesis to enhance chilling resistance in fruit[J]. Plant Biotechnology Journal,2023,22(2):413-426
[45]孔洁玙,杨妮,罗微,等. 茶树NAC转录因子基因CsNAC79和CsNAC9鉴定及其对非生物胁迫的响应[J]. 西北植物学报,2024,44(4):572-581
(责任编辑 闵芝智)

