紫菜薹叶绿体全基因组序列及其系统发育分析


摘" 要" 紫菜薹(Brassica rapa var. purpuraria)是十字花科芸薹属白菜亚种中的一个变种,通过Illumina高通量测序平台,对其叶绿体基因组进行测序、组装和注释。结果表明,紫菜薹叶绿体基因组为典型的四分环状结构,大小为153 483 bp,GC含量为36.36%,由长度为83 282 bp的大单拷贝区(LSC)、17 775 bp的小单拷贝区(SSC)和1对各26 213 bp的反向重复区(IRa/IRb)组成。基因组注释共得到132个基因,包括:87个蛋白编码基因,37个tRNA和8个rRNA。共鉴定到313个简单重复序列(SSR)和37个散在重复序列。密码子偏好性分析发现存在偏好使用A/U碱基结尾的现象。边界分析和序列变异分析显示,芸薹属叶绿体基因组非常保守,其中SSC区差异性最大,变异程度最高,IR区差异性最小,最为保守。基于叶绿体基因组序列的系统发育分析表明,紫菜薹在分类上与白菜的其他变种在一个小分支中。研究结果可为芸薹属植物,特别是白菜种的分类学、系统学和生物地理学研究提供参考。
关键词" 白菜;紫菜薹;叶绿体基因组;系统发育分析
doi:10.7606/j.issn.1004-1389.2024.08.009
https://doi.org/10.7606/j.issn.1004-1389.2024.08.009
收稿日期:2023-12-04" 修回日期:2024-01-16
基金项目:宿州学院国家级大学生创新创业训练计划(202310379012);宿州学院博士(后)科研启动基金(2023BSK022);宿州学院分子育种中心横向课题(2022xhx235,2023xhx129);安徽省教育厅重点项目(2022AH051384)。
第一作者:王传之,男,博士,高级农艺师,主要从事分子植物遗传育种与栽培研究。E-mail:jfwcz@163.com
通信作者:周贤玉,女,硕士,农艺师,主要从事蔬菜遗传育种研究。E-mail:1562783858@qq.com
任海龙,男,博士,副研究员,主要从事蔬菜遗传育种研究。E-mail:renhailong_2006@163.com
紫菜薹(Brassica rapa var. purpuraria)又名红菜苔,属十字花科芸薹属一、二年生草本植物[1-2],因其茎部呈紫色而得名。紫菜薹是由原始的菘(中国古代指白菜)从中国南方向北,至长江流域逐渐演化而来的一个变种,距今已有一千多年的栽培历史[3]。目前,紫菜薹在全国各地均有种植,在南方蔬菜生产和消费中有重要地位[4-5]。花茎和叶是紫菜薹的主要食用器官,研究表明:紫菜薹的花茎和叶中含有丰富的花色苷类物质,具有抗氧化、抗突变、抗癌及抗病毒等生物活性。同时,该物质对植物的生长发育也发挥着重要作用,因而受到广泛关注[6-7]。
叶绿体是绿色植物特有的半自主型细胞器,是能量转化和光合作用的重要场所,也是母系遗传信息的重要载体[8]。与核基因组相比,叶绿体基因组的结构更为稳定,基因组的大小、种类及密码子组成等均十分保守,还具有分子进化速率适宜、基因密集度高,拷贝数多等特点[9-10]。在高等植物中,基于上述特点的叶绿体基因组已成为种质鉴别分析的有效分子手段[11],在系统发育关系分析[12]、物种鉴定、物种演变等研究中得到广泛应用[13]。
近年来,随着高通量测序技术的不断完善及测序成本的降低,越来越多的植物叶绿体基因组被收录到基因库中[11]。十字花科是被子植物最大的科之一,包括已知的340个属的4 636个种,其中的芸薹属物种具有极高的经济价值,包括了许多重要的蔬菜、油料、饲料作物,如白菜、甘蓝、油菜、芥菜、芜菁和甘蓝等[14]。基于芸薹属物种的重要性,已有多个物种的叶绿体基因组完成了测序[15-21]。本研究首次报道了紫菜薹叶绿体全基因组序列,并进行了系统发育分析,为深入了解紫菜薹的系统发育关系提供了科学依据。
1" 材料与方法
1.1" 材" 料
紫菜薹线粒体基因组测序材料为‘优选洪山’,是提纯后的常规品系,由广州市农业科学研究院提供。紫菜薹在室内种植,光暗交替周期及温度为16 h、25 ℃/8 h、10 ℃,光照强度为250 μmol/(m2·s),光照光谱为白∶红=3∶1。播种后28 d,取紫菜薹幼嫩叶片,无菌水冲洗擦干后,液氮速冻5~10 min,存放于-80" ℃冰箱。
1.2" 方" 法
1.2.1" DNA提取及测序" 使用改良的CTAB法[22]从叶片样品中提取总基因组DNA,样品基因组DNA检测合格后,用机械打断的方法(超声波)将DNA片段化,然后对片段化的DNA进行片段纯化、末端修复、3′端加A、连接测序接头,再用琼脂糖凝胶电泳进行片段大小选择,进行PCR扩增形成测序文库,建好的文库先进行文库质检,质检合格的文库用Illumina 6000平台进行双端(PE)测序,测序读长为150 bp。
1.2.2" 叶绿体全基因组序列组拼接与注释" 使用fastp(version 0.20.0,https://github.com/OpenGene/fastp)软件[23]对原始数据进行过滤,得到的高质量的clean data。使用SPAdes v3.10.1[24](http://cab.spbu.ru/software/spades/)软件组装叶绿体基因组,kmer分别使用55、87、121。随后,将组装好的序列用Prodigal v2.6.3[25](https://www.github.com/hyattpd/Prodigal)注释CDS(Coding sequence),用Hmmer v3.1b2[26](http://www.hmmer.org)预测rRNA,用Aragorn v1.2.38[27](http://130.235.244.92/ARAGORN)预测tRNA。以Brassica rapa subsp rapa的叶绿体基因组为参考基因组(登录号:NC_049891),使用OGDRAW软件[28](https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制叶绿体全基因组图谱。最终注释的叶绿体基因组提交至NCBI,获得登录号:OP729397。
1.2.3" 叶绿体基因组特征分析" 使用微卫星检索工具MISA[29](https://webblast.ipk-gatersleben.de/misa/)鉴定叶绿体基因组中的简单序列重复序列(SSR),参数分别设置为8、5、3、3、3、3(单核苷酸至六核苷酸),且2个SSRs之间的最小距离为100 bp。使用vmatch v2.3.0[30](http://www.vmatch.de/)软件结合Perl脚本鉴定散在重复序列,其参数设置为:最小长度(minimum length)=30 bp,海明距离(hamming distance)=3。使用mafft软件[31](--auto模式)对不同物种的同源基因序列进行全局比对,使用dnasp5[32]计算每个基因的Pi值,使用IRscope[33](https://github.com/xul962464/perl-IRscope)进行边界分析作图。
1.2.4" 系统发育分析" 从NCBI(https://www.ncbi.nlm.nih.gov)下载18种十字花科植物的叶绿体基因组,分别为:Camelina anomala(NC_061686.1)[34],Boechera stricta(NC_049599.1),Capsella bursa-pastoris(NC_009270.1),Crucihimalaya himalaica(NC_061290.1)[35],Turritis glabra OhmiShirahama(LC325490.1),Cardamine bipinnata(NC_060865.1)[36],Schrenkiella parvula(NC_028726.1)[37],Sinapis arvensis(NC_035303.1),Raphanus sativus(NC_024469.1),Arabidopsis thaliana(MN603471.1)[38],B.carinata(NC_059807.1),B.juncea(NC_028272.1),B.napus(NC_016734.1)[39],B.oleracea(MN396847.1)[40],B.rapa subsp.rapa(NC_049891.1),B.nigra(NC_030450.1)[16],B.rapa subsp. pekinensis(NC_015139.1)和B.rapa subsp. chinensis (KX681648.1),番木瓜科番木瓜属Carica papaya(NC_010323.1)作为外类群。使用MAFFT软件(v7.427)[41]完成不同物种间的多序列比对。使用RAxML v8.2.10软件[42](https://cme." h-its.org/exelixis/software.html)GTRGAMMA模型进行Rapid Bootstrap分析(bootstrap=" 1 000),并构建最大似然发进化树。
2" 结果与分析
2.1" 紫菜薹叶绿体基因的基本特征
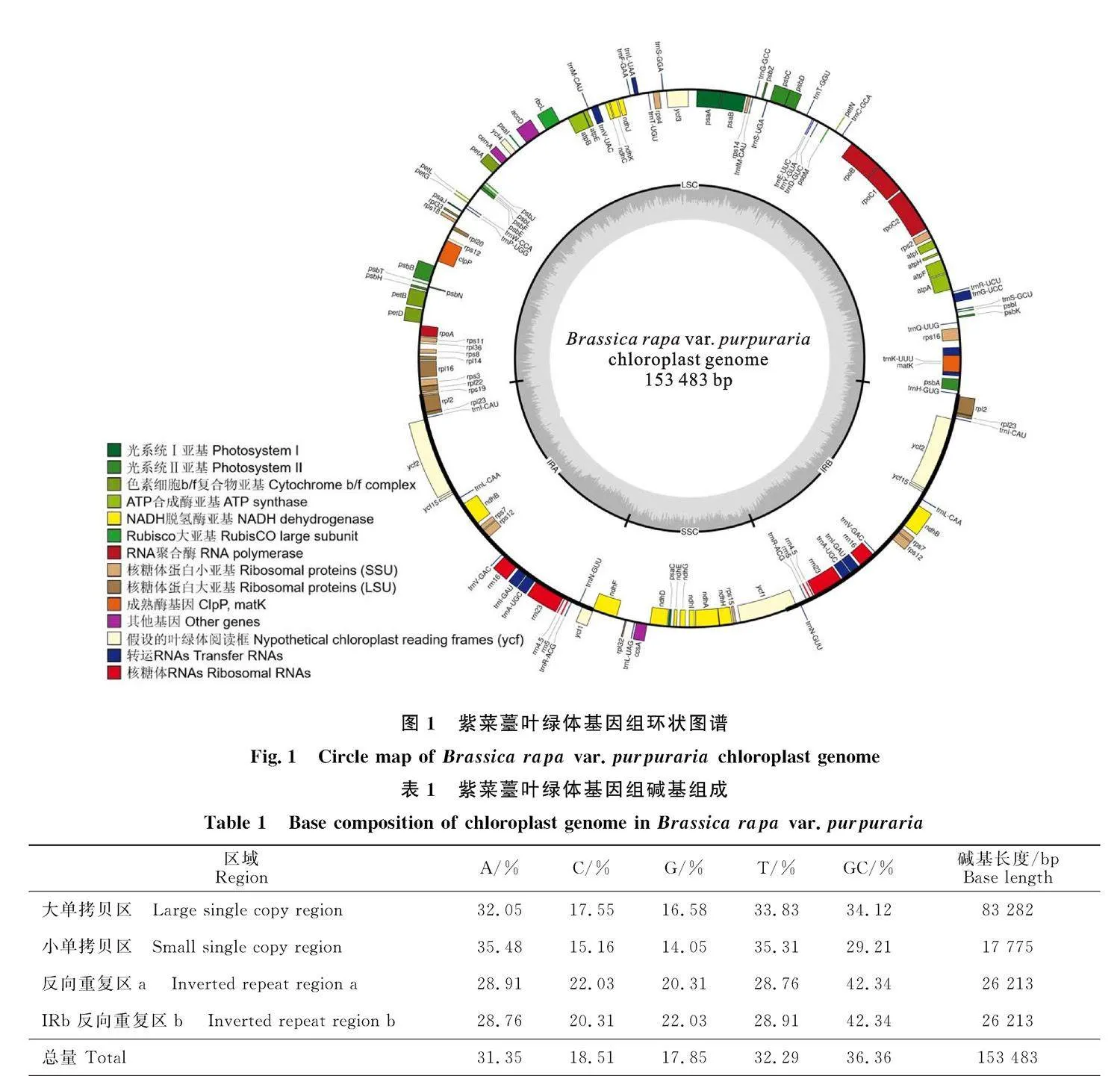
紫菜薹叶绿体基因组共测得3.29 G数据,质控后获得19 543 592条高质量的clean reads,组装、拼接后获得叶绿体基因组环状序列(图1)。如图1所示,紫菜薹叶绿体基因组为典型的四分环状结构,基因组整体GC含量为36.36%;序列长度为153 483 bp,包括1个83 282 bp的大单拷贝区(Large single copy region,LSC)、1个17 775 bp的小单拷贝区(Small single copy region,SSC)和1对各26 213 bp的反向重复区(Inverted repeat region A,IRA/Inverted repeat region B,IRB)(表1)。
2.2" 紫菜薹叶绿体基因的功能注释
紫菜薹叶绿体基因组共注释得到132个基因,分别为87个蛋白编码基因、37个tRNA和8个rRNA。基因功能主要为光合合成、自我复制等,此外还有一些功能未知的基因(表2)。其中,6个tRNA基因(trnA-UGC、trnG-UCC、trnI-GAU、trnK-UUU、trnL-UAA和trnV-UAC)和10个蛋白编码基因(ndhA、ndhB、petB、petD、" rpl16、 rpl2、 rpoC1、 rps12和 rps16)中各包含1个内含子,2个蛋白编码基因(clpP和 ycf3)各包含2个内含子。12个蛋白编码基因(ndhB、 rpl2、 rpl23、 rps12、 rps7、 rrn16、 rrn23、 rrn4.5、 rrn5、 ycf1、 ycf15和 ycf2)和7个tRNA基因(trnA-UGC、trnI-CAU、trnI-GAU、trnL-CAA、trnN-GUU、trnR-ACG和trnV-GAC)有2个拷贝。
2.3" 密码子偏好性分析
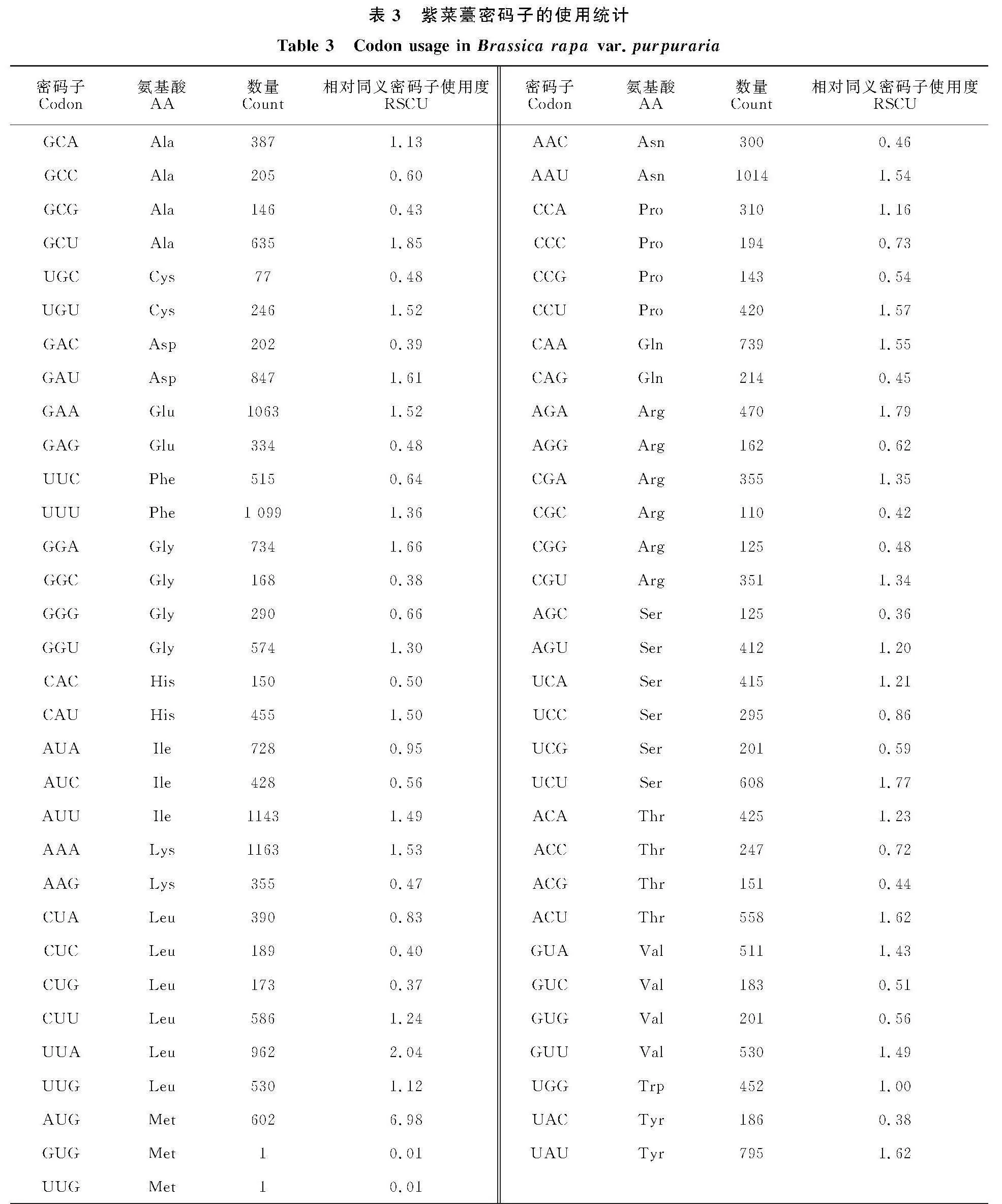
由于密码子的简并性,每个氨基酸最少对应1种密码子,最多对应6种密码子。不同物种、不同生物体的基因组密码子使用率存在着很大的差异,这种偏好性被认为是自然选择、物种突变和遗传漂变的综合结果。紫菜薹叶绿体基因组中,亮氨酸(Leu)使用最为频繁,其数量为2 830个;其次是异亮氨酸(Ile)和丝氨酸(Ser),数量分别为" 2 299个和2 056个;使用次数最少的是半胱氨酸(Cys),数量仅为323个。其中,使用最多的同义密码子是编码赖氨酸(Lys)的AAA,数量为" 1 163个,使用最少的是编码甲硫氨酸(Met)的GUG和UUG,仅为1个(表3)。
相对同义密码子使用度(RSCU)是一个较好的衡量密码子偏好性的指标。当RSCUgt;1时,表明密码子存在明显的偏好性。在紫菜薹中,RSCUgt;1的有30个密码子,且多以A或U结尾。其中RSCU值最高的是编码甲硫氨酸(Met)的AUG,为6.98,因为AUG既编码甲硫氨酸,又作为起始密码子,因此具有较高的密码子使用偏好。RSCU值最低的是编码甲硫氨酸(Met)的GUG和UUG,仅为0.01。随机遗传漂变、突变压力、自然选择和重组等均可导致密码子使用偏好性。因此,研究密码子使用偏好性对于理解基因表达水平及生物的遗传与演化具有重要意义。
2.4" 重复序列检测
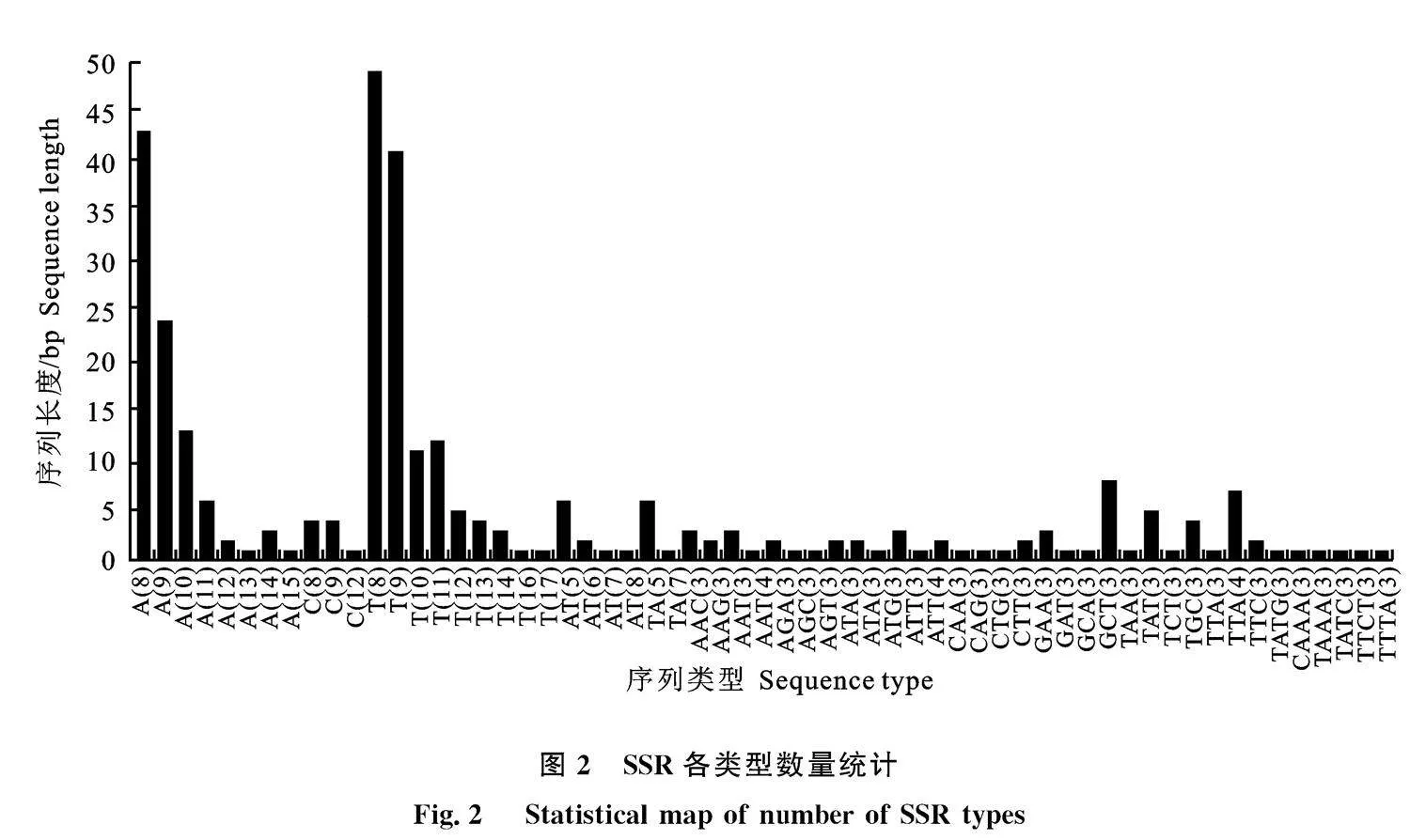
简单重复序列SSR(Simple Sequence Repeats),是一类由几个核苷酸(一般为1~6个)为重复单位组成的长达几十个核苷酸的串联重复序列,其广泛存在于叶绿体基因组中,常用于植物物种鉴定的研究中。本研究中,紫菜薹叶绿体基因组中共检测到315个SSRs,总长度3 364 bp,包括229个单核苷酸、17个二核苷酸、63个三核苷酸和6个四核苷酸(图2)。进一步分析发现,315个SSRs中比例最高的前4种类型(A/T、AAT/ATT、AAG/CTT和AT/AT),分别占SSR总数的69.84%(220个)、7.62%(24个)、6.03%(19个)和5.40%(17个),这些SSRs分别位于LSC(197)、SSC(72)和IRs(46)。
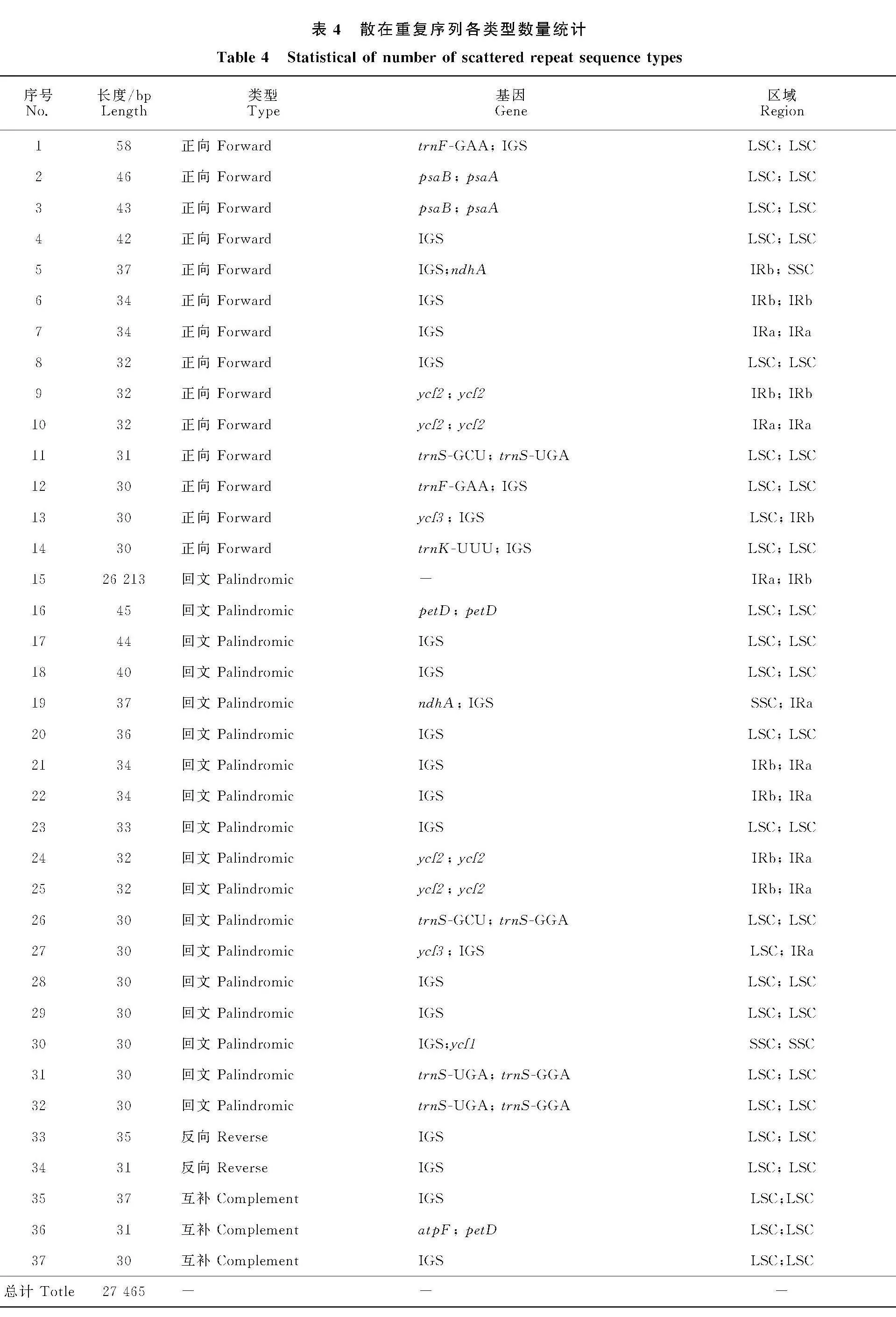
散在重复序列是与串联重复序列不同的另一种重复序列,在基因组中呈分散式分布,以正向(forward)、回文(palindromic)、反向(reverse)、互补(complement)4种形式存在。在紫菜薹叶绿体基因组中共鉴定出37个散在重复序列(表4),总长度27 465 bp,正向、回文、反向和互补4种散在重复序列分别有14个、18个、2个和3个。
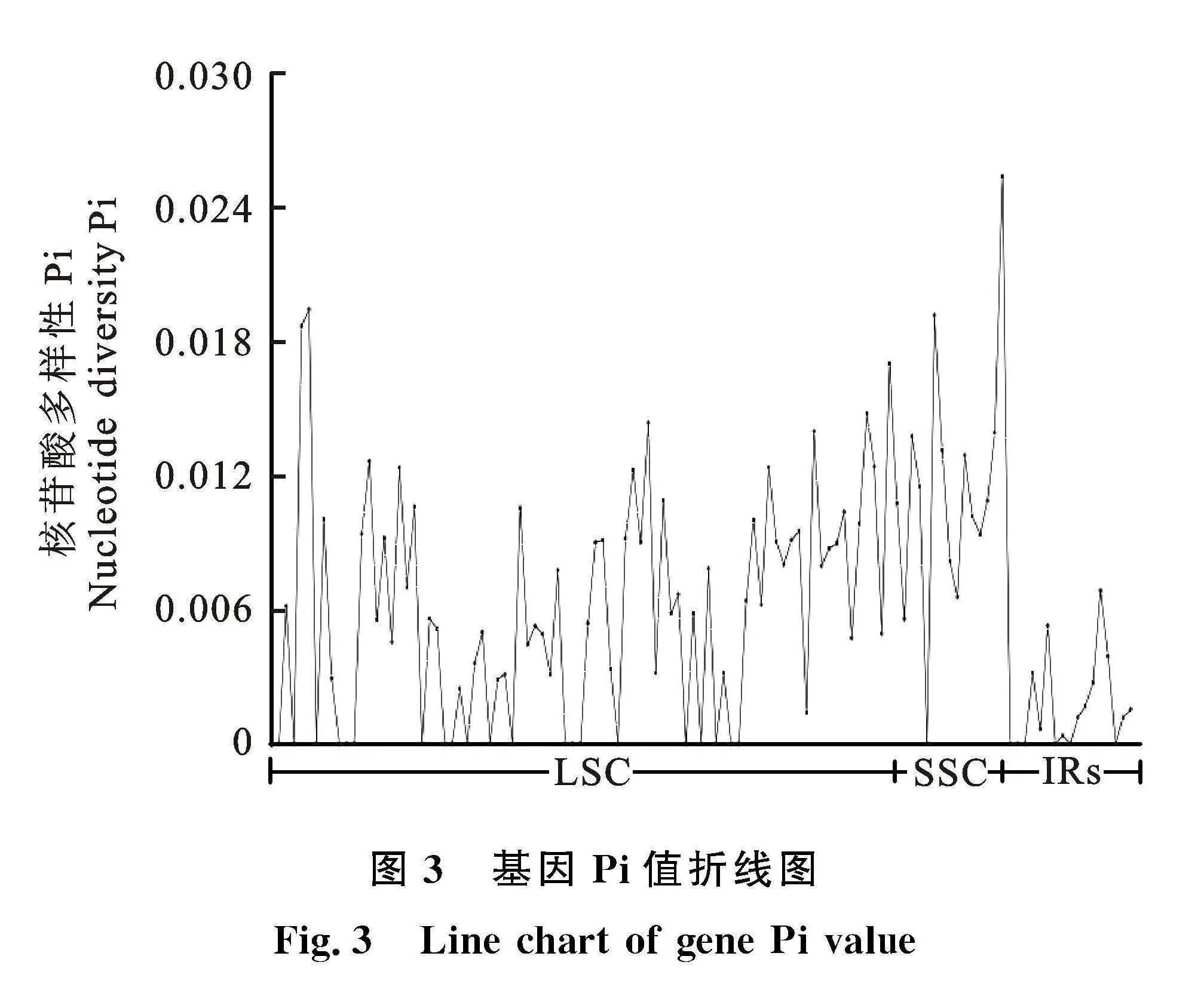
2.5" 核酸多样性Pi和边界分析提供潜在的分子标记。本研究选取Brassica rapa var.purpuraria(OP729397.1)、Brassica rapa" subsp.chinensis(KX681648.1)、Brassica rapa" subsp.pekinensis(NC_015139.1)、Brassica oleracea(MN396847.1)、Arabidopsis thaliana(MN603471.1)、Brassica napus(NC_016734.1)、Raphanus sativus(NC_024469.1)、Brassica juncea(NC_028272.1)、Brassica nigra(NC_030450.1)、Brassica rapa subsp.rapa(NC_049891.1)和Brassica carinata(NC_059807.1)等11种十字花科植物的叶绿体基因组。核酸多样性分析表明,11个物种的平均核苷酸多样性为0.006 2。叶绿体基因组不同区域核苷酸多样性有明显不同,LSC的核苷酸多样性在0~0.019 7之间,平均为0.006 2;SSC的核苷酸多样性在0~0.025 7之间,平均为0.011 6;而IR的核苷酸多样性在" 0~0.006 9之间,平均仅为0.001 7。由此可知,十字花科11个物种的叶绿体基因组中SSC的核苷酸多样性最高,而IR的核苷酸多样性明显低于其他2个区域(图3),说明IR相比其他2个区域更为保守。
在基因组进化过程中,IR边界会发生扩张与收缩,使某些基因进入IR区或单拷贝区,这是植物叶绿体基因组大小差异的主要原因。本研究同样选取上述11种十字花科植物进行叶绿体基因组边界分析,结果显示(图4),所选取植物的叶绿体基因组均有4个边界,即LSC/IRb、IRb/SSC、SSC/IRa和IRa/LSC。其中,JLB(LSC/IRb)均位于rps19基因编码区内,且向IRb区有113~114 bp的扩张;JSB(IRb/SSC)边界区均位于" ycf1和ndhF基因编码区内,ycf1基因向IRb区有" 1 027~1 030 bp的扩张,ndhF基因向IRb区有36~37 bp的扩张;JSA(SSC/IRa)边界均位于ycf1基因内,长度向IRa区域扩张程度不同,为" 1 027~1 030 bp。从以上分析结果来看,十字花科不同属植物在进化过程中IR边界区中存在一定的收缩和扩张。但总体来说,IR区的变化幅度非常小,十字花科植物的叶绿体基因组比较" 保守。
2.6" 系统发育分析
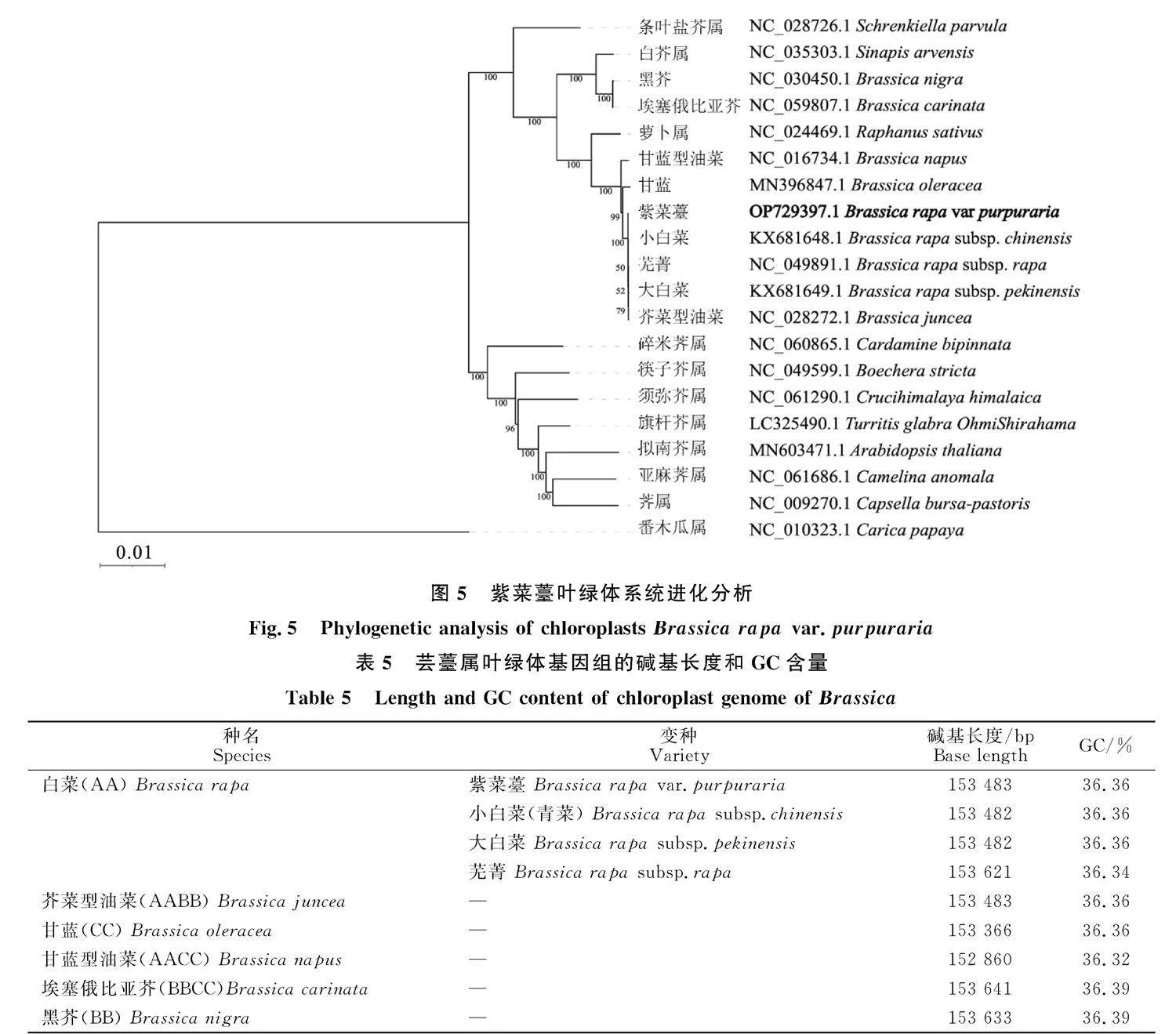
以20种十字花科植物为内类群,番木瓜为外类群,采用最大似然法(ML法)构建植物系统发育树(图5)。结果表明,20种十字花科植物以100%的支持率分为两个分支,第一个分支包括条叶盐芥属、白芥属、萝卜属和芸薹属全部,其中紫菜薹Brassica rapa var.purpuraria(OP729397.1)与小白菜(青菜)Brassica rapa subsp.chinensis(KX681648.1)、芜菁Brassica rapa subsp.rapa(NC_049891.1)、大白菜Brassica rapa subsp. pekinensis(NC_015139.1)和芥菜型油菜Brassica juncea(NC_028272.1)以100%支持率聚在" 一个小分支;第二个分支包括亚麻荠属、筷子芥属、荠属、须弥芥属、旗杆芥属、碎米荠属和拟南" 芥属。
3" 讨论与结论
芸薹属(Brassica)是十字花科最重要的一个属[43]。该属有6个种,包括白菜(AA,2n=2x=20)、甘蓝(CC,2n=2x=18)和黑芥(BB,2n= "2x=16)3个二倍体基本种以及甘蓝型油菜(AACC,2n=4x=38)、芥菜型油菜(AABB,2n=4x=36)和埃塞俄比亚芥(BBCC,2n=4x=34)" 3个四倍体复合种[44],且四倍体复合种是由3个基本种通过两两相互杂交和自然加倍而形成,这种关系被定义为“禹氏三角”[14]。其中,白菜种内变种繁多且名称混乱,相互之间可以自由杂交,紫菜薹就是经长期驯化培育出的一个白菜变种。因此,搞清楚不同变种间的亲缘关系,对于白菜类蔬菜的品种选配十分必要。叶绿体基因组大小、结构和基因种类一般较为保守,且为单亲遗传,现已成为研究植物系统进化及进行物种鉴定的有力工具[45]。
本研究发现芸薹属叶绿体基因组长度为" 152 860~153 641,GC含量为36.32%~36.39%(表5)。其中,紫菜薹叶绿体基因组长度与小白菜、大白菜的芥菜型油菜几乎相同,为" 153 482 bp或153 483 bp;GC含量与小白菜、大白菜、芥菜型油菜和甘蓝相同,均为36.36%,结果与朱惠霞等[46]在甘蓝上的研究相似,说明芸薹属种内叶绿体基因组的碱基数量和成分比较保守。进一步分析发现,造成紫菜薹叶绿体基因组长度比小白菜和大白菜多一个碱基的原因主要是受LSC长度的影响。此外,本研究还发现,芜菁与白菜的其他变种(紫菜薹、小白菜和大白菜)在基因组长度和GC含量上存在一定的差异,但在系统发育树中仍聚在一起,说明叶绿体基因组在十字花科植物系统进化及进行物种鉴定的研究中是有效的。
奚丹丹等[47]利用普通绿叶紫菜薹、紫叶紫菜薹和绿叶绿菜薹不同组织的Illumina高通量转录组测序数据,分析了unigene序列中SSR的组成、分布频率和特性,同样发现单核苷酸重复" (31.23%)和三核苷酸重复(19.74%)为主要的重复类型,但所占比例与本研究结果有明显的区别,可能与叶绿体基因组的特点及序列长度较短有关。十字花科植物叶绿体基因组的聚类分析表明,白菜(紫菜薹、小白菜、大白菜和芜菁)与芥菜型油菜为一个小分支,关系最近,这与从序列特征及IR边界角度分析得出的结果相一致,从而证实了紫菜薹是白菜的变种。
本研究对紫菜薹进行了叶绿体基因组的组装与注释,解析了紫菜薹叶绿体基因组的基本结构、重复序列和SSR位点等基本特征,发现紫菜薹的叶绿体基因组较保守,未出现基因丢失及基因组大片段缺失现象;且含有丰富的重复序列和SSR位点,可作为潜在的DNA条形码用于遗传多样性分析、种质鉴定及系统发育研究。在比较紫菜薹与其近缘属物种之间的关系时,系统发育分析表明,白菜不同亚种间及芥菜型油菜的亲缘关系较近,与Xue等[48]在线粒体基因组中的研究,发现芥菜型油菜的母系来源于白菜的结果一致。本研究为进一步理解紫菜薹的系统发育关系及叶绿体基因组的变异情况提供了有价值的信息。
参考文献" Reference:
[1]" ZHANG X,ZHANG K,WU J,et al.QTL-Seq and sequence assembly rapidly" mapped the gene BrMYBL2.1 for the" purple trait in Brassica rapa[J].Scientific Reports,2020,10:2328.
[2]LI G H,CHEN H C,LIU J L,et al.A high-density genetic map developed by specific-locus amplified fragment (SLAF) sequencing and identification of a locus controlling anthocyanin pigmentation in stalk of Zicaitai (Brassica rapa L.ssp.chinensis var.purpurea)[J].BMC Genomics,2019,20(1):343.
[3]聂启军,李金泉,董斌峰,等.紫菜薹名优品种——洪山菜薹[J].湖北农业科学,2020,59(22):133-135.
NIE Q J,LI J Q,DONG B F,et al.A famous variety of purple Caita——Hongshan Caitai[J].Hubei Agricultural Sciences,2020,59(22):133-135.
[4]吴朝林,陈文超.中国紫菜薹地方品种初步研究[J].作物品种资源,1997(3):8-10.
WU CH" L,CHEN W CH.A preliminary study on local Zicaitai in China[J].Crop Variety Resources,1997(3):8-10.
[5]邝敏杰,齐敏玉,何静仁,等.紫菜薹花色苷组分鉴定及其稳定性和抗氧化性[J].中国农业科学,2014,47(20):4067-4077.
KUANG M J,QI M Y,HE J R,et al.Identification of anthocyanins in Brassica campestris L.and their stability and" antioxidant" activity[J].Scientia Agricultura Sinica,2014,47(20):4067-4077.
[6]王建军,徐园园,刘同坤,等.紫菜薹BrbHLH49基因克隆与功能分析[J].南京农业大学学报,2021,44(3):421-427.
WANG J J,XU Y Y,LIU T K,et al.Cloning and function analysis of BrbHLH49 gene in purple tsai-tai[J].Journal of Nanjing Agricultural University,2021,44(3):421-427.
[7]GUO N,WU J,ZHENG S,et al.Anthocyanin profile characterization and quantitative trait locus mapping in zicaitai (Brassica rapa L.ssp.chinensis var.purpurea)[J].Molecular Breeding,2015,35:113.
[8]陶晓丽,王彦荣,刘志鹏.牧草叶绿体基因组研究进展[J].草业科学,2015,32(6):978-987.
TAO X L,WANG Y R,LIU ZH P.Progress in chloroplast genome analysis of herbage[J].Pratacultural Science,2015,32(6):978-987.
[9]孙志轩,敖平星,毕玉芬,等.‘德钦’紫花苜蓿叶绿体基因组序列及特征分析[J].草地学报,2022,30(2):320-328
SUN ZH" X,AO P X,BI Y F,et al.Complete chloroplast genomes sequence and characteristics analsis of Medicago sativa ‘Deqin’[J].Acta Agrestia Sinica,2022,30(2):320-328.
[10]" 陈晓颖,胡本祥,史嘉周,等.茜草叶绿体全基因组序列及其系统发育分析[J].西北植物学报,2023,43(11):1855-1865.
CHEN X Y,HU B X,SHI J ZH,et al.Complete chloroplast genome and phylogenetic analysis of Rubia cordifolia[J].Acta Botanica Boreali-Occidentalia Sinica,2023,43(11):1855-1865.
[11]郝新艳,赵淑文,刘嘉伟,等.杂花苜蓿叶绿体基因组特征及系统发育分析[J].草地学报,2023,31(6):1665-1672.
HAO X Y,ZHAO SH W,LIU J W,et al.Characteristics and phylogenetic analysis of chloroplast genome of Medicago varia[J].Acta Agrestia Sinica,2023,31(6):1665-1672.
[12]马孟莉,张" 薇,孟衡玲,等.草果叶绿体基因组特征及系统发育分析[J].中草药,2021,52(19):6023-6031.
MA M L,ZHANG W,MENG H L,et al.Characterization and phylogenetic analysis of the complete chloroplast genome of Amomum tsao-ko[J].Chinese Traditional and Herbal Drugs,2021,52(19):6023-6031.
[13]赵" 明,张宏斌,李" 伟,等.祁连圆柏叶绿体基因组序列特征分析[J].植物资源与环境学报,2023,32(3):1-11.
ZHAO M,ZHANG H B,LI W,et al.Analysis on chloroplast genome sequence characteristics of" Juniperus przewalskii[J].Journal of Plant Resource and Environment,2023,32(3):1-11.
[14]WU J,LIANG J,LIN R,et al.Investigation of Brassica and its relative genomes in the post-genomics era[J].Horticulture Research,2022:182.
[15]PRABHUDAS S K,RAJU B,THODI S K,et al.The complete chloroplast genome sequence of Indian mustard (Brassica juncea L.)[J].Mitochondrial DNA,2015, 27(6):4622-4623.
[16]SEOL Y J,KIM K,KANG S H,et al.The complete chloroplast genome of two Brassica species,Brassica nigra and B.Oleracea[J].Mitochondrial DNA Part A,2017," 28(2):167-168.
[17]KIM C K,SEOL Y J,PERUMAL S,et al.Re-exploration of U’s" triangle Brassica species" based on chloroplast genomes and 45S nrDNA sequences[J].Scientific Reports,2018,8(1):7353.
[18]DU X,ZENG T,FENG Q,et al.The complete chloroplast genome sequence of yellow mustard (Sinapis alba L.) and its phylogenetic relationship to other Brassicaceae species[J].Gene,2020,731:144340.
[19]HAN R,TIAN M,ZHAN G,et al.Complete chloroplast genome sequence of turnip (Brassica rapa. ssp. rapa):genome structure and phylogenetic analysis[J].Mitochondrial DNA B Resources,2020,5(3):3555-3557.
[20]WU J Y,MA X C,Ma L,et al.Complete chloroplast genome sequence and phylogenetic analysis of winter oil rapeseed (Brassica rapa L.)[J].Mitochondrial DNA B Resources,2021,6(3):723-731.
[21]LIU H,ZHAO W,HUA W,et al.A large-scale population based organelle pan-genomes construction and phylogeny analysis reveal the genetic diversity and the evolutionary origins of chloroplast and mitochondrion in Brassica napus L[J].BMC Genomics,2022,23(1):339.
[22]SAHU S K,THANGARAJ M,KATHIRESAN K.DNA extraction" protocol for" plants with high" levels of secondary" metabolites and" polysaccharides without" using" liquid" nitrogen and" phenol[J].ISRN" Molecular Biology,2012,6(4):1-6.
[23]CHEN S,ZHOU Y,CHEN Y,et al.Fastp:An ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[24]BANKEVICH A,NURK S,ANTIPOV D,et al.SPAdes:a new genome assembly algorithm and its applications to single-cell sequencing[J].Journal of Computational Biology,2012,19(5):455-477.
[25]HYATT D,CHEN G L,LOCASCIO P F,et al.Prodigal:prokaryotic gene recognition and translation initiation site identification[J].BMC Bioinformatics,2010,11:119.
[26]POTTER S C,LUCIANI A,EDDY S R,et al.HMMER web server:2018 update[J].Nucleic Acids Research,2018,46(W1):W200-W204.
[27]LASLETT D,CANBACK B.ARAGORN,a program to detect tRNA genes and tmRNA genes in nucleotide sequences[J].Nucleic Acids Research,2004,32(1):11-16.
[28]GREINER S,LEHWARK P,BOCK R.OrganellarGenomeDRAW (OGDRAW) version1.3.1:Expanded toolkit for the graphical visualization of organellar genomes[J].Nucleic Acids Research,2019,47:59-64.
[29]BEIER S,THIEL T,MVNCH T,et al.MISA-web:a web server for microsatellite prediction[J].Bioinformatics,2017,33(16):2583-2585.
[30]KURTZ S.The Vmatch large scale sequence analysis software-A Manual[J].Center for Bioinformatics,2010,170(24):391-392.
[31]KATOH K,MISAWA K,KUMA K,et al.MAFFT:a novel method for rapid multiple sequence alignment based on fast Fourier transform[J].Nucleic Acids Research,2002,30(14):3059-3066.
[32]LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25:1451-1452.
[33]AMIRYOUSEFI A,HYVONEN J,POCZAI P.IRscope:an online program to visualize the junction sites of chloroplast genomes[J].Bioinformatics,2018,34(17):3030-3031.
[34]BROCK J R,MANDAKOVA T,MCKAIN M,et al.Chloroplast phylogenomics in Camelina (Brassicaceae) reveals multiple origins of polyploid species and the maternal lineage of C.sativa[J].Horticulture Research,2022,9:uhab050.
[35]CHEN H L,AL-SHEHBAZ I A,QIAN L S,et al.Pulvinatusia (Brassicaceae),a new cushion genus from China and its systematic position[J].Phyto" Keys,2022,189:9-28.
[36]RU Y,SCHULZ R,KOCH M A.Successful without sex-the enigmatic biology and evolutionary origin of coralroot bittercress (Cardamine bulbifera,Brassicaceae)[J].Perspectives in Plant Ecology Evolution and Systematics,2020,46:125557.
[37]HE Q,HAO G,WANG X,et al.The complete chloroplast genome of Schrenkiella parvula (Brassicaceae)[J].Mitochondrial DNA A DNA Mapp Seq Anal,2016,27(5):3527-3528.
[38]ZHANG M,ZHAO J,LI W,et al.Increased photosystem ii translation efficiency as an important photoprotective mechanism in an arabidopsis thaliana ecotype (tibet-0)"" adapted to high light environments[J].Environmental and Experimental Botany,2020,183:104350.
[39]HU Z Y,HUA W,HUANG S M,et al.Complete chloroplast genome sequence of rapeseed (Brassica napus L.) and its evolutionary implications[J].Genetic Resources and Crop Evolution,2011,58:875-887.
[40]PERUMAL S,WAMINAL N E,LEE J,et al.Nuclear and chloroplast genome diversity revealed by low-coverage whole-genome shotgun sequence in 44 Brassica oleracea breeding lines[J].Horticultural Plant Journal,2021,7:539-551.
[41]KATOH K,STANDLEY D M.Mafft multiple sequence ""alignment software version 7:improvements in performance and usability[J].Molecular Biology and Evolution,2013,30(4):772-780.
[42]STAMATAKIS A.RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J].Bioinformatics,2014,30(9):1312-1313.
[43]ESSOH A P,MONTEIRO F,PENA A R,et al.Exploring glucosinolates diversity in Brassicaceae:a genomic and chemical assessment for deciphering abiotic stress tolerance[J].Plant Physiology and Biochemistry,2020,150:151-161.
[44]KOH,J C O,BARBULESCU D M,NORTON S,et al.A multiplex PCR for rapid identification of Brassica species in the triangle of U[J].Plant Methods,2017,13:49.
[45]高娜娜,赵志礼,倪梁红.植物叶绿体" ycf15基因应用于药用植物鉴定的前景展望[J].中草药,2017,48(15):3210-3217.
GAO N N,ZHAO ZH L,NI L H.Prospect:Identification of medicinal plant based on chloroplast gene"" ycf15[J].Chinese Traditional and Herbal Drugs,2017,48(15):3210-3217.
[46]朱惠霞,陶兴林,刘明霞.彩色花椰菜叶绿体全基因组特征分析[J].种子,2023,42(4):64-71,157.
ZHU H X,TAO X L,LIU M X.Characterization of the complete chloroplast genome of" Brassica oleracea L.var.botrytis[J].Seed,2019,42(4):64-71,157.
[47]奚丹丹,高" 璐,李晓锋,等.基于转录组测序的菜薹SSR分子标记开发及初步验证[J/OL],分子植物育种,2022-09-17[2023-12-04].http://kns.cnki.net/kcms/detail/46.1068.S.20220916.1333.050.html.
XI D D,GAO L,LI X F,et al.Development and identification of SSR" molecular markers based on transcriptome sequencing of caitai[J/OL].Molecular Plant Breeding,2022-09-17[2023-12-04].http://kns.cnki.net/kcms/detail/46.1068.S.20220916.1333.050.html.
[48]XUE J Y,WANG Y,CHEN M,et al.Maternal" inheritance of U’s triangle and" evolutionary" process of Brassica" mitochondrial" genomes[J].Frontiers in Plant Science,2020,11:805.
Chloroplast" Genome Sequence and Phylogenetic Analysis of Brassica rapa var.purpuraria
WANG Chuanzhi1,ZHOU Xianyu2,LI Yangmei1,
XIAO Wanyu2,WANG Yufeng1 and" REN Hailong3
(1.School of Biology and Food Engineering,Suzhou University,Suzhou" Anhui" 234000,China; 2.Guangzhou
Institute of Agricultural Science,Guangzhou" 510308,China; 3.Institute of Crop Research,
Guangdong Academy of Agricultural Sciences/Key Laboratory of Crop Genetic Improvement of
Guangdong Province,Guangzhou" 510640,China)
Abstract" Zicaitai (Brassica rapa var.purpuraria) is a variety of Brassica" rapa subspecies belonging to the" Brassicaceae" family.The chloroplast genome of Brassica rapa var.purpuraria was sequenced,assembled,and annotated using the Illumina high-throughput sequencing platform.The results indicated that the chloroplast genome of Brassica rapa var.purpuraria had a typical quartered circular structure,with a size of 153 483 bp and GC content of 36.36%.It comprises a large single copy region (LSC) of 83 282 bp,a small single copy region (SSC) of 17 775 bp,and a pair of reverse repeat regions (IRa/IRb) of 2×26 213 bp.Genome annotation predicted 132 genes,including 87 protein coding genes,37 tRNAs,and 8 rRNAs.Additionally,313 simple repeat sequences (SSRs) and 37 scattered repeat sequences were identified.Codon bias analysis found that there was a bias for"" using A/U base endings.Boundary analysis and sequence variation analysis demonstrated the chloroplast genome of Brassica to be highly conservative,SSC region of chloroplast genome had the largest difference and the highest degree of variation,while the IR region had the smallest difference and was the most conservative.Phylogenetic analysis based on the chloroplast genome sequence indicated that Brassica rapa var.purpuraria belongs to the same branch as other varieties of Brassica rapa.This study provides a reference for the taxonomic,systematic,and biogeographical studies of Brassica genus,particularly Brassica rapa species.
Key words" Brassica rapa; Brassica rapa var.purpuraria; Chloroplast genome; Phylogenetic analysis
Received"" 2023-12-04""" Returned" 2024-01-16
Foundation item" National College Student Innovation and Entrepreneurship Training Project of Suzhou"" University (No.202310379012); Suzhou University (Post-doctoral) Research Fund (No.2023BSK022); Horizontal Project of Molecular Breeding Center of Suzhou University (No.2022xhx235,No.2023xhx129); Key Project of Education Department of Anhui Province (No.2022AH051384).
First author" WANG Chuanzhi,male,Ph.D,senior agronomist.Research area:molecular plant genetics,breeding and cultivation.E-mail:jfwcz@163.com
Corresponding"" author" ZHOU Xianyu,female,master,agronomist.Research area:vegetable genetics and breeding.E-mail:1562783858@qq.com
REN Hailong,male,Ph.D,associate research fellow.Research area:vegetable genetics and breeding.E-mail:renhailong_2006@163.com
(责任编辑:潘学燕" Responsible editor:PAN Xueyan)

