染色质阅读器蛋白EBS和SHL的诱导表达和体外SUMO化修饰鉴定


【摘" "要】" "EBS和SHL是植物特有的一类染色质阅读器蛋白,通过特异性识别并结合两类功能相互拮抗的组蛋白修饰调控靶基因表达和相关生物学过程,但目前未见有关翻译后修饰调控其蛋白功能的报道。利用大肠杆菌SUMO化修饰重建体系发现,EBS和SHL都存在SUMO化修饰,且不论以哪种SUMO分子亚型为供体都只有一条SUMO化条带,根据SUMO化条带和与本底蛋白分子量差值推测均为单位点的单SUMO化修饰。进一步利用定点突变实验证实,K216是EBS唯一且关键的SUMO化位点,而SHL的SUMO化位点有待继续研究,但至少可以排除K111、K178和K220三个位点,为下一步探讨SUMO化修饰调控EBS和SHL的蛋白功能奠定了基础。
【关键词】" "染色质阅读器蛋白;EBS;SHL;SUMO化修饰;SUMO
【Abstract】" " EBS and SHL are plant-specific chromatin reader proteins, which regulate target gene expression and related biological processes by specifically recognizing and binding to two antagonistic histone modifications. However, there have been no reports on the regulation of their protein functions by post-translational modifications. In this study, after the E. coli SUMOylation reconstruction system is used, it is found that both EBS and SHL have SUMOylation, and there is only one SUMOylation band, no matter which SUMO molecular subtype is used as the donor. According to the difference of molecular weight between the SUMOylation band and the background protein band, it is inferred that both EBS and SHL are single-site single SUMOylation. Further site-directed mutagenesis experiments confirm that K216 is the single critical SUMOylation site for EBS, while the SUMOylation site for SHL remains to be further explored, but at least K111, K178, and K220 can be excluded. This study lays a foundation for further exploration of the regulation of EBS and SHL protein functions by SUMOylation modification.
【Key words】" " "chromatin reader; EBS; SHL; SUMOylation; SUMO
〔中图分类号〕 Q943" " " " " " " " "〔文献标识码〕" A
〔文章编号〕 1674 - 3229(2024)04 - 0041 - 08" " " " " " " " " " " "DOI:10.20218/j.cnki.1674-3229.2024.04.006
0" " "引言
SUMO化修饰(SUMOylation)是一种重要的蛋白质翻译后修饰,在真核生物中广泛存在,该修饰与泛素化修饰的过程类似,依次需要 E1 激活酶、E2 偶联酶、E3 连接酶顺序催化,最终将活性 SUMO (Small Ubiquitin-like Modifier,小泛素样修饰蛋白)分子共价连接到底物蛋白特定赖氨酸残基(Lys,K)侧链的ε-NH2上,通过影响底物蛋白的活性、稳定性、亚细胞定位、蛋白质相互作用以及蛋白复合体的形成或解离等,最终影响底物蛋白的生物功能及其参与的生物学过程[1-5]。在泛素化修饰中,底物蛋白泛素化的特异性往往由数量庞大的泛素E3连接酶决定,且泛素化通常导致底物蛋白降解[6]。与泛素化不同的是,底物蛋白SUMO化的特异性不是由SUMO E3连接酶决定,且SUMO化通常不会直接诱导底物蛋白降解,另外SUMO化有时候不需要SUMO E3连接酶参与也能完成,E3只是增强了SUMO化的效率[2,6-7]。
SUMO化修饰以SUMO分子为供体,新翻译出来的SUMO分子一般以前体的形式存在,此时还没有活性,不能直接作为SUMO化底物的供体[1-3]。SUMO蛋白酶利用其内肽酶的活性切掉前体SUMO分子C末端一小段序列后暴露出两个连续的甘氨酸序列(GG),此时才形成成熟有活性的SUMO分子[1-3]。SUMO分子在不同物种中通常存在多种亚型和差异性的时空表达模式,因此不同SUMO分子亚型在SUMO化形成和功能发挥上会呈现一定的差异性和特异性。比如在哺乳动物中据报道有4个SUMO分子亚型,在拟南芥中则有9个亚型,但这9个亚型中确定存在转录本的只有4个,即SUMO1、SUMO2、SUMO3和SUMO5[1-2]。
SUMO化修饰的位点在底物蛋白特定序列的K上,这些特定序列存在一定的规律性和保守性,故而被称为“共有基序”,最经典的共有基序为“[Ψ]-[K]-[x]-[α]”,其中Ψ为疏水氨基酸,x为任意氨基酸,α为天冬氨酸(Asp,D)或谷氨酸(Glu,E)[1-2]。后续研究发现,SUMO化有时候也发生在一些不完整的共有基序中,如“[K]-[x]-[α]”,或者是反向的,如“[α]-[x]-[K]-[Ψ]”[8-9]。可见,SUMO 化基序不是单一的,存在一定的变异性,具有多样化的倾向。
拟南芥 EBS(Early Bolting in Short Days)和 SHL(Short Life)是两个高度同源的染色质阅读器蛋白,存在一定的功能冗余,已被证实共同参与调控拟南芥的开花时间[10-12]、种子休眠与萌发[13]、高温介导的萌发抑制[14]、根的发育[15]等过程。EBS和SHL都含有N端BAH和C端PHD两个功能结构域,BAH结构域特异性识别并结合抑制性组蛋白修饰H3K27me2/3,PHD结构域特异性识别并结合激活型组蛋白修饰H3K4me2/3,BAH-H3K27me2/3和PHD-H3K4me2/3这两种相互作用模块是独立发生的事件,而且是相互排斥的[10-11,16]。EBS和SHL一旦结合H3K27me2/3或H3K4me2/3后,就能够招募其他表观遗传组分或转录因子等形成转录抑制或激活复合物,进而抑制或激活靶基因或靶位点的转录,调控相关生物学过程[10-11,14-16]。
目前有关EBS和SHL功能与机制的报道均未涉及翻译后修饰的调控。前人通过酵母双杂交技术批量筛选发现,SHL与SUMO E2偶联酶SCE1(SUMO conjugating enzyme1)能够直接结合[17],暗示SHL可能存在SUMO化修饰。因此,本研究利用大肠杆菌体外SUMO化重建体系[18]和生化手段,检测 SHL及其同源蛋白EBS是否存在SUMO化修饰,并寻找其可能的SUMO化位点,为探讨SUMO化调控EBS和SHL的蛋白功能奠定基础。
1" " "材料与方法
1.1" "菌种与载体
基因克隆用的是大肠杆菌DH5α菌株,蛋白原核表达用的是大肠杆菌BL21(DE3)菌株,载体构建进行目的蛋白诱导表达用的是改造的商品化pET28a载体(Novagen),即在N端6×His标签之后引入了两个连续的FLAG标签(2×FLAG),改造成pET28a-6×His-2×FLAG,简写为pET28a-FLAG,引入FLAG标签的目的是方便后续蛋白免疫印迹检测。大肠杆菌体外SUMO化修饰重建体系由编码3个不同抗性基因的原核表达载体组成:pCDFDuet、pACYCDuet以及改造的pET28a-FLAG。pCDFDuet载体含有壮观霉素抗性基因(Spe+),引入了SUMO分子和SUMO E2偶联酶SCE1的全长蛋白编码序列,使得SUMO分子和E2分别带有N端6×His标签和C端S-Tag标签;pACYCDuet载体含有氯霉素抗性基因(Chl+),引入了组成SUMO E1激活酶全酶的两个亚基SAE1和SAE2的全长蛋白编码序列,使得SAE1、SAE2分别带有C端S-Tag标签和N端6×His标签;pET28a-FLAG含有卡那霉素抗性基因(Kan+),可引入待检测蛋白的全长或部分编码序列,如EBS和SHL,使其N端带有2×FLAG标签。
1.2" "实验试剂与药品
基因扩增、载体构建和定点突变使用诺唯赞的试剂(Vazyme)。基因扩增用的高保真酶Phanta Max Super-Fidelity DNA Polymerase(P505-d3),载体构建使用一步法克隆试剂盒ClonExpress Ultra One Step Cloning Kit(C112-02),菌落PCR使用诺唯赞的2×Rapid Taq Master Mix(P222-04),定点突变用的是Mut Express II Fast Mutagenesis Kit V2(C214-02),质粒提取试剂盒(DC201)和胶回收/DNA纯化试剂盒(DC301)也都购自诺唯赞,载体构建用的限制性内切酶如BamHI、StuI、EcoRI等购自NEB和Thermo Fisher。
大肠杆菌生长用的胰蛋白胨(Tryptone,LP0042)和酵母提取物(Yeast Extract,LP0021)购自OXOID公司;蛋白诱导表达诱导试剂IPTG (isopropyl-β-D-thiogalactopyranoside,I5502-10G)、PAGE胶促凝剂过硫酸铵(AP,A3678-100G)、蛋白还原剂DTT(DL-Dithiothreitol,D9163-25G)和蛋白酶抑制剂PMSF(P7626-5G)均购自sigma公司;缺乏EDTA的蛋白酶抑制剂片剂Cocktail(4693132001)购自Roche;Tween20(A600560-0500)、脱脂奶粉(A600669-0250)、NaCl(A501218-0001)等购自上海生工;ECL化学发光底物试剂盒购自Biosharp(BL520A)和Thermo Fisher(SuperSignal West Atto 超敏化学发光底物,A38555);PVDF膜(Immobilon® -P PVDF Membrane)购自Millipore(IPVH00010);其他常用的生化试剂和药品主要购自Sigma、Amresco、BBI、Solarbio、上海生工或上海国药等公司。
壮观霉素(Spectinomycin,S742-25G)、氯霉素(Chloramphenicol,C252-100G)、羧苄青霉素(Carbenicillin,C346-25G)、氨苄青霉素(Ampicillin,A116-25G)均购自PhytoTechnology;硫酸卡那霉素(Kanamycin Sulfate,K9316)购自lablead/兰博利德。
鼠源FLAG抗体Anti-DYKDDDDK 单克隆抗体购自Abmart公司(M20008L)和Sigma公司(F1804);山羊抗鼠的二抗Goat Anti-Mouse IgG-HRP(M21001L)购自Abmart公司;带HRP的抗His标签的鼠单克隆抗体(HRP Conjugated Anti His-Tag Mouse Monoclonal Antibody,CW0285M)购自康为试剂;抗S-Tag标签的鼠多克隆抗体 S-Tag Monoclonal Antibody(3B3)(YM3017)购自ImmunoWay。
1.3" "实验方法
1.3.1" "原核表达载体的构建
目的基因需要连接到改造的pET28a-FLAG载体上,使其带有N端2×FLAG标签方便后续免疫印迹检测。利用BamHI和StuI双酶切使pET28a-FLAG载体线性化,将目的基因扩增后得到的PCR产物进行纯化,然后与线性化的pET28a-FLAG载体用一步法重组克隆试剂盒(C112-02)重组,转化卡那霉素抗性平板(LB,50 mg/L Kan+)筛选阳性菌落,用2×Rapid Taq Master Mix(P222)进行阳性菌落鉴定,鉴定出来的阳性克隆送测序,确保目的基因序列完全无误且与FLAG标签正确融合。
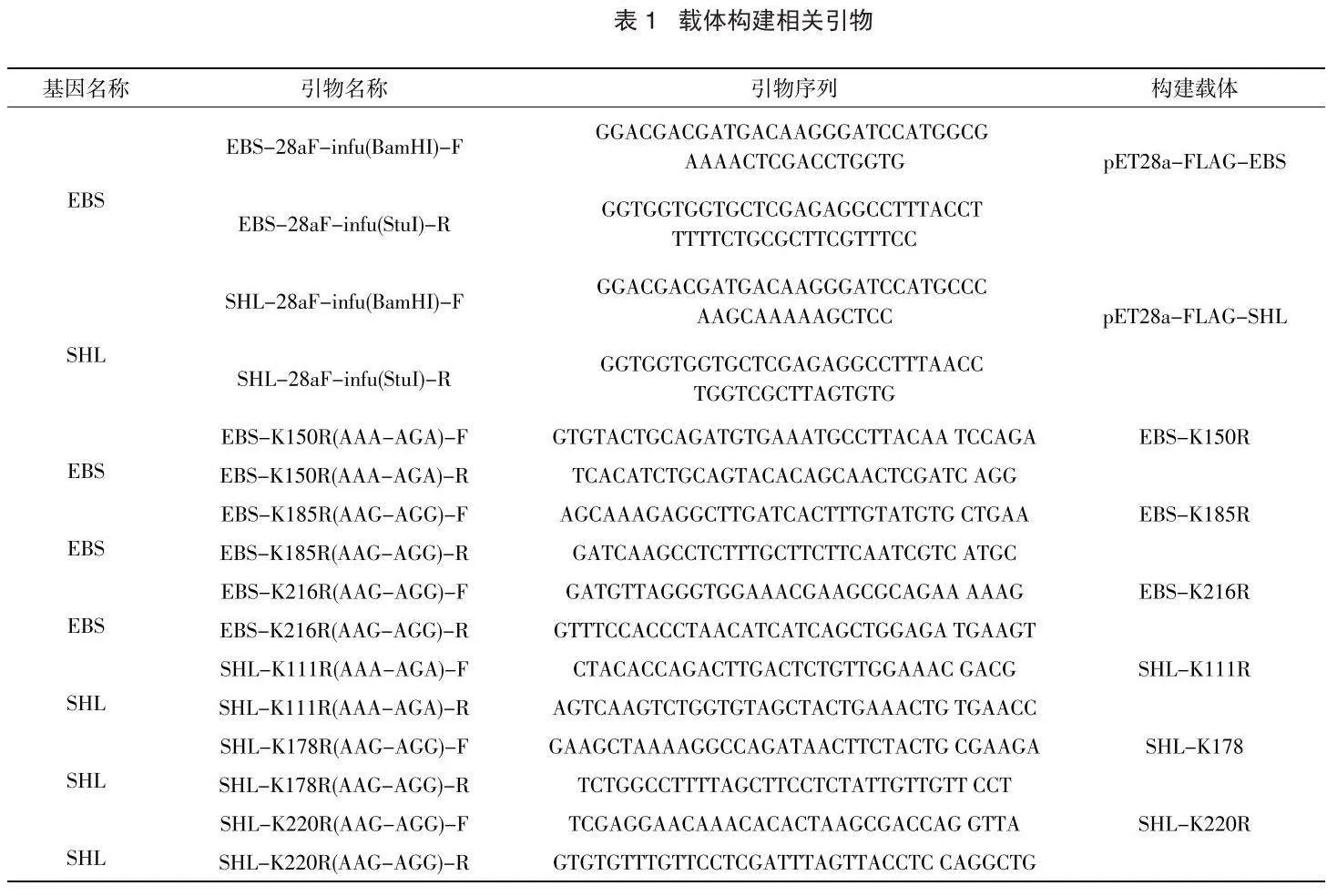
载体构建涉及到的引物合成和测序均由苏州金唯智生物科技有限公司完成,引物合成方式为tPAGE或hPAGE,引物序列及用途见表1。
1.3.2" "目的蛋白的诱导表达与提取
将构建好的融合表达载体pET28a-Target-FLAG转化BL21(DE3)大肠杆菌感受态,在卡那霉素抗性平板上(LB,50 mg/L Kan+)筛选阳性克隆,然后进行蛋白诱导表达。
挑取阳性单克隆过夜摇培,摇培起来的菌液按照体积比1:200进行二次摇培,两次摇培的条件均为37 ℃,200 r/min。待二次摇培菌液的OD600值在 0.6~0.8 范围时,加入终浓度为0.5 mM 的IPTG进行蛋白诱导表达,诱导条件为22 ℃,220 r/m,诱导12~18 h。收集添加和不添加 IPTG 的菌液各100 μL,离心收集菌体后加入100 μL蛋白提取裂解液(50 mM Tris-HCl(pH 7.6),150 mM NaCl,5 mM MgCl2,10%甘油,0.5 mM DTT,1 mM PMSF)重悬菌体,接着加入100 μL 蛋白上样缓冲液(2×SDS loading buffer),混合均匀后在100 ℃的金属浴中变性5~10 min,期间颠倒混匀几次,使细菌裂解充分。变性后的蛋白在冰上静置 5 min,使用4 ℃预冷的离心机于12000 r/min条件下离心 10 min,将上清转移至新的EP管中,上样、电泳、转膜,然后进行蛋白免疫印迹检测(Western blot,WB)和考马斯亮蓝染色(Coomassie Brilliant Blue,CBB)。
1.3.3" "蛋白电泳与WB检测
制备合适浓度的SDS-PAGE蛋白胶,浓度大小根据目标蛋白分子量大小而定,一般为8%或10%。待分离胶充分凝固后制备4%的浓缩胶,选取上样量适宜的制胶梳子,一般为10孔或15孔。待浓缩胶充分凝固后拔掉梳子,将制备好的蛋白胶置于含有1×蛋白电泳缓冲液的电泳槽中,清理干净浓缩胶胶孔中的残胶后上样。
样品在浓缩胶中首先以80 V的电压电泳,使所有样品浓缩成整齐的一条线,电泳约30 min待所有样品进入分离胶后,调整电压至120 V继续电泳,之后的电泳时长依据目的蛋白分子量大小决定。电泳结束后,用半干转膜仪选择合适的电压和时长转膜,一般为22 V,20~50 min,转膜时长依据目的蛋白分子量大小决定。转膜时需要注意PVDF膜需要提前用甲醇浸泡约1 min后才能使用,转膜时从下到上的顺序为:厚滤纸-PVDF膜-蛋白胶-厚滤纸,各层之间不能有气泡。
转膜结束后,用5% 的脱脂牛奶对PVDF膜进行封闭,封闭时间至少1 h。封闭结束后,用1×PBST漂洗3次,每次5 min,然后加入适当稀释浓度的一抗(如FLAG抗体,1:5000稀释)于4 ℃孵育过夜或室温孵育2 h。孵育结束后回收一抗以备后续重复使用,然后用 1×PBST 漂洗PVDF膜3次,每次10 min。漂洗结束后加入二抗,于室温继续孵育30~60 min,孵育结束后回收二抗备用,然后用1×PBST漂洗PVDF膜3~5次,每次5 min。漂洗结束后,将混合好的ECL发光底物均匀涂抹在PVDF膜上,在凝胶成像仪下扫描WB结果。
1.3.4" "大肠杆菌SUMO化修饰体系的介绍及改进
大肠杆菌SUMO化修饰体系主要包含有三个载体。
(1) pCDFDute-AtSUMO1/2/3/5 GG (AA)-AtSCE1∶
该载体含有拟南芥SUMO化发生的必备组分AtSUMO1/2/3/5和SUMO E2结合酶AtSCE1。GG代表有活性的SUMO分子,其C端带有两个连续的甘氨酸基序。当两个连续的甘氨酸(GG)都突变为丙氨酸(AA)后,相应SUMO分子的活性就完全丧失。同理,E2的活性位点为第94位的半胱氨酸(C94),当突变为丝氨酸(C94S)后E2活性丧失。
(2) pACYCDute-AtSAE1-AtSAE2∶该载体含有构成拟南芥SUMO E1激活酶全酶的两个亚基SAE1和SAE2。
(3) pET28a-His-Target:该载体含有待检测的目的基因,其N端含有6×His标签。为了方便后续WB检测,将其改造为pET28a-FLAG-Target。
以上3种质粒分别带有壮观霉素(Spe+)抗性基因、氯霉素(Chl+)抗性基因和卡那霉素(Kan+)抗性基因,便于后期转化BL21(DE3)感受态后利用抗生素筛选阳性克隆。
实验中发现,3种质粒同时转化BL21(DE3)感受态非常困难,为了提高转化效率,本研究使用了分步转化策略。首先将pACYDute-AtSAE1-AtSAE2转入BL21(DE3),并制备成感受态细胞,命名为E1-BL21;接着将pCDFDute-AtSUMO-AtSCE1、pCDFDute-AtSUMO(GG-AA)-AtSCE1、pCDFDute-
AtSUMO-AtSCE1(C94S)分别转入E1-BL21感受态,并进一步制备成新的感受态细胞,分别命名为E1-E2-SUMO-BL21、E1-E2-SUMO(AA)-BL21、E1-E2(C94S)-SUMO-BL21;最后将构建有目的基因的pET28a-FLAG-Target转入上述3种感受态细胞中,在同时含有3种不同抗性的培养基上筛选阳性克隆,之后进行目的蛋白的诱导表达和WB检测等实验。
1.3.5" "潜在 SUMO 化修饰位点的预测
SUMO 化修饰的预测工具常用的有3个: GPS-SUMO、SUMOplot、JASSA,其对应的网站依次为 http: //sumosp. biocuckoo. org/online.php/、 https://www.abcepta.com/tools/、http://www.jassa.fr/。3个网站的预测结果经常不一致,多数情况下存在差异甚至相差甚远,其准确性和可靠性都不是很高,只能作为参考,其预测位点是否精准往往需要借助质谱鉴定结合定点突变实验进行验证和分析。
2" " "实验结果
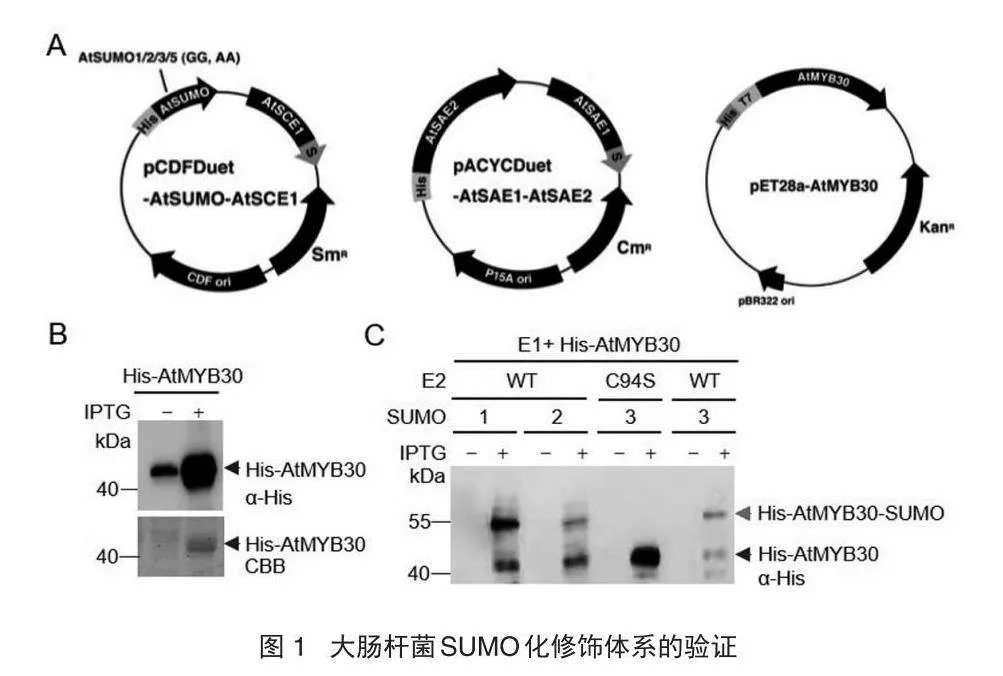
2.1" "大肠杆菌 SUMO 化重建体系的验证
前人已经在大肠杆菌中重建了拟南芥SUMO化修饰体系[18](图1A),为了验证其可靠性,本研究首先重复了前人的部分实验。结果发现,作为拟南芥中已知的SUMO化修饰底物,AtMYB30能够被正常诱导(图1B),且不论以SUMO1、SUMO2还是SUMO3为供体,在AtMYB30本底蛋白上方均出现了一条高分子量的条带,该条带即为AtMYB30的SUMO化条带(图1C)。但当E2活性位点突变后(C94S),高分子量的修饰条带完全消失(图1C)。证明AtMYB30的确存在SUMO化修饰,同时也验证了大肠杆菌SUMO化修饰重建体系的特异性和可靠性。
2.2" "EBS和SHL在大肠杆菌 SUMO 化体系中存在SUMO化修饰
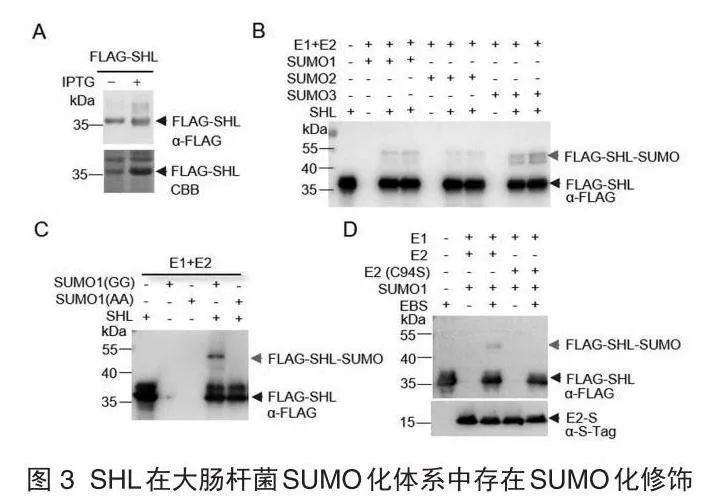
前人以SUMO E2偶联酶SCE1和SUMO蛋白酶ESD4(Early in Short Days4)为诱饵筛选酵母双杂交文库,发现SHL能够直接结合SCE1[17],为SHL存在SUMO化修饰提供了线索和可能性。由于EBS与SHL高度同源,因此本研究用大肠杆菌 SUMO 化重建体系一起进行了检测。
将EBS和SHL分别构建到pET28a-FLAG载体上,蛋白诱导表达和WB结果显示, EBS和SHL都能够正常表达,且分子量都略高于35 kDa,符合预期大小(图2A和图3A)。将pET28a-EBS-FLAG和pET28a-SHL-FLAG分别导入同时含有E1、E2和不同亚型SUMO分子的大肠杆菌SUMO化体系中,发现在EBS和SHL本底蛋白上方均出现了一条高于本底蛋白的特异性条带,分子量介于40~55 kDa之间(图2B和图3B),表明这些高分子量条带很可能为EBS和SHL的SUMO化条带。为了确定高分子量条带为EBS和SHL的SUMO化条带,将pET28a-EBS-FLAG和pET28a-SHL-FLAG分别导入含有SUMO分子活性缺失突变体(SUMO GG-AA)或E2活性缺失突变体(E2 C94S)中进行检测,结果发现,高分子量SUMO化条带在这些活性缺失突变体中都完全消失(图2C、图2D、图3C、图3D)。以上结果表明,EBS和SHL的确存在SUMO化修饰,同时也再次佐证了大肠杆菌SUMO化重建体系的特异性和可靠性。
由于一个成熟SUMO分子的分子量大约为11~12 kDa,从SUMO化条带与本底蛋白的分子量差值推断,EBS和SHL的SUMO化很可能都是单位点的单SUMO化修饰。接下来就需要寻找EBS和SHL具体的SUMO化位点。
2.3" "EBS和SHL 的蛋白结构与SUMO化位点预测
EBS和SHL隶属于染色质阅读器蛋白家族成员,都有N端BAH和C端PHD两个功能结构域,BAH和PHD分别特异性识别并结合抑制型组蛋白修饰H3K27me2/3和激活型组蛋白修饰H3K4me2/3[10-11,16]。EBS和SHL都存在不同的转录本,但不同转录本的蛋白编码产物都包括完整的BAH和PHD两个功能结构域。EBS和SHL主要转录本的蛋白编码产物的长度分别为224aa和228aa,EBS和SHL的蛋白结构和各结构域大小和位置如图4A所示。
常用的SUMO化预测工具有3个,即GPS-SUMO、SUMOplot和JASSA。将EBS和SHL的全长蛋白序列分别用这3个SUMO化预测工具综合分析发现,EBS和SHL都存在SUMO化修饰,且最有可能发生SUMO化的位点也都各有3个:EBS是K150、K185和K216,SHL是K111、K178和K220(图4B)。在EBS 3个可能的SUMO化位点中,K150和K185均位于EBS的PHD结构域内部,K216则位于PHD结构域后面的区域(图4B)。在SHL的3个预测位点中,K111位于SHL的BAH结构域内部,K178位于SHL的PHD结构域内部,K220则位于其PHD结构域后面的区域(图4B)。所有这些位点中到底哪个是真实的SUMO化位点需要定点突变结合WB实验进行验证和分析。
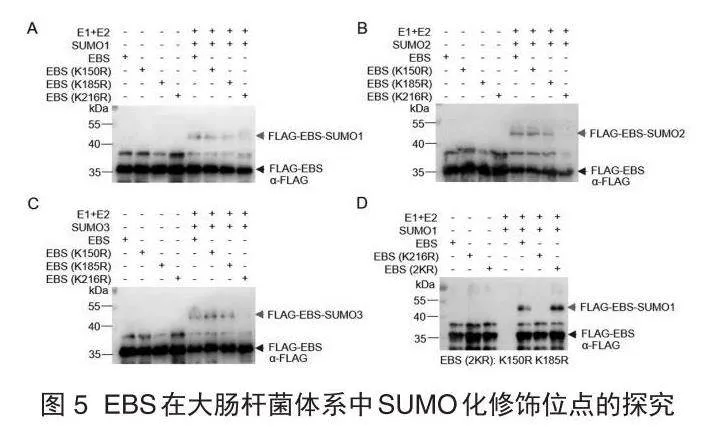
2.4" "K216是 EBS唯一的SUMO化修饰位点
为探究EBS的SUMO化位点,将K150、K185和K216 3个精氨酸残基分别定点突变为赖氨酸残基,即分别突变为K150R、K185R和K216R,以模拟这些位点的SUMO化修饰缺失突变体,然后利用大肠杆菌SUMO化重建体系进行分析。
首先以SUMO1为供体进行研究。发现K150和K185分别点突变后,在EBS本底蛋白上方仍然存在一条特异性高分子量SUMO化条带,位置和大小与无突变的EBS对照完全一致, 而K216R的点突变使得EBS本底蛋白上方的高分子量条带消失(图5A),可见K150R和K185R的点突变不影响EBS的SUMO化修饰,K216R的点突变导致EBS的SUMO化修饰丧失。结果表明,K150和K185都不是EBS真实的SUMO化位点,而K216则可能是EBS的关键SUMO化位点。接着以SUMO2和SUMO3分别为供体开展类似实验,得到与以SUMO1为供体类似的结果,即只有K216R能够造成EBS的SUMO化条带消失,其他两个位点的点突变则不影响(图5B、图5C)。最后,将K150R和K185R同时引入EBS,构建成EBS-2KR的双点突变(K150R/K185R),以无突变的EBS和K216R点突变的EBS为对照,利用大肠杆菌SUMO化重建体系重复上述实验。结果发现,2KR也不影响EBS的SUMO化修饰带型和强弱,只有K216R能够造成EBS的SUMO化条带消失(图5D)。以上实验证实,K216是EBS唯一且关键的SUMO化位点。
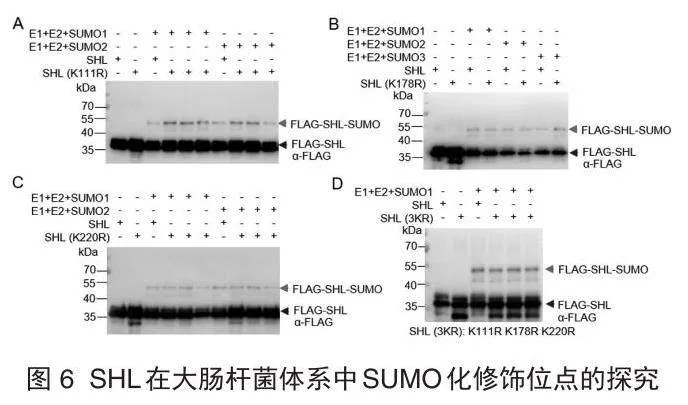
2.5" "探究SHL的SUMO化修饰位点
与探究EBS的SUMO化修饰位点类似,本研究也用同样的手段探究了SHL可能的SUMO化位点。将K111R、K178R和K220R 3个点突变分别导入SHL,发现不论以哪种SUMO分子为供体,这些单点突变都不影响SHL的SUMO化修饰情况(图6A、图6B、图6C),表明这些位点可能不是SHL真实的SUMO化位点。由于SUMO化可能存在修饰位点冗余,于是将这3个点突变全部导入SHL,构建成SHL-3KR,即K111R/K178R/K220R。同时选取3个独立的SHL-3KR单克隆一起进行检测。结果发现,3个独立的3KR单克隆都不影响SHL的SUMO化带型和强弱(图6D)。以上结果表明,K111、K178和K220都不是SHL的SUMO化位点,SHL的SUMO化位点有待继续研究。
3" " "结论与讨论
EBS和SHL是一类同时含有BAH和PHD结构域的植物特有的染色质阅读器蛋白,通过特异性识别并结合H3K27me2/3和H3K4me2/3这两类功能相互拮抗的组蛋白修饰,参与调控植物开花时间[10-12]、种子休眠与萌发[13]、高温介导的萌发抑制[14]、植物根的发育[15]等重要生物学过程。这些重要功能和机制的解析主要基于EBS和SHL基因缺失突变体的发现与表型分析、遗传学实验、生化分析、结构解析、转录组和染色质免疫共沉淀偶联的高通量测序(ChIP-seq)等组学数据分析等。目前有关EBS和SHL的报道都是基于基因缺失或功能结构域存在缺陷的证据,未见有蛋白质翻译后修饰调控其功能的报道。
蛋白质翻译后修饰通过影响蛋白质构象、活性、稳定性、相互作用、亚细胞定位等方式调控蛋白功能,EBS和SHL是否存在翻译后修饰以及这些修饰是否影响、如何影响其功能未知。前人通过酵母双杂交实验高通量筛选发现,SUMO E2偶联酶SCE1与SHL能够直接结合[17],为SHL可能存在SUMO化修饰提供了线索。
为了探究SHL及其同源蛋白EBS是否存在SUMO化修饰,利用大肠杆菌SUMO化重建体系[18]展开研究。结果发现,SHL和EBS都存在SUMO化修饰,且都有一条特异性SUMO化条带,根据分子量推测二者均为单位点的单SUMO化修饰。进一步通过定点突变结合生化实验证实,K216R是EBS发生SUMO化修饰的唯一关键位点,而SHL的SUMO化位点有待进一步研究,但至少可以排除K111、K178和K220这3个位点。未来需要借助质谱技术结合定点突变继续研究SHL的SUMO化位点。
[参考文献]
[1] Miura K,Jin JB,Hasegawa PM. Sumoylation,a post-translational regulatory process in plants [J]. Current Opinion in Plant Biology, 2007,10(5):495-502.
[2] G.Wilson V. SUMO Regulation of Cellular processes [M]. Springer Nature,2017:1-412.
[3] Augustine RC,Vierstra RD. SUMOylation: re-wiring the plant nucleus during stress and Development [J]. Current Opinion in Plant Biology, 2018,45(Pt A):143-154.
[4] Rytz TC,Miller MJ,McLoughlin F,et al. SUMO ylome Profiling Reveals a Diverse Array of Nuclear Targets Modified by the SUMO Ligase SIZ1 during Heat Stress [J]. Plant Cell,2018,30(5):1077-1099.
[5] Han DL,Lai JB,Yang CW. SUMOylation: A critical transcription modulator in plant cells [J]. Plant Science, 2021,310(110987).
[6] Singh M,Singh A,Yadav N,et al. Current perspectives of ubiquitination and SUMOylation in abiotic stress tolerance in plants [J]. Frontiers in Plant Science,2022,13(993194).
[7] Jmii S,Cappadocia L. Plant SUMO E3 Ligases: Function,Structural Organization,and Connection With DNA [J]. Frontiers in Plant Science, 2021,12(652170).
[8] Matic I,Schimmel J,Hendriks IA,et al. Site-Specific Identification of SUMO-2 Targets in Cells Reveals an Inverted SUMOylation Motif and a Hydrophobic Cluster SUMOylation Motif [J]. Molecular Cell, 2010,39(4):641-652.
[9] Hendriks IA,Lyon D,Young C,et al. Site-specific mapping of" the human SUMO proteome reveals co-modification with phosphorylation [J]. Nat Struct Mol Biol, 2017,24(3):325-336.
[10] Yang ZL,Qian SM,Scheid RN,et al. EBS is a bivalent histone reader that regulates floral phase transition in Arabidopsis [J]. Nature Genetics, 2018,50(9):1247-1253.
[11] Qian SM,Lv XC,Scheid RN,et al. Dual recognition of H3K4me3 and H3K27me3 by a plant histone reader SHL [J]. Nature Communications, 2018,9(1):2425.
[12] López-González L,Mouriz A,Narro-Diego L,et al. Chromatin-Dependent Repression of the Floral Integrator Genes Involves Plant Specific PHD-Containing Proteins [J]. Plant Cell , 2014,26(10):3922-3938.
[13] Narro-Diego L,López-González L,Jarillo JA,et al. The PHD-containing protein EARLY BOLTING IN SHORT DAYS regulates seed dormancy in Arabidopsis [J]. Plant Cell and Environment, 2017,40(10):2393-2405.
[14] Li P,Zhang QL,He DN,et al. AGAMOUS-LIKE67 Cooperates with the Histone Mark Reader EBS to Modulate Seed Germination under High Temperature [J]. Plant Physiology,2020,184(1):529-545.
[15] Huang C,Wang D,Yang YQ,et al. Suppressor of Frigida 4 cooperates with the histone methylation reader EBS to positively regulate root development [J]. Plant Physiology,2024:321.
[16] Li ZC,Fu X,Wang Y Z,et al. Polycomb-mediated gene silencing by the BAH-EMF1 complex in plants [J]. Nature Genetics,2018,50(9):1254-1261.
[17] Elrouby N,Coupland G. Proteome-wide screens for small ubiquitin-like modifier (SUMO) substrates identify Arabidopsis proteins implicated in diverse biological processes [J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(40):17415-17420.
[18] Okada S,Nagabuchi M,Takamura Y,et al. Reconstitution of Arabidopsis thaliana SUMO pathways in E. coli: functional evaluation of SUMO machinery proteins and mapping of SUMOylation sites by mass spectrometry [J]. Plant amp; Cell Physiology,2009,50(6):1049-1061.
责任编辑" "吕荣荣
[收稿日期]" " 2024-09-27
[基金项目]" "河北省自然科学基金项目(C2022205017);河北师范大学博士基金项目(L2019B23)
[作者简介]" "魏建(1994- ),男,河北师范大学硕士,研究方向:SUMO化修饰。
[通信作者]" "韩永峰(1982- ),男,博士,河北师范大学副教授,研究方向:SUMO化修饰。

