玉米穗行数分子遗传研究进展


摘要:玉米作为全球重要的粮食作物之一,其产量高低直接关系到全球粮食安全和农业经济发展。在玉米产量构成因素中,穗行数是一个至关重要的因素,直接影响玉米的籽粒数量和单产水平,因此,对玉米穗行数的研究具有重要的理论价值和实践意义。从玉米雌穗的发育进程、穗行数与产量及驯化的关系、QTL定位、基因克隆及基因编辑技术在玉米穗行数遗传改良中的应用5个方面对玉米穗行数遗传基础进行了综述,同时结合现在的研究现状和存在问题,对未来研究方向进行展望,以期为玉米高产分子育种和生产提供理论参考。
关键词:玉米;产量;穗行数;QTL定位;基因克隆;基因编辑;分子育种
Progress in Molecular Genetics Studies of the Kernel Row Number in Maize
SU Yujie1,LIU Pingli2,SONG Junfeng1,LU Hongwei1,WANG Bangtai1,QIN Guiwen1,
XU Ling3,GAO Keke4,ZHANG Shuli5,ZHANG Xiaochun1
(1Hebi Academy of Agricultural Sciences,Hebi 458031,Henan;2Hebi Agriculture and Rural Bureau,Hebi 458030,Henan;
3Henan Modern Seed Industry Co.,Ltd.,Zhengzhou 450000;4Sinograin Zhumadian Depot Co.,Ltd.,Zhumadian 463000,
Henan;5Hebi Agricultural and Rural Development Service Center,Hebi 458031,Hehan)
在我国粮食作物中,玉米种植面积及产量均居首位,其中的70%用于饲料生产,因此,不断提高玉米产量对我国的粮食安全和经济发展都有重要影响。然而,近年来,由于耕地面积减少、作物结构调整、水资源短缺等因素的限制,我国玉米的种植面积开始呈现下降的趋势,要依靠增加播种面积来提高我国玉米产量的空间已经非常有限,在有效耕地面积难以增加的情况下,提高单产是保障我国粮食生产的根本出路。玉米单位面积产量的三大构成因子为单位面积穗数、穗粒数和百粒重[1]。杨坤等[2]研究结果表明,增加玉米穗行数可以提高产量,通过选育一定范围内的穗行数是提高玉米产量的有效途径之一。穗行数作为重要的产量构成因子之一,在玉米的驯化和改良过程中,与玉米的祖先大刍草、农家种和现代杂交种相比,穗行数是穗部形态方面最显著的变化之一。因此,对穗行数进行深入的遗传分析不仅有助于推动高产玉米育种的发展,还能为玉米的驯化历程以及花序结构的演变提供新的见解。
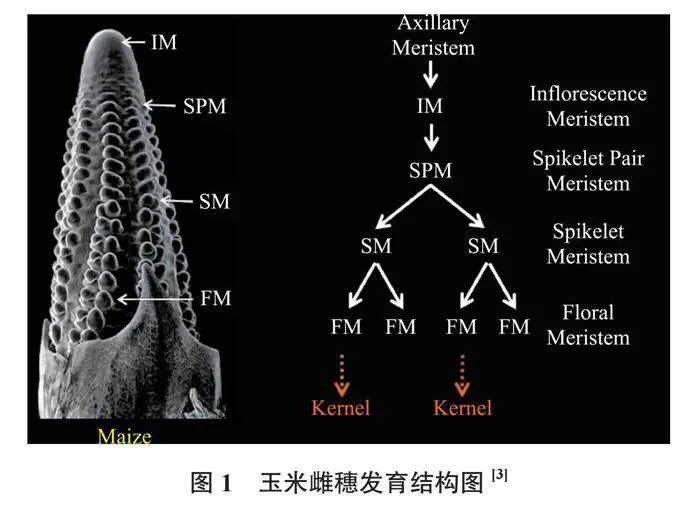
1 玉米雌穗的发育
穗花的发育是玉米由营养生长向生殖生长转变的主要表现,大约在6~8叶期完成,雄穗和雌穗的发育都需要经过不同时期的细胞变化与功能转变,首先雄穗最先开始分化发育,其次是雌穗,雄穗是从顶端分生组织开始进行细胞分裂形成花序分生组织(IM,Inflorescence meristem),而雌穗的花序分生组织是由叶腋处的腋生分生组织(AM,Axillary meristem)分化形成的,然后IM经过一定时期的细胞分裂与形态转变发育成一系列的成对小穗分生组织(SPM,Spikelet-Pair meristem),SPM经过细胞分裂发育成2个小穗分生组织(SM,Spikelet meristem),这样的发育方式决定了玉米偶数穗行的特性,最后SM经过细胞分裂形成2个小花分生组织(FM,Floral meristem),分别是上位花和下位花,只有上位花发育形成籽粒,下位花则败育退化
(图1)[3-6]。
可见,雌穗发育过程中IM的长度、宽度和活性最重要,它决定了SPM的数目和持续分化SPM的能力,是玉米穗行数最终形成的关键因素[7]。所以了解玉米雌穗的形态建成与分子生物学机制,对玉米育种和产量提高具有重要意义。
图1 玉米雌穗发育结构图[3]
2 玉米穗行数与产量及驯化的关系
穗行数是玉米种质改良的重要目标性状之一,同时也是玉米重要的产量构成因子,适宜的穗行数可以提高产量。徐宝峰等[8]研究表明穗行数属于数量性状,广义遗传力较高且与玉米产量呈显著的正相关,这表明对穗行数的遗传改良是提高玉米产量的一种有效途径;另一方面,玉米是一种驯化作物,约9000年前,玉米由墨西哥的大刍草(Zea mays ssp.parviglumis)驯化而来[9-10]。整个驯化过程使现代玉米的植株和果穗形态相比于原始祖先大刍草发生了巨大的变化。随着分子生物学的发展,研究人员克隆了一些主效的驯化基因,例如,基因TGA1(Teosinte glume architecture1)决定籽粒的颖壳有无[11];基因TB1(Teosinte branched1)在现代玉米中表达量高,使现代玉米没有了分蘖[12];基因GT1(Grassy tillers1)在现代玉米中的高表达使玉米同一节上只有一个变大的雌穗[13]。除了这些性状,雌穗的形态,包括穗行数的多少也是玉米重要的驯化性状(图2)。在玉米驯化过程中,其整体形态结构(尤其在花序结构上)发生了很大的改变[14],现代玉米从小穗到大穗的转变伴随着穗行数(KRN,Kernel row number)的急剧增加[15],大刍草有2列穗行,而农家种和现代玉米杂交种的穗行数明显变多,这是现代玉米富集大量与穗行数相关的优异等位基因的结果。但到目前为止,已经定位的QTL有数百,但是已经克隆的仅有3个,数量极少。因此,有必要对穗行数进行更为深入的遗传结构解析,定位和克隆更多的QTL,为玉米产量性状的遗传改良提供靶标基因。
图2 玉米和大刍草对比图
3 玉米穗行数QTL的研究进展
现代栽培玉米的穗行数集中在 8~20行,是一个复杂的数量性状。尽管已经对玉米雌穗发育和穗行数形成的遗传调控有了一定的了解,但大部分通过突变体材料所克隆的雌穗穗行数发育相关基因,其突变体都携带有雌穗扁化、畸形或穗行数紊乱等不利特征,很难直接用于生产实践。QTL定位是剖析复杂数量性状遗传结构的经典方法,利用双亲连锁群体和自然群体等开展连锁分析和关联分析,能够有效挖掘控制玉米穗行数数量性状位点的自然变异。利用QTL定位手段挖掘玉米穗行数的自然变异,不仅可以进一步了解玉米雌穗发育和穗行数形成的遗传调控基础,还可以实现对雌穗分生组织和穗行数进行微调操作,从而有助于将有利的遗传变异基因直接应用于玉米的遗传改良中。
3.1 连锁分析在穗行数QTL定位中的应用 玉米穗行数为典型的数量性状,因此采用分子标记技术与连锁作图是进行穗行数QTL定位和解析玉米穗行数遗传机制的有效方法之一。即通过生物统计学方法,分析分离群体穗行数QTL与分子标记之间的连锁关系,进而确定QTL的具体位置,并估计其效应[16]。目前,已有上百个穗行数QTL被相继报道,但是不同研究中所用到的亲本材料、群体类型和规模、标记密度不尽相同,定位的结果也存在较大差异。这表明穗行数的遗传变异和主效遗传调控位点受到遗传背景的强烈干预。如1994年Veldboom等[17]利用150个F2:3家系进行了玉米穗行数遗传定位研究,结果共鉴定到11个QTL与穗行数有关。2006年Yan等[18]利用266个F2:3家系进行了玉米穗行数遗传定位研究,结果在2个环境条件下共定位到9个与穗行数相关的QTL。2007年Ma等[19]
利用一套RIL群体在全基因组上鉴定到13个穗行数QTL。2010年Tang等[20]率先在玉米QTL定位中引入永久F2群体(IF2),共鉴定到11个穗行数QTL位点。2011年Lu等[21]利用自交系掖478和丹340开发的一套F2:3群体,在7个环境中检测到13个穗行数QTL。2015年Yang等[22]选用一个4行玉米材料与B73杂交构建F2:3群体,检测到3个穗行数QTL。2014年Li等[23]利用一套单片段导入系对玉米穗行数进行了遗传定位研究,结果共鉴定到11个QTL与穗行数有关。2016年Liu
等[24]利用300个家系F2群体,共检测到4个穗行数QTL。2017年Zhang等[25]利用365个RILs家系对玉米穗行数进行了遗传定位研究,结果定位到10个与穗行数相关的QTL。2021年赵强等[26]采用高密度SNP标记对118份F2:3家系进行了连锁分析,结果鉴定到4个与穗行数相关的QTL位点。2022年Fei等[27]对NAM群体的1617个RILs家系进行了连锁和关联分析,结果鉴定到2个新的QTL位点,分别是qKRN4.2和Qkrn9.1。2022年赵鹏斌等[28]对283份重组自交系(RIL)行了连锁分析,结果筛选到19个与玉米穗行数有关的QTL。2023年聂蕾等[29]利用150份F2、F2:3家系对玉米穗部性状进行了连锁分析,结果在2个环境条件下筛选到46个QTL,其中19个QTL与穗行数相关。
以上研究所利用的群体都是由现代玉米自交系发展而来的群体,驯化降低了现代玉米的遗传多样性,优良的等位基因在驯化过程中可能丢失,这其中也包含一些对玉米遗传改良有效的优良位点,近些年来,研究者开始利用玉米与大刍草杂交构建的群体进行穗行数的遗传定位,例如,Liu等[30]利用10份大刍草材料(parviglumis)与一个共同亲本B73构建了一套包含928个近等基因系(BC4)的NAM(NAM,Nested association mapping)群体,在1、2、4、5号染色体各鉴定到1个控制穗行数的QTL;Chen等[31]利用5份大刍草材料(4份parviglumis与1份mexicana)与一个共同亲本W22构建了一套包含1257个RILs(BC1S4)的NAM群体,在全基因组共鉴定到24个穗行数QTL。
玉米穗行数的遗传受多基因控制,且存在复杂的遗传互作。近年来,通过分子标记和基因组学技术的应用,研究人员已经定位到一些与穗行数相关的基因和QTL。这些基因和QTL的发现,为玉米穗行数的遗传改良提供了重要的分子标记和候选基因。
3.2 关联分析在穗行数QTL定位中的应用 关联分析是利用自然变异群体研究作物复杂农艺性状和表型变异的主要方法之一,它以连锁不平衡为基础,通过分子标记构建的遗传图谱对目标农艺性状进行关联分析;自然关联群体的遗传基础比较宽泛,因此可同时对同一基因座的多个等位基因进行检测分析,这是双亲连锁群体无法实现的,因此一些研究人员利用关联分析的方法对玉米穗行数这个产量性状进行了研究和定位[32-34]。2011年Brown
等[35]对NAM群体的4892个RILs家系进行了关联分析,结果筛选到261个QTL与穗行数紧密相关。2014年张焕新等[36]对203份材料进行了关联分析,结果鉴定出9个与穗行数显著关联的SNP,其中8个SNP位于已定位的数量性状座位(QTL)区间内。2015年Liu 等[37]利用关联分析对513份材料进行了定位分析,结果筛选到31个SNPs与穗行数相关。2020年An等[38]利用关联分析对639份材料进行了定位研究,结果在多环境条件下筛选到7个一致性高的SNPs。2021年Ma等[39]利用关联分析对309份材料进行了穗部性状关联分析,定位到8个与穗行数相关的SNP。
与连锁分析相比关联分析的应用提高了玉米农艺性状研究的精度和深度,但是在应用的时候关联分析也会存在一定的局限性,比如群体结构、遗传漂变和自然选择等都会导致假阳性结果的出
现[40-42]。采用关联分析与连锁分析相结合,可以更快筛选鉴定QTL位点,采用区域关联分析缩小QTL区间,更快明确候选基因,2015年Liu等[43]利用该方法在KRN4区间内进行区域关联分析,不仅定位到了UB3的功能位点,还检测出4个与穗行数显著关联的位点。
4 玉米穗行数基因的研究进展
由于基因组学和生物技术的快速发展,研究人员通过突变体克隆了一些玉米调控穗行数的基因,如转录因子(tsh4、ub2、ub3基因)、激酶(td1、fea2和fea3)、小RNA(cg1和ts4基因)、romasa类家族基因(ra1、ra2和ra3)、其他酶类(GTP酶、Gro/Tup1酶、短肽)等,这些基因在CLAVATA-WUSCHEL(CLV-WUS)负反馈循环信号调控途径、激素调控途径、microRNA调控途径和RAMOSA调控途径中发挥着重要作用[7]。但是这些突变体表型都过于极端,常伴随不利表型,难以利用。
利用双亲构建的定位群体或关联群体克隆的穗行数QTL也非常稀少。尽管已经定位了大量的QTL位点,但是调控穗行数的QTL克隆方面进展缓慢。截至目前,已经克隆的QTL有3个(表1),一个是华中农业大学张祖新课题组利用连锁群体克隆到了玉米穗行数的主效QTL——KRN4,KRN4被定位到一个3kb的基因间区,该区间位于UB3基因的下游60kb,负调控籽粒行数;3kb区间内一个1.2kb的PAV变异和UB3基因的第3个外显子上一个SNP的变异加性调控KRN,并且这2个位点都在玉米改良中受到选择[43]。在水稻和玉米中,UB3基因表达蛋白结合在参与细胞分裂途径的基因上,编码花序相关转录因子。在玉米中,UB3蛋白还参与生长素途径和CLV-WUS途径。另外一个是来自中国农业大学林中伟课题组的KRN1,结合基于图谱的克隆和关联分析鉴定到另外一个穗行主效QTL——KRN1,克隆发现,该基因是已经克隆的基因ids1/ts6,该基因编码与小麦关键驯化基因产物同源的AP2结构域蛋白;过表达KRN1能够增加小穗分生组织的数量,进而增加籽粒行数,DNA序列分析结果表明,KRN1在玉米和小麦育种过程中受到选择,RNA-seq分析结果显示,KRN1参与调控小穗分生组织发育的多条途径,能够影响到雌穗小花发育相关的多个关键基因,如fea3、fea4和ra3等[44]。杨晓红课题组以穗行数多的材料B73和穗行数少的大刍草渐渗系MT-6为亲本构建重组自交系群体,结果在2号染色体上鉴定到一个主效QTL——qKRN2,通过图位克隆的方法克隆了该QTL的功能基因KRN2,进一步研究发现在玉米中降低KRN2的表达量,可以使玉米的穗行数增加[45-46]。
5 基因编辑技术助力玉米穗行数遗传改良
基因编辑技术(CRISPR-Cas9)是当前农业生物遗传改良过程中的一项颠覆性技术。已经利用基因编辑技术在玉米、番茄、水稻、大豆等作物中开展了品种和品质的遗传改良,高油酸的大豆已经在美国上市,基因编辑高产糯玉米和抗旱玉米已经研发成功,这些基因编辑产品的共同特点都是对单个基因进行改造。在穗行数的遗传改良方面,2021年冷泉港David Jackson研究组利用基因编辑技术,对2个CLAVATA3同源基因ZmCLE7和ZmFCP1的调控区域(启动子)进行基因编辑,创制出ZmCLE7和ZmFCP1的优良等位基因。研究结果表明,创制出的优良等位基因能够精细调控ZmCLE7和ZmFCP1的表达水平,数个启动子编辑等位基因可使表达量水平降低45%~69%,平衡了其在花序发育中的功能,最终强化玉米产量性状表现。产量测定数据表明,编辑后的新材料在自交系背景及杂交种背景下,均能显著提升产量。这些等位基因对产量的提升,主要体现在果穗增粗、穗行数增加、单穗粒数增加,与对照相比,在穗行数上的提升十分显著,提升了约6行,且粒重未出现显著变化,最终单穗产量提升约26%[47] 。这些研究结果表明,通过克隆出产量相关性状的基因位点,利用基因编辑技术对这些基因进行精准编辑,能够鉴定出显著增产的最优等位基因。
6 玉米穗行数研究存在的问题及未来展望
近年来,随着玉米研究的深入,玉米穗行数研究取得了显著成果。在遗传育种方面,研究者通过分子标记技术、基因定位等方法,发现了多个与玉米穗行数相关的基因,为玉米穗行数遗传改良提供了重要材料。在栽培生理方面,研究者揭示了玉米穗行数形成的分子机理和环境因素影响,为制定玉米高产优质栽培技术提供了理论依据。
尽管在玉米穗行数研究方面取得了一定的成果,但目前仍存在一些问题。首先,玉米穗行数遗传规律尚未完全明确,部分基因功能和调控机制尚不清晰。其次,尽管研究者已经发现了多个与玉米穗行数相关的基因,但将这些基因应用于实际育种中的效果并不理想。此外,栽培环境中气象、土壤等条件的变化对玉米穗行数的影响规律尚不明确,缺乏针对不同生态区的玉米穗行数调控技术。
针对上述问题,未来玉米穗行数研究应从以下几个方面展开:首先加强玉米穗行数遗传规律研究,发掘新的基因资源,利用高通量测序、基因组学和基因编辑等先进技术,深入研究玉米穗行数遗传规律,挖掘新的调控基因,为育种提供更多优质资源。其次揭示基因与环境互作机制,提高育种效果,深入研究玉米穗行数相关基因与环境因素的互作关系,为基因型与环境的协同育种提供理论支持,综合考虑穗行数与其他产量构成因素的关系。同时在玉米高产育种中,要综合考虑穗行数与其他产量构成因素的关系,通过多性状综合选择育种策略,培育出高产、优质、抗逆性强的玉米新品种。最后,加强生物学、生态学、计算机科学等领域的跨学科交叉合作,利用大数据、人工智能等先进技术,为玉米穗行数研究提供新思路。
参考文献
[1]谭巍巍,李永祥,王阳,刘成,刘志斋,彭勃,王迪,张岩,孙宝成,石云素,宋燕春,杨德光,王天宇,黎裕.在干旱和正常水分条件下玉米穗部性状QTL分析.作物学报,2011,37(2):235-248
[2]杨坤,段正凤,李福星,陈芝能.玉米穗部性状及产量间的通径分析.农技服务,2016,33(1):69-71
[3] Bennetzen J L,Hake S C.Handbook of maize:its biology.New York:Springer,2009
[4]白胜双,彭勃,王超楠.玉米穗行数遗传基础的研究进展.天津农业科学,2016,22(5):8-10
[5]安怡昕.玉米穗行数主效位点qKRN5.04的精细定位与候选基因挖掘.北京:中国农业科学院,2018
[6]安怡昕.玉米穗行数的遗传解析及主效位点的候选基因挖掘与功能分析.北京:中国农业科学院,2022
[7]殷芳冰,王成,龙艳,董振营,万向元.玉米雌穗性状遗传分析与形成机制.中国生物工程杂志,2021,41(12):30-46
[8]徐宝峰,许正学,邬生辉,曲海涛,王越人,李光发.玉米穗行数的遗传研究.农业科技通讯,2019(2):85-86
[9] Piperno D R,Flannery K V.The earliest archaeological maize(Zea mays L.)from highland Mexico:new accelerator mass spectrometry dates and their implications.Proceedings of the National Academy of Sciences,USA,2001,98(4):2101-2103
[10] Matsuoka Y,VIouroux Y,Goodman M M,Sanchez G J,Buckler E,Doebley J.A single domestication for maize shown by multilocus microsatellite genotyping.Proceedings of the National Academy of Sciences,USA,2002,99(9):6080-6084
[11] Wang H,Nussbaum-Wagler T,Li B L,Zhao Q,Vigouroux Y,Faller M,Bomblies K,Lukens L,Doebley J F.The origin of the naked grains of maize.Nature,2005,436(7051):714-719
[12] Studer A,Zhao Q,Ross-Ibarra J,Doebley J.Identification of a functional transposon insertion in the maize domestication gene tb1.Nature Genetics,2011,43(11):1160-1163
[13] Wills D M,Whipple C J,Takuno S,Kursel L E,Shannon L M,Ross-Ibarra J,Doebley J F.From many,one:genetic control of prolificacy during maize domestication.Plos Genetics,2013,9(6):e1003604
[14] Iltis H H.Homeotic sexual translocations and the origin of maize
(Zea mays,Poaceae):A new look at an old problem.Economic Botany,2000,54(1):7-42
[15] Doebley J.The genetics of maize evolution.Annual Review of Genetics,2004,38(38):37-59
[16]席章营,朱芬菊,台国琴,李志敏.作物QTL分析的原理与方法.中国农学通报,2005,21(1):88-92,99
[17] Veldboom L R,Lee M,Woodman W L.Molecular marker-facilitated studies in an elite maize population:I.Linkage analysis and determination of QTL for morphological traits.Theoretical and Applied Genetics,1994,88(1):7-16
[18] Yan J B,Tang H,Huang Y Q,Zheng Y L,Li J S.Quantitative trait loci mapping and epistatic analysis for grain yield and yield components using molecular markers with an elite maize hybrid.Euphytica,2006,149(1):121-131
[19] Ma X Q,Tang J H,Teng W T,Yan J B,Meng Y J,Li J S.Epistatic interaction is an important genetic basis of grain yield and its components in maize.Molecular Breeding,2007,20(1):41-51
[20] Tang J H,Yan J B,Ma X Q,Teng W T,Wu W R,Dai J R,Dhillon B S,Melchinger A E,Li J S.Dissection of the genetic basis of heterosis in an elite maize hybrid by QTL mapping in an immortalized F2 population.Theoretical and Applied Genetics,2010,120(2):333-340
[21] Lu M,Xie C X,Li X H,Hao Z F,Li M S,Weng J F,Zhang D G,Bai L,Zhang S H.Mapping of quantitative trait loci for kernel row number in maize across seven environments.Molecular Breeding,2011,28:143-152
[22] Yang C,Liu J,Rong T Z.Detection of quantitative trait loci for ear row number in F2 populations of maize.Genetics and Molecular Research,2015,14(4):14229-14238
[23] Li F,Jia H T,Liu L,Zhang C X,Liu Z J,Zhang Z X.Quantitative trait loci mapping for kernel row number using chromosome segment substitution lines in maize.Genetics and Molecular Research,2014,13(1):1707-1716
[24] Liu C L,Zhou Q,Dong L,Wang H,Liu F,Weng J F,Li X H.Genetic architecture of the maize kernel row number revealed by combining QTL mapping using a high-density genetic map and bulked segregant RNA sequencing.BMC Genomics,2016,17(1):915
[25] Zhang C S,Zhou Z Q,Yong H J,Zhang X C,Hao Z F,Zhang F J,Li M S,Zhang D G,Li X H,Wang Z H,Weng J F.Analysis of the genetic architecture of maize ear and grain morphological traits by combined linkage and association mapping.Theoretical and Applied Genetics,2017,130(5):1011-1029
[26]赵强,陈柔屹,王安贵,郭向阳,刘鹏飞,祝云芳,吴迅,陈泽辉.基于高密度SNP标记对玉米穗部相关性状的QTL定位及候选基因分析.玉米科学,2021,29(3):36-41
[27] Fei X H,Wang Y F,Zheng Y X,Shen X M,E L Z,Ding J Q,Lai J S,Song W B,Zhao H M.Identification of two new QTLs of maize(Zea mays L.)underlying kernel row number using the HNAU-NAM1 population.BMC Genomics,2022,23(1):593
[28]赵鹏斌,贾晓艳,马江睿,祝丽英,赵永锋.基于RIL群体的玉米穗行数相关QTLs定位与分析.种子,2022,41(12):54-58
[29]聂蕾,何玥,郭爽,王栋,涂亮,刘鹏飞,王安贵,祝云芳,吴迅,陈泽辉,郭向阳.玉米穗部性状QTL定位与候选基因分析.种子,2023,42(7):24-30
[30] Liu Z B,Cook J,Melia-Hancock S,Guill K,Bottoms C,Garcia A,Ott O,Nelson R,Recker J,Balint-Kurti P,Larsson S,Lepak N,Buckler E,Trimble L,Tracy W,McMullen M D,Flint-Garcia S A.Expanding maize genetic resources with predomestication alleles:maize-teosinte introgression populations.Plant Genome,2016,9(1):1-11
[31] Chen Q Y,Yang C J,York A M,Xue W,Daskalska L L,DeValk C A,Krueger K W,Lawton S B,Spiegelberg B G,Schnell J M,Neumeyer M A,Perry J S,Peterson A C,Kim B,Bergstrom L,Yang L,Barber I C,Tian F,Doebley J F.TeoNAM:A nested association mapping population for domestication and agronomic trait analysis in maize.Genetics,2019,213(3):1065-1078
[32]杨小红,严建兵,郑艳萍,余建明,李建生.植物数量性状关联分析研究进展.作物学报,2007,33(4):523-530
[33]王荣焕,王天宇,黎裕.植物基因组中的连锁不平衡.遗传,2007,29(11):1317-1327
[34]任生林,吴才文,经艳芬,刘家勇.全基因组关联分析在作物中的研究进展.分子植物育种,2024,22(11):3594-3602
[35] Brown P J,Upadyayula N,Mahone G S,Tian F,Bradbury P J,Myles S,Holland J B,Flint-Garcia S,McMullen M D,Buckler E S,Rocheford T R.Distinct genetic architectures for male and female inflorescence traits of maize.PLoS Genetics,2011,7(11):e1002383
[36]张焕新,翁建峰,张晓聪,刘昌林,雍洪军,郝转芳,李新海.玉米穗行数全基因组关联分析.作物学报,2014,40(1):1-6
[37] Liu L,Du Y F,Huo D G,Wang M,Shen X M,Yue B,Qiu F Z,Zheng Y L,Yan J B,Zhang Z X.Genetic architecture of maize kernel row number and whole genome prediction.Theoretical and Applied Genetics,2015,128(11):2243-2254
[38] An Y X,Chen L,Li Y X,Li C H,Shi Y S,Zhang D F,Li Y,Wang T Y.Genome-wide association studies and whole-genome prediction reveal the genetic architecture of KRN in maize.BMC Plant Biology,2020,20(1):490
[39] Ma J,Cao Y Y.Genetic dissection of grain yield of maize and yield-related traits through association mapping and genomic prediction.Frontiers in Plant Science,2021,12:690059
[40] Rafalski J A.Association genetics in crop improvement.Current Opinion in Plant Biology,2010,13(2):174-180
[41] Yan J.Association mapping for enhancing maize genetic improvement.Crop Science,2013,51(2):433-449
[42]岳庆春,傅迦得,章辰飞,吴月燕.植物关联分析应用研究进展.江苏农业科学,2019,47(18):24-30
[43] Liu L,Du Y F,Shen X M,Li M F,Sun W,Huang J,Liu Z J,Tao Y S,Zheng Y L,Yan J B,Zhang Z X.KRN4 controls quantitative variation in maize kernel row number.PLoS Genetics,2015,11(11):e1005670
[44] Wang J,Lin Z L,Zhang X,Liu H Q,Zhou LN,Zhong S Y,Li Y,Zhu C,Lin Z W.krn1,a major quantitative trait locus for kernel row number in maize.New Phytologist Foundation,2019,223(3):1634-1646
[45] Chen W K,Chen L,Zhang X,Yang N,Guo J H,Wang M,Ji S H,Zhao X Y,Yin P F,Cai L C,Xu J,Zhang L L,Han Y J,Xiao Y N,Xu G,Wang Y B,Wang S H,Wu S,Yang F,Jackson D,Cheng J H,Chen S H,Sun C Q,Qin F,Tian F,Fernie A R,Li J S,Yan J B,Yang X H.Convergent selection of a WD40 protein that enhances grain yield in maize and rice.Science,2022,375(6587):eabg7985
[46]李晨,王方.玉米和水稻趋同选择的奥秘.中国科学报,2022-03-28(001)
[47] Liu L,Gallagher J,Arevalo E D,Chen R,Skopelitis T,Wu Q Y,Bartlett M,Jackson D.Enhancing grain-yield-related traits by CRISPR-Cas9 promoter editing of maize CLE genes.Nature Plants,2021,7(3):287-294
(收稿日期:2024-07-08)

