沙塘鳢肠道致病菌G-2全基因组测序及信息分析



摘要:为探明造成养殖沙塘鳢肠道出血的病原菌G-2的特性,对G-2进行全基因组测序和分析。基因组组分分析和基因功能注释结果显示:G-2的总基因数为5 134个,其中蛋白编码基因4 977个,非蛋白编码基因157个,非编码基因中tRNA基因107个,rRNA基因42个,sRNA基因8个;分别有5 109、3 339、3 362、2 543个基因在对应的Nr、SwissProt、COG、KEGG数据库中获得注释;毒力基因分析表明G-2含有多种肠毒素和溶血素基因、InhA蛋白、磷脂酶及蛋白质毒素,解释了该菌造成养殖沙塘鳢肠道出血的原因。基于G-2菌株的16S rRNA序列信息构建系统进化树,确定其为苏云金芽孢杆菌。
关键词:沙塘鳢;全基因组测序;苏云金芽孢杆菌
中图分类号:S943 文献标识码:A 文章编号:1006-060X(2024)09-0008-07
Whole Genome Sequencing and Bioinformatics Analysis of a Pathogenic Strain G-2 in the Gut of Odontobutis potamophila
ZHAO Yan-hua1,2,XIA Ai-jun1,2,JIANG Hu-cheng1,2,XUE Hui1,2,LIU Guo-xing1,2
[1. Jiangsu Provincial Freshwater Fisheries Research Institute, Nanjing 210017, PRC; 2. Low-Temperature Germplasm Bank of
Important Economic Fish of Jiangsu Provincial Science and Technology Resources (Agricultural Germplasm Resources)
Coordination Service Platform, Nanjing 210017, PRC]
Abstract: Whole genome sequencing and bioinformatics analysis were carried out to characterize the pathogenic strain G-2 causing gut bleeding of cultured Odontobutis potamophila. The results showed that the genome of strain G-2 contained 5 134 genes, including 4 977 coding genes and 157 non-coding genes (107 tRNA genes, 42 rRNA genes, and 8 sRNA genes). In Nr, SwissProt, COG, and KEGG, 5 109, 3 339, 3 362, and 2 543 genes, respectively, were annotated. Strain G-2 carried multiple enterotoxin and hemolysin genes and the genes encoding InhA protein, phospholipase, and protein toxins, which explained the gut bleeding caused by this strain. The phylogenetic tree built based on the 16S rRNA gene sequence identified strain G-2 as Bacillus thuringiensis.
Key words: Odontobutis potamophila; whole genome sequencing; Bacillus thuringiensis
全基因组测序(Whole genome sequencing)是对未知基因组序列的物种进行个体的基因组测序,通过将生物的基因组打断、测序和拼接获得较为完整的基因组序列,继而进行序列分析和功能基因注释,最终从基因水平深入了解生物的各项机理[1-2]。1986年,Renato Dulbecco等科学家最早提出人类基因组测序计划。1995年,流感嗜血杆菌(Haemophilus influenzae)成为第一个完成全基因组测序的自由生存生物[3]。随着测序技术的发展和测序成本的降低,目前全基因组测序已经经历了三代技术发展,对任何生物的基因组进行测序成为可能[4-5]。在病害防控领域,全基因组测序技术可以有效获取病原菌较为完整的基因组序列,再结合功能基因组学和生物信息学手段,为快速了解致病菌的致病机理和耐药机制提供了极大的帮助[3]。
苏云金芽孢杆菌(Bacillus thuringiensis)是一种革兰氏阳性细菌,可做微生物源杀虫剂,以胃毒作用为
主[6]。其中,苏云金芽孢杆菌以色列亚种(Bacillus thuringiensis subsp. israelensis)是一种低毒杀虫剂,会在芽孢形成过程中产生名为δ-内毒素的杀虫伴孢晶体蛋白,杀虫晶体蛋白分为Cry和Cyt两大类[7]。Cyt蛋白具有溶血和溶细胞特性,对脊椎动物和无脊椎动物均有溶解细胞的毒害作用[8-10]。苏云金芽孢杆菌也是典型的昆虫病原菌,对多种昆虫、线虫和螨类等具有特异性的毒杀活性,在生物农药领域占据重要地位,是目前世界上研究最深入、应用最广泛的微生物杀虫剂[11-12]。
为探明造成养殖沙塘鳢肠道出血的病原菌G-2的特性,解析G-2的致病机理,该研究结合三代PacBio RS II测序技术和二代Illumina HiSeq 2000测序技术,对病原菌G-2进行全基因组测序分析,并对获得的数据进行Nr(Non-Redundant Protein Sequence
Database)、Swiss-Prot、KEGG(Kyoto Encyclopedia of Genes and Genomes)、GO(Gene Ontology)、COG(Cluster of Orthologous Groups of proteins)、VFDB(Virulence Factors of Pathogenic Bacteria)数据库注释及分析,明确其基因组遗传特性,为沙塘鳢的病害防控提供研究基础。
1 材料与方法
1 供试材料
1.1.1 致病菌 菌株G-2来自江苏省扬中市沙塘鳢养殖基地,由江苏省淡水水产研究所病害防治研究室分离、鉴定及保存。
1.1.2 主要试剂和仪器 CTAB试剂(广州化学试剂厂),Luria-Bertani(LB)培养基、脑心浸液肉汤培养基(广东环凯生物科技有限公司),DNA提取试剂盒[生工生物工程(上海)股份有限公司],琼脂糖凝胶电泳仪(北京六一生物科技有限公司),NanoDrop微量分光光度计(美国赛默飞世尔科技公司),移液器(德国艾本德股份公司)。
1.2 研究方法
1.2.1 G-2的复苏及扩大培养 将实验室冻存的G-2菌株于室温条件下解冻,并用移液枪吸取100 mL菌液加入脑心浸液肉汤培养基中,置于30℃恒温培养箱中培养24 h;划线接种于LB固体培养基,置于同样培养条件下培养24 h;挑取长势良好的单菌落接种于LB液体培养基,进行扩大培养,1 000 r/min转速下离心10 min,收集菌体。
1.2.2 基因组提取、测序及组装 使用CTAB法提取基因组DNA,将基因组DNA置于-20℃冰箱保存。对提取的DNA进行质量检测,DNA样品检测合格后,进行文库构建和文库检测,库检合格后,送至广州基迪奥科技服务有限公司进行测序。结合三代PacBio RS II测序技术和二代Illumina HiSeq 2000测序技术[13],对病原菌G-2进行全基因组测序分析。使用三代测序数据进行组装,保证基因组组装的完整性;使用二代测序数据对组装结果进行校正,保证组装结果的准确性;使用Fastp软件对二代测序数据进行质控,基于校正后的组装结果进行基因组组分分析。
1.2.3 基因组组分分析 使用NCBI数据库(National Center for Biotechnology Information)进行基因预测,使用RNAmmer软件预测基因组中的rRNA基因[14],使用tRNAscan-SE软件预测基因组中的tRNA基因,基于Rfam数据库使用CMScan软件预测基因组中的sRNA基因[15-16],使用tRNAscan-SE软件预测tRNA区域和tRNA的二级结构[17],使用RepeatMasker软件预测基因组重复序列,使用TRF软件预测基因组的串联重复序列[18],使用CRISPRfinder软件预测基因组的CRISPR区域,使用在线工具IslandViewer 4对基因组进行基因岛预测。
1.2.4 基因功能注释 使用KEGG数据库(http://www.genome.jp/kegg/)进行编码基因功能预测,查找基因所在的生物学通路[19-20];使用GO数据库(http://geneontology.org/)对基因和蛋白质功能进行限定和描述[21];基于COG数据库(http://www.ncbi.nlm.
nih.gov/COG/)使用DIAMOND软件进行数据比对,进行蛋白功能分类;利用VFDB数据库(http://www.mgc.ac.cn/)进行BLAST比对,预测毒力因子[22]。
1.2.5 G-2系统发育树的构建 基于G-2菌株的16S rRNA序列信息,从GenBank中选取亲缘关系较近的10个序列,利用MEGA 4软件进行序列比对,采用邻接法分析同源性,构建系统发育树。
2 结果与分析
2.1 G-2基因组测序及组装分析
使用三代测序平台(PacBio RS II)对G-2进行测序,G-2过滤后的有效reads数为1 618 433条,平均测序读长为4 359.5 bp,N50为6 698 bp,组装后总长度为5 270 034 bp,GC含量为35.36%。
2.2 G-2基因分析及预测
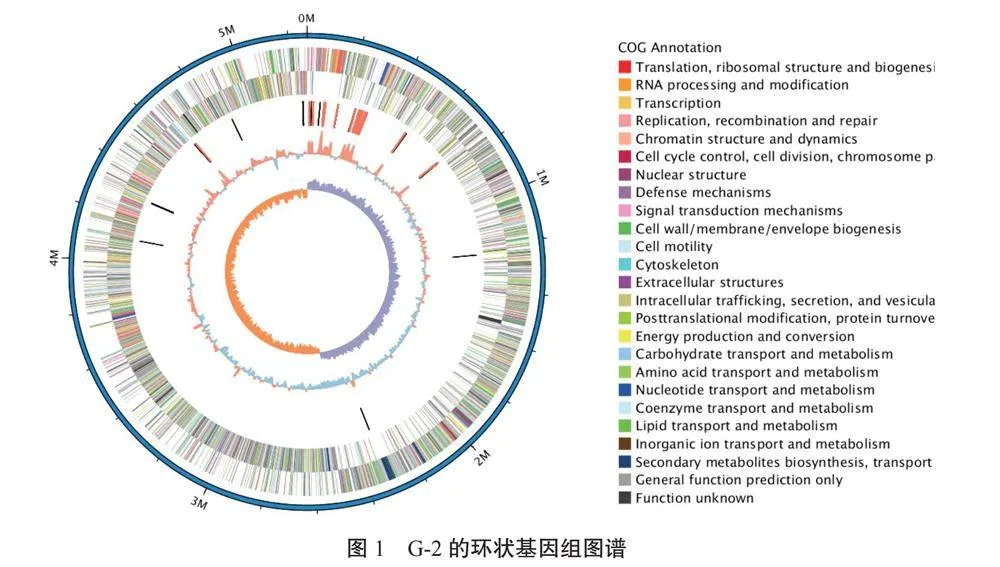
使用二代测序对三代测序数据进行校正,最终获得一条4 400 748 bp的环状染色体基因组(图1)。该基因组的总基因数为5 134个,其中蛋白编码基因4 977个,编码区域长度为4 327 191 bp,占总长度的98.3%;非蛋白编码基因157个,其中tRNA基因107个,rRNA基因42个,sRNA基因8个。rRNA基因中23S_rRNA基因14个,16S_rRNA基因14个,5S_rRNA基因14个。基因间隔区域长度为73 557 bp,占总长度的1.7%。菌株G-2基因组的GC-depth分布图如图2所示,发现其散点分布较为集中,无杂菌污染。由G-2的基因长度分布图(图3)可知,基因长度小于1 000 bp的基因出现频率较高,基因长度为2 500~3 000 bp的基因出现的频率较低。
2.3 基因功能注释分析
2.3.1 数据库注释比对 菌株G-2全基因组测序共获得5 134个编码基因,具有明确生物学功能的编码基因5 110个。如图4所示,将获得的编码基因与相应数据库进行比对,分析结果显示:有5 109个基因在对应的Nr数据库获得注释,有3 339个基因在对应的SwissProt数据库获得注释,有3 362个基因在对应的COG数据库获得注释,有2 543个基因在对应的KEGG数据库获得注释。
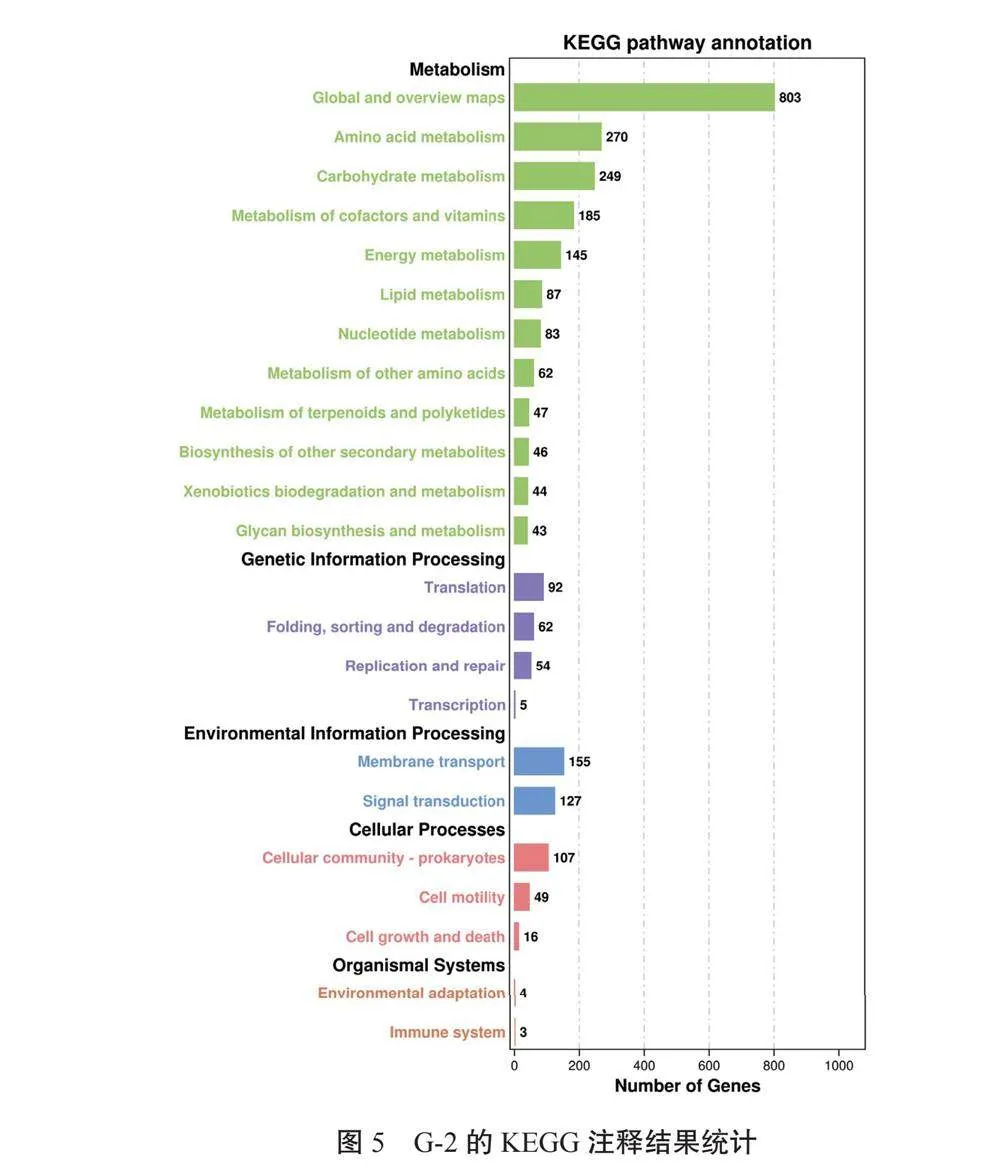
2.3.2 KEGG代谢通路分析 菌株G-2在KEGG数据库中得到注释的基因有2 543个。由图5可知,在代谢类中,有12种通路得到注释,其中全局总览的基因个数最多,有803个;其次为氨基酸代谢通路和碳水化合物代谢通路,分别有270和249个基因;甘氨酸生物合成和代谢通路最少,只有43个基因。在遗传信息处理类中,有4种通路得到注释,其中翻译通路的基因有92个,转录通路的基因有5个。在环境信息处理类中,有2种通路得到注释,其中膜运输通路有155个基因。在细胞过程类中,有3种通路得到注释,其中原核生物基因数量最多,为107个,细胞生长和死亡通路最少,只有16个基因。2种生物系统类通路得到注释,环境适应性和免疫系统的基因分别为4、3个。
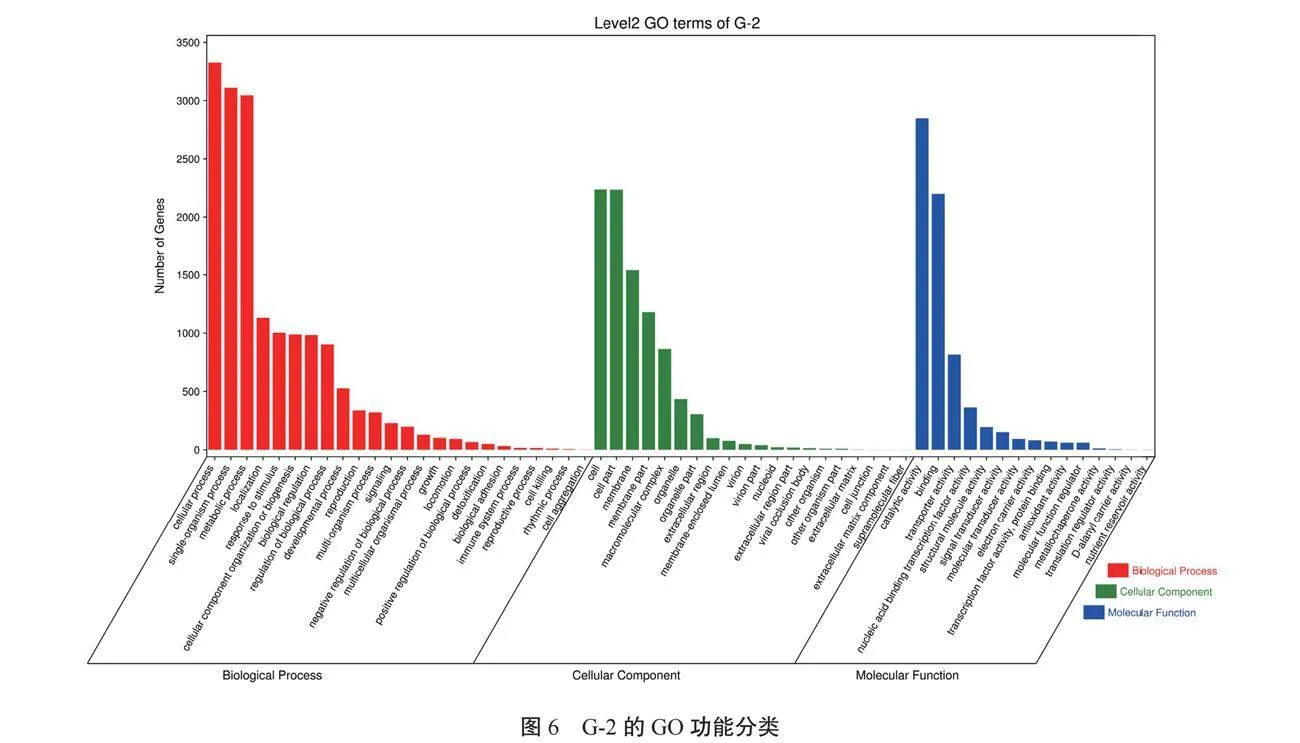
2.3.3 GO注释 GO数据库是用于描述基因和蛋白质功能的标准化分类系统,包括生物过程、细胞组分和分子功能3种基因功能[23]。对苏云金芽孢杆菌G-2的基因进行GO注释和分类,判断其基因参与的主要功能。如图6所示,共有4 130个基因在GO数据库中得到注释,在生物过程中,有3 792个基因得到注释,分类到细胞过程中的基因占比最多;在细胞组分分类中,有2 706个基因得到注释,分类到细胞和细胞组成中的基因占比最多;在分子功能分类中,有3 767个基因得到注释,分类到催化活性中的基因占比最多。
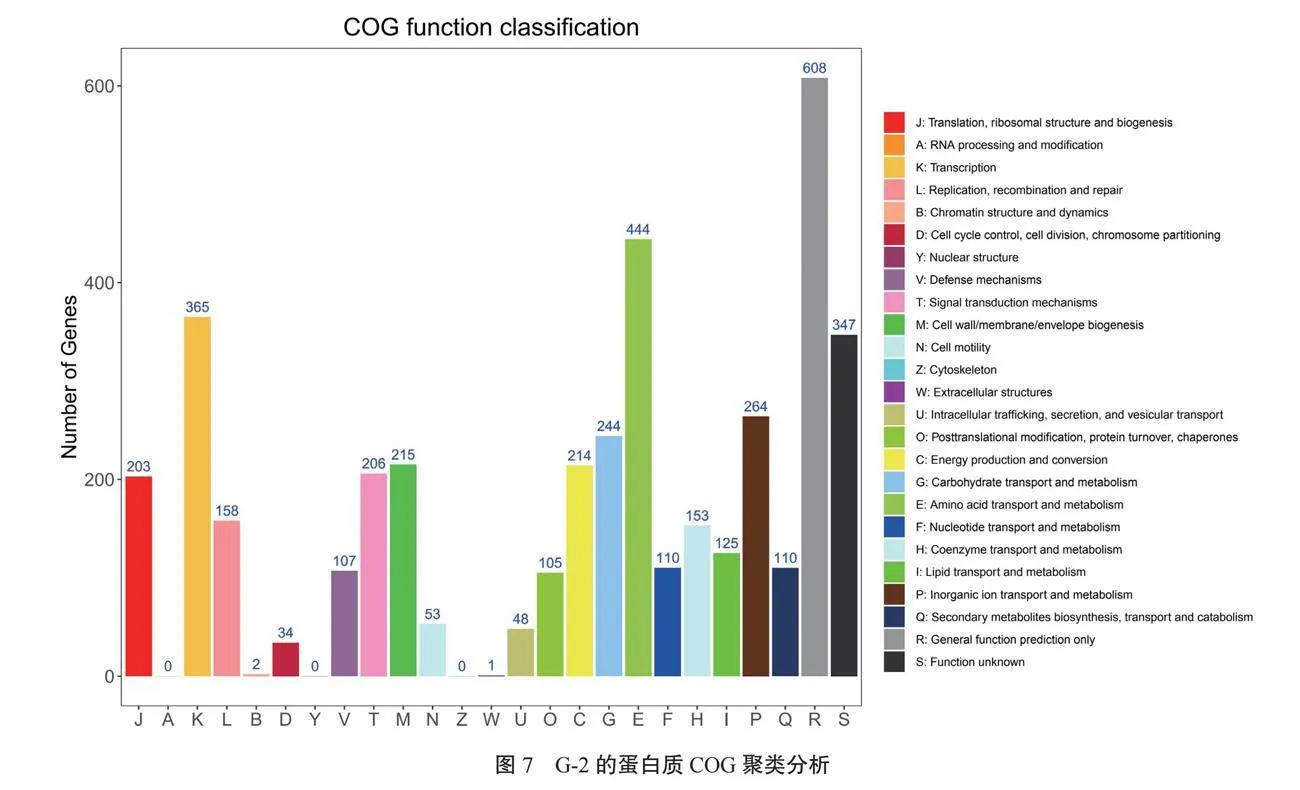
2.3.4 COG功能分类 由图7可知,COG分类的蛋白编码基因共3 362个,其中,常规功能预测类基因608个,氨基酸转运和代谢类基因444个,转录类基因365个,无机离子转运和代谢类基因264个,辅酶转运和代谢类基因244个。
2.3.5 VFDB注释 通过VFDB数据库对比,发现G-2含有多种肠毒素及溶血素基因,如溶血素BL、非溶血性肠毒素基因A、肠毒素C等,还含有InhA蛋白(即免疫抑制因子A)、磷脂酶Phospholipase C precursor和蛋白质毒素Perfringolysin O precursor。溶血素通过溶解红细胞损害宿主,InhA蛋白通过水解宿主的抗细菌蛋白来增强自身的毒性,磷脂酶则通过水解磷脂酞胆碱、磷脂酞丝氨酸和磷脂酞乙醇破坏宿主肠道,蛋白质毒素Perfringolysin O的编码基因主要存在于产气夹膜梭菌中,具有膜穿孔的能力[24]。
2.4 G-2系统发育树分析
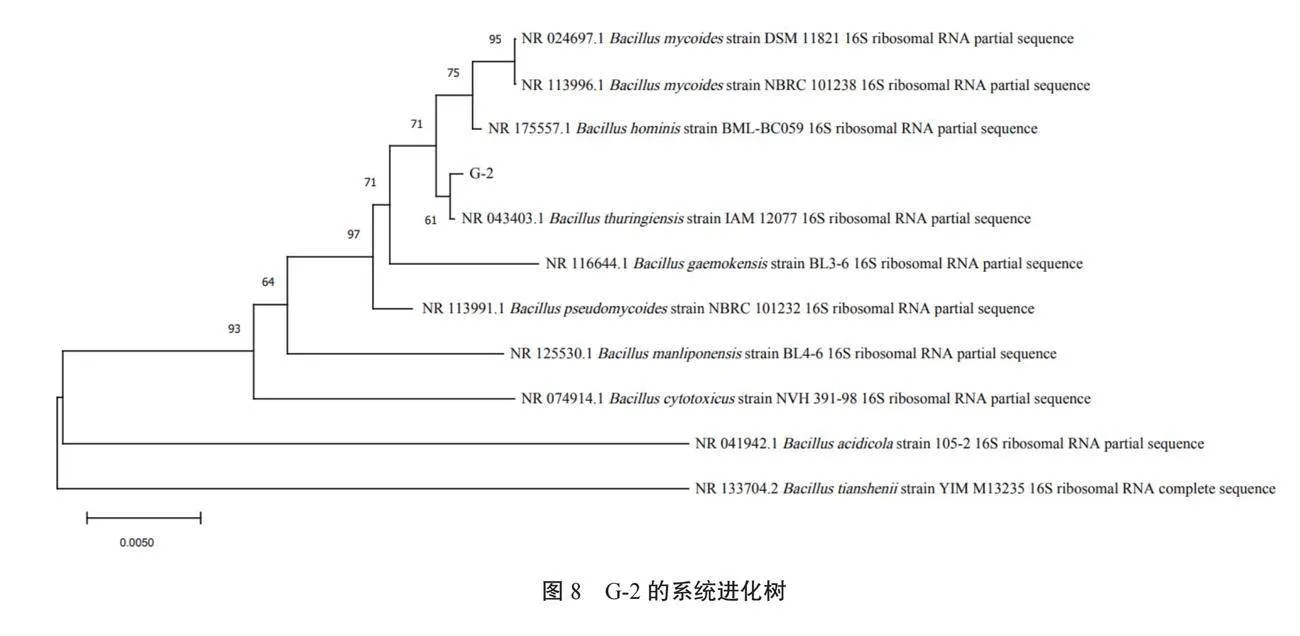
由图8可知,G-2与苏云金芽孢杆菌IAM 12077聚为一支,自展值为61%,因此可以确定菌株G-2为苏云金芽孢杆菌。
3 讨论与结论
随着水产养殖的集约化发展,高密度养殖和抗生素的大量使用造成了养殖鱼类的细菌性病害日益严重。黄志坚等[25]研究发现苏云金芽孢杆菌与凡纳滨对虾肝胰腺坏死症暴发密切相关,凡纳滨对虾感染苏云金芽孢杆菌后,其鳃和肠道的Lv-JAK基因和Lv-STAT基因的相对表达量均显著上调,而JAK基因和STAT基因与天然免疫密切相关。杨湾等[26]研究表明,苏云金芽孢杆菌能够感染甲鱼,造成甲鱼脖子和脾脏肿胀并使其肝脏呈土黄色。
该研究中苏云金芽孢杆菌G-2会造成养殖沙塘鳢肠道出血,鳃部及头部溃烂[27],对G-2进行全基因组测序,获得了一条4 400 748 bp的环状染色体基因组,总基因数为5 134个,其中蛋白编码基因
4 977个,非蛋白编码基因157个,非蛋白编码基因中tRNA基因107个,rRNA基因42个,sRNA基因8个。rRNA中23S_rRNA基因14个,16S_rRNA基因1 4个,5S_rRNA基因14个。Schnepf等[7]发现苏云金芽孢杆菌染色体为环状DNA,GC含量一般为32%~35%,基因组大小2.4~5.7 Mb不等,与该研究测序得到的结果相似。
苏云金芽孢杆菌G-2有2 543个基因在KEGG数据库中得到注释,代谢类的子条目和基因数量都是最多的,其中氨基酸代谢通路和碳水化合物代谢通路分别有270和249个基因。总体而言,菌株G-2代谢活动比较丰富,需要与外界进行能量交换来维持自身生命活动。GO注释结果表明,菌株G-2蛋白功能主要集中在生物过程中的代谢过程和分子功能中的催化活性功能。蛋白质COG分析结果表明G-2碳水化合物代谢和氨基酸的转运活动较多,表明该菌株的基础代谢较高、生理活动比较丰富。VFDB注释可以分析不同病原菌的毒力因子构成以及基因组的分布特点,从基因水平上解释病原菌的入侵机制。通过数据库对比发现,G-2含有多种肠毒素和溶血素基因,这也解释了为什么感染该菌株的沙塘鳢的主要症状为肠道出血。同时该菌株还含有InhA蛋白、磷脂酶和蛋白毒素;与华中农业大学测序的苏云金芽孢杆菌YBT-1520比较发现,YBT-1520和G-2都含有肠毒素、溶血素、磷脂酶和InhA蛋白;但YBT-1520还含有蛋白酶Enhancin和几丁质酶Chitinase,可以提高杀虫活性,G-2则含有蛋白质毒素Perfringolysin O,能够溶解红细胞。
目前国内针对苏云金芽孢杆菌的全基因组测序研究相对较少,为探明造成养殖沙塘鳢肠道出血的病原菌苏云金芽孢杆菌G-2的特性,该研究对G-2进行了全基因组测序,从基因组学上分析了该菌的基因功能,并进行毒力基因分析,解释了该菌造成养殖沙塘鳢肠道出血的原因,以期为沙塘鳢的病害防控提供科研基础。
参考文献:
[1] GOODWIN S,MCPHERSON J D,MCCOMBIE W R. Coming of age:ten years of next-generation sequencing technologies[J]. Nature Reviews Genetics,2016,17(6):333-351.
[2] GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al. Microbial genome analysis:the COG approach[J]. Briefings in Bioinformatics,2019,20(4):1063-1070.
[3] 刘贵明. 苏云金芽孢杆菌YBT-1520全基因组测序和比较基因组学研究[D]. 杭州:浙江大学,2008.
[4] MARGUERAT S,WILHELM B T,BÄHLER J. Next-generation sequencing:applications beyond genomes[J]. Biochemical Society Transactions,2008,36(Pt 5):1091-1096.
[5] 谭寿湖,张文飞,叶大维. 苏云金芽孢杆菌基因组研究概况[J]. 基因组学与应用生物学,2009,28(1):202-208.
[6] BEN-DOV E. Bacillus thuringiensis subsp. israelensis and its dipteran-specific toxins[J]. Toxins,2014,6(4):1222-1243.
[7] SCHNEPF E,CRICKMORE N,VAN RIE J,et al. Bacillus thuringiensis and its pesticidal crystal proteins[J]. Microbiology and Molecular Biology Reviews:MMBR,1998,62(3):775-806.
[8] WAALWIJK C,DULLEMANS A M,VAN WORKUM M E,et al. Molecular cloning and the nucleotide sequence of the Mr 28000 crystal protein gene of Bacillus thuringiensis subsp. israelensis[J]. Nucleic Acids Research,1985,13(22):8207-8217.
[9] PROMDONKOY B,ELLAR D J. Membrane pore architecture of a cytolytic toxin from Bacillus thuringiensis[J]. The Biochemical Journal,2000,350(Pt 1):275-282.
[10] AKIBA T,ABE Y,KITADA S,et al. Crystal structure of the parasporin-2 Bacillus thuringiensis toxin that recognizes cancer cells[J]. Journal of Molecular Biology,2009,386(1):121-133.
[11] CRICKMORE N,ZEIGLER D R,FEITELSON J,et al. Revision of the nomenclature for the Bacillus thuringiensis pesticidal crystal proteins[J]. Microbiology and Molecular Biology Reviews:MMBR,1998,62(3):807-813.
[12] 喻子牛,孙明,刘子铎,等. 苏云金芽孢杆菌的分类及生物活性蛋白基因[J]. 中国生物防治,1996,12(2):85-89.
[13] GAN Y Q,ZHANG T,GAN Y Q,et al. Complete genome sequences of two Enterococcus faecium strains and comparative genomic analysis[J]. Experimental and Therapeutic Medicine,2020,19(3):2019-2028.
[14] LAGESEN K,HALLIN P,RØDLAND E A,et al. RNAmmer:consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research,2007,35(9):3100-3108.
[15] GARDNER P P,DAUB J,TATE J G,et al. Rfam:updates to the RNA families database[J]. Nucleic Acids Research,2009,37:D136-D140.
[16] NAWROCKI E P,KOLBE D L,EDDY S R. Infernal 1.0:inference of RNA alignments[J]. Bioinformatics,2009,25(10):1335-1337.
[17] LOWE T M,EDDY S R. tRNAscan-SE:a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research,1997,25(5):955-964.
[18] BENSON G. Tandem repeats finder:a program to analyze DNA sequences[J]. Nucleic Acids Research,1999,27(2):573-580.
[19] KANEHISA M,GOTO S,KAWASHIMA S,et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research,2004,32(Database issue):D277-D280.
[20] KANEHISA M,GOTO S,HATTORI M,et al. From genomics to chemical genomics:new developments in KEGG[J]. Nucleic Acids Research,2006,34(Database issue):D354-D357.
[21] ASHBURNER M,BALL C A,BLAKE J A,et al. Gene ontology:tool for the unification of biology. The Gene Ontology Consortium[J]. Nature Genetics,2000,25(1):25-29.
[22] CHEN L,YANG J,YU J,et al. VFDB:a reference database for bacterial virulence factors[J]. Nucleic Acids Research,2005,33(suppl_1):D325-D328.
[23] 王媛. 猕猴桃展青霉素产生菌的识别分析及控制机制研究[D]. 杨凌:西北农林科技大学,2017.
[24] HEUCK A P,HOTZE E M,TWETEN R K,et al. Mechanism of
membrane insertion of a multimeric beta-barrel protein:perfringolysin
O creates a pore using ordered and coupled conformational changes[J]. Molecular Cell,2000,6(5):1233-1242.
[25] 黄志坚,陈勇贵,翁少萍,等. 多种细菌与凡纳滨对虾肝胰腺坏死症(HPNS)爆发有关[J]. 中山大学学报(自然科学版),2016,55(1):1-11.
[26] 杨湾,王琳,朱雅琳,等. 中华鳖源苏云金芽孢杆菌的分离鉴定及毒力因子检测[J]. 浙江万里学院学报,2022,35(6):85-93.
[27] 赵彦华,史杨白,刘国兴,等. 一株养殖沙塘鳢肠道溶血性苏云金芽孢杆菌的分离鉴定[J]. 水产养殖,2021,42(4):16-19.
(责任编辑:王婷)

