铁十字秋海棠R2R3-MYB基因家族的鉴定及表达分析







摘要:【目的】对铁十字秋海棠(Begonia masoniana)R2R3-MYB基因家族进行鉴定分析,并检测R2R3-MYB基因家族成员在铁十字秋海棠叶片不同生长发育时期中叶斑区和非叶斑区的表达模式,为铁十字秋海棠叶斑分子调控机制和叶色改良及新品种培育提供理论参考。【方法】以铁十字秋海棠3个关键生长发育时期的叶片为试验材料,参考拟南芥R2R3-MYB家族蛋白序列,利用生物信息学方法从铁十字秋海棠基因组数据库中鉴定出R2R3-MYB基因家族成员,并对其理化性质、染色体定位、系统发育、基因结构、保守基序及启动子顺式作用元件等进行预测分析,并通过实时荧光定量PCR检测7个BmaMYBs基因在铁十字秋海棠3个生长发育期中叶斑区和非叶斑区的表达模式。【结果】从铁十字秋海棠基因组中鉴定出110个R2R3-MYB基因家族成员,该基因家族成员的氨基酸数量为128~870个,分子量为14660.85~97435.93 Da,理论等电点(pI)为4.92~10.10,成员之间差异较大,大部分为不稳定的疏水性蛋白,均定位在细胞核。110个R2R3-MYB家族基因在15条染色体上均有分布,部分基因在同一染色体上集中分布,形成基因簇;BmaMYBs含有0~10个内含子;约94%的成员含有Motif 1、Motif 2和Motif 3基序;R2R3-MYB基因家族成员启动子均含有大量光响应元件;BmaMYB10、BmaMYB18、BmaMYB38、BmaMYB53、BmaMYB97、BmaMYB99和BmaMYB109基因在叶片发育过程中叶斑区与非叶斑区均有表达,叶斑区BmaMYB53基因的相对表达量较非叶斑区高达19.41~131.99倍。【结论】在铁十字秋海棠基因组中鉴定出110个R2R3-MYB基因家族成员,叶斑区与非叶斑区BmaMYB53基因的相对表达量差异最大,推测该基因是调控铁十字秋海棠叶斑形成的关键基因。
关键词:铁十字秋海棠;R2R3-MYB基因家族;花青苷;叶斑
中图分类号:S682.36文献标志码:A文章编号:2095-1191(2024)09-2665-14
Identification and expression analysis of R2R3-MYB gene family in Begonia masoniana
LIU Ya-zhi,DENG Hui-min,PENG Jia-hui,YU Yi-xun,LIU Juan-xu*
(College of Horticulture,South China Agricultural University,Guangzhou,Guangdong 510642,China)
Abstract:【Objective】Identification and analysis of the R2R3-MYB gene family in Begonia masoniana was con-ducted,and the expression patterns of the R2R3-MYB gene family members in different growth stages of leaf spots and non-leaf spot regions was detected,providing theoretical reference for understanding the molecular regulatory mecha-nisms underlying leaf spot formation in B.masoniana,as well as for leaf color improvement and new variety breeding.【Method】Using leaves from 3 key growth and development periods of B.masoniana as experimental materials,with Ara-bidopsis thaliana R2R3-MYB family proteins as reference sequences,members of the R2R3-MYB gene family from the B.masoniana genome database were identified using bioinformatics methods.Then predicted and analyzed their physico-chemical properties,chromosomal localization,phylogenetic relationships,gene structures,conserved motifs and pro-motercis-acting elements.Additionally,examined the expression patterns of 7 BmaMYBs genes in the leaf spot region and non-leaf spot region during 3 growth and development periods of B.masoniana through real-time fluorescence quantita-tive PCR.【Result】Atotal of 110 members of the R2R3-MYB gene family were identified from the genome of B.masoniana.These gene family members had amino acid quantity ranging from 128 to 870,molecular weights ranging from 14660.85 to 97435.93 Da,and theoretical isoelectric points(pI)ranging from 4.92 to 10.10.There were great differences among the members,with most being unstable hydrophobic proteins,and all of them located in the nucleus.The 110 R2R3-MYB family genes were distributed across all 15 chromosomes,with some genes concentrated on the same chro-mosome forming gene clusters.BmaMYBs contained 0-10 introns;approximately 94%of the members contained Motif 1,Motif 2 and Motif 3;and the promoter regions of R2R3-MYB gene family members contained a large number of light-responsive elements.During leaf development,BmaMYB10,BmaMYB18,BmaMYB38,BmaMYB53,BmaMYB97,Bma-MYB99 and BmaMYB109 genes were expressed in both leaf spot and non-leaf spot regions;relative expression level ofthe BmaMYB53 gene in the leaf spot region was as high as 19.41 to 131.99 times compared to that in non-leaf spot region.【Conclusion】In the genome of B.masoniana,110 members of the R2R3-MYB gene family are identified.The relative ex-pression level difference between the leaf spot region and non-leaf spot region is the greatest for BmaMYB53 gene,sug-gesting that this gene is a key regulator in the formation of leaf spots in B.masoniana.
Key words:Begonia masoniana;R2R3-MYB gene family;anthocyanins;leaf spot
Foundation items:National Natural Science Foundation of China(32271939,31870692)
0引言
【研究意义】铁十字秋海棠(Begonia masoniana)作为原产于我国的重要观叶植物,因其叶片中央分布有红褐色十字形斑纹而得名,叶斑随发育时期不同而呈现粉红、红褐和深褐色,然而其彩斑形成机理仍不清楚。研究表明,花青苷的积累可能会影响秋海棠属植物叶斑的形成(Wang et al.,2018)。在植物体内众多转录因子家族中,MYB被认为是数量最多的转录因子家族之一,广泛参与调节植物生长发育、次生代谢、逆境胁迫响应等各项生命活动,同时还能够调控花青苷的生物合成(左鑫,2023)。MYB转录因子的特征是在N端有一段高度保守的MYB-DNA结合结构域,包含1~4个串联的、不完全重复的MYB重复区域(R1、R2和R3)。根据MYB结构域的数量和位置将MYB基因家族分为四大类,分别是1R-MYB(或MYB相关)、2R-MYB(R2R3-MYB)、3R-MYB(R1R2R3-MYB)和4R-MYB(Dubos et al.,2010)。R2R3-MYB是植物所特有,在MYB基因家族中数量最多,已被证实与植物组织生长发育(Paz-Ares et al.,1987)、激素信号转导(Stracke et al.,2001)、调节花青苷生物合成(Davies et al.,2012)、响应逆境胁迫(崔晓娜等,2023)等过程相关。因此,对铁十字秋海棠R2R3-MYB基因家族进行鉴定并分析其表达模式,对探究铁十字秋海棠叶斑形成的分子机制和叶色改良及新品种培育具有重要意义。【前人研究进展】研究发现,花青苷的合成在铁十字秋海棠斑叶发育过程中发挥重要作用(杨婷等,2021)。花青苷是植物界中最大的一类水溶性色素,属于类黄酮化合物家族,主要储存于植物的中央液泡中,使得多种植物组织如根、茎、叶、花、果实和种子呈现不同的颜色(梁平和宋洪元,2014;梁玉镯等,2022)。而花青苷的合成主要受两类基因的调控:一类是编码花青苷生物合成各种酶的结构基因,另一类是起转录调控作用的调节基因(Tanaka and Ohmiya,2008;Zhang et al.,2014)。目前已报道的关键调节基因主要包括MYB、bHLH和WD40,其中MYB和bHLH蛋白既可以单独调控花青苷的生物合成,也可以通过MYB-bHLH-WD40(MBW)形成三元复合物抑制或者激活相关基因的表达,调控花青苷的合成和分布(Ramsay and Glover,2005)。植物中第一个R2R3-MYB转录因子于1987年首次在玉米(Zea mays)中被鉴定,证实其功能与花青苷的合成密切相关(Paz-Ares et al.,1987)。随着研究的深入,MYB基因家族被鉴定的物种数量日益增多,R2R3-MYB基因家族在不同物种中的成员数量也呈现出多样性,例如在拟南芥(Arabidopsis thaliana)中有126个(Stracke et al.,2001),水稻(Oryza sativa)有102个(Chen et al.,2006),大豆(Glycine max)有244个(许玲等,2015),番茄(Solanum lycopersicum)有127个(Li et al.,2016)。在大量植物中已发现存在对花青苷生物合成具有正向调控的R2R3-MYB转录因子,例如对拟南芥的研究发现,S6亚组的成员(AtMYB75、AtMYB90、AtMYB113和AtMYB114)能正向调控花青苷的生物合成(Gonzalez et al.,2008);菊花(Chrysanthemum morifolium Ramat.)的CmMYB6和CmbHLH2转录因子互作,可上调CmDFR基因的表达诱导花青苷积累,将CmMYB6基因转入烟草中过表达,结果发现烟叶中花青苷含量急剧增加,从而证实CmMYB6基因是调控菊花花青素生物合成的重要基因(Liu et al.,2015);文心兰(Oncidium Gower Ramsay)中OgMYB1可以激活CHI和DFR基因的转录,从而诱导花青苷的合成(Chiou and Yeh,2008)。此外,矮牵牛(Petunia hybrida)的AN2(Quattrocchioetal.,1999)和AN4(Walker et al.,1999)以及牡丹(Paeonia suffruticosa)的PsMYB12(Gu etal.,2019)都是调控花青苷生物合成的转录激活子。另外,研究还揭示了能够抑制花青苷生物合成的R2R3-MYB转录因子,此类抑制子在模式植物拟南芥中已有广泛研究,如S4亚组的成员AtMYB4、AtMYB7和AtMYB32(Kranz etal.,1998),在其他植物种类中此类抑制子亦被发现,例如矮牵牛的PhMYB27(Albert et al.,2014)、郁金香(Tulipa gesne-riana)的TgMYB4(Hu etal.,2024)等。【本研究切入点】目前,已对拟南芥(Stracke et al.,2001)、水稻(Chen et al.,2006)、大豆(许玲等,2015)等许多物种开展R2R3-MYB基因家族鉴定分析研究,尚未见有关铁十字秋海棠R2R3-MYB基因家族鉴定的相关报道。【拟解决的关键问题】以铁十字秋海棠3个关键生长发育时期的叶片为试验材料,参考拟南芥R2R3-MYB家族蛋白序列,利用生物信息学方法从铁十字秋海棠基因组数据库中鉴定出R2R3-MYB基因家族成员,对其理化性质、染色体定位、系统发育、基因结构、保守基序及启动子顺式作用元件等进行预测分析,并通过实时荧光定量PCR检测7个BmaMYBs基因在铁十字秋海棠3个生长发育期中叶斑区和非叶斑区的表达模式,进而探究R2R3-MYB基因家族在铁十字秋海棠叶斑形成过程中的分子机制,为铁十字秋海棠叶色改良及新品种培育提供科学依据。
1材料与方法
1.1试验材料
样品采自华南农业大学园艺学院培养室的2年生铁十字秋海棠健康植株,分别选取叶片3个关键生长发育时期,即S1:叶长4 cm;S2:叶长8 cm;S3:叶长12 cm(图1)。主要试剂:FastPure Universal Plant Total RNA Isolation Kit、HiScript II Q RT Super-Mix for qPCR+gDNA wiper RNA逆转录试剂盒和AceQ®Universal SYBR qPCR Master Mix定量PCR检测试剂盒均购自南京诺唯赞生物科技股份有限公司。
1.2铁十字秋海棠R2R3-MYB基因家族成员鉴定
从国家基因库生命大数据平台(https://db.cngb.org/search/assembly/CNA0013975/)下载获得铁十字秋海棠基因组数据(Li et al.,2022)(基因组编号:CNA0013975),从TAIR数据库(https://www.arabi-dopsis.org/browse/genefamily/index.jsp)下载获得拟南芥R2R3-MYB家族蛋白序列。随后,使用拟南芥数据库中的R2R3-MYB蛋白序列进行本地Blast搜索,查询铁十字秋海棠基因组数据库得到MYB候选基因。然后通过SMART(http://smart.embl.de/)和Pfam(http://pfam.xfam.org/)数据库验证候选基因中MYB结构域,排除不包含完整MYB保守结构域的序列,确定最终成员。
1.3铁十字秋海棠R2R3-MYB家族蛋白理化性质分析
利用ExPASy(https://web.expasy.org/protparam/)对R2R3-MYB家族成员进行理化性质预测分析,使用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)进行亚细胞定位预测,通过TMHMM网站(https://services.healthtech.dtu.dk/services/TMH MM-2.0/)预测蛋白跨膜结构域。
1.4铁十字秋海棠R2R3-MYB基因家族染色体定位
通过分析基因组GFF文件获取铁十字秋海棠的R2R3-MYB家族基因在染色体上的具体位置信息,并进行整理统计。利用TBtools中Gene Location Visua-lize from GTF/GFF功能将铁十字秋海棠R2R3-MYB家族基因的起始位置、终止位置以及相应染色体的总长度信息结合,绘制染色体定位模式图。
1.5系统发育进化树构建
通过MEGA 11.0中Clustal W对铁十字秋海棠与拟南芥的R2R3-MYB蛋白序列进行多重比对。基于比对结果,采用邻接法(Neighbor-joining,NJ)构建系统发育进化树,Bootstrap设为1000次。最后,利用在线工具iTOL(https://itol.embl.de/)对系统发育进化树进行可视化、优化和美化处理。
1.6铁十字秋海棠R2R3-MYB家族基因结构及蛋白保守基序分析
将铁十字秋海棠R2R3-MYB家族蛋白序列上传到Multiple Expectation Maximization for Motif Elicitation在线分析平台(http://meme-suite.org/meme/tools/meme),然后利用TBtools对结果进行可视化处理。
1.7铁十字秋海棠R2R3-MYB基因家族启动子分析
使用TBtools中的gtf/gff3 Sequences Extract工具提取铁十字秋海棠R2R3-MYB基因家族起始密码子ATG上游2000 bp的启动子序列,然后通过PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测其顺式作用元件,分析整理后再利用TBtools将结果可视化。
1.8实时荧光定量PCR检测
利用FastPure Universal Plant Total RNA Isola-tion Kit分别提取铁十字秋海棠叶片叶斑区和非叶斑区的3个关键生长发育时期(S1~S3)的总RNA,并使用HiScript II Q RT SuperMix for qPCR(+gDNA wiper)RNA反转录试剂盒合成cDNA第一链,以其为模板进行实时荧光定量PCR检测,分析铁十字秋海棠叶片叶斑区和非叶斑区的3个关键生长发育时期相关BmaMYBs基因的表达模式。利用Primer Premier 5.0设计基因的编码区(CDS)特异性引物(表1),引物由生工生物工程(上海)股份有限公司合成。反应体系和扩增程序参照AceQ®Universal SYBR qPCR Master Mix定量PCR检测试剂盒说明书进行,每样本设3个生物学重复以及3次技术重复。选取Actin7为内参基因(杨婷等,2021),所有基因的表达水平以内参基因Actin7进行均一化,使用2−ΔΔCt法计算目的基因的相对表达量。
1.9统计分析
采用SPSS 25.0进行显著性分析,使用GraphPad Prism 9.0制图。
2结果与分析
2.1铁十字秋海棠R2R3-MYB基因家族成员鉴定结果
通过比对分析拟南芥和铁十字秋海棠R2R3-MYB基因家族蛋白的氨基酸序列和保守结构域,从铁十字秋海棠中共鉴定出130个MYB基因家族成员,依据结构域类型进行分类,其中属于1R-MYB类型有13个,R2R3-MYB类型有110个,3R-MYB类型有6个,而4R-MYB类型仅有1个。由于R2R3-MYB是MYB基因家族中数量最多的一类,并且在植物转录调控研究中扮演重要角色,因此,本研究选择铁十字秋海棠110个R2R3-MYB基因家族成员作为研究对象,为方便识别和引用,根据在基因组中的序列号顺序将其命名为BmaMYB1~BmaMYB110。
2.2铁十字秋海棠R2R3-MYB基因家族蛋白理化性质分析结果
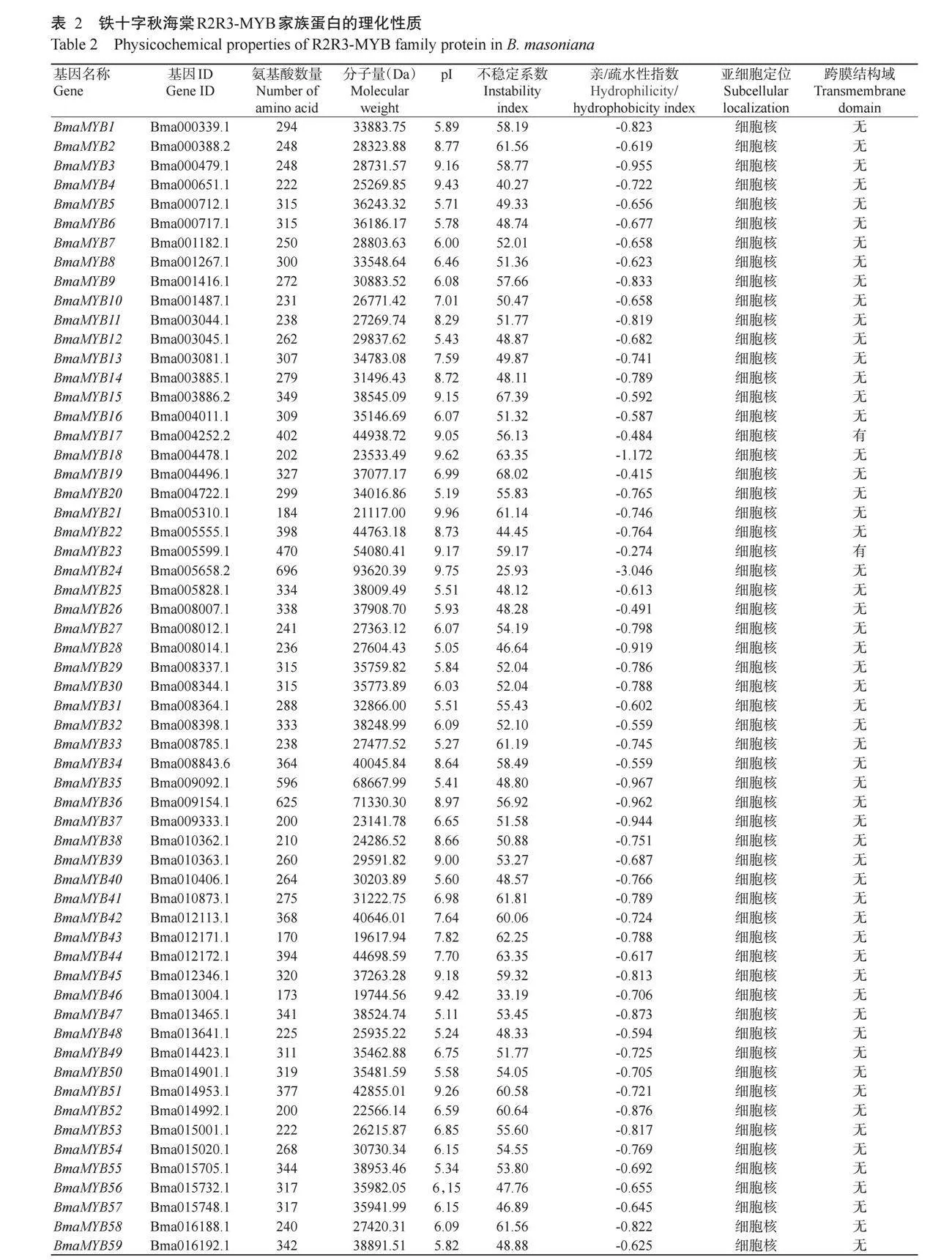
通过ExPASy对铁十字秋海棠110个R2R3-MYB家族蛋白进行理化性质预测分析,结果如表2所示。铁十字秋海棠R2R3-MYB基因家族成员编码的氨基酸数量为128~870个,差异较大;分子量为14660.85~97435.93 Da;理论等电点(pI)为4.92~10.10,其中64个BmaMYBs蛋白的pI小于7,为酸性蛋白;108个BmaMYBs蛋白不稳定系数大于40,为不稳定蛋白;102个BmaMYBs蛋白亲/疏水性指数小于-0.5,表明绝大多数为疏水性蛋白。根据Cell-PLoc 2.0预测结果显示,110个BmaMYBs均定位在细胞核。通过TMHMM对110个BmaMYBs蛋白进行蛋白跨膜结构域预测结果显示,有108个蛋白无跨膜结构域,仅2个蛋白(BmaMYB17和BmaMYB23)存在明显的跨膜结构域。
2.3铁十字秋海棠R2R3-MYB基因家族染色体定位
为探究铁十字秋海棠R2R3-MYB家族基因的进化关系,利用TBtools对110个R2R3-MYB家族基因进行染色体定位,根据基因位置信息获得R2R3-MYB基因在铁十字秋海棠染色体上的定位图(图2),铁十字秋海棠110个R2R3-MYB家族基因在15条染色体上均有分布,但每条染色体上分布的基因数量各不相同,数量范围为3~13。部分R2R3-MYB家族基因在同一染色体上的相同区域集中分布,例如BmaMYB29、BmaMYB30、BmaMYB31和BmaMYB32基因在12号染色体(Chr12)下端紧密分布。
2.4铁十字秋海棠R2R3-MYB家族基因系统发育进化树分析结果
通过MEGA 11.0中Clustal W对铁十字秋海棠R2R3-MYB家族110个蛋白氨基酸序列和拟南芥R2R3-MYB家族(Dubos etal.,2010)126个蛋白氨基酸序列进行多重比对,并采用NJ构建系统发育进化树,结果(图3)显示,根据拟南芥MYB转录因子的分组方法(Dubos etal.,2010),将铁十字秋海棠R2R3-MYB划分为33个亚组,用B1~B33表示。铁十字秋海棠B1亚组(BmaMYB15、BmaMYB68、BmaMYB76、BmaMYB86和BmaMYB100)与拟南芥S21亚组(AtMYB52、AtMYB54和AtMYB69)聚为一类;B19亚组(BmaMYB10、BmaMYB18、BmaMYB38、Bma-MYB53、BmaMYB97、BmaMYB99和BmaMYB109)与拟南芥S6亚组(AtMYB75、At-MYB90、AtMYB113和AtMYB114)属于同一分支;B21亚组(BmaMYB55和BmaMYB73)与拟南芥S7亚组(AtMYB12和AtMYB111)聚为一类。
2.5铁十字秋海棠R2R3-MYB家族基因结构及其编码蛋白保守基序分析结果
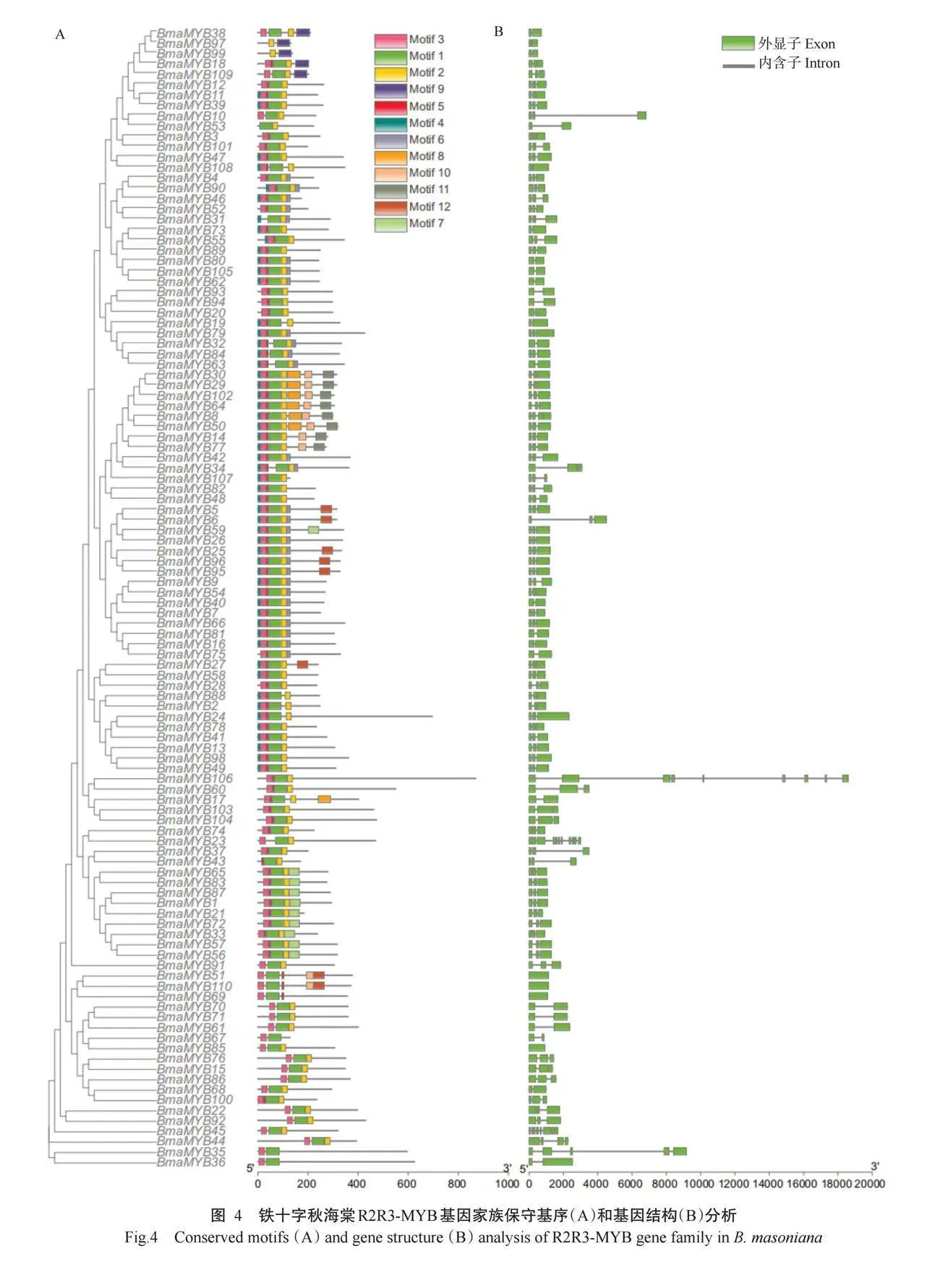
对铁十字秋海棠R2R3-MYB基因家族成员进行基因结构分析结果如图4-B所示。由于铁十字秋海棠GFF文件并未对其非编码区(UTR)进行注释,因此图中仅展示了各基因家族成员之间的CDS和内含子区域。铁十字秋海棠R2R3-MYB基因家族的内含子数量为0~10个,约63%的成员有2个内含子,3个成员(BmaMYB51、BmaMYB69和BmaMYB110基因)无内含子结构。
蛋白保守基序分析结果(图4-A)显示,特异性基序数量设置为12,分别为Motif 1~Motif 12。约92%成员的Motif分布在蛋白序列的N端,少数成员的Motif分布在序列中间以及C端。另外,约94%的成员均含有Motif 1、Motif 2和Motif 3,表明这3个Motifs是铁十字秋海棠R2R3-MYB家族中较为保守的基序。
2.6铁十字秋海棠R2R3-MYB基因家族启动子顺式作用元件分析结果
由图5可知,所有启动子均含有大量光响应元件,推测其对光诱导会产生响应。此外,该基因家族成员启动子的顺式作用元件还包含激素响应元件和逆境胁迫响应元件。说明R2R3-MYB基因家族的表达受到复杂机制的调控。
2.7 BmaMYBs基因表达模式分析结果
研究表明,花青素苷的合成在铁十字秋海棠叶斑生长发育过程中发挥重要作用(杨婷等,2021)。对位于B19亚组中的7个可能参与调控铁十字秋海棠花青苷积累的基因BmaMYB10、BmaMYB18、Bma-MYB38、BmaMYB53、BmaMYB97、BmaMYB99和Bma-MYB109进行实时荧光定量PCR检测,分析这7个基因在铁十字秋海棠叶片3个关键生长发育时期中叶斑区和非叶斑区的相对表达量,结果(图6)显示,BmaMYB10基因在叶片3个关键生长发育时期叶斑区和非叶斑区的相对表达量均无显著差异(Pgt;0.05,下同)。在S1时期,叶斑区和非叶斑区BmaMYB18、BmaMYB38和BmaMYB99基因的相对表达量无显著差异;在S2和S3时期,叶斑区BmaMYB18和Bma-MYB38基因的相对表达量均显著高于非叶斑区(Plt;0.05,下同),而BmaMYB99基因在叶斑区的相对表达量却显著低于非叶斑区。S1~S3时期中,Bma-MYB53和BmaMYB97基因在叶斑区的相对表达量从S1到S3时期呈逐渐升高趋势,且在3个时期叶斑区2个基因的相对表达量均显著高于非叶斑区,叶斑区BmaMYB53基因的相对表达量较非叶斑区高达19.41~131.99倍。3个时期叶斑区BmaMYB109基因的相对表达量均显著高于非叶斑区,在S2时期差异最大,为非叶斑区的3.16倍。
3讨论
本研究通过生物信息学方法,首次对铁十字秋海棠R2R3-MYB基因家族成员进行鉴定,共鉴定出110个R2R3-MYB家族基因,其数量少于拟南芥(126个)(Stracke et al.,2001)、玉米(200个)(Dias et al.,2003)和番茄(127个)(Li etal.,2016),多于水稻(102个)(Chen et al.,2006)和黄瓜(99个)(Li etal.,2012)。研究表明,在马铃薯(李元铭,2021)、假俭草(蒋宇佳等,2023)和黄瓜(郭玉婷和杜长霞,2024)中大多数R2R3-MYB家族基因分布在染色体的两端,这种现象可能跟基因的串联复制有关。本研究发现铁十字秋海棠中也存在类似现象,110个家族成员分布在15条染色体上,且大部分基因家族成员聚集在染色体的两端形成基因簇,推测通过基因串联复制可扩大基因家族数量以便发挥更为丰富的功能。
R2R3-MYB转录因子在植物功能研究中发挥重要作用,尤其在调控花青苷生物合成方面(Dubos et al.,2010)。Zuluaga等(2008)研究发现拟南芥中属于不同亚族的R2R3-MYB转录因子能激活或抑制花青苷生物合成途径中的关键基因,而S6亚组成员对花青苷的生物合成具有正调控。宋杨等(2015)对越橘(Vaccinium corymbosum)中21个R2R3-MYB基因家族构建系统发育进化树,结果发现在S4、S5和S6亚组中有6个基因与花青苷生物合成有关。本研究将铁十字秋海棠110个R2R3-MYB家族成员与拟南芥的126个R2R3-MYB家族成员建立系统发育关系,并参照拟南芥R2R3-MYB转录因子的分组方式,将铁十字秋海棠110个R2R3-MYB分为33个亚组,其中有23个亚组成员可以归类到拟南芥亚家族中。铁十字秋海棠B1亚组(BmaMYB15、BmaMYB68、BmaMYB76、BmaMYB86和BmaMYB100)与拟南芥S21亚组(AtMYB52、AtMYB54和AtMYB69)聚为一类,拟南芥S21亚组成员是致力于纤维细胞细胞壁增厚的正调节因子(Zhong et al.,2008),因属于同一亚组的蛋白通常具有相似且保守的功能,因此推测铁十字秋海棠B1亚组成员也具有相似功能。拟南芥中S7亚组(AtMYB12和AtMYB111)参与调控所有组织中黄酮醇的生物合成,铁十字秋海棠B21亚组(BmaMYB55和BmaMYB73)与拟南芥S7亚组聚为一类,表明铁十字秋海棠B21亚组成员也可能参与调控黄酮醇的生物合成(Dubos et al.,2010)。位于铁十字秋海棠B19亚组中的BmaMYB53等转录因子与拟南芥中调控花青苷合成的MYB转录因子(S6亚组)聚为一类。位于同一分支两个物种的基因家族成员可能具有部分相同功能,拟南芥S6亚组可调控花青苷的合成(Gonzalez et al.,2008),推测铁十字秋海棠B19亚组也参与花青苷物质的合成。崔卫华和管开云(2013)研究发现,花青素苷的合成分布可能会影响秋海棠叶片的斑纹结构,因此推测B19亚组成员是影响铁十字秋海棠叶斑形成的关键调控因子。
铁十字秋海棠R2R3-MYB基因家族成员启动子顺式作用元件主要包含3类:光响应元件、激素响应元件和逆境胁迫响应元件。光响应元件可能与铁十字秋海棠为阴生植物,在弱光环境中更适合生存等因素有关。薛珍珍等(2023)对铁十字秋海棠GASA基因家族启动子顺式作用元件进行分析,发现除了光响应元件以外,GASA基因家族启动子还包含与环境信号有关的低温和干旱胁迫响应元件,以及与植物激素响应有关的顺式作用元件,本研究结果与之类似,推测R2R3-MYB基因家族在铁十字秋海棠生长发育及抵御非生物胁迫等方面发挥重要作用。
花青苷生物合成途径中调控转录因子高表达能导致植物呈现深色。桃叶的红色是由于花青苷积累造成,通过分析在红色和绿色叶片相关PpMYBs基因的相对表达量差异,发现PpMYB10.4基因能调节桃叶中花青苷色素沉着(Yinget al.,2014);对蝴蝶兰(Phalaenopsis orchids)的黑色花斑进行研究,发现在PeMYB11基因启动子中存在1个可以插入到PeMYB11基因上游调控区的反转座子,造成PeMYB11基因表达量增加超过2倍,从而使黑色花瓣中花青苷高度积累(Hsu et al.,2019);在矮牵牛中,PaMYB33/AN2基因属于P11(S6)亚组,参与类黄酮生物合成,在色素沉着过程中该基因的表达水平升高(Chen et al.,2021)。本研究中,对可能与铁十字秋海棠花青苷生物合成有关的7个BmaMYBs基因进行实时荧光定量PCR检测,结果发现BmaMYB10基因在铁十字秋海棠叶片叶斑区和非叶斑区的相对表达量无显著差异,BmaMYB18、BmaMYB38、BmaMYB97、BmaMYB99和BmaMYB109基因在叶斑区和非叶斑区的相对表达量虽然有差异,但5个基因的相对表达量较低,推测这5个基因并非关键调控基因。BmaMYB53基因在叶斑区的表达量随着生长发育阶段发生明显变化,其相对表达量逐渐升高,叶斑区该基因的相对表达量较非叶斑区高19.41~131.99倍,且与铁十字秋海棠叶片生长发育过程中,叶斑的颜色呈逐渐由浅变深的变化趋势一致,因此推测Bma-MYB53在铁十字秋海棠叶斑形成过程中发挥重要作用,具有调控花青苷生物合成的功能,可为进一步探究铁十字秋海棠叶斑形成机理提供理论依据。
4结论
在铁十字秋海棠基因组中鉴定出110个R2R3-MYB基因家族成员,通过对铁十字秋海棠与拟南芥的R2R3-MYB家族成员构建系统发育进化树获得的7个与花青苷合成分布有关的BmaMYBs基因,其中,3个叶片关键生长发育时期的叶斑区与非叶斑区BmaMYB53基因的相对表达量差异最大,推测该基因是调控铁十字秋海棠叶斑形成的关键基因。
参考文献(References):
崔卫华,管开云.2013.中国秋海棠属植物叶片斑纹多样性研究[J].植物分类与资源学报,35(2):119-127.[Cui W H,Guan K Y.2013.Diversity of leaf variegation in Chinese begonias[J].Plant Diversity and Resources,35(2):119-127.]doi:10.7677/ynzwyj201312118.
崔晓娜,陈美君,曹园园,周舒浩,李潇楠,张海荣.2023.泛素化与SUMO化修饰在脱落酸信号转导中的调控作用研究进展[J].河南农业大学学报,57(5):726-735.[Cui X N,Chen M J,Cao Y Y,Zhou S H,Li X N,Zhang H R.2023.Research progress on the regulatory roles of ubiqui-tylation and SUMOylation in abscisic acid signaling[J].Journal of Henan Agricultural University,57(5):726-735.]doi:10.16445/j.cnki.1000-2340.20230505.001.
郭玉婷,杜长霞.2024.黄瓜R2R3-MYB亚家族鉴定及生物信息学分析[J].浙江农林大学学报,41(2):286-296.[Guo Y T,Du C X.2024.Identification and bioinformatics analysis of R2R3-MYB subfamily in cucumber[J].Journal of Zhejiang Aamp;F University,41(2):286-296.]doi:10.11833/j.issn.2095-0756.20230278.
蒋宇佳,于元平,孙向一,周敏,吴春妍,李玉华,刘明稀.2023.假俭草R2R3-MYB基因家族的鉴定及其在干旱胁迫下的表达模式分析[J].草地学报,31(9):2628-2641.[Jiang Y J,Yu Y P,Sun X Y,Zhou M,Wu C Y,Li Y H,Liu M X.2023.Identification of R2R3-MYB gene family and analysis of its expression pattern in centipedegrass under drought stress[J].ActaAgrestia Sinica,31(9):2628-2641.]doi:10.11733/j.issn.1007-0435.2023.09.007.
李元铭.2021.马铃薯块茎花色素苷生物合成相关MYB转录因子的挖掘及调控机理研究[D].兰州:甘肃农业大学.[Li Y M.2021.Identification and regulatory mechanism of MYB transcription factors on anthocyanin biosynthesisin potato tuber[D].Lanzhou:Gansu Agricultural Univer-sity.]doi:10.27025/d.cnki.ggsnu.2021.000271.
梁平,宋洪元.2014.植物花青素生物合成转录调控研究进展[J].南方农业学报,45(8):1375-1379.[Ling P,Song H Y.2014.Advances on the transcriptional regulation of anthocyanin biosynthesis in plant[J].Journal of Southern Agriculture,45(8):1375-1379.]doi:10.3969/j.issn.2095-1191.2014.8.1375.
梁玉镯,陈新娜,陈东亮,王森,李彦慧,黄丛林.2022.MYB转录因子调控植物花青素生物合成研究进展[J].江苏农业科学,50(22):55-64.[Liang YZ,Chen X N,Chen D L,Wang S,Li Y H,Huang C L.2022.Research progress on MYB transcription factor regulating plant anthocyanin bio-synthesis[J].Jiangsu Agricultural Sciences,50(22):55-64.]doi:10.15889/j.issn.1002-1302.2022.22.007.
宋杨,刘红弟,张红军,窦连登.2015.越橘果实转录组及R2R3-MYB转录因子分析[J].园艺学报,42(12):2383-2394.[Song Y,Liu H D,Zhang H J,Dou L D.2015.Analysis of transcriptome and R2R3-MYB transcriptionfactors in blueberry fruit[J].Acta Horticulturae Sinica,42(12):2383-2394.]doi:10.16420/j.issn.0513-353x.2015-0373.
许玲,卫培培,张大勇,徐照龙,何晓兰,黄益洪,马鸿翔,邵宏波.2015.大豆转录因子GmMYB111的克隆及功能分析[J].中国农业科学,48(15):3079-3089.[Xu L,Wei P P,Zhang D Y,Xu Z L,He X L,Huang Y H,Ma H X,Shao H B.2015.Expression and function analysis of the transcrip-tion factor GmMYB111 in soybean[J].Scientia Agricul-tura Sinica,48(15):3079-3089.]doi:10.3864/j.issn.0578-1752.2015.15.019.
薛珍珍,关鸿发,李娜,李凌飞,钟春梅.2023.铁十字秋海棠GASA家族全基因组鉴定及叶斑形成的初步探索[J].园艺学报,50(7):1482-1494.[Xue Z Z,Guan H F,Li N,Li L F,Zhong C M.2023.Genome-wide identification of GASA family and preliminary exploration of leaf variega-tion formation in Begonia masoniana[J].Acta Horticul-turae Sinica,50(7):1482-1494.]doi:10.16420/j.issn.0513-353x.2022-0472.
杨婷,薛珍珍,李娜,郎校安,李凌飞,钟春梅.2021.铁十字秋海棠斑叶发育过程内参基因筛选及验证[J].园艺学报,48(11):2251-2261.[Yang T,Xue Z Z,Li N,Lang X A,Li L F,Zhong C M.2021.Reference genes selection and validation in Begonia masoniana leaves of different deve-lopmental stages[J].Acta Horticulturae Sinica,48(11):2251-2261.]doi:10.16420/j.issn.0513-353x.2021-0397.
左鑫.2023.MYB转录因子调控3种地黄属植物花青素合成的分子机制研究[D].郑州:河南农业大学.[Zuo X.2023.Molecular mechanism of MYB transcription factor regulating anthocyanin biosynthesis in three Rehmannia species[D].Zhengzhou:Henan Agricultural University.]doi:10.27117/d.cnki.ghenu.2022.000556.
Albert N W,Davies K M,Lewis D H,Zhang H B,Montefiori M,Brendolise C,Boase M R,Ngo H,Jameson P E,Sch-winn K E.2014.A conserved network of transcriptional activators and repressors regulates anthocyanin pigmenta-tion in eudicots[J].The Plant Cell,26(3):962-980.doi:10.1105/tpc.113.122069.
Chen G Q,He W Z,Guo X X,Pan J S.2021.Genome-wide identification,classification and expression analysis of the MYB transcription factor family in Petunia[J].Interna-tional Journal of Molecular Sciences,22(9):4838.doi:10.3390/ijms22094838.
Chen Y H,Yang X Y,He K,Liu M H,Li J G,Gao Z F,Lin Z Q,Zhang Y F,Wang X X,Qiu X M,Shen Y P,Zhang L,Deng X H,Luo J C,Deng X W,Chen Z L,Gu H Y,Qu L J.2006.The MYB transcription factor superfamily of Ara-bidopsis:Expression analysis and phylogenetic compari-son with the rice MYB family[J].Plant Molecular Bio-logy,60(1):107-124.doi:10.1007/s 11103-005-2910-y.
Chiou C Y,Yeh K W.2008.Differential expression of MYB gene(OgMYB1)determines color patterning in floral tis-sue of Oncidium Gower Ramsey[J].Plant Molecular Bio-logy,66(4):379-388.doi:10.1007/s 11103-007-9275-3.
Davies K M,Albert N W,Schwinn K E.2012.From landinglights to mimicry:The molecular regulation of flower colouration and mechanisms for pigmentation patterning[J].Functional Plant Biology,39(8):619-638.doi:10.1071/FP12195.
Dias A P,Braun E L,McMullen M D,Grotewold M M.2003.Recently duplicated maize R2R3 MYB genes provide evi-dence for distinct mechanisms of evolutionary divergence after duplication[J].Plant Physiology,131(2):610-620.doi:10.1104/pp.012047.
Dubos C,Stracke R,Grotewold E,Weisshaar B,Martin C,Lepiniec L.2010.MYB transcription factors in Arabidop-sis[J].Trends in Plant Science,15(10):573-581.doi:10.1016/j.tplants.2010.06.005.
Gonzalez A,Zhao M Z,Leavitt J M,Lloyd A M.2008.Regula-tion of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seed-lings[J].The Plant Journal,53(5):814-827.doi:10.1111/j.1365-313X.2007.03373.x.
Gu Z Y,Zhu J,Hao Q,Yuan Y W,Duan Y W,Men S Q,Wang Q Y,Hou Q Z,Liu Z A,Shu Q Y,Wang L S.2019.A novel R2R3-MYB transcription factor contributes to petal blotch formation by regulating organ-specific expression of PsCHS in tree peony(Paeonia suffruticosa)[J].Plantamp;Cell Physiology,60(3):599-611.doi:10.1093/pcp/pcy 232.
Hsu C C,Su C J,Jeng M F,Chen W H,Chen H H.2019.A HORT1 retrotransposon insertion in the PeMYB11 pro-moter causes harlequin/black flowers in Phalaenopsis orchids[J].Plant Physiology,180(3):1535-1548.doi:10.1104/pp.19.00205.
Hu X M,Liang Z H,Sun T X,Huang L,Wang Y P,Chan Z L,Xiang L.2024.The R2R3-MYB transcriptional repressor TgMYB4 negatively regulates anthocyanin biosynthesis in tulips(Tulipagesneriana L.)[J].International Journal of Molecular Sciences,25(1):563.doi:10.3390/ijms2501 0563.
Kranz H D,Denekamp M,Greco R,Jin H,Leyva A,Meissner R C,Petroni K,Urzainqui A,Bevan M,Martin C,Smeek-ens S,Tonelli C,Paz-Ares J,Weisshaar B.1998.Towards functional characterisation of the members of the R2R3-MYB gene family from Arabidopsis thaliana[J].The Plant Journal,16(2):263-276.doi:10.1046/j.1365-313x.1998.0 0278.x.
Li L F,Chen X L,Fang D M,Dong S S,Guo X,Li N,Campos-Dominguez L,Wang W G,Liu Y,Lang X A,Peng Y,Tian D K,Thomas D C,Mu W X,Liu M,Wu C Y,Yang T,Zhang S Z,Yang L L,Yang J F,Liu Z J,Zhang L S,Zhang X T,Chen F,Jiao YN,Guo Y L,Hughes M,Wang W,Liu X F,Zhong C M,LiAR,Sunil K S,Yang H M,Ernest W,Sharbrough J,Lisby M,Liu X,Xu X,Soltis D E,de Peer Y V,Kidner C,Zhang S Z,Liu H.2022.Genomes shedlight on the evolution of Begonia,a mega-diverse genus[J].The New Phytologist,234(1):295-310.doi:10.1111/nph.17949.
Li Q,Zhang C J,Li J,Wang L N,Ren Z H.2012.Genome-wide identification and characterization of R2R3MYB fa-mily in Cucumis sativus[J].PLoS One,7(10):e47576.doi:10.1371/journal.pone.0047576.
Li Z J,Peng R H,Tian Y S,Han H J,Xu J,Yao Q H.2016.Genome-wide identification and analysis of the MYB tran-scription factor superfamily in Solanum lycopersicum[J].Plantamp;Cell Physiology,57(8):1657-1677.doi:10.1093/pcp/pcw091.
Liu X F,Xiang L L,Yin X R,Grierson D,Li F,Chen K S.2015.The identification of a MYB transcription factor con-trolling anthocyanin biosynthesis regulation in Chrysanthe-mum flowers[J]Scientia Horticulturae,194:278-285.doi:10.1016/j.scienta.2015.08.018.
Paz-Ares J,Ghosal D,Wienand U,Peterson P A,Saedler H.1987.The regulatory c1 locus of Zea mays encodes a pro-tein with homology to MYB proto-oncogene products and with structural similarities to transcriptional activators[J].The EMBO Journal,6(12):3553-3558.doi:10.1002/j.1460-2075.1987.tb02684.x.
Quattrocchio F,Wing J,van der Woude K,Souer E,de Vetten N,Mol J,Koes R.1999.Molecular analysis of the anthocy-anin2 gene of petunia and its role in the evolution of flower color[J].The Plant Cell,11(8):1433-1444.doi:10.1105/tpc.11.8.1433.
Ramsay N A,Glover B J.2005.MYB-bHLH-WD40 protein complex and the evolution of cellular diversity[J].Trends in Plant Science,10(2):63-70.doi:10.1016/j.tplants.2004.12.011.
Stracke R,Werber M,Weisshaar B.2001.The R2R3-MYB gene family in Arabidopsis thaliana[J].Current Opinionin Plant Biology,4(5):447-456.doi:10.1016/S1369-5266(00)00199-0.
Tanaka Y,Ohmiya A.2008.Seeing is believing:Engineering anthocyanin and carotenoid biosynthetic pathways[J].Cur-rent Opinion in Biotechnology,19(2):190-197.doi:10.1016/j.copbio.2008.02.015.
Walker A R,Davison P A,Bolognesi-Winfield A C,James C M,Srinivasan N,Blundell T L,Esch J J,Marks M D,Gray J C.1999.The TRANSPARENT TESTA GLABRA1 locus,which regulates trichome differentiation and antho-cyanin biosynthesis in Arabidopsis,encodes a WD40 repeat protein[J].The Plant Cell,11(7):1337-1350.doi:10.1105/tpc.11.7.1337.
Wang J W,Gao M L,Li Y H,Wu R H,Zhang K M.2018.High-throughput transcriptome sequencing reveals the role of anthocyanin metabolism in Begonia semperflorens under high light stress[J].Photochemistry and Photobio-logy,94(1):105-114.doi:10.1111/php.12813.
Ying Z,Hui Z,Wang K L,Vimolmangkang S,Espley R V,Wang L,Allan A C,Han Y P.2014.Transcriptome analysis and transient transformation suggest an ancient duplicated MYB transcription factor as a candidate gene for leaf red coloration in peach[J].BMC Plant Biology,14:388.doi:10.1186/s 12870-014-0388-y.
Zhang Y,Butelli E,Martin C.2014.Engineering anthocyanin biosynthesis in plants[J].Current Opinion in Plant Bio-logy,19:81-90.doi:10.1016/j.pbi.2014.05.011.
Zhong R Q,Lee C H,Zhou J L,McCarthy R L,Ye Z H.2008.A Battery of transcription factors involved in the regula-tion of secondary cell wall biosynthesis in Arabidopsis[J].The Plant Cell,20(10):2763-2782.doi:10.1105/tpc.108.061325.
Zuluaga D L,Gonzali S,Loreti E,Pucciariello C,Degl’Inno-centi E,Guidi L,AlpiA,Perata P.2008.Arabidopsis thalia-na MYB75/PAP1 transcription factor induces anthocya-nin production in transgenic tomato plants[J].Functional Plant Biology,35(7):606-618.doi:10.1071/FP08021.
(责任编辑 李洪艳)

