棉花种间杂种F1纤维中关键代谢路径和基因鉴定

摘要 为在表型和遗传上揭示远缘杂种F1纤维的杂种优势并解析产量性状杂种优势不明显的原因,分别对远缘杂种F1与陆地棉亲本发育10 d 的纤维组织样品进行混池取样,利用转录组学解析材料间在遗传转录水平上的差异,采用生物信息学方法鉴定材料间存在明显差异的代谢路径和关键基因。结果显示,杂种F1长纤维细胞数量少,并且种子表面有绿色短绒;陆地棉亲本和远缘杂种F1纤维转录组数据共鉴定到1 128 个上调基因,678 个下调基因;通过基因富集分析发现,差异基因主要富集在苯丙烷生物合成及其分支类黄酮生物合成路径;差异基因的偏亲表达分析显示,杂种F1纤维等位基因偏向海岛棉表达,其中杂种F1的类黄酮代谢路径的二氢黄酮醇-3′, 5′-羟化酶(F3′5′H)基因只表达海岛棉的等位基因。总体来看,海岛棉-陆地棉远缘杂种F1纤维产量性状具有负向杂种优势并且具有明显的绿色短绒,这可能与苯丙烷生物合成和类黄酮生物合成路径所产生的次生代谢物有关。本研究在遗传转录水平挖掘陆地棉-海岛棉远缘杂交F1纤维的杂种优势及其重要的代谢路径和基因,并建立了表型性状与遗传转录及遗传转录与代谢合成的联系。
关键词 棉花; 杂种优势; 纤维; 转录组; 类黄酮代谢; 二氢黄酮醇-3′,5′-羟化酶(F3′5′H)
中图分类号 S562 文献标志码 A 文章编号 1000-2421(2024)06-0191-10
棉花是世界上重要的经济作物之一,棉纤维是最主要的纺织原材料,纤维产量和品质是棉花育种家最关注的性状。目前,陆地棉以纤维高产而占据棉花总产的95% 以上;海岛棉因纤维优质也具有一定的生产面积,在我国,海岛棉主要在新疆的南疆部分地区集中种植。将这2 个栽培种的优势有效结合起来并应用于生产是广大棉花育种家致力于实现的目标。棉花育种家主要采取2 种策略将两者的优异等位基因聚合应用。一种方法是通过经典的种间杂交和连续回交的方式产生遗传渐渗系,从而将海岛棉优异基因导入到陆地棉中[1-4],由于纤维产量和品质性状均属于典型的数量性状,通过渐渗少数几个遗传片段改良纤维产量和品质的效果不佳;另一种是直接通过经典种间杂交的方法产生海岛棉-陆地棉杂种F1,然而该种间杂种优势主要体现在棉株的生物量上,在纤维产量上的优势并不明显[5-6]。
解释杂种优势主要有3 种经典模型:显性模型、超显性模型以及上位互作模型[7]。随着分子生物学和生物技术的快速发展,研究人员从不同角度解析了杂种优势,比如基因组水平、转录组水平、蛋白组水平、代谢组水平等[8-9]。众多研究证据表明种间特异性基因不平衡的表达在杂种优势中起着重要作用,Shao 等[10]在水稻中鉴定到3 270 个对杂种优势有贡献的特异等位基因。最近有学者研究发现生物学通路的表达互补调控拟南芥生物量杂种优势[11]。Li等[12]利用转录组、代谢组和表观组学技术解析了马铃薯的杂种优势现象,研究发现马铃薯在淀粉和糖代谢中表现出显著的显性互补优势,杂交种将更多的能量投入到初生代谢中从而产生杂种优势,其中马铃薯杂交种花朵的颜色也表现出明显的杂种显性优势,表明花青素类物质在杂种马铃薯中积累,因此次生代谢物质的产生和积累也可能是杂种优势的一种表现形式。
棉花纤维的主要成分是纤维素、半纤维素、木质素和果胶等,因此,糖代谢是棉花纤维中的第一大代谢路径。棉花纤维中苯丙烷代谢是仅次于糖代谢的第二大代谢路径[13],据报道,苯丙烷代谢路径主要负责棉花纤维木质素、木聚素和类黄酮等次生代谢物的合成,因此苯丙烷代谢路径与棉花纤维产量及品质密切相关[14-15]。苯丙烷代谢路径中存在3 个关键酶:苯丙氨酸解氨酶(phenylalanine ammonia lyase,PAL)、肉桂酸-4-羟化酶(cinnamic acid 4-hydroxylase,C4H)和4-香豆酸-CoA 连接酶(4-coumarate-CoA ligase, 4CL),分别催化生成肉桂酸、对羟基肉桂酸和对香豆酰辅酶A,这些化合物是下游代谢反应的底物[16]。类黄酮代谢路径也属于苯丙烷代谢路径的主要分支,负责合成黄酮类代谢物,与植物颜色的形成相关,该路径与纤维产量和品质的形成显著相关[17]。一般栽培棉纤维长而颜色洁白,野生棉纤维短且偏棕色,类黄酮代谢路径被认为是棉花在驯化过程中受到选择的一条主要代谢路径。在野生棉和彩棉中,类黄酮代谢物质含量也相对丰富,从而导致纤维呈现出一定的彩色。类黄酮相关的结构基因往往在彩棉中高表达[18],彩棉颜色的深浅往往与其产量品质呈现负相关的关系[19]。Sun 等[20]通过绿棉和白棉的转录组和代谢组数据分析发现,绿棉中苯丙烷代谢路径发挥重要作用,其中4CL 基因显著表达,表明该基因可能参与了相关色素的合成。Li 等[21]鉴定到咖啡酸衍生物、木质素和木酚素在绿色棉中富集。
目前,陆地棉种内杂交在产量上可以表现出一定的杂种优势,中棉所63 和杂交棉H318 都属于陆地棉种内杂交品种,中棉所63 在单株结铃数、铃质量和叶面积载铃量上都要显著高于亲本[22],H318 的杂种优势主要体现在棉铃数、较快的生长速度和干物质积累量方面[23]。而海岛棉与陆地棉远缘杂种的杂种优势主要表现在生物量上,杂种F1的种子大小、株高和叶片显著大于亲本的叶片[24]。理论上利用种间杂交来提高杂种F1 的纤维产量具有可行性,但是在实践中却存在杂种优势不明显并且呈负向效应的现象。为从表型和遗传转录水平鉴定出种间杂种F1纤维的杂种优势因子并解析产量性状杂种优势不明显的原因,本研究将远缘杂种F1 与陆地棉亲本发育10 d 的纤维组织样品分别进行混样测序,转录组测序后进行生物信息学分析,以期鉴定控制海陆杂种F1纤维产量的关键代谢路径以及关键基因,为棉花遗传改良奠定基础。
1 材料与方法
1.1 试验材料
以陆地棉(Gossypium hirsutum L.)新陆中20 号(以下简称LDM)为母本,海岛棉3-79(G. bar⁃badense L.)为父本,2020 年7 月在江西农业大学试验田进行杂交形成F1。2021 年在江西高安原种场棉花试验田进行材料种植和性状考察,本试验采用育苗移栽,等行种植的方式,分别种植母本、父本和F1,行距1 m,株距0.2 m,亲本和F1分别种植20 株,田间管理采用大田常规管理方式。
1.2 表型调查与统计分析
于2021 年10 月13 日收获亲本和F1。收回的棉花进行晾晒,晒干后于11 月22 日进行室内考种,考种项目有单铃籽棉质量、单铃皮棉质量、百粒质量、短绒颜色和衣分。使用Excel 软件对材料各类性状的数据进行统计分析。
中亲优势(mid-parent heterosis,Hm)和最优亲本优势(best parent heterosis,Hb)通过杂种F1的表型与最优亲本表型(Fb)或是双亲(P1,P2)的表型平均值(Mv)进行比较计算:Mv=(P1+P2)/2;Hm=(F1-Mv)/Mv;Hb=(F1-Fb)/Fb。
1.3 转录组数据分析
2021 年8 月10 日分别对陆地棉LDM 和F1材料发育10 d 的纤维材料进行取样,每个材料2 个重复,每个重复为包含3 个单株的混池样品。取样后迅速置于干冰中送往上海美吉生物医药科技公司,样品经过研磨,提取RNA,建立RNA 测序文库后,利用HiSeq2000 平台进行测序,将获得的原始数据使用fastp 软件进行质控获得过滤后数据,通过生物信息学软件Bowtie2 将该数据比对到陆地棉参考基因(https://cottonfgd.org/about/download.html)。基于所选参考基因组序列,使用StringTie 或Cufflinks 软件对测序数据进行拼接,并与原有的基因组注释信息进行比较,寻找以前未被注释的转录区,发掘该物种的新转录本和新基因,从而补充和完善基因组注释信息。将基因/转录本与6 大数据库(NR、Swiss-Prot、Pfam、EggNOG、GO 和KEGG)进行比对,全面获得基因/转录本的注释信息并对各数据库注释情况进行统计。使用软件(RSEM,http://deweylab.github. io/RSEM/; Kallisto, https://pachterlab.github. io/kallisto/; Salmon, https://combine-lab.github.io/salmon/)分别对基因和转录本的表达水平进行定量分析,以便后续分析不同样本间基因/转录本的差异表达情况,并结合序列功能信息揭示基因的调控机制。采用软件Goatools 对基因集中的基因进行GO(gene ontology,基因本体)富集分析,从而获得该基因集中的基因主要具有哪些GO 功能。使用Fisher 精确检验,当经过校正的Plt;0.05 时,认为此GO 功能存在显著富集情况。采用R 脚本对基因集中的基因/转录本进行KEGG 通路富集分析,计算原理同GO 功能富集分析,当经过校正的Plt;0.05 时,认为此KEGG 路径存在显著富集情况。
1.4 等位基因偏亲表达和RT-PCR检测
将转录组原始数据过滤后比对到参考基因组上,利用Bowtie2 软件比对到陆地棉参考基因组上[25],然后利用GATK 软件(https://software.broadinstitute.org/gatk/download/)获得SNP 和Indel变异信息,再利用VCFtools 软件提取等位位点信息,只保留变异位点质量大于500 并且在F1材料中的测序深度大于10,在亲本中属于纯合的位点进行等位基因的表达检测。偏亲表达计算公式:R=(P1-P2)/(P1+P2),其中,P1 表示亲本1 的等位位点在F1中的测序深度,P2 表示亲本2 的等位位点在F1中的测序深度,若Rgt;0.33 则该基因偏向P1 表达,若Rlt;−0.33,则该基因偏向P2 表达,此处阈值设置为0.33 相当于两亲本间的表达差异大于2 倍。
对鉴定到的目标基因,通过CottonFGD(https://cottonfgd.org)数据库获得其在陆地棉中的转录本和CDS(coding sequence)序列,利用DNAMAN软件分别将2 个基因各自的转录本和CDS 序列进行比对,确定2 个基因的UTR(un-translated region)序列,利用Prier Primer 5.0 软件分别在每个基因的5′UTR 和3′UTR 设计正向引物和反向引物,其中Ubq7 基因为参照(表1)。利用UTR 引物分别在陆地棉和海岛棉-陆地棉F1 的纤维材料RNA 反转录的cDNA 中进行RT-PCR 检测。PCR 扩增所需的试剂由上海Yeasen 公司提供(产品货号:1010ES03)。PCR 扩增体系为反应总体积为50 μL 的体系:模板cDNA 10 μL,Primer 正向 1.0 μL( 浓度为0.1μmol/L),Primer 反向 1.0 μL,2×Hieff®RobustPCR Master Mix 13.0 μL。PCR 反应条件为:94 ℃预变性5 min;94 ℃变性10 s,55 ℃退火20 s,72 ℃延伸90 s,35 个循环;72 ℃再延伸5 min,4 ℃保温。PCR产物用1% 的琼脂糖凝胶电泳检测。
2 结果与分析
2.1 表型观测与统计
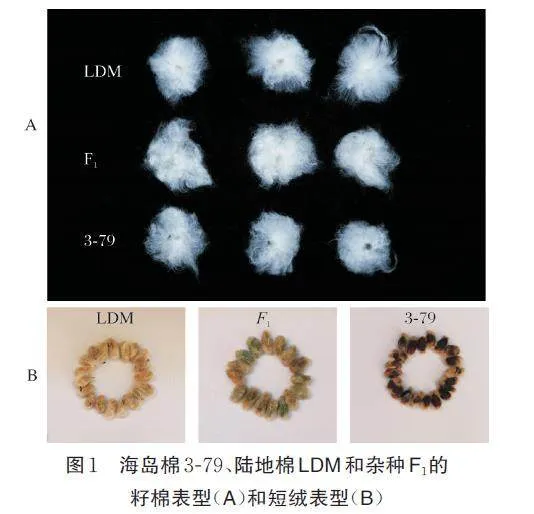
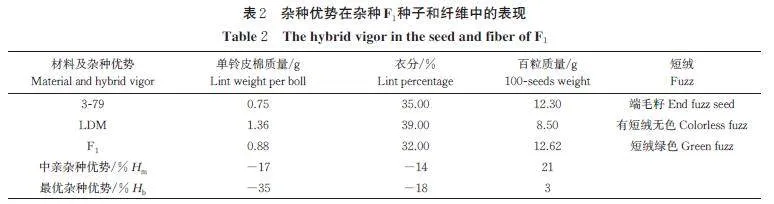
陆地棉LDM、海岛棉3-79 和杂种F1的籽棉表型鉴定结果显示,陆地棉LDM 具有短绒,长纤维数量比F1和海岛棉3-79 要多;F1具有明显的绿色短绒表型;而海岛棉3-79 属于端毛籽材料(图1)。考种结果显示,杂种F1 在百粒质量上具有显著的中亲优势和3% 的优亲优势,在单铃皮棉质量上反而呈现负向中亲优势(−7%)和负向优亲优势(−35%),在衣分上也表现出负向中亲优势(−14%)和负向超亲优势(−18%)(表2)。因此,杂种F1在纤维产量性状上呈现负向杂种优势,而在纤维短绒的有无及颜色上呈现出杂种显性的特征。
2.2 差异基因鉴定
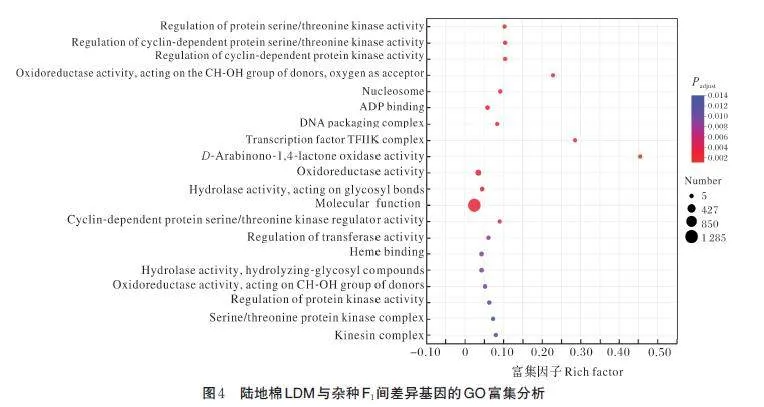
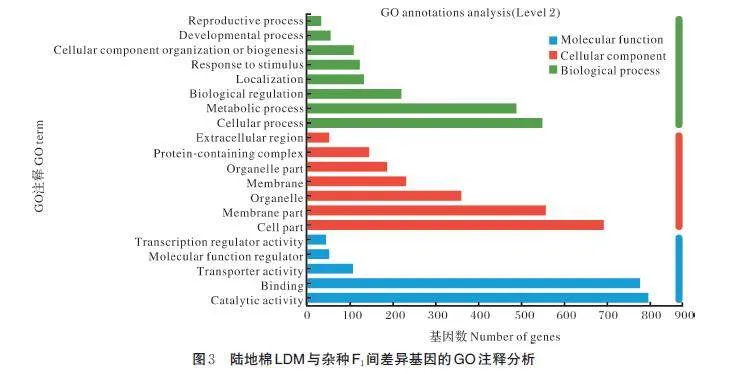
为了比较陆地棉亲本LDM 和杂种F1 遗传上的差异,分别取2 个材料发育10 d 的纤维进行转录组测序。利用比对到参考基因的表达谱数据进行主成分分析发现LDM 和F1的样本间可以明显地区分开,而LDM 和F1的材料内部的2 个重复在横坐标上(PC1)的位置差异不大,表明通过混池取样的方法可以保证组内样本间良好的重复性(图2A)。转录组数据经过比较分析,共在LDM 和F1间鉴定到1 806 个差异基因(differential expression genes ,DEGs),其中1 128 个基因在F1 发育10 d 的纤维中上调表达,678个基因在F1 发育10 d 的纤维中下调表达(图2B)。通过差异基因的基因本体注释发现在分子功能(molecular function)中,有796 个DEGs 具有催化活性的功能,有777 个DEGs 具有绑定的功能;在细胞组分(cellular component)中,692 个基因在细胞部位发挥功能,557 个基因在细胞膜部位发挥功能;在生物过程中,549 个基因参与细胞过程,488 个基因参与代谢过程(图3)。差异基因通过GO 富集分析鉴定到具有激酶活性的一类基因被显著富集;富集因子数据表明D-阿拉伯糖-1,4-内酯氧化酶活性的一类基因富集最显著,另外还有转录因子TFIIK 复合体和氧化还原酶活性的一类基因被显著富集;在富集数量上,分子功能相关的一类基因富集最多(图4)。
2.3 差异基因的代谢路径富集分析
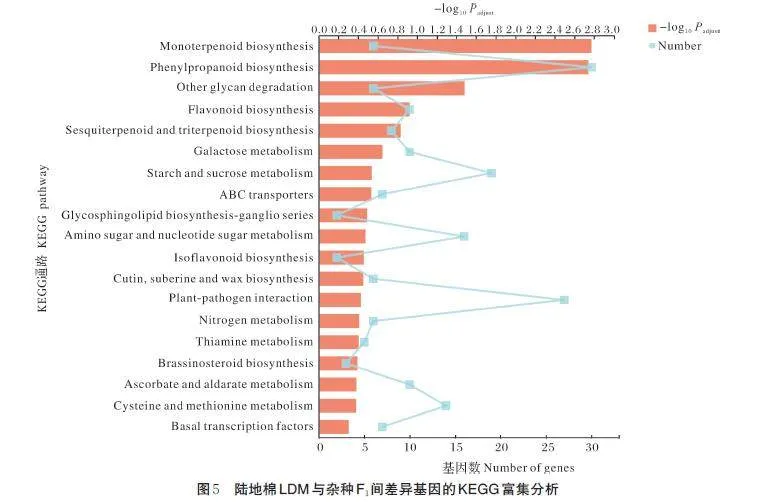
对1 806 个DEGs 进行KEGG 分析,鉴定到6 个DEGs 被富集到单萜类化合物生物合成代谢路径(map00902),30 个DEGs 被富集到苯丙烷生物合成代谢路径(map00940),6 个DEGs 被富集到其他多糖降解代谢路径(map00511),10 个DEGs 被富集到类黄酮代谢路径(map00941),8 个DEGs 被富集到倍半萜和三萜类化合物生物合成(图5)。从代谢路径富集结果中可以看到苯丙烷代谢和类黄酮代谢所富集的基因数量较多,并且类黄酮代谢路径属于苯丙烷代谢路径的分支,杂种F1和陆地棉LDM 显著的差异可能来自于这2 条代谢路径的差异表达。
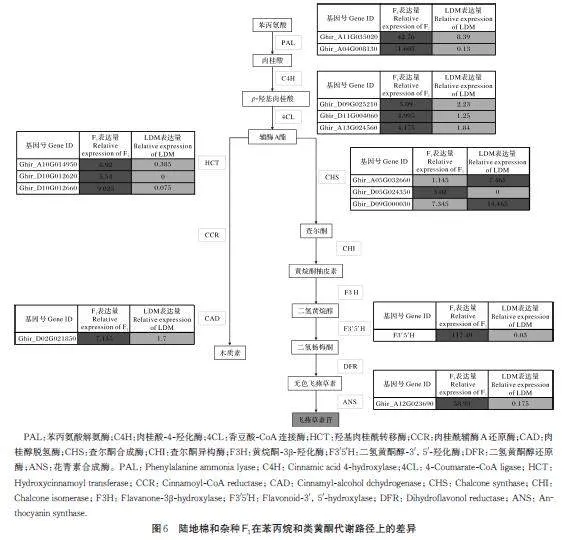
进一步分析苯丙烷和类黄酮代谢分支路径结构基因,结果显示,苯丙烷代谢路径的2 个PAL 和3 个4CL 主干基因分别在F1 中显著上调表达,合成木质素分支路径的3 个HCT 基因和2 个CAD 基因也在F1中显著上调表达;类黄酮代谢路径分析表明,有1个CHS 基因,1 个F3'5'H 基因和1 个ANS 基因分别在F1中显著上调表达,2 个CHS 基因在亲本LDM 中上调表达(图6)。综合来看,由于CAD 基因是木质素合成的关键基因,而F3'5'H 基因是飞燕草素合成的关键基因,因此,杂种F1纤维中的苯丙烷代谢和类黄酮代谢路径的代谢产物可能分别主要流向木质素和花青素中飞燕草素的生物合成。
2.4 等位基因偏亲表达和关键基因鉴定
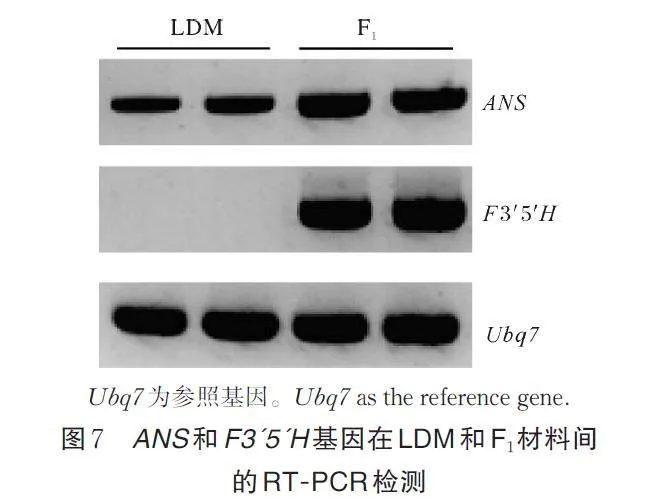
经过筛选过滤,在1 806 个DEGs 中,共鉴定到1 053 个DEGs 中存在3 833 个高质量SNP 位点,对获得的每个SNP 等位变异的测序深度进行等位基因偏亲表达分析,发现有317 个DEGs 的等位基因在杂种F1中处于相对平衡的表达状态,736 个DEGs 呈现出偏亲表达的现象,其中有515 个基因偏向海岛棉3-79 表达,221 个基因偏向陆地棉LDM 表达。本研究鉴定到苯丙烷代谢路径中的HST、PAL 和CAD1 基因偏向海岛棉等位位点表达,类黄酮代谢路径的ANS 和F3'5'H 偏向海岛棉表达。本研究重点对陆地棉LDM 和杂种F1中的ANS 和F3'5'H 进行RT-PCR检测,结果表明ANS 和F3'5'H 在2 个材料间表达差异显著,尤其是F3'5'H 基因只在杂种F1中表达,对F1中扩增得到的产物进行测序,经序列比对表明该基因来自于海岛棉3-79(图7)。
3 讨论
3.1 苯丙烷代谢路径影响种间杂种F1纤维发育
杂种优势是植物中普遍存在的现象,目前为止还没有模型或假说可以完美地解释杂种优势现象[26]。本研究从表型和遗传转录水平充分挖掘种间杂种F1纤维中的杂种优势。研究表明虽然杂种F1在纤维产量上呈现负向杂种优势,但是杂种F1纤维在苯丙烷和类黄酮代谢路径上优势表达(表2 和图5)。分析代谢通路介导的作物农艺性状表型是分析作物杂种优势的有效方法之一[27],前人研究表明,绿色纤维的表型和苯丙烷代谢路径的主干基因4CL 及其代谢呈显著相关[20-21],因此,本研究中杂种F1的负向杂种优势和绿色短绒表型极有可能和优势表达的苯丙烷代谢路径有关。
苯丙烷代谢路径是众多次生代谢物质产生的主要代谢路径,其中木质素和类黄酮类物质是最主要的2 类代谢物[28]。Fan 等[29]研究表明CAD 基因与木质素合成相关,一般在纤维发育的次生壁形成过程中表达,而木质素的多少和纤维的长度一般呈负相关;C4H 是在纤维次生壁加厚时期表达[30];4CL 基因的超表达可以促进木质素沉积[31]。前人研究表明,黄酮类化合物柚皮素的富集会导致棉花纤维的变短[32],而彩棉颜色深浅往往与纤维产量和品质呈负相关[19]。孙君灵等[33]通过QTL 定位方法,发现棕色纤维基因(Lc1)在关于纤维强度、长度、细度等的QTL,能够降低纤维强度、长度和细度,这反映了Lc1 基因与纤维品质成负相关。对于棉花来说,纤维细胞发育的速度和数量至关重要,有研究表明陆地棉开花后1 d 纤维突起的密度比海岛棉多[34],这可能是导致陆地棉长纤维多而产量有所提升的原因。
本研究发现类黄酮相关基因在杂种F1中显著上调表达(图6),可能造成了F1纤维中类黄酮物质的积累而影响纤维的发育,尤其短绒具有肉眼可见的绿色,表明有大量色素的积累,这些色素可能抑制了部分纤维细胞发育为长纤维。
3.2 杂种F1的种间等位基因的偏亲表达
双亲杂交后产生的杂交种与亲本比较,性状或者基因的表达往往可以呈现出显性或者超显性等表达模式[35-37],但是对于具体某一等位基因在F1中的表达不能很直观地呈现出来。转录组测序不仅可以快速检测样本间全基因组的差异基因,同时为研究人员检测杂种F1等位基因的特异表达提供了便捷的途径。Shao 等[10]、Fu 等[38]曾利用转录组测序鉴定到特异等位基因的表达可能介导了杂种优势形成的机制。本研究对差异基因进行检测发现1 053 个差异基因中有736 个差异基因是偏亲表达的,并且偏亲表达的差异基因中有515 个基因是偏向海岛棉表达,这也可能是造成杂种F1纤维的表型偏向海岛棉的原因之一,从而导致其产量较低。等位基因的偏亲表达现象在类黄酮代谢路径的结构基因中尤其明显。本研究在类黄酮代谢路径中鉴定到F1 杂种中表达的F3'5'H 等位基因来自于海岛棉,该基因是产生飞燕草素的关键基因,也是花卉呈现蓝色的一种主要色素[39],该基因可以为将来的彩棉基因工程育种提供重要的基因来源。
本研究结果表明,杂种F1 的纤维产量呈负向杂势,具有明显的绿色短绒;差异基因富集分析表明苯丙烷代谢路径及其分支类黄酮代谢路径影响了杂种F1纤维的表现;杂种F1的等位基因偏向海岛棉等位基因表达,这也可能是杂种F1纤维产量低的原因;本研究鉴定到的苯丙烷代谢和类黄酮路径结构基因是杂种F1 在次生代谢中具有杂种优势的有力证明,尤其F3'5'H 基因可以为将来彩棉遗传改造提供基因来源。
参考文献 References
[1] ZHANG J F,PERCY R G,MCCARTY J C.Introgression geneticsand breeding between upland and Pima cotton:a review[J].Euphytica,2014,198(1):1-12.
[2] CHEN Y,LIU G D,MA H H,et al. Identification of introgressedalleles conferring high fiber quality derived from Gossy⁃pium barbadense L.in secondary mapping populations of G.hir⁃sutum L.[J/OL]. Frontiers in plant science,2018,9:1023[2024-05-27].https://doi.org/10.3389/fpls.2018.01023 .
[3] BROWN N,KUMAR P,SINGH R,et al. Evaluation of achromosome segment from Gossypium barbadense harboringthe fiber length QTL qFL-Chr. 25 in four diverse upland cottongenetic backgrounds[J].Crop science,2019,59(6):2621-2633.
[4] ZHU D,LI X M,WANG Z W,et al.Genetic dissection of anallotetraploid interspecific CSSLs guides interspecific geneticsand breeding in cotton[J/OL].BMC genomics,2020,21(1):431 [2024-05-27]. https://doi. org/10.1186/s12864-020-06800-x .
[5] 华兴鼐,周行,黄骏麒,等. 海岛棉与陆地棉杂种一代优势利用的研究[J]. 作物学报,1963,2(1):1-28.HUA X, ZHOUH, HUANG J, et al.The use of heterosis in the interspecificF1 hybrids between G. hirsutum and G. barbadense[J]. Actaagronomica sinica,1963, 2(1):1-28 (in Chinese with Englishabstract).
[6] 菲力申. 陆地棉与海岛棉杂种优势利用研究[D]. 南宁:广西大学,2008.FEI L S.Studies on heterosis utilisation betweenupland cotton and island cotton[D].Nanning:Guangxi University,2008( in Chinese with English abstract).
[7] BIRCHLER J A,YAO H,CHUDALAYANDI S,et al.Heterosis[J].The plant cell,2010,22(7):2105-2112.
[8] HUANG X H,YANG S H,GONG J Y,et al.Genomic architectureof heterosis for yield traits in rice[J].Nature,2016,537(7622):629-633.
[9] LIU J,LI M J,ZHANG Q,et al.Exploring the molecular basisof heterosis for plant breeding[J]. Journal of integrative plantbiology,2020,62(3):287-298.
[10] SHAO L,XING F,XU C H,et al.Patterns of genome-wideallele-specific expression in hybrid rice and the implications onthe genetic basis of heterosis[J].PNAS,2019,116(12):5653-5658.
[11] LIU W W,HE G M,DENG X W.Biological pathway expressioncomplementation contributes to biomass heterosis in Ara⁃bidopsis[J/OL].PNAS,2021,118(16):e2023278118[2024-05-27]. https://doi.org/10.1073/pnas.2023278118.
[12] LI D W,LU X Y,ZHU Y H,et al.The multi-omics basis ofpotato heterosis[J].Journal of integrative plant biology,2022,64(3):671-687.
[13] GOU J Y,WANG L J,CHEN S P,et al.Gene expression andmetabolite profiles of cotton fiber during cell elongation andsecondary cell wall synthesis[J].Cell research,2007,17(5):422-434.
[14] AL-GHAZI Y,BOUROT S,ARIOLI T,et al.Transcript profilingduring fiber development identifies pathways in secondarymetabolism and cell wall structure that may contribute tocotton fiber quality[J].Plant amp; cell physiology,2009,50(7):1364-1381.
[15] HAN L B,LI Y B,WANG H Y,et al.The dual functions ofWLIM1a in cell elongation and secondary wall formation in developingcotton fibers[J].The plant cell,2013,25(11):4421-4438.
[16] 尚军,吴旺泽,马永贵. 植物苯丙烷代谢途径[J]. 中国生物化学与分子生物学报,2022,38(11):1467-1476.SHANG J,WU W Z,MA Y G.Phenylpropanoid metabolism pathway inplants[J].Chinese journal of biochemistry and molecular biology,2022,38(11):1467-1476 (in Chinese with English abstract).
[17] 涂礼莉,谭家福,郭凯,等. 类黄酮代谢途径与棉花纤维发育[J]. 中国科学(生命科学),2014,44(8):758-765.TU L L,TAN J F,GUO K,et al.Flavonoid pathway in cotton fiber development[J]. Scientia sinica (vitae),2014,44(8):758-765(in Chinese with English abstract).
[18] FENG H J,TIAN X H,LIU Y C,et al.Analysis of flavonoidsand the flavonoid structural genes in brown fiber of upland cotton[J/OL]. PLoS One,2013,8(3):e58820[2024-05-27].https://doi.org/10.1371/journal.pone.0058820.
[19] WEN T W,WU M,SHEN C,et al.Linkage and associationmapping reveals the genetic basis of brown fibre (Gossypiumhirsutum)[J].Plant biotechnology journal,2018,16(9):1654-1666.
[20] SUN S C,XIONG X P,ZHU Q H,et al.Transcriptome sequencingand metabolome analysis reveal genes involved inpigmentation of green-colored cotton fibers[J/OL]. Internationaljournal of molecular sciences,2019,20(19):4838[2024-05-27].https://doi.org/10.3390/ijms20194838.
[21] LI Z H,SU Q,XU M Q,et al. Phenylpropanoid metabolismand pigmentation show divergent patterns between brown colorand green color cottons as revealed by metabolic and geneexpression analyses[J/OL].Journal of cotton research,2020,3(1):27[2024-05-27]. https://doi. org/10.1186/s42397-020-00069-x.
[22] 葛勇,李威,刘艳改,等. 中棉所63 棉铃对位叶光合生理性能的杂种优势研究[J]. 棉花学报,2016,28(3):250-259.GE Y,LI W,LIU Y G,et al.Heterosis studies on photosynthetic andphysiological performance of the subtending leaf of cotton bollof CCRI 63[J].Cotton science,2016,28(3):250-259( in Chinesewith English abstract).
[23] DING Y H,ZHANG R,ZHU L F,et al.An enhanced photosynthesisand carbohydrate metabolic capability contributes toheterosis of the cotton (Gossypium hirsutum) hybrid‘ HuazaMian H318’,as revealed by genome-wide gene expressionanalysis[J/OL].BMC genomics,2021,22(1):277[2024-05-27].https://doi.org/10.1186/s12864-021-07580-8.
[24] LI T Y,WANG F Q,YASIR M,et al.Expression patterns divergenceof reciprocal F1 hybrids between Gossypium hirsutumand Gossypium barbadense reveals overdominance mediatinginterspecific biomass heterosis[J/OL]. Frontiers in plant science,2022,13:892805 [2024-05-27]. https://doi. org/10.3389/fpls.2022.892805 .
[25] WANG M J,TU L L,YUAN D J,et al.Reference genome sequencesof two cultivated allotetraploid cottons,Gossypiumhirsutum and Gossypium barbadense[J]. Nature genetics,2019,51(2):224-229.
[26] HOCHHOLDINGER F,BALDAUF J A.Heterosis in plants[J].Current biology,2018,28(18):1089-1092.
[27] 向丽媛,徐凯,苏静,等. 基于通路分析剖析水稻农艺性状配合力和杂种优势[J]. 作物学报,2019,45(9):1319-1326.XIANG L Y,XU K,SU J,et al.Genetic dissection of combiningability and heterosis of rice agronomic traits based on pathwayanalysis[J].Acta agronomica sinica,2019,45(9):1319-1326( in Chinese with English abstract).
[28] VOGT T. Phenylpropanoid biosynthesis[J]. Molecular plant,2010,3(1):2-20.
[29] FAN L,SHI W J,HU W R,et al.Molecular and biochemicalevidence for phenylpropanoid synthesis and presence of walllinkedphenolics in cotton fibers[J].Journal of integrative plantbiology,2009,51(7):626-637.
[30] NI Z Y,LI B,NEUMANN M P,et al.Isolation and expressionanalysis of two genes encoding cinnamate 4-hydroxylasefrom cotton (Gossypium hirsutum)[J]. Journal of integrativeagriculture,2014,13(10):2102-2112.
[31] 孟颖,邢蕾蕾,曹晓红,等. 小麦Ta4CL1 基因的克隆及其在促进转基因拟南芥生长和木质素沉积中的功能[J]. 作物学报,2022,48(1):63-75.MENG Y,XING L L,CAO X H,etal. Cloning of Ta4CL1 and its function in promoting plantgrowth and lignin deposition in transgenic Arabidopsis plants[J].Acta agronomica sinica,2022,48(1):63-75 (in Chinesewith English abstract).
[32] TAN J F,TU L L,DENG F L,et al.A genetic and metabolicanalysis revealed that cotton fiber cell development was retardedby flavonoid naringenin[J].Plant physiology,2013,162(1):86-95.
[33] 孙君灵,霍曙明,周忠丽,等. 棉纤维颜色与品质性状杂种优势关系研究[J]. 棉花学报,2010,22(3):267-272.SUN J L,HUO S M,ZHOU Z L,et al.Analysis on the relationship betweenheterosis of quality traits fiber colors[J].Cotton science,2010,22(3):267-272( in Chinese with English abstract).
[34] 李成奇,郭旺珍,张天真. 棉花4 个栽培种纤维初始发育的比较研究[J]. 作物学报,2007,33(8):1346-1351.LI C Q,GUOW Z,ZHANG T Z.Comparative studies on fiber initiation developmentof four cultivated cotton species[J].Acta agronomicasinica,2007,33(8):1346-1351( in Chinese with English abstract).
[35] ZHU X X,ZHU Y C,AI N J,et al.Gene differential expressionat seedling stage in four cotton combinations hybridizedby CRI-12 and its pedigree-derived lines[J].Acta agronomicasinica,2009,35(9):1637-1645.
[36] LI S J,JAYASINGHEGE C P A,GUO J,et al.Comparativetranscriptomic analysis of gene expression inheritance patternsassociated with cabbage head heterosis[J/OL]. Plants,2021,10 (2) : 275 [2024-05-27]. https://doi. org/10.3390/plants10020275.
[37] LIN T Z,ZHOU C,CHEN G M,et al. Heterosis-associatedgenes confer high yield in super hybrid rice[J].Theoretical andapplied genetics,2020,133(12):3287-3297.
[38] FU J,ZHANG Y L,YAN T Z,et al.Transcriptome profilingof two super hybrid rice provides insights into the genetic basisof heterosis[J/OL]. BMC plant biology,2022,22(1):314[2024-05-27]. https://doi. org/10.1186/s12870-022-03697-4.
[39] SHIMADA Y,OHBAYASHI M,NAKANO-SHIMADA R,et al.Genetic engineering of the anthocyanin biosynthetic pathwaywith flavonoid-3′,5′-hydroxylase:specific switching ofthe pathway in petunia[J]. Plant cell reports,2001,20(5):456-462.
(责任编辑:张志钰)
基金项目:江西省科技计划项目(20212BAB215009);江西省教育厅科技计划项目(GJJ200440)

