N-乙酰转移酶对罗非鱼无乳链球菌毒力相关特性的影响




摘要 为阐明N-乙酰转移酶(NAT)在无乳链球菌致病过程中的作用,利用同源重组法构建野生株HN016的nat 基因缺失株Δnat,并比较野生株和缺失株的生长、形态、黏附入侵细胞能力、抗吞噬能力、全血存活能力和致病力。结果显示,Δnat 的溶血能力降低,并且nat 缺失导致无乳链球菌对罗非鱼脑细胞的黏附和入侵能力下降,并增加了细菌对血液杀伤和吞噬细胞吞噬的敏感性。罗非鱼感染试验结果显示,nat 基因缺失株Δnat(2×108 CFU/尾,致死率为36%)对罗非鱼的致死力显著低于野生株HN016(2×108 CFU/尾,致死率为85%)。罗非鱼感染12 h 后,与野生对照株相比,Δnat 突变株在血液、脾脏和脑组织中载菌量显著降低。研究表明,N-乙酰转移酶参与无乳链球菌抵抗宿主血液和吞噬细胞的清除,并协助细菌对罗非鱼组织的入侵和定植。
关键词 无乳链球菌; N-乙酰转移酶; 基因缺失; 生物学特性; 罗非鱼
中图分类号 S917.1 文献标识码 A 文章编号 1000-2421(2024)06-0289-08
无乳链球菌(Streptococcus agalactiae)是一种革兰氏染色呈紫色的链状球菌,又称B 族链球菌(group B streptococcus,GBS)。近年来,无乳链球菌已成为水生环境中的主要病原体之一,能感染许多养殖鱼类,导致败血症和脑膜炎[1],给养殖业尤其是罗非鱼(Oreochromis spp.)养殖业造成了巨大的经济损失。
细菌发挥致病作用的前提是能够响应宿主体内微环境的变化,而这种响应依赖于蛋白翻译后的修饰[2]。乙酰化修饰是蛋白翻译后修饰中最保守的方式之一。乙酰化可以调节RcsB 蛋白的活性,从而影响大肠杆菌(Escherichia coli)在酸胁迫下的存活和鞭毛合成[3]。乙酰化的过程需要乙酰转移酶的参与,乙酰转移酶可以催化乙酰基从乙酰辅酶A 转移到底物,并通过催化底物的乙酰化,协调细菌的各种生物过程[7]。已鉴定的乙酰转移酶被分为3 个家族,分别为GCN5 (general control non-repressed 5 protein,GCN5)、CBP/p300 (cyclic-AMP response bindingprotein-binding protein, CBP) 和MYST (Moz,Ybf2, Sas2 和Tip60, MYST),其中GCN5 家族乙酰转移酶是细菌中最主要的乙酰转移酶[5]。GNAT(GCN5-related N-acetyltransferase,GNAT)将乙酰辅酶A 的乙酰基转移到蛋白质的N-氨基上,因此称为N-乙酰转移酶。
N-乙酰转移酶在细菌耐药性和代谢等多种生理活动中发挥重要作用。例如,在结核分枝杆菌(My⁃cobacterium tuberculosis)中,具有N-乙酰转移酶活性的Eis 蛋白可以通过乙酰化修饰氨基糖苷类抗生素,从而削弱其与核糖体的结合,导致耐药性[6];在炭疽芽孢杆菌(Bacillus anthraci)中,N-乙酰转移酶可以乙酰化和灭活链霉素,导致细菌抵抗链霉素的抑制和杀灭[7];在牙龈卟啉单胞菌(Porphyromonas gingi⁃valis)中,N-乙酰转移酶能够调节牙龈痛蛋白酶的激活和成熟[8]。目前,在链球菌中关于乙酰转移酶的研究并不多见。已报道的相关研究包括氯霉素乙酰转移酶CAT 可以赋予粪肠球菌(Enterococcus faecalis)和A 族链球菌氯霉素抗性,并且将CAT 基因重组到质粒上用于科学研究[9];由无乳链球菌的neuD 基因编码的O-乙酰转移酶是细菌荚膜多糖唾液酸所必需的[10],而无乳链球菌中的N- 乙酰转移酶尚未见报道。
本研究构建无乳链球菌nat 基因缺失株,探究该基因缺失对细菌溶血能力、全血存活能力、黏附入侵能力、抗吞噬能力、组织定植能力以及对罗非鱼致死率的影响,以期阐明N-乙酰转移酶在无乳链球菌致病过程中的作用,促进对无乳链球菌致病机制的了解。
1 材料与方法
1.1 菌株、质粒、细胞和试剂
无乳链球菌野生株HN016 分离自广东省某养殖场患脑膜炎的罗非鱼。大肠杆菌DH5α 购自北京全式金生物技术有限公司。温敏型自杀载体pSET4s由华中农业大学周洋教授惠赠。TiB 细胞是一种来自罗非鱼大脑的成纤维细胞系,由佛山科学技术学院曾伟伟教授赠送。小鼠单核细胞巨噬细胞(RAW264.7)由笔者所在实验室保存并传代。
DNA 聚合酶混合液、核酸Marker、限制性核酸内切酶、核酸连接酶、pMD9-T 载体、反转录试剂盒和荧光定量PCR 酶混合物购自宝生物工程有限公司(大连);质粒提取、琼脂糖凝胶核酸回收和原核生物的RNA 提取试剂盒购自Tiangen 公司;DL-苏氨酸购自Invitrogen 公司;蔗糖、磷酸氢二钾、氢氧化钾和甘油购自国药集团化学试剂有限公司;革兰氏染色试剂盒和PBS(磷酸盐缓冲液)购自Solarbio 公司;麻醉剂MS-222 购自Macklin 公司;药敏片购自杭州微生物有限公司;壮观霉素(spectinomycin,Spc)购自德国Biofroxx 公司;胎牛血清和DMEM(Dulbecco’smodified Eagle medium)培养基购自Gibco 公司;THB (Todd-Hewitt broth) 和LB (Luria-Bertanibroth)培养基购自青岛海博生物技术有限公司。
1.2 菌株及细胞的培养
无乳链球菌在THB 或含1.5%(m/V)琼脂的THB 培养基中培养,温度为37 ℃。大肠杆菌DH5α在LB 或含1.5%(m/V)琼脂的LB 培养基上生长,温度为37 ℃。必要时将壮观霉素加入固体培养基或肉汤中,终质量浓度为100 μg/mL。小鼠单核细胞巨噬细胞(RAW264.7)在37 ℃、5% 二氧化碳条件下用含10% 胎牛血清的DMEM 培养基培养。罗非鱼脑细胞系(TiB)在含10% 胎牛血清的DMEM 培养基中培养,培养温度为28 ℃。
1.3 Δnat 缺失株的构建
1)基因缺失质粒的构建。以HN016 基因组为模板,分别用目的基因上游引物和下游引物扩增上下游同源臂。用融合PCR 的方法将2 个同源片段连接,再与pMD19-T 载体连接测序,之后用相应的限制性内切酶酶切后克隆至质粒pSET4s 的合适位置,采用热击方法转入大肠杆菌感受态细胞中,提取质粒备用。
2)感受态细胞的制备。将过夜培养菌液按1∶100 转接至THY(含40 mmol/L DL- 苏氨酸的THB)培养液中,37 ℃,180 r/min 培养至OD600 nm 为0.4~0.6;冰浴30 min 后,12 000 r/min,4 ℃离心10min 收集菌体;用EB 缓冲溶液(10% 蔗糖、2 mmol/L磷酸氢二钾)重悬细菌,同上离心;再用超纯水洗2 次,之后用15% 甘油溶液重悬细菌,洗2 次。最后用15% 甘油重悬菌体,按每管80~100 μL 分装,于-80 ℃冻存待用。
3)电转化。将该菌的感受态细胞与基因缺失质粒以体积比10∶1 混合(质粒最终的剂量控制在l μg)后加到电转化杯中,冰上放置0.5 h。电转化时,仪器设置:电压2.35 kV/cm、电阻200 Ω 和电容25 μF。电转化结束后立即将l mL 复苏液加入电击杯中。将电转化后的无乳链球菌在28 ℃和180 r/min 振荡培养3 h,然后加入壮观霉素至终质量浓度100 μg/mL,继续培养过夜。
4)缺失株的筛选。将培养过夜的菌液涂布在固体THB 培养基上,直至长出单克隆。挑取单克隆接种至含有100 μg/mL 壮观霉素的THB 液体培养基中。之后,在不含抗生素的THB 培养液中,28 ℃、180 r/min 连续传代培养,在温度和抗性的双重选择下促使重组缺失质粒与基因组上具有相同序列的基因片段发生同源重组。将传代后的菌液梯度稀释后涂在含有壮观霉素抗性和无抗性的THB 平板过夜培养。当无抗性的平板上长的菌落明显多于抗性平板时挑选无抗性平板上的菌落,用缺失基因的内部引物和上下游同源臂引物进行PCR 筛选。
5)缺失株的验证。用同源臂引物PCR 扩增疑似缺失株相应片段,并通过DNA 胶回收试剂盒纯化PCR 产物,测序,测序结果只含有缺失基因上下游片段,则说明缺失株构建成功。另外,将培养至对数生长期的无乳链球菌野生株和缺失株进行RNA 的提取,用DNase Ⅰ去除基因组DNA 干扰。用反转录试剂盒将RNA 反转录为cDNA。使用SYBR premixEx TaqTM Kit,采用说明书两步法qRT-PCR 检测目的基因的mRNA 水平,以16S rRNA 基因作为内参。所用引物见表1,均由北京擎科生物公司合成。采用2-ΔΔCt法分析mRNA 水平的差异[11]。
1.4 表型测定
1)生长曲线。将过夜培养的菌液按1∶100 稀释到新鲜的THB 培养基中,培养至对数中期(OD600 nm=0.4~0.5)。培养物在新鲜的THB 培养基中稀释至OD600 nm为0.1。取稀释后的培养物 200 μL至96 孔微孔板中,37 ℃孵育,每小时摇动10 s 后测量OD600 nm 值,设置4 个技术重复。根据重复的平均值绘制OD600 nm随时间的变化曲线图。
2)溶血性。将过夜培养的细菌划线接种于血平板上,37 ℃倒置培养48 h,使其形成明显溶血圈,观察溶血情况并拍照。
3)革兰氏染色。先涂片固定,草酸铵结晶紫染1 min,蒸馏水冲洗;再加碘液覆盖涂面染约1 min,水洗,用吸水纸吸去水分;再加95% 乙醇数滴,轻轻摇动脱色,20 s 后水洗,吸去水分;番红染色液染1 min后用蒸馏水冲洗;干燥,镜检。
1.5 全血存活试验
试验参照文献[12]描述的方法,并有所修改。在罗非鱼的尾静脉取血,立即加入0.1% 肝素钠,备用。准备好已经生长到对数期的无乳链球菌,稀释至合适浓度加到血液中,控制细菌最终的浓度为1×103 CFU/mL,将混合液置于37 ℃培养箱并保持微翻转。分别于0 和1.5 h 取一定量的混合液,涂布到THB 固体培养平板上,记录长出的菌落数。
1.6 黏附和入侵试验
试验参照文献[12]描述的方法,并有所修改。
1)黏附试验。用0.25% 胰酶处理单层TiB 细胞后,显微镜观察并计算细胞数量,再加到24 孔细胞培养板培养。无乳链球菌野生株HN016 和缺失株培养至对数期(OD600 nm 为0.6),洗菌液3 次,调整到浓度为4×106 CFU/mL,将菌液加入细胞孔(复合感染比MOI=10),微速离心后将细胞板放入37 ℃培养箱培养1 h。取出后用PBS 缓冲液洗3~5 次,再加入纯水裂解细胞并进行平板计数菌落数。黏附率是衡量细菌黏附细胞的一个重要指标,黏附率(裂解后菌落数量/加入细菌数量)越高说明细菌黏附细胞的能力越强。
2)入侵试验。方法同黏附试验,不同的是PBS洗涤后,需加入青霉素G 至终质量浓度为100μg/mL,继续培养细胞1 h。入侵率是衡量细菌侵入细胞的一个重要指标,入侵率(裂解后菌落数量/加入细菌数量)越高说明细菌侵入细胞的能力越强。
1.7 抗吞噬试验
试验参照文献[12]描述的方法。RAW264.7 细胞用含10% 胎牛血清的DMEM 营养液在5% CO2和37 ℃条件下传代培养,以4×105 细胞/孔传代至24 孔板上,形成单层贴壁细胞。以MOI 为10,即4×106 细胞/孔的细菌悬液感染RAW264.7 细胞。接种后将24 孔板在室温下1 000 r/min 离心15 min 使细菌与细胞充分接触。24 孔板置37 ℃、5% CO2作用1 h。用PBS 洗3~5 遍,每孔加入l mL 含100 μg/mL 庆大霉素和5 μg/mL 青霉素的DMEM,以彻底清除巨噬细胞外残余细菌。加入抗生素1 h 后用无菌PBS 清洗2 遍再加入去离子水裂解细胞。将该裂解液梯度稀释后涂平板计数菌落数。吞噬率是衡量细菌抵抗吞噬的一个重要指标,吞噬率(裂解后菌落数量/加入细菌数量)越低说明细菌抗吞噬的能力越强。
1.8 致死率试验
试验参照文献[12]描述的方法,并有所修改。试验用罗非鱼均购自广东粤强丰水产养殖场,体质量(10±1) g,于实验室暂养2 周后进行相关试验。通过2 次离心收集对数后期的细菌,并用PBS 重悬,备用。将120 尾鱼随机分为3 组(野生株组、突变株组和对照组),每组40 尾。对照组注射无菌PBS 0.1mL,试验组罗非鱼用90 mg/L 的MS-222 麻醉后腹腔注射0.1 mL 细菌悬液(每尾鱼注射2×108 CFU)。对被注射感染的鱼进行监测,连续14 d,从所有死鱼的脑组织中重新分离出细菌并进行鉴定。试验重复2 次。
1.9 组织载菌量试验
试验参照文献[19]描述的方法,并有所修改。将20 尾鱼随机分为2 组,每组10 尾。分别腹腔注射野生株HN016 和突变株,每尾鱼注射2×108 CFU。麻醉的处理方法同本文“材料与方法1.8”。感染后12 h,采集脑、脾脏和血液样本,称测各组织质量。匀浆后用PBS 连续稀释至10-2、10-3、10-5 和10-6 进行平板计数。计数时选取菌落数在30~300 的平板。
1.10 数据处理
采用Graphpad Prism 6.0 软件做图,利用t 检验进行统计学分析,以Plt;0.05 (*)为有统计学差异,Plt;0.01 (**)为有显著统计学差异,Plt;0.001 (***)为有极显著的统计学差异。除了特殊说明,所有试验均进行3 次重复。
2 结果与分析
2.1 无乳链球菌nat 基因缺失株的构建
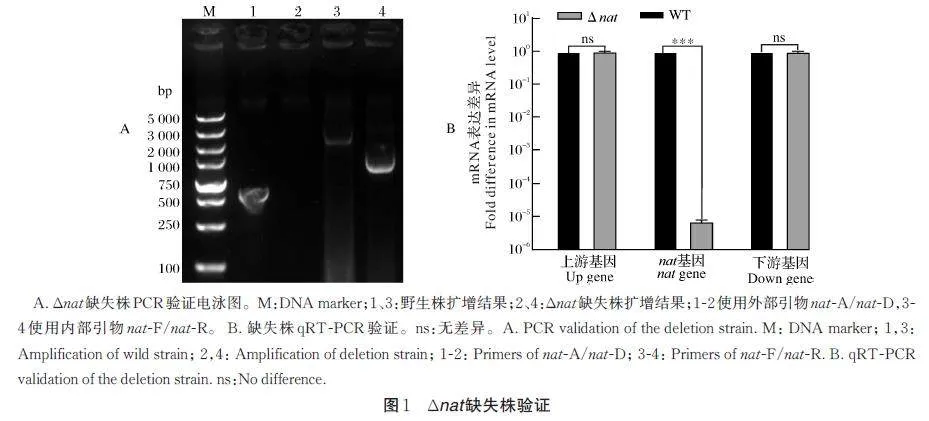
如图1A 所示,用nat 基因内部引物(nat-F/nat-R)进行PCR 扩增,以缺失株Δnat 基因组为模板,无法获得扩增产物,以野生株HN016 基因组为模板能获得约500 bp 的产物;以nat 基因外部引物(nat-A/nat-D)进行PCR 扩增,以缺失株Δnat 基因组为模板,PCR 扩增产物大小为上下游同源臂的大小之和约1 000 bp,而以野生株HN016 基因组为模板,PCR扩增产物大小为上下游同源臂加nat 基因长度约2 200 bp。结果表明缺失株Δnat 构建成功。用qRTPCR检测nat 基因mRNA 的表达水平,结果(图1B)显示,nat 基因的mRNA 在野生菌株中正常表达,而在缺失株Δnat 中未检测到表达;另外,用qRT-PCR检测nat 基因的上游基因(SAHN016_RS06510)和下游基因(SAHN016_RS06520)的mRNA 表达水平,结果(图1B)显示在缺失株Δnat 和野生株HN016 中nat 上下游基因的mRNA 表达水平没有显著差异。因此,nat 基因缺失不影响上下游基因的表达。
2.2 nat 基因缺失对无乳链球生长特性、菌落形态和溶血能力的影响
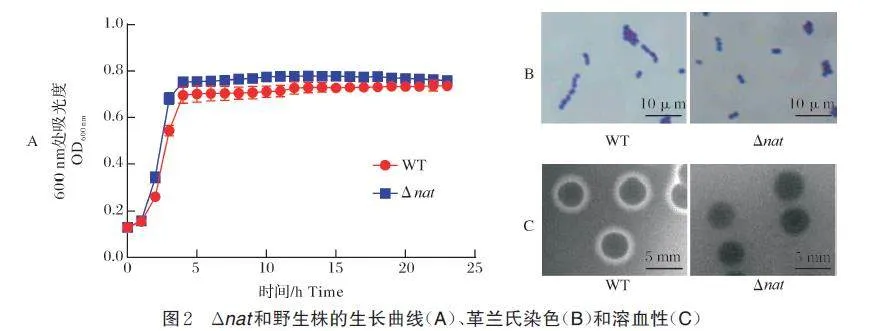
如图2A 所示,缺失株Δnat 和野生株HN016 均在2 h 左右进入对数生长期,4 h 左右进入平台期,表明缺失株Δnat 和野生株HN016 生长无显著差异,这表明nat 基因缺失不影响细菌生长。同时革兰氏染色结果也表明基因缺失不影响细菌革兰氏染色(图2B)。另外通过比较缺失株Δnat 和野生株HN016 在血平板上的溶血环,nat 基因缺失使无乳链球菌的溶血能力减弱(图2C)。
2.3 nat 基因缺失对无乳链球菌在血液中生存能力的影响
如图3 所示,与罗非鱼全血孵育1.5 h 后,野生株HN016 增长了14.6 倍,缺失株Δnat 增长了11.9 倍,Δnat 在血液中的生存能力显著弱于野生株HN016。说明nat 基因缺失显著降低无乳链球菌在罗非鱼全血中的生存能力。
2.4 nat 基因缺失对无乳链球菌黏附和入侵罗非鱼脑细胞能力的影响
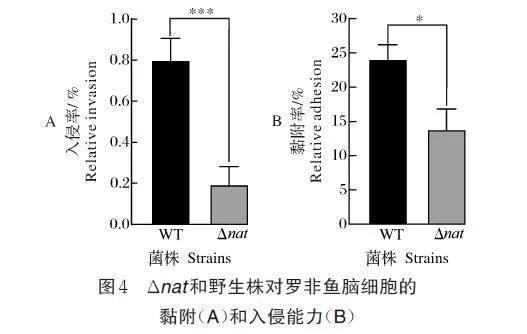
如图4A 所示,野生型HN016 对TiB 细胞的入侵率为0.8%,缺失株Δnat 对TiB 细胞的入侵率为0.19%,Δnat 的入侵率比野生型下降了75.6%(图4A)。如图4B 所示,野生型HN016 对TiB 细胞的黏附率为24.1%,缺失株Δnat 对TiB 细胞的黏附率为13.8%,Δnat 对TiB 细胞的黏附性下降了42.7%(图4B)。这些结果表明nat 基因缺失显著降低无乳链球菌黏附和入侵罗非鱼细胞的能力。
2.5 nat 基因缺失对无乳链球菌抗吞噬能力的影响
用RAW264.7 评价Δnat 的抗吞噬能力,突变株Δnat 和野生株HN016 的吞噬率分别为64.5%和38.2%,Δnat 比HN016 更容易被吞噬(图5)。结果表明,Δnat 对RAW264.7 的吞噬作用更敏感,nat 基因缺失显著降低无乳链球菌对巨噬细胞吞噬作用的抗性。
2.6 nat 基因缺失对无乳链球菌致病力的影响
对培养至对数期的野生株和缺失株以2×108CFU/尾的剂量,腹腔注射的方式攻毒罗非鱼,并在之后的14 d 内观察存活情况。结果如图6 所示,HN016 感染罗非鱼3 d 内可造成85% 死亡率,第4~14 天内没有出现死鱼。Δnat 感染罗非鱼8 d 后造成36% 罗非鱼死亡,第8~14 天内没有出现死鱼。PBS对照组在试验周期内并没有造成罗非鱼的死亡。感染HN016 的罗非鱼表现出典型的出眼症、角膜混浊、鳃帽下缘出血等症状,而感染Δnat 的罗非鱼症状较轻或无明显症状(图6B)。这些结果表明nat 基因缺失显著降低了无乳链球菌的致病力。
2.7 缺失株Δnat 感染组织载菌量的变化
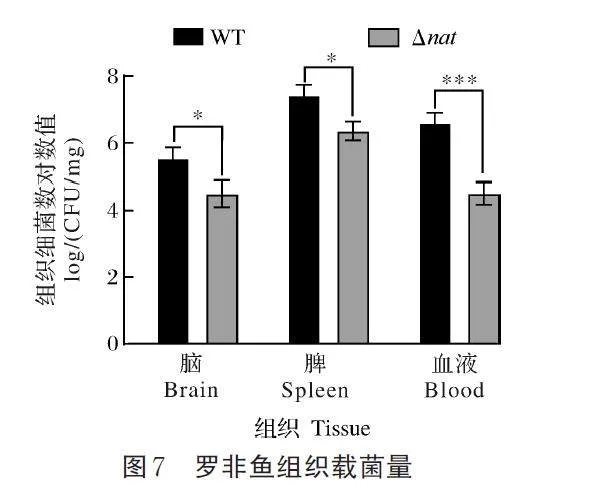
如图7 所示,罗非鱼感染野生株12 h 后,脑、脾脏和血液载菌量分别为4.81×105、3.77×107 和4.5×106 CFU/mg,感染突变株Δnat 后,脑、脾脏和血液载菌量分别为3×104、2.86×106 和2.6×104 CFU/mg(图7),Δnat 感染后的组织载菌量较野生株显著下降,其中在大脑减少93.8%,在脾脏减少92.4%,在血液中减少了99.4%。这些结果进一步表明nat 基因缺失降低了无乳链球菌毒力。
3 讨论
本研究通过同源重组构建了无乳链球菌nat 基因缺失菌株,发现与野生株相比,Δnat 的革兰氏染色和在培养基中的生长无明显差异,表明nat 基因缺失对细菌生长和形态的影响不大。但nat 缺失明显削弱细菌在全血中的存活能力,表明nat 基因有助于无乳链球菌在血液中生存。溶血性试验结果表明,nat基因的缺失导致了无乳链球菌溶血能力减弱,这与大肠杆菌中的乙酰转移酶缺失的表型一致[13],大肠杆菌中的溶血素需要乙酰化才能发挥溶血活性,这表明nat 基因的缺失,可能导致了溶血素不能被乙酰化,最终导致无乳链球菌溶血性降低。荚膜多糖是细菌关键毒力因子,对细菌在血液中存活至关重要[14]。在大肠杆菌中,乙酰化的荚膜多糖可以使细菌逃避血液的杀伤,避免宿主的免疫防御[15]。因此,我们推测nat 缺失导致无乳链球菌在血液中生存能力的下降可能是因为细菌荚膜多糖不能被乙酰化修饰,这还需要进一步的实验验证。
脑膜炎是无乳链球菌感染最常见的临床综合征,细菌导致脑膜炎需要黏附和入侵脑细胞。本研究利用罗非鱼大脑成纤维细胞TiB 评估基因缺失对细菌黏附入侵能力的影响。结果显示Δnat 黏附和入侵TiB 的比率远低于野生株,表明nat 基因参与无乳链球菌入侵大脑过程。与上述结果相似,CheY 也是一种乙酰化蛋白,Yao 等[16]的研究表明CheY 促进单增李斯特菌(Listeria monocytogenes)与上皮细胞的初步黏附并有助于单增李斯特菌对上皮细胞的入侵。插入沉默空肠弯曲菌(Campylobacter jejuni)的cheY 基因可导致该细菌获得较高的体外黏附和入侵能力,但无法在体内定植和引起疾病[11]。抵抗吞噬细胞吞噬是无乳链球菌发挥致病作用的关键,因此我们还探究了nat 缺失对无乳链球菌抗RAW264.7吞噬能力的影响,结果表明缺失株对吞噬细胞的敏感性显著增加。这说明致病菌的乙酰转移酶在抗吞噬过程中发挥重要作用。
黏附和入侵减少以及吞噬敏感性增加是否会导致无乳链球菌对罗非鱼致病力的降低?为了回答这个问题,我们利用罗非鱼感染模型验证了缺失株和野生株对罗非鱼的致死率和在器官中的定植能力。当使用2×108 CFU/尾的剂量攻毒罗非鱼时,野生株死亡率为85%,而nat 基因缺失株仅为36%,同时感染野生株的罗非鱼死亡速度更快,在感染最初的2 d发生集中性大量死亡,但缺失株没有发生集中性死亡,表明nat 基因缺失导致无乳链球菌对罗非鱼致死率发生明显下降。为了更全面了解细菌致病力,我们采集了受感染罗非鱼的脾脏、血液和大脑,并统计了不同组织和血液中的细菌载量。结果显示缺失株在器官和血液中的载量均低于野生株,表明nat 基因的缺失导致无乳链球菌更容易被免疫系统清除,血液和脾脏中的细菌密度显著降低可以证明这一点。而在大脑中观察到的细菌密度的减少再次证明Δnat进入大脑的能力受损。由此可见,nat 基因表达的N-乙酰转移酶是无乳链球菌关键毒力因子,nat 基因缺失导致无乳链球菌毒力减弱。乙酰转移酶介导的乙酰化修饰可调节多种细菌毒力。在沙门氏菌中,乙酰转移酶可以通过乙酰化调控PhoP 的活性,而PhoP是细菌毒力的重要转录调节因子,因此,推测乙酰转移酶具有调控细菌毒力的功能[17]。在铜绿假单胞菌(Pseudomonas aeruginosa)中,kdsA 基因产物被乙酰化,参与脂多糖的生物合成,是铜绿假单胞菌毒力所必需的[18]。因此,我们推测Δnat 毒力的减弱可能是细菌中某些毒力因子乙酰化程度降低导致的,需要进一步进行实验验证。
综上,本研究初步探究了无乳链球菌N-乙酰转移酶的功能,发现N-乙酰转移酶参与无乳链球菌通过血液运输并感染罗非鱼器官的过程,并且能够协助无乳链球菌抵抗吞噬细胞和血液的清除,最终导致无乳链球菌对罗非鱼的高致病力。这一结果阐明了N-乙酰转移酶在无乳链球菌中的作用,为进一步探索无乳链球菌致病机制提供了理论依据。本研究中,我们成功地以HN016 的基因组为模板,PCR 扩增并纯化出nat 基因片段,并通过尝试酶切酶连或者重叠PCR 的方法将nat 基因连接到回补质粒pSET4s上,但是,目前我们还没有获得正确的回补质粒,后续会继续尝试获得回补株,并检测回补株的表型和毒力。
参考文献References
[1] CHEN M,LI L P,WANG R,et al.PCR detection and PFGEgenotype analyses of streptococcal clinical isolates from tilapiain China[J]. Veterinary microbiology,2012,159(3/4):526-530.
[2] 陆雨星. 氨基酸小分子变构调控ACT-GNAT 型乙酰转移酶功能研究[D]. 上海:华东理工大学,2017.LU Y X.Study onthe function of ACT-GNAT acetyltransferase regulated byamino acid small molecule allosteric regulation[D].Shanghai:East China University of Science and Technology,2017 (inChinese with English abstract).
[3] CASTAÑO-CEREZO S,BERNAL V,POST H,et al.Proteinacetylation affects acetate metabolism,motility and acidstress response in Escherichia coli[J/OL].Molecular systemsbiology,2014,10(11):762[2023-08-17]. https://doi. org/10.15252/msb.20145227.
[4] SHIRMAST P,GHAFOORI S M,IRWIN R M,et al.Structuralcharacterization of a GNAT family acetyltransferase fromElizabethkingia anophelis bound to acetyl-CoA reveals a newdimeric interface[J/OL].Scientific reports,2021,11(1):1274[2023-08-17].https://doi.org/10.1038/s41598-020-79649-5.
[5] BERNDSEN C E,DENU J M.Catalysis and substrate selectionby histone/protein lysine acetyltransferases[J]. Currentopinion in structural biology,2008,18(6):682-689.
[6] CHEN W J,BISWAS T,PORTER V R,et al.Unusual regioversatilityof acetyltransferase Eis,a cause of drug resistance inXDR-TB[J].PNAS,2011,108(24):9804-9808.
[7] BURCKHARDT R M,ESCALANTE-SEMERENA J C.Insightsinto the function of the N-acetyltransferase SatA that detoxifiesstreptothricin in Bacillus subtilis and Bacillus anthracis[J/OL]. Applied and environmental microbiology,2019,85(6):e03029-e03018[2023-08-17]. https://doi.org/10.1128/AEM.03029-18.
[8] MISHRA A,ROY F,DOU Y T,et al.Role of acetyltransfer‐ase PG1842 in gingipain biogenesis in Porphyromonas gingi⁃valis[J/OL]. Journal of bacteriology,2018,200(24) :e00385-e00318 [2023-08-17]. https://doi. org/10.1128/jb.00385-18.
[9] ALBERTÍ S,ASHBAUGH C D,WESSELS M R.Structureof the has operon promoter and regulation of hyaluronic acidcapsule expression in group A Streptococcus[J].Molecular microbiology,1998,28(2):343-353.
[10] LEWIS A L,HENSLER M E,VARKI A,et al.The group Bstreptococcal sialic acid O-acetyltransferase is encoded byneuD,a conserved component of bacterial sialic acid biosyntheticgene clusters[J].Journal of biological chemistry,2006,281(16):11186-11192.
[11] DONS L,ERIKSSON E,JIN Y X,et al.Role of flagellin andthe two-component CheA/CheY system of Listeria monocyto⁃genes in host cell invasion and virulence[J].Infection and immunity,2004,72(6):3237-3244.
[12] DONG Y H,MA K,CAO Q,et al. CRISPR-dependent endogenousgene regulation is required for virulence in piscineStreptococcus agalactiae[J].Emerging microbes amp; infections,2021,10(1):2113-2124.
[13] HACKER J,HUGHES C.Genetics of Escherichia coli hemolysin[J] Current topics in microbiology and immunology,1985,118:139-162.
[14] LEYING H,SUERBAUM S,KROLL H P,et al.The capsularpolysaccharide is a major determinant of serum resistancein K-1-positive blood culture isolates of Escherichia coli[J].Infection and immunity,1990,58(1):222-227.
[15] COLINO J,OUTSCHOORN I.The form variation of the capsularpolysaccharide K1 is not a critical virulence factor ofEscherichia coli in a neonatal mouse model of infection[J].Microbialpathogenesis,1999,27(4):187-196.
[16] YAO R,BURR D H,GUERRY P. CheY-mediated modulationof Campylobacter jejuni virulence[J].Molecular microbiology,1997,23(5):1021-1031.
[17] REN J,SANG Y,QIN R,et al.Metabolic intermediate acetylphosphate modulates bacterial virulence via acetylation[J].Emerging microbes amp; infections,2019,8(1):55-69.
[18] OUIDIR T,COSETTE P,JOUENNE T,et al. Proteomicprofiling of lysine acetylation in Pseudomonas aeruginosa revealsthe diversity of acetylated proteins[J].Proteomics,2015,15(13):2152-2157.
(责任编辑:边书京)
基金项目:武汉市知识创新专项-基础研究项目(2023020201010103);中央高校基本科研业务费专项(2662023PY021);国家现代农业产业技术体系专项(CARS-46);国家自然科学基金面上项目(32173015);岭南现代农业实验室科研项目(NT2021008)

