高粱PLC基因家族全基因组鉴定及其对非生物胁迫的响应表达分析

摘要:磷脂酶C(PLC)是磷酸肌醇代谢中重要的水解酶之一,在调节植物生长发育、应对逆境胁迫和多种信号转导过程中发挥着重要功能。鉴定高粱的PLC基因家族,能够为磷脂酶C基因功能研究提供理论依据,有利于筛选出植物抗逆发育生长的有效基因。基于高粱全基因组数据,鉴定得到高粱PLC基因家族,通过生物信息学方法分析并研究高粱PLC基因家族成员的理化性质、染色体定位、基因结构、保守结构域、顺式作用元件、共线性关系和系统发育关系,并用qRT-PCR技术探究高粱PLC基因家族在不同组织时期、胁迫处理下的表达模式。结果表明,共鉴定到11个SbPLC基因,分为2个亚家族,各亚家族内的SbPLC具有典型的蛋白基序和基因结构特征,在一定程度上支持分类预测。此外,这11个SbPLC基因在高粱的6条染色体上不均匀分布并且均富集了生长发育、激素响应相关的顺式元件。通过对SbPLC共线性关系的研究发现,基因复制事件可能导致一些SbPLC基因的产生,片段复制比串联重复更有助于SbPLC基因家族的扩展。qRT-PCR结果显示,SbPLC基因不同程度地受到非生物胁迫的影响,并且在花器官、种子中的表达相对活跃。综上所述,本研究结果和信息为进一步研究高粱PLC基因的生物学功能和进化提供了良好基础,同时也为高粱的抗逆性分子育种提供了一些候选基因。
关键词:高粱;磷脂酶C(PLC);基因家族;非生物胁迫;表达分析
中图分类号:S514.03" 文献标志码:A
文章编号:1002-1302(2024)23-0038-11
简子林,王自力,秦" 畅,等. 高粱PLC基因家族全基因组鉴定及其对非生物胁迫的响应表达分析[J]. 江苏农业科学,2024,52(23):38-48.
doi:10.15889/j.issn.1002-1302.2024.23.005
收稿日期:2023-11-26
基金项目:贵州省科技计划(编号:黔科合支撑[2022]重点026);贵州省十大工业(优质烟酒)产业振兴专项资金(编号:黔财工[2020]198);贵州省省级绿色高产高效建设项目(编号:S2023-002)。
作者简介:简子林(2001—),男,贵州贵阳人,硕士,从事高粱遗传育种方面的研究。E-mail:1395488225@qq.com。
通信作者:任明见,教授,博士生导师,从事小麦、高粱等作物的遗传育种与配套栽培技术、品质分析、抗病性鉴定及分子生物学方面的研究。E-mail:rmj72@163.com。
磷脂是构成细胞膜的重要成分,同时也是细胞内信号转导物质的重要组分[1]。磷脂酶可以将磷脂水解成二酰基甘油(DAG)、磷脂酸(PA)、游离脂肪酸(FFA)等多种次级产物,这些产物将参与细胞调节与新陈代谢并涉及细胞内信号传递过程[2]。根据磷脂水解位点的差异,植物中的磷脂酶可分为磷脂酶A1(PLA1)、磷脂酶A2(PLA2)、磷脂酶D(PLD)和磷脂酶C(PLC)[3]。这些不同类型的磷脂酶在反应条件和底物选择上存在差异,但它们在植物生长发育和应对非生物胁迫的过程中都发挥着重要的调控作用。特别是PLC在植物中被认为是重要的磷脂水解酶,对膜脂的重塑、细胞内信号转导具有重要影响。根据底物特异性范围的不同,植物PLC可分为磷脂酰肌醇特异性PLC(PI-PLC)、非特异PLC(NPC)2个类型[1,4],这些不同类型的PLC在植物生物学中扮演着独特且重要的角色。
一般而言,典型的植物PI-PLC在结构上至少包含2个结构域,即催化性的PI-PLC-X结构域和PI-PLC-Y结构域[5]。这2个结构域可以共同形成1个扭曲的磷酸三糖异构酶(TIM)桶状结构,其中包含活性位点残基[6]。因此,PI-PLC的X/Y结构域具有较高保守性。此外,PI-PLC还具有1个C端的C2结构域和1个N端的EF手型结构域[6]。EF手型结构通常由4个螺旋-环-螺旋折叠组成,这也是钙结合蛋白的显著特征[7]。然而,植物中 PI-PLC 的EF手型(EF-hand)结构并不完整[8],研究发现,某些植物中保守的N末端结构域可能代表催化活性所需的EF手型结构域,但并不是脂质结合所必需的[9]。在本研究中,高粱PLC基因家族成员仅有1个包含EF结构域的蛋白,但其功能尚不明确。据报道,植物PI-PLC除了含有PI-PLC-X结构域、PI-PLC-Y结构域外,还都包含C2结构域,这是PLC发挥功能所必需的重要组成部分[10]。尽管人们已对某些植物中的PI-PLC结构进行了研究,但仍然没有对NPC结构的研究。现有研究显示,所有的NPC都含有1个磷酸酯酶(phosphoesterase)结构域,这是磷脂酶功能所必需的[11-13]。
植物PLC家族成员在植物的生长、发育和胁迫反应等各种生物过程中发挥着重要作用。在拟南芥中,AtNPC4的表达是由高浓度NaCl诱导的,敲除AtNPC4基因的植株对盐的敏感性增强,且AtNPC3、AtNPC4在根发育中起着重要的调控作用[4]。AtPLC2参与生长素的生物合成和信号传导,从而调节拟南芥雄性、雌性胚胎的发育[14]。此外,PLC基因在多种非生物胁迫下表现出响应差异。拟南芥AtPI-PLC成员都可以在高盐度、寒冷等各种非生物胁迫下被诱导[15]。在其他植物中,OsPLC1可引发应激诱导的Ca2+调节耐盐性的信号,揭示了在盐胁迫响应期间由PLC介导的磷酸肌醇与Ca2+之间的联系[16]。ZmPI-PLC3a、ZmPI-PLC3b的表达量经冷处理后上调,经Cu2+处理后下调。ZmNPC4、ZmNPC5的表达量在渗透处理下下调,但高盐度可迅速诱导其表达[17]。大多数PLC成员的表达量在非生物胁迫(盐、寒冷)下及不同发育阶段表现出显著差异,具有在一定阶段、处理下重叠表达的特点。上述结果表明,不同PLC成员在植物生长发育阶段和逆境胁迫中发挥着重要作用。
高粱(Sorghum bicolor L.)是我国五大粮食作物之一,被广泛应用于酿造、畜牧、制糖等多个产业[18]。近年来,复杂多变的盐、碱、干旱等外部环境因素严重限制了高粱产量和品质[19]。因此,本研究对高粱PLC基因家族进行全面分析,研究结果可为SbPLC在高粱生长发育和非生物胁迫响应中发挥的作用奠定理论基础,并为高粱的抗逆育种提供新的思路。
1" 材料与方法
1.1" 植物材料
本研究所用材料为高粱品种 BTx623,于2023年从国家小麦改良中心贵州分中心收集。将BTx623种子置于24 ℃、光—暗周期14 h—10 h、相对湿度为75%的人工气候箱中培养,待植株生长至3叶1心期后取样,然后进行后续试验。
1.2" 基因家族成员的鉴定
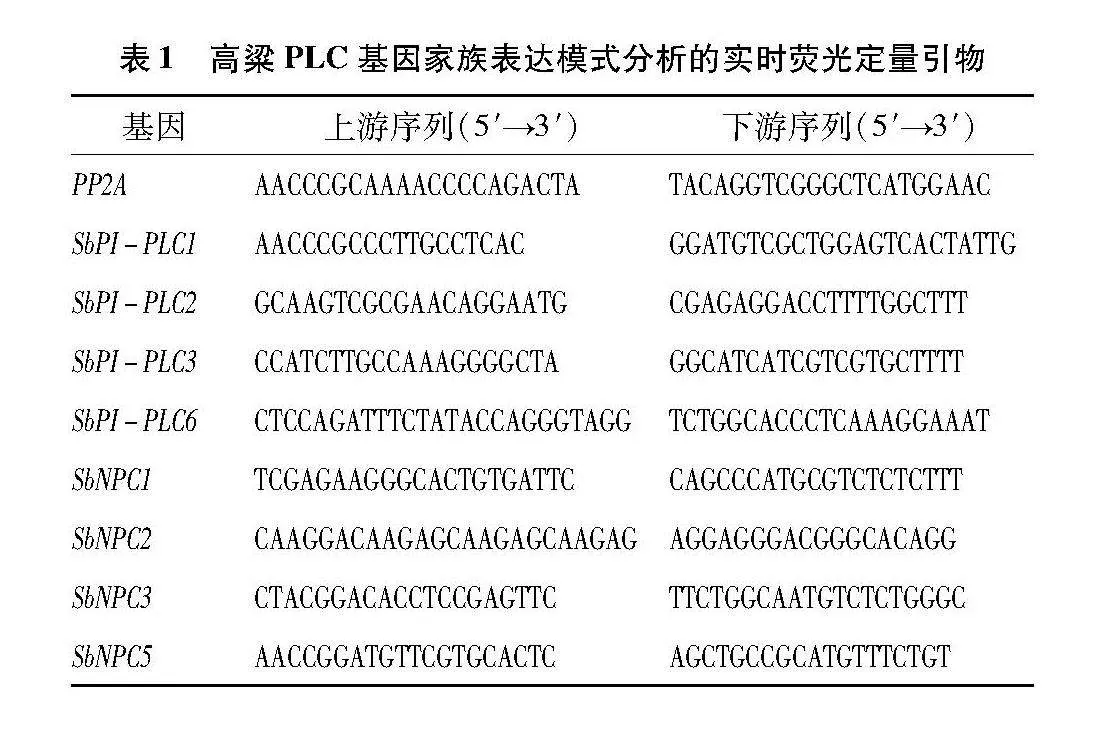
本研究从Ensembl Plants网站(http://plants.ensembl.org/index.html)下载高粱全基因组数据;在pfam数据库(http://pfam.xfam.org/)中下载PLC家族特征的隐马尔科夫模型(PI-PLC-X、PI-PLC-Y),作为本地HMM搜索的查询序列,设置E值lt;0.01,初步鉴定得到高粱PLC成员。为了鉴定可能通过HMM搜索而缺失的SbPLC蛋白,利用模式植物拟南芥、水稻中PLC家族成员的公开ID,从NCBI数据库(http://www.ncbi.nlm.nih.gov/)中提取相应的蛋白序列,与高粱全基因组数据进行BLASTP比对以去除冗余。结合2种方式鉴定得到的PLC蛋白序列,用pfam-search(http://pfam.xfam.org/search#tabview=tab1)与Smart(http://smart.embl.de/)对PLC序列进行验证,最终保留含有PLC特征结构域的基因家族成员。
1.3" 理化性质分析
将SbPLC的蛋白序列提交至ExPASy(https://web.expasy.org/protparam/),预测总平均亲水性、相对分子量和等电点及氨基酸数目等理化性质,并用WolF PSORT Prediction(https://wolfpsort.hgc.jp/)预测亚细胞的位置。
1.4" 系统进化树分析
用MEGA 11软件的clustalW功能对筛选得到的高粱PLC基因家族成员及上述根据拟南芥、水稻PLC家族成员公开ID提取的序列进行氨基酸多序列比对,采用邻近法(neighbor-joining,NJ)构建系统进化树。最后通过EvolView(https://web.expasy.org/protparam/)在线软件对系统进化树进行美化。
1.5" 染色体定位、基因结构和保守基序分析
从Ensembl Plants网站(http://plants.ensembl.org/index.html)下载高粱基因组结构注释信息GFF3文件,用TBtools软件的Gene Location Visualize from GTF/GFF功能绘制基因在染色体上的位置、基因结构[20]。用MEME(http://meme.sdsc.edu/meme4_3_0/intro.html)对高粱PLC基因家族的保守基序进行分析。
1.6" 顺式作用元件分析
为了确定可能参与SbPLC转录调控的顺式元件,用PlantCARE[PlantCARE,adatabase of plant promoters and their cis-acting regulatory elements(ugent.be)]在SbPLC上游2 000 bp的启动子区域对可能响应逆境胁迫和激素作用的顺式作用元件进行分析预测。这些顺势作用元件可通过TBtools软件的Basic Biosequence View功能来实现可视化。
1.7" 共线性分析
从Ensembl Plants数据库(http://plants.ensembl.org/index.html)中下载水稻、拟南芥的全基因组序列和基因注释文件,用TBtools分析PLC基因家族在高粱种内和不同物种间的共线性区段及其基因复制事件,分别鉴定得到高粱与拟南芥、水稻之间的共连基因对,以及高粱物种内的同源基因,结果通过Advanced Circos功能实现可视化。
1.8" 高粱PLC时空表达模式分析
为了研究高粱PLC家族成员在不同组织下的时空表达差异,从已公布的高粱参考基因组数据库中下载相关RNA-seq数据。所有高粱PLC基因的表达水平通过FPKM(fragmentsper kilobase of exon model per milion mapped fragments)方法表示。SbPLC基因的时空表达热图利用ClusyVis网站的ClustVis:a web tool for visualizing clustering of multivariate data (BETA) (ut.ee)进行分析绘制。
1.9" 非生物胁迫和激素处理下高粱幼苗表达模式分析
为了理解SbPLC在不同胁迫下的表达模式,以高粱BTx623作为试验材料,在24 °C、光—暗周期为14 h—10 h、相对湿度为75%的人工气候箱中培养,待植株生长至3叶1心期时,分别用 100 μmol/L ABA、200 mmol/L NaCl在4 ℃条件下处理BTx623幼苗,并分别于0、3、12、24 h取样(每个时间节点设立3次生物学重复)。用液氮速冻然后保存在 -80 ℃ 冰箱中,用于RNA的提取,所用引物见表1。通过qRT-PCR技术验证部分SbPLC在不同胁迫处理和激素作用下的表达水平,试验数据通过2-ΔΔCT法(其中:C表示循环数;T表示阈值)计算相对表达量[21],使用Graphpad Prism 9.5绘制成图。
2" 结果与分析
2.1" 高粱PLC基因家族成员的鉴定与理化性质的分析
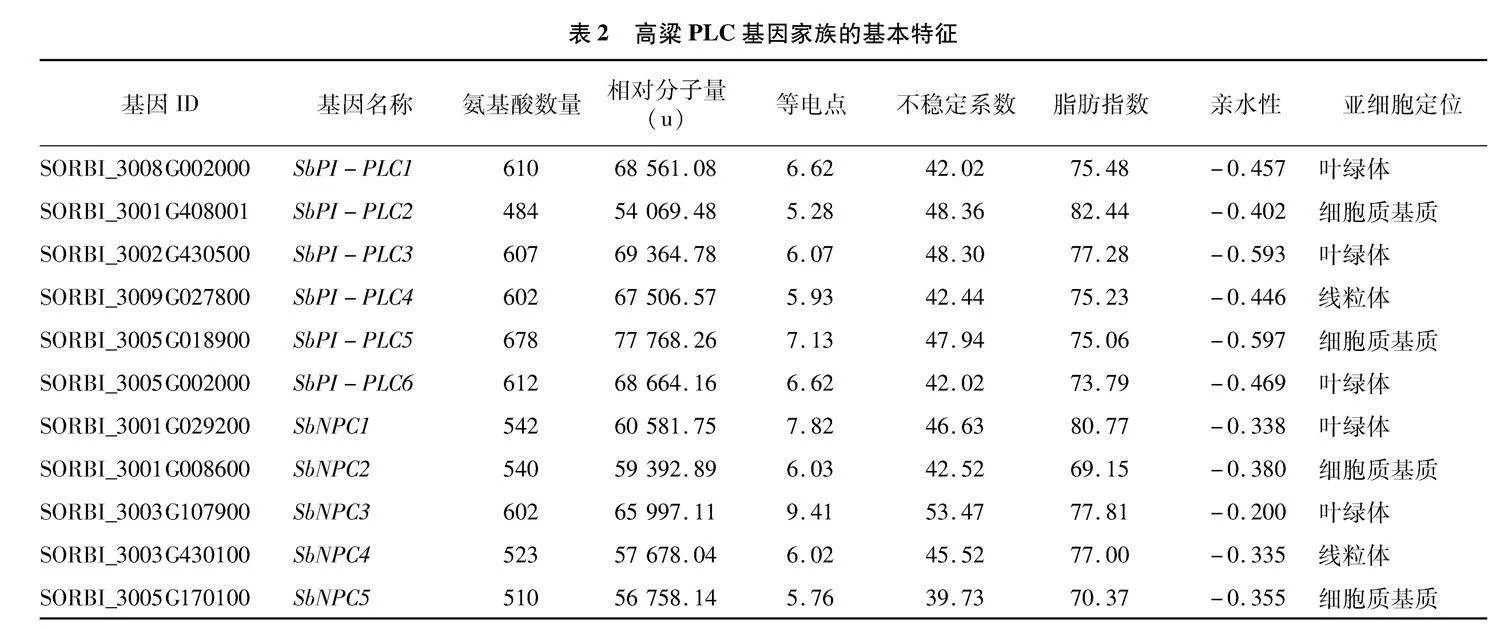
基于生物信息学方法,得到11个SbPLC基因。根据不同类型的保守结构域,将SbPLC分为SbPI-PLC和SbNPC等2类。其中:SbPI-PLC包含6个成员,分别命名为SbPI-PLC1、SbPI-PLC2、SbPI-PLC3、SbPI-PLC4、SbPI-PLC5和SbPI-PLC6。这些编码蛋白的氨基酸数量介于484~678个之间,相对分子量介于54.0~77.7 ku之间,等电点在5.28~7.13之间,不稳定系数在42.02~48.36之间。SbNPC包含5个成员,分别命名为SbNPC1、SbNPC2、SbNPC3、SbNPC4和SbNPC5。这些编码蛋白的氨基酸数量介于510~602个之间,相对分子量介于56.7~65.9 ku之间,等电点在5.76~9.41之间,不稳定系数在39.73~53.47之间。所有SbPLC的平均亲水性值都小于0,表明SbPLC家族成员都被预测为亲水性蛋白。根据WolF PSORT Prediction预测的亚细胞定位,SbPI-PLC1、SbPI-PLC3、SbPI-PLC6、SbNPC1和SbNPC3定位在叶绿体中,SbPI-PLC4和SbNPC4定位在线粒体中,其他SbPLC都定位在细胞质基质中(表2)。SbPLC能够响应多种非生物胁迫和激素作用,可能与SbPLC作用于多种细胞器及细胞质基质有关。此外,不同SbPLC成员具有不同的理化性质,推测SbPLC可能参与了复杂的细胞内信号转导过程。
2.2" SbPLC染色体定位与基因结构分析
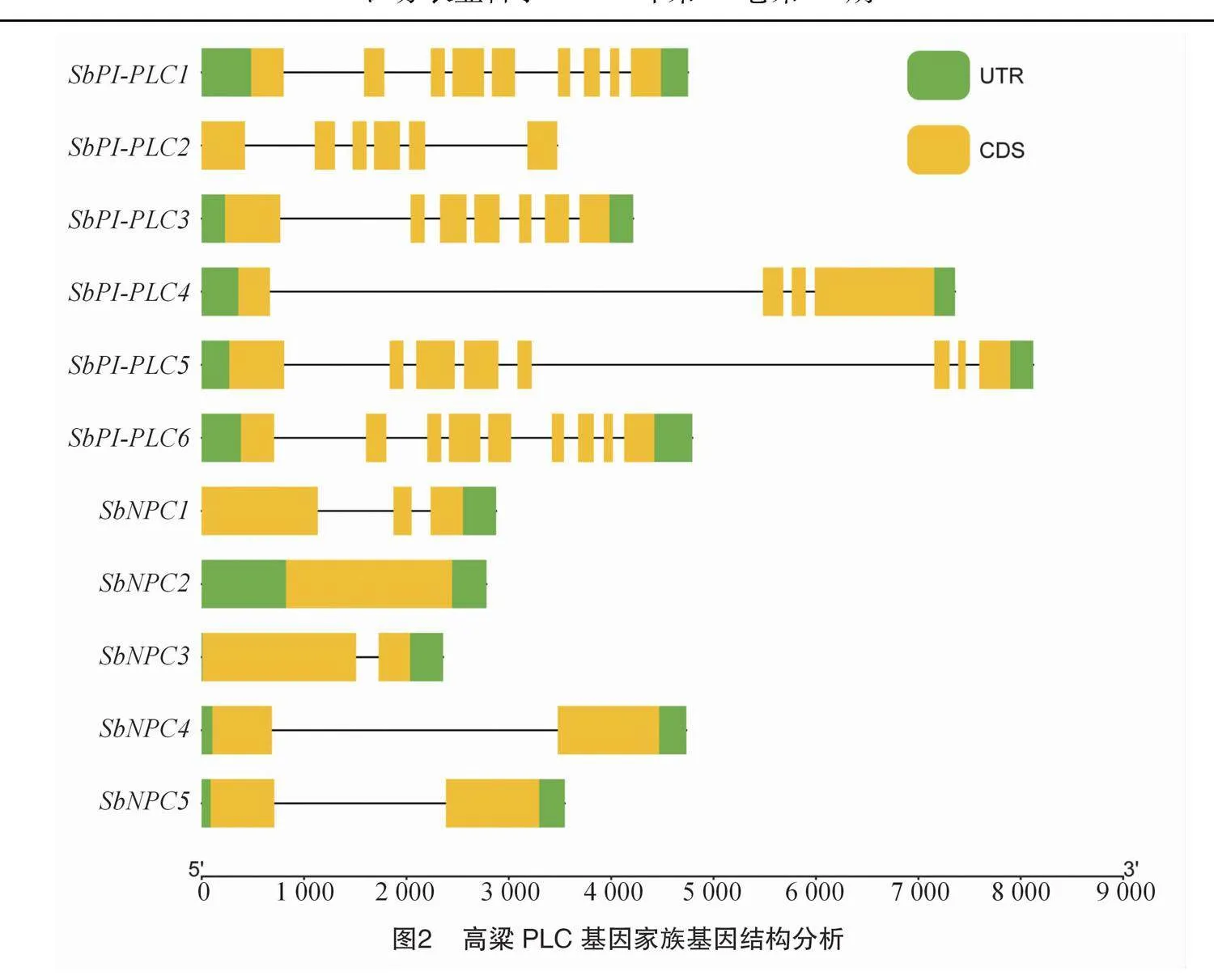
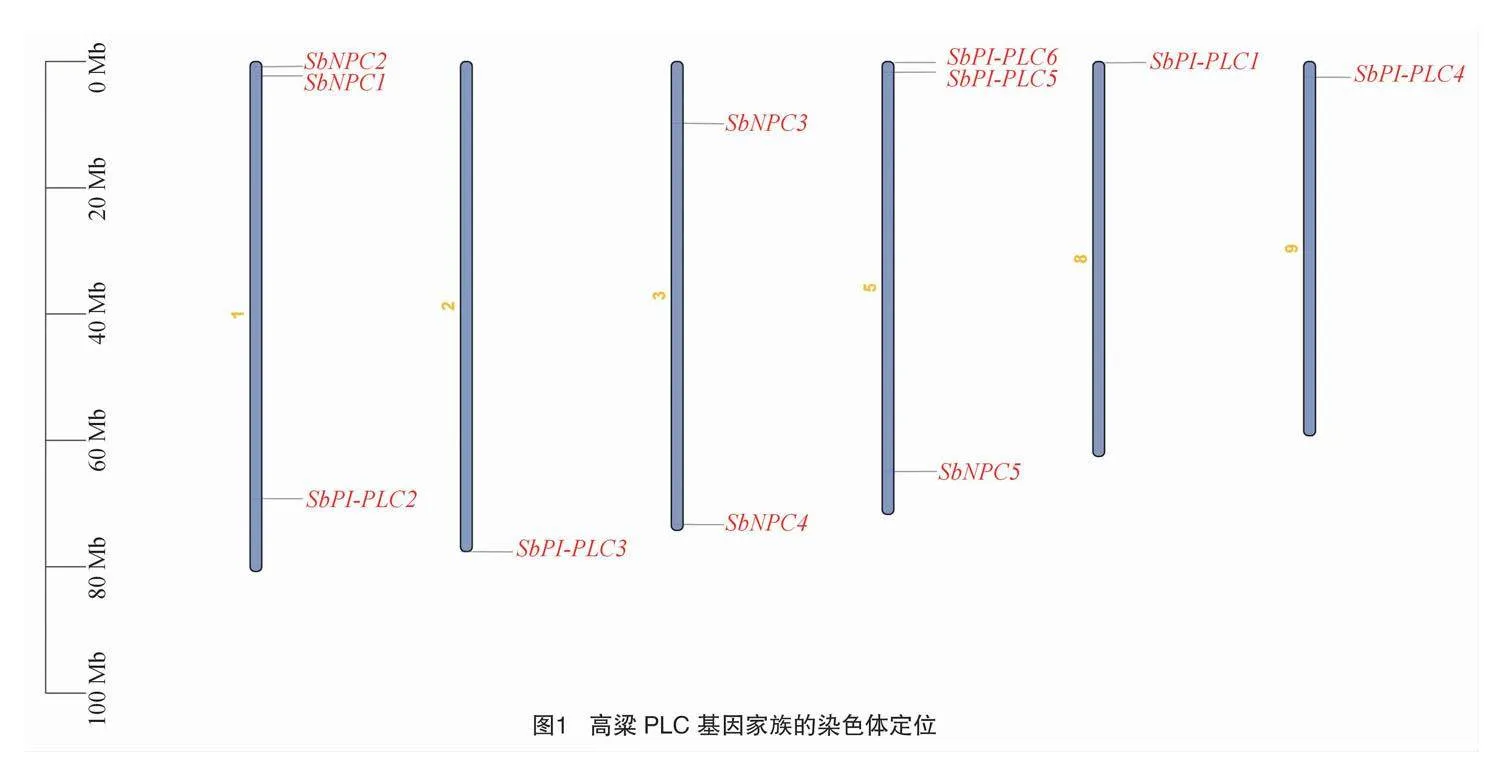
根据基因组注释文件绘制SbPLC染色体定位图,结果(图1)显示,除了4号、6号、7号和10号染色体外,SbPLC的11个家族成员较均匀地分布在高粱的6条染色体两端。其中,SbNPC1、SbNPC2和SbPI-PLC2定位于1号染色体,SbNPC3和SbNPC4定位于3号染色体,SbNPC5、SbPI-PLC5和SbPI-PLC6定位于5号染色体两端,SbPI-PLC1、SbPI-PLC3和SbPI-PLC4分别定位于2号、8号和9号染色体上。为了获得有关高粱PLC家族成员进化特征的线索,对SbPLC的基因结构进行分析,结果(图2)表明,SbPLC之间存在较大的结构差异,可能对应于SbPLC相关成员之间的功能多样性。SbNPC分支通常包含1~3个编码区域(CDS),而SbPI-PLC分支大部分基因均包含8个以上的编码区域(CDS),在每个分支中都存在相似的CDS、UTR模式。
2.3" SbPLC系统进化树分析与保守基序分析
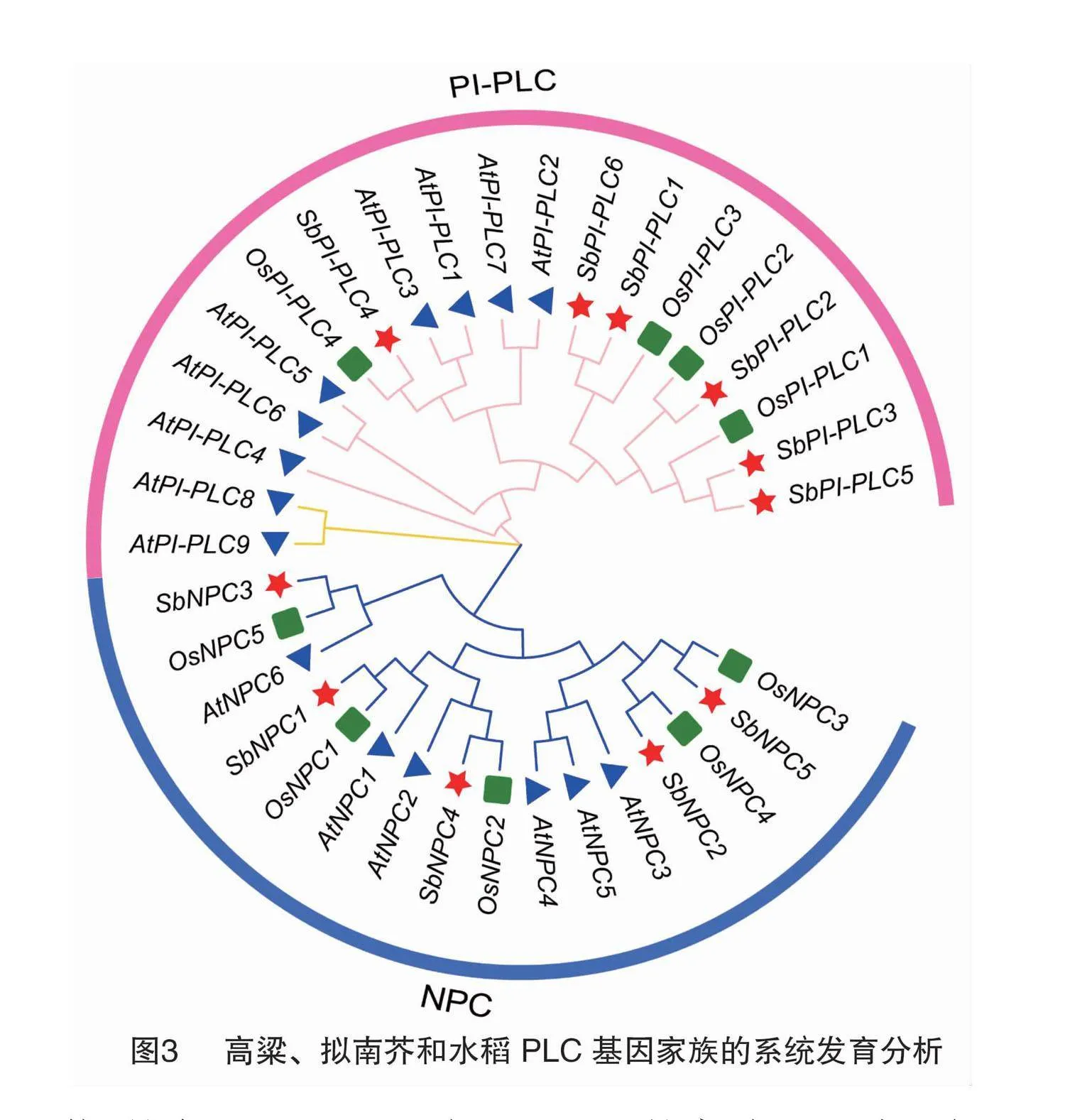
为了深入了解植物PLC的进化历程,并确定高粱中PLC的多样性是否在植物间保守,笔者对几种不同植物的NPC蛋白序列进行了系统发育分析,结
果见图3。通过NJ方法构建系统进化树,将9个水稻OsPLC家族成员、15个拟南芥AtPLC家族成员及11个高粱SbPLC家族成员分为2个亚家族,即PI-PLC和NPC,与之前的CDS/UTR结构分析结果相符。系统发育分析结果显示,SbPLC不同家族成员间具有很高的同源性,特别是SbPI-PLC1和SbPI-PLC6及SbPI-PLC3和SbPI-PLC5形成了独立的小分支。此外,SbPI-PLC2与OsPI-PLC2、SbPI-PLC4与OsPI-PLC4、SbNPC3与OsNPC5、SbNPC1与OsNPC1、SbNPC4与OsNPC2、SbNPC2与OsNPC4、SbNPC5与OsNPC3也形成了独立的分支。上述结果表明,高粱与水稻之间在NPC亚家族中具有非常高的同源性,推测它们可能具有相似的进化路径。
为了进一步研究SbPLC的特征区域,笔者使用MEME在线网站在SbPLC中鉴定出10个保守基序(图4),发现同一分支的成员在结构域方面呈现相似的结构和长度。在PI-PLC分支中,成员具有保守基序1、2、3、4、5、6、8、10,分别注释为 PI-PLC-X、PI-PLC-Y和Ca2+与磷脂结合C2结构域。而在NPC分支中,成员具有保守基序3、5、7、8和9,被注释为磷酸酯酶结构域。保守基序分析结果表明,SbPLC具有高度保守性。
2.4" SbPLC顺式作用元件分析
为了理解SbPLC的表达调控模式及潜在功
能,笔者对SbPLC上游2 000 bp的启动子区域进行分析,并预测了顺式作用元件。图5显示,从SbPLC中共鉴定得到17种顺式作用元件,分别为厌氧诱导响应、光响应、脱落酸响应、水杨酸响应、缺氧特异性响应、种子特异性调节、胚乳表达、分生组织表达、生长素响应、玉米醇溶蛋白代谢调控、防御和应激响应、 昼夜节律控制调节、低温响应、创面响应、干
旱响应、茉莉酸甲酯响应、赤霉素响应元件。这些顺式作用元件预示着SbPLC在高粱的生殖生长发育阶段可能扮演重要角色。大量非生物胁迫响应元件及生长激素类元件的存在也可能在高粱的生长发育阶段、逆境胁迫应对中发挥着至关重要的作用。然而,顺式作用元件的类型、数量与亚家族成员的分布之间没有明显的相关性,同一亚家族的不同成员可能呈现不同的反应模式。
2.5" SbPLC共线性分析
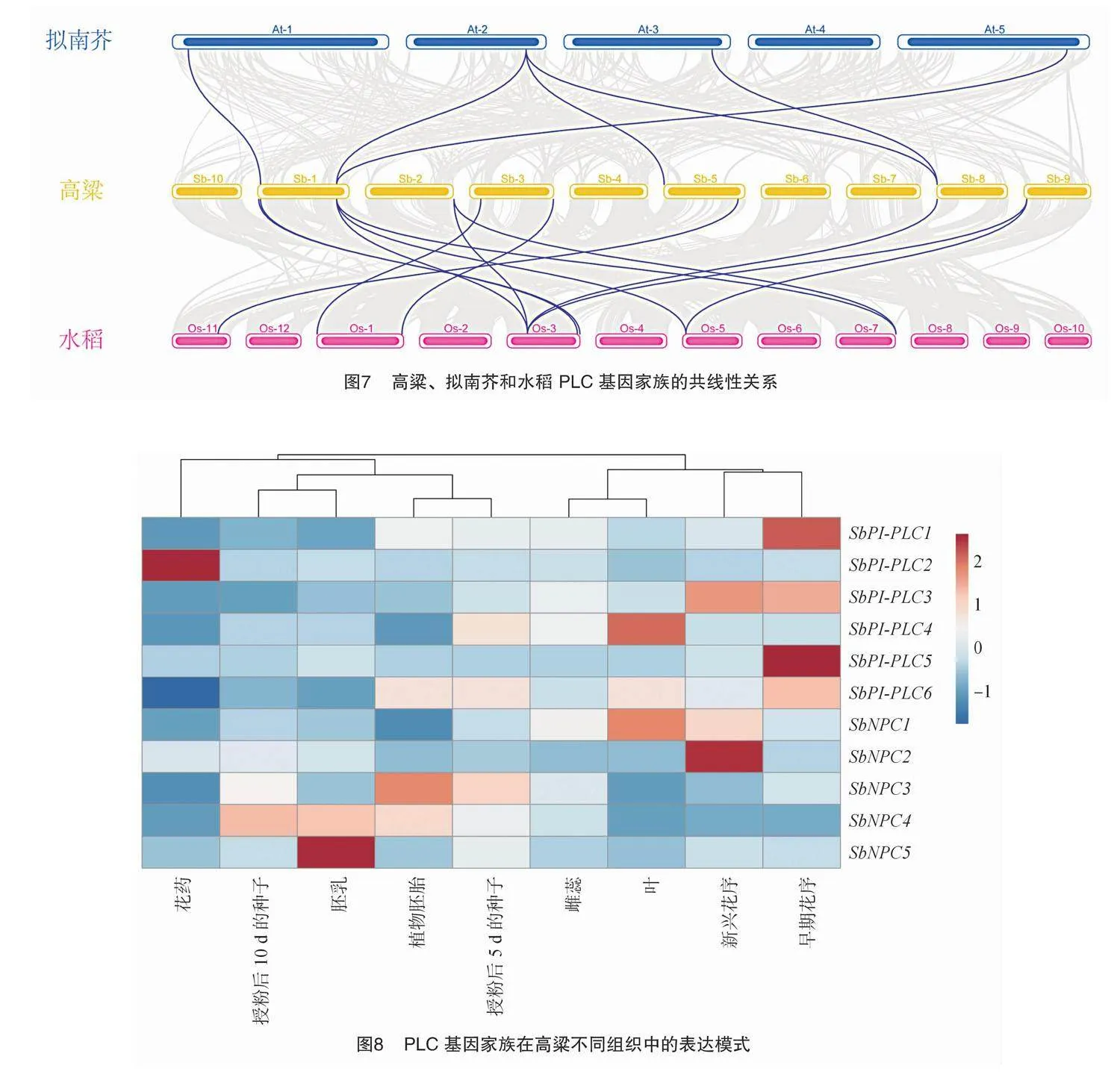
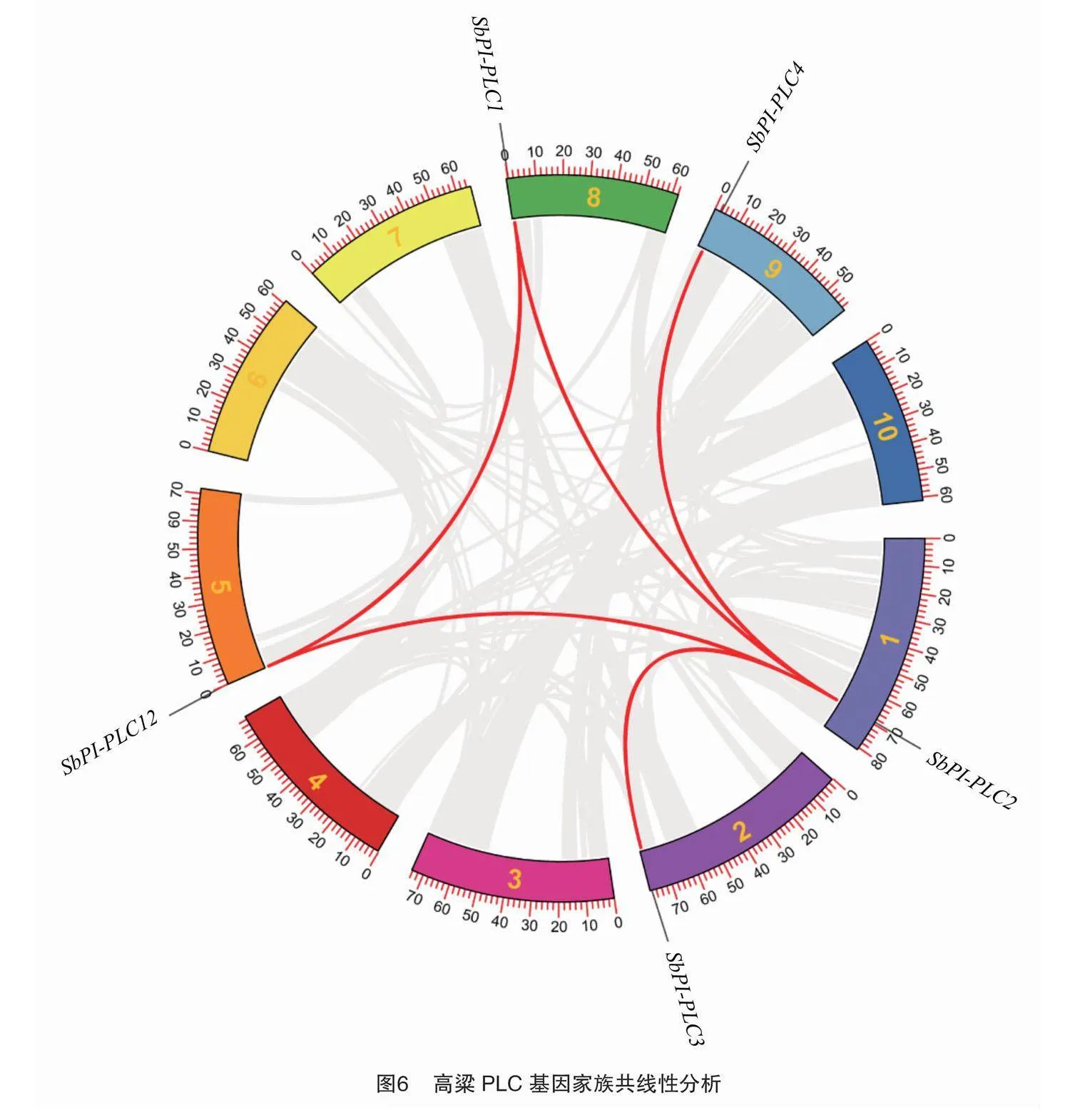
基因组复制事件一直被认为是新颖性进化的起源。在进化过程中,基因的串联复制和大片段重复往往会引发基因家族的形成。因此,笔者基于对高粱基因组的注释和序列比对结果,分析了SbPLC共线性区段和基因复制事件。分析结果用TBtools进行可视化展示,结果如图6所示,在SbPLC中的11个家族成员中,并未发现基因的串联复制,但发现了5对共线性关系,全部发生在PI-PLC分支中。在NPC分支中未发现基因的串联复制和共线性区段,推测SbNPC基因可能是独立进化的。综上,在高粱中基因的串联复制并不是导致PLC基因扩张和进化的主要原因,而更大范围的片段重复才起着关键作用。为进一步了解SbPLC的系统发育关系,笔者还对高粱与拟南芥和水稻等其他2种植物进行了共线性分析,结果(图7)显示,高粱与拟南芥间存在6对共线性关系,而与水稻存在13对共线性关系,表明高粱与水稻间具有更相近的亲缘关系和更相似的进化路径。此外,PLC基因的演化应该发生在单子叶植物和双子叶植物分化之前。
2.6" SbPLC对高粱生长发育和非生物胁迫响应的功能分析
鉴定SbPLC在组织和器官中的表达水平和空间特性,可为深入揭示其在植物生长发育中的作用提供有效手段。笔者利用RNA-Seq数据库分析了SbPLC在不同组织中的表达模式,并通过ClusyVis制作成热图。RNA-Seq数据库包括9个样本,分别为花药、胚乳、植物胚胎、授粉后5 d的种子、授粉后10 d的种子、雌蕊、叶片、新兴花序、早期花序。11个SbPLC基因在所有样本组织中展现出不同的表达模式(图8)。例如,SbPI-PLC2、SbPI-PLC5、SbNPC2、SbNPC5在花药、早期花序、新兴花序和胚乳中表现出特异性;SbPI-PLC1和SbPI-PLC3在花序中显著表达;SbPI-PLC4和SbNPC1在叶片中的相对表达量高于其他组织;SbNPC3和SbNPC4在植物胚胎中的相对表达量相对较高;SbPI-PLC6在花药中表现为低表达。上述结果表明,在高粱的不同组织和发育阶段, SbPLC发挥了重要作用, 主要
在花器官、种子中活跃表达,可能与胚乳、种皮区域是已知的主要脂质储存和代谢位点有关。
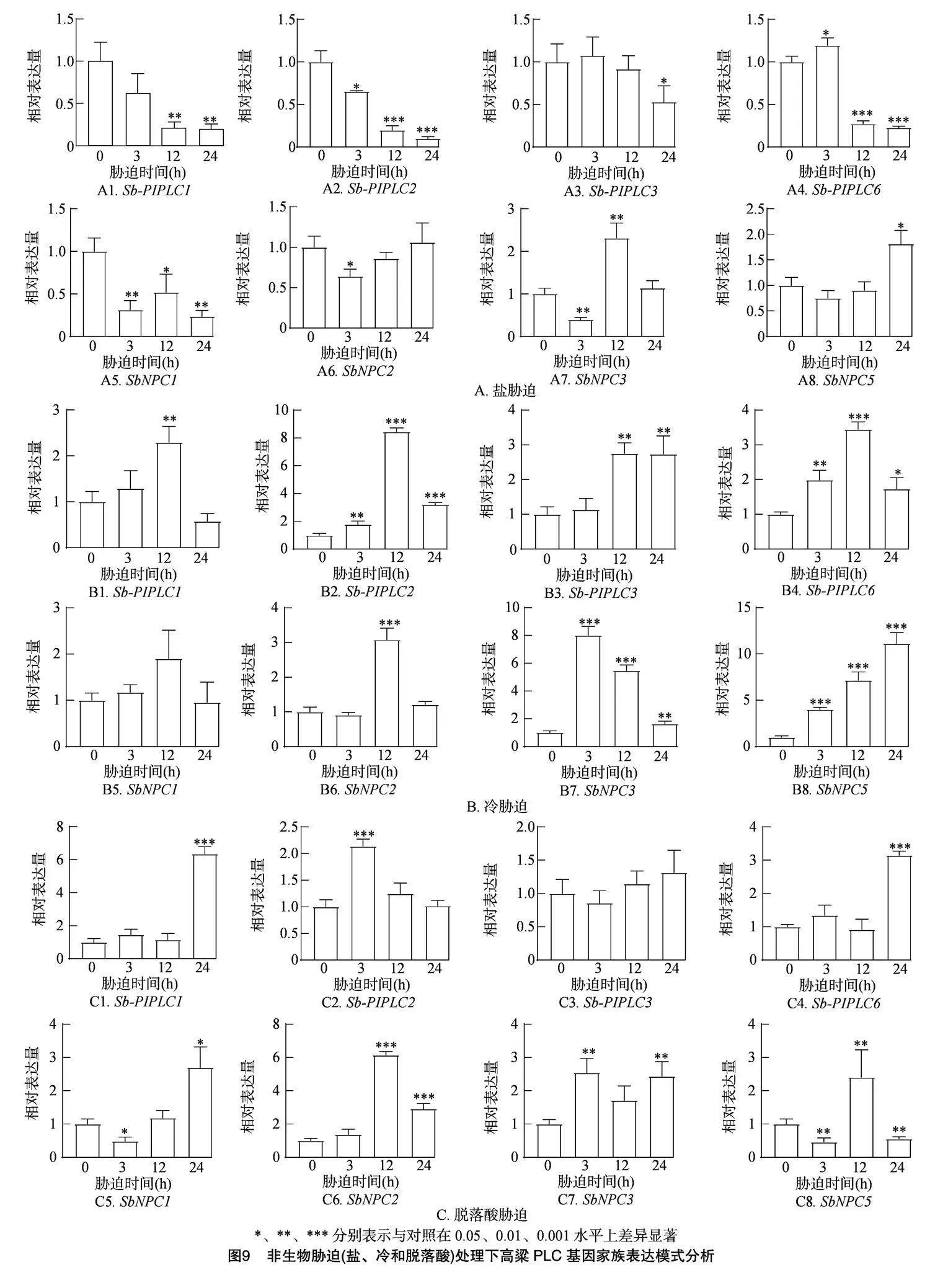
为了进一步阐明SbPLC可能存在的潜在功能,本试验研究了其在胁迫、激素处理下的表达变化。通过qRT-PCR分析,本试验对高粱样品进行了不同的胁迫处理,包括20%PEG-8000、100 μmol/L ABA、200 mmol/L NaCl和4 ℃。首先,通过对系统发育关系和启动子区顺式作用元件的分析,从不同的亚家族中选取8个SbPLC家族成员。结果(图9)显示,在不同的胁迫处理和激素作用下,这8个SbPLC表达出现差异。除了SbNPC2、SbNPC3和SbNPC5外,其余SbPLC基因在 200 mmol/L NaCl处理下被不同程度地抑制表达,其相对表达量随时间的增加而逐渐降低,在处理后24 h其相对表达量达到最低水平。SbNPC3和SbPLC5分别在处理后12、24 h显著表达,而SbNPC2在处理后3 h时达到较低的表达水平,其在处理后12、24 h的表达特征与其余SbPLC基因的表达特征不一致。冷处理几乎立即诱导了所有SbPLC基因表达,除SbNPC3、SbNPC5外,SbPLC基因的相对表达量在处理后12 h达到峰值,同时SbNPC3、SbNPC5的相对表达量也受到显著诱导,但其表达量随时间的推移分别呈下降、上升趋势,其中SbNPC1几乎不受影响。 在 100 μmol/L ABA处理,SbPI-PLC2、SbNPC2、SbNPC3
和SbNPC5在处理24 h内被诱导,其中SbPI-PLC2、SbNPC3在ABA处理3 h时表现出较高的表达水平,而SbNPC2、SbNPC5的相对表达量则在ABA处理12 h时达到峰值。SbPI-PLC1、SbPI-PLC6、SbNPC1也在ABA作用下被诱导,并在处理24 h时达到较高的表达水平。相比之下,SbPI-PLC3没有显示出显著的表达差异。综上所述,SbPLC可能在应对各种非生物胁迫中发挥重要作用。
3" 讨论
测序技术和生物分析工具的改进能更好地反映物种的演化史,为植物基因家族的鉴定和其相关功能的挖掘提供途径,有助于改进其种植方法、管理方法、育种过程。本研究利用生物信息学分析技术,共从高粱中鉴定到11个PLC基因家族成员,这11个基因在高粱中是通过大范围的片段重复事件产生的,基因的串联复制并不是引起PLC基因扩张和进化的主要原因。在SbPI-PLC分支中鉴定得到5对共线性关系,而在SbNPC分支中未鉴定得到基因串联复制及共线性区段,分析SbPLC中的NPC分支发现可能具有独立的进化模式。在后续的研究中,NPC基因也表现出了更强的对复杂多变环境的适应能力和较为不同的表达特征。而SbPLC基本特征在氨基酸数量、分子量、等电点等理化性质上存在较大差异,但总平均亲水性值都小于0,皆被预测为亲水性蛋白。另外,由于编码区(CDS)不同的分布特征,SbPLC基因结构也被发现有较不同的进化模式,SbNPC分支通常包含1~3个CDS,而SbPI-PLC分支的大部分基因均包含8个以上的CDS,每个分支存在各自相似的CDS和UTR模式。在系统发育分析中,笔者通过构建拟南芥、水稻和高粱PLC基因家族进化树,发现PLC基因家族明显被聚类为2个分支(PI-PLC及NPC),而其中水稻、高粱PLC基因普遍形成独立的小分支,表明它们有更高的同源性。通过研究PLC基因高粱与拟南芥、水稻的共线性关系发现,水稻与高粱之间存在更多的共连基因对。结合系统发育分析结果发现,小麦、水稻中的PLC基因具有较高的同源性。进一步的染色体定位分析结果显示,11个SbPLC基因在6条染色体上的定位较为规律,基本都靠近染色体的末端区域。在4、6、7、10号染色体上未发现SbPLC基因,可能是高粱进化过程中基因复制事件的结果。
目前,PLC基因在不同组织中的表达模式已在拟南芥、水稻、大豆等不同的植物中被描述。笔者利用RNA-Seq数据库分析SbPLC在不同组织中的表达模式发现,SbPI-PLC2、SbPI-PLC5、SbNPC2、SbNPC5分别在花药、早期花序、新兴花序及胚乳中有较高的表达水平。已有的研究结果表明,AtPLC1、AtPLC7在茎中的表达水平高于其他器官,AtPLC3在叶、茎中的表达水平高于在花、根中的表达水平。OsPLC2、OsPLC3在穗发育阶段显著表达,而OsNPC4在种子发育阶段显著表达。高粱PLC基因的表达模式不同,说明这些基因可能参与了高粱不同阶段或器官的发育。
非生物胁迫可诱导植物一系列反应模式,从转录调控到信号转导,以及功能特异性的表达和激活。磷脂酶基因在应对环境胁迫中发挥着重要调控作用[22-34]。由于其重要性,PLC基因家族在高粱中还未得到深入研究。在本研究中,qRT-PCR结果显示,SbPLC基因对干旱、盐和冷胁迫及脱落酸处理有不同程度的响应。在这几种非生物胁迫处理中,SbPLC基因对冷胁迫更为敏感,特别是SbNPC5在冷胁迫下的表达水平增加了近11倍。本研究结果表明,SbPLC基因的表达在1个或多个非生物胁迫下发生了变化,表明它们可能在响应环境不利条件中发挥了重要作用。
越来越多的研究结果表明,PLC是植物中发挥重要作用的基因家族,其表达水平的抑制或上升取决于刺激的性质及其时空特征。笔者通过生物信息学方法分析了高粱中PLC家族参与控制细胞生理功能的相关证据,并通过qRT-PCR技术验证了SbPLC调节影响细胞代谢响应非生物胁迫和激素感知,研究结果可为SbPLC与其他植物磷脂酶一起作为参与细胞信号传递的关键因素提供一定的支持。然而,需要进一步的研究来阐明PLC基因家族作用的分子性质及与细胞中其他信号系统的相互作用,以丰富PLC基因家族在高粱生长和发育过程中的作用。
参考文献:
[1]Pokotylo I,Kolesnikov Y,Kravets V,et al. Plant phosphoinositide-dependent phospholipases C:variations around a canonical theme[J]. Biochimie,2014,96:144-157.
[2]Hong Y Y,Zhao J,Guo L,et al. Plant phospholipases D and C and their diverse functions in stress responses[J]. Progress in Lipid Research,2016,62:55-74.
[3]Tuteja N,Sopory S K. Plant signaling in stress[J]. Plant Signaling amp; Behavior,2008,3(2):79-86.
[4]Kocourková D,Krcˇková Z,Pejchar P,et al. The phosphatidylcholine-hydrolysing phospholipase C NPC4 plays a role in response of Arabidopsis roots to salt stress[J]. Journal of Experimental Botany,2011,62(11):3753-3763.
[5]Hicks S N,Jezyk M R,Gershburg S,et al. General and versatile autoinhibition of PLC isozymes[J]. Molecular Cell,2008,31(3):383-394.
[6]Chen G Q,Snyder C L,Greer M S,et al. Biology and biochemistry of plant phospholipases[J]. Critical Reviews in Plant Sciences,2011,30(3):239-258.
[7]Kumar M,Ahmad S,Ahmad E,et al. In silico prediction and analysis of Caenorhabditis EF-hand containing proteins[J]. PLoS One,2012,7(5):e36770.
[8]Essen L O,Perisic O,Cheung R,et al. Crystal structure of a mammalian phosphoinositide-specific phospholipase C delta[J]. Nature,1996,380(6575):595-602.
[9]Otterhag L,Sommarin M,Pical C.N-terminal EF-hand-like domain is required for phosphoinositide-specific phospholipase C activity in Arabidopsis thaliana[J]. FEBS Letters,2001,497(2/3):165-170.
[10]Zen K C,Shikano T,Nakamura Y,et al. The role of EF-hand domains and C2 domain in regulation of enzymatic activity of phospholipase Czeta[J]. The Journal of Biological Chemistry,2005,280(22):21015-21021.
[11]Pokotylo I,Pejchar P,Potock M,et al. The plant non-specific phospholipase C gene family.Novel competitors in lipid signalling[J]. Progress in Lipid Research,2013,52(1):62-79.
[12]Gleadow R M,McKinley B A,Blomstedt C K,et al. Regulation of dhurrin pathway gene expression during Sorghum bicolor development[J]. Planta,2021,254(6):119.
[13]Cui H Y,Chen J Q,Liu M J,et al. Genome-wide analysis of C2H2 zinc finger gene family and its response to cold and drought stress in sorghum [Sorghum bicolor (L.)] moench[J]. International Journal of Molecular Sciences,2022,23(10):5571.
[14]Li L,He Y Q,Wang Y R,et al. Arabidopsis PLC2 is involved in auxin-modulated reproductive development[J]. The Plant Journal,2015,84(3):504-515.
[15]Tasma I M,Brendel V,Whitham S A,et al. Expression and evolution of the phosphoinositide-specific phospholipase C gene family in Arabidopsis thaliana[J]. Plant Physiology and Biochemistry,2008,46(7):627-637.
[16]Li L,Wang F W,Yan P W,et al. A phosphoinositide-specific phospholipase C pathway elicits stress-induced Ca2+ signals and confers salt tolerance to rice[J]. The New Phytologist,2017,214(3):1172-1187.
[17]Zhu J T,Zhou Y Y,Li J L,et al. Genome-wide investigation of the phospholipase C gene family in Zea mays[J]. Frontiers in Genetics,2021,11:611414.
[18]de Morais Cardoso L,Pinheiro S S,Martino H S D,et al. Sorghum (Sorghum bicolor L.):nutrients,bioactive compounds,and potential impact on human health[J]. Critical Reviews in Food Science and Nutrition,2017,57(2):372-390.
[19]刘欢欢. 高粱驯化相关性状遗传结构的解析[D]. 北京:中国农业大学,2016.
[20]Chen C J,Chen H,Zhang Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[21]Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408.
[22]Rhee S G,Bae Y S. Regulation of phosphoinositide-specific phospholipase C isozymes[J]. The Journal of Biological Chemistry,1997,272(24):15045-15048.
[23]Nakamura Y,Koizumi R,Shui G H,et al. Arabidopsis lipins mediate eukaryotic pathway of lipid metabolism and cope critically with phosphate starvation[J]. Proceedings of the National Academy of Sciences of the United States of America,2009,106(49):20978-20983.
[24]Okabe A,Shimizu T,Hayashi H. Cloning and sequencing of a phospholipase C gene of Clostridium perfringens[J]. Biochemical and Biophysical Research Communications,1989,160(1):33-39.
[25]Singh A,Kanwar P,Pandey A,et al. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice[J]. PLoS One,2013,8(4):e62494.
[26]Paponov I A,Paponov M,Teale W,et al. Comprehensive transcriptome analysis of auxin responses in Arabidopsis[J]. Molecular Plant,2008,1(2):321-337.
[27]Tautz D. Evolution of transcriptional regulation[J]. Current Opinion in Genetics amp; Development,2000,10(5):575-579.
[28]Béziau D M,Toussaint F,Blanchette A,et al. Expression of phosphoinositide-specific phospholipase C isoforms in native endothelial cells[J]. PLoS One,2015,10(4):e0123769.
[29]Goda H,Shimada Y,Asami T,et al. Microarray analysis of brassinosteroid-regulated genes in Arabidopsis[J]. Plant Physiology,2002,130(3):1319-1334.
[30]van Leeuwen W,Okrész L,Bgre L,et al. Learning the lipid language of plant signalling[J]. Trends in Plant Science,2004,9(8):378-384.
[31]Dowd P E,Coursol S,Skirpan A L,et al. Petunia phospholipase C1 is involved in pollen tube growth[J]. The Plant Cell,2006,18(6):1438-1453.
[32]Wang X M. Plant phospholipases[J]. Annual Review of Plant Physiology and Plant Molecular Biology,2001,52:211-231.
[33]Riao-Pachón D M,Ruzicic S,Dreyer I,et al. PlnTFDB:an integrative plant transcription factor database[J]. BMC Bioinformatics,2007,8:42.
[34]Nishizuka Y. Protein kinase C and lipid signaling for sustained cellular responses[J]. FASEB Journal,1995,9(7):484-496.

