薄壳山核桃SSR-PCR体系优化及引物筛选
摘 要:【目的】为建立薄壳山核桃SSR-PCR的最佳反应体系,筛选薄壳山核桃高多态性SSR引物,为薄壳山核桃构建指纹图谱、亲缘关系分析、品种鉴定等后续的相关研究提供有力工具。【方法】采用单因素试验与L16 (45)正交试验设计相结合的方法,以薄壳山核桃DNA作为模板,对影响薄壳山核桃SSR-PCR反应体系中的6个影响因素(DNA、Mg2+、10×PCR Buffer、引物、Taq酶、dNTPs)进行单因素试验,根据单因素试验的结果确定各影响因素的适宜用量范围,再据此设计正交试验。【结果】根据正交试验扩增结果,确立薄壳山核桃的最佳反应体系(10 μL)为:Taq酶0.15 U,Mg2+ 2.0 mmol/L,dNTPs 0.1 mmol/L,引物1.0 μmol/L,50 ng模板DNA 1.0 μL,10×PCR Buffer 1.0 μL,ddH2O 5.8 μL。5个因素影响薄壳山核桃SSR-PCR反应体系的扩增效果,其影响程度大小依次为Taq酶>引物=DNA>Mg2+>dNTPs。以8种薄壳山核桃DNA为模板,4对薄壳山核桃引物对优化后的薄壳山核桃SSR-PCR反应体系进行验证,均扩增出明亮清晰的条带,证明优化后的反应体系稳定可靠。利用优化后的反应体系在283对核桃及山核桃引物中筛选出高多态性薄壳山核桃引物12对,其多态性位点信息数均高于0.5。【结论】优化后的反应体系及筛选出的12对薄壳山核桃引物可直接用于后续的SSR分子标记试验,为薄壳山核桃亲缘关系鉴定、交配系统分析等研究奠定理论基础。
关键词:薄壳山核桃;SSR-PCR反应体系;引物筛选;单因素试验;正交设计试验
中图分类号:S722 文献标志码:A 文章编号:1673-923X(2024)09-0138-10
基金项目:安徽省自然科学基金青年项目(2208085QC92);安徽农业大学引进与稳定人才项目(rc372109);校级大学生创新创业训练计划项目(X202310364233);安徽省高等学校科学研究项目(2024AH050484);省级大学生创新创业训练计划项目(S202310364160)。
Optimization of SSR-PCR reaction system and selection of primers in Carya illinoinensis
LUO Xiaolei1, HUANG Dan1, PENG Bingyang1, WANG Leibin2, BI Huihui2, HE Deming2, Lü Jiabin1
(1.a. College of Forestry and Landscape Architecture; b. Key Laboratory of Forest Resource Cultivation in Anhui Province, Anhui Agricultural University, Hefei 230036, Anhui, China; 2. Anhui Jia Ye Agriculture Co., Ltd, Hefei 231200, Anhui, China)
Abstract:【Objective】In order to optimize the SSR-PCR reaction system of Carya illinoinensis, we conducted a screening of highly polymorphic SSR primers specific to C. illinoinensis. This screening process has provided a valuable tool for various subsequent research activities, including fingerprint construction, genetic relationship analysis, and variety identification of C. illinoinensis.【Method】A combination of univariate experiments and L16 (45) orthogonal experimental designs was used. The DNA of C. illinoinensis was used as a template. Firstly, the effects of six factors (DNA, Mg2+, 10×PCR Buffer, primer, Taq enzyme, dNTPs) on the SSR-PCR reaction system of C. illinoinensis were tested individually. Based on the results of the single factor tests, the appropriate dosage range for each factor was determined, and an orthogonal test was designed accordingly.【Result】Based on the results amplified by orthogonal test, the optimal reaction system of C. illinoinensis was determined as follows: Taq enzyme 0.15 U, Mg2+ 2.0 mmol/L, dNTPs 0.1 mmol/L, primer 1.0 μmol/L, 50 ng DNA template 1.0 μL, 10×PCR Buffer 1.0 μL, and ddH2O 5.8 μL. The extremum difference analysis of orthogonal test revealed that the five factors affecting the amplification effect of the SSR-PCR reaction system of C. illinoinensis ranked in the following order: Taq enzyme > primer = DNA > Mg2+ > dNTPs. To validate the optimized SSR-PCR reaction system of C. illinoinensis, four pairs of C. illinoinensis primers were used with the DNA of eight species of C. illinoinensis as a template. The results showed the successful amplification of bright and clear bands, confirming the stability and reliability of the optimized reaction system. Out of the 283 pairs of walnut and C. cathayensis primers, 12 pairs of highly polymorphic primers were selected using the optimized reaction system. The number of polymorphic sites exceeded 0.5.【Conclusion】The optimized reaction system and 12 pairs of primers are screened and can be directly utilized for subsequent SSR molecular marker experiments. This lays a solid foundation for the identification of genetic relationships and mating system analysis of C. illinoinensis.
Keywords: Carya illinoinensis; SSR-PCR reaction system; selection of primers; single factor test; orthogonal design experiment
薄壳山核桃Carya illinoinensis,又名美国山核桃、长山核桃,原产地是美国和墨西哥,属于胡桃科山核桃属植物,是一种落叶乔木[1]。薄壳山核桃的果仁含油量高,其种仁含油量在70%以上,是优质的木本油料树种[2]。薄壳山核桃的果仁口感松脆,壳薄易取,营养丰富,果实富含蛋白质、氨基酸、维生素B1和B2等多种对人体有益的物质[3],具有降低血脂、延缓衰老、保护肠胃、预防心血管疾病等作用,是优良的保健食品[4]。薄壳山核桃的木质纹理清晰,树干通直,可作为良好的庭院绿化树种[2],亦可作为用材林生产家具用品。薄壳山核桃不仅可以提供营养丰富、口感美味的干果,还能绿化荒漠,改善环境,集生态价值、经济价值于一体,具有重要的研究意义。
简单重复序列(simple sequence repeat,SSR),又称为微卫星分子标记,是大量存在于真核生物的基因组中,以1~6个核苷酸为基本单位的重复串联序列[5]。与其他分子标记相比,SSR分子标记分析所需的DNA用量少,对DNA质量要求低,呈孟德尔共显性遗传,信息含量丰富,多态性高[6]。目前,微卫星分子标记技术已广泛应用于植物基因型鉴定、遗传图谱构建、基因流检测[7]以及遗传多样性和遗传进化等方面的研究[8]。由于SSR分子标记是以PCR技术为核心,确定优良的PCR反应体系,对后续的标记开发和研究具有基础性意义。
目前,已有大量研究对不同植物的SSR-PCR反应体系进行了优化。如黄彬等[9]采用L16(45)正交试验对观赏海棠Malus crabapple的SSR-PCR反应体系进行优化,得到了观赏海棠的最优SSRPCR反应体系,并利用优化后的反应体系筛选出15对中高度多态性引物,可直接用于观赏海棠的后续试验研究。于肖夏等[10]采用单因素试验与正交试验设计相结合的方法对冰草Agropyron cristatum的SSR-PCR反应体系进行优化,筛选出最佳的反应体系,为冰草后续的相关研究如指纹图谱构建、基因定位及分子标记辅助育种等研究奠定基础。目前,薄壳山核桃的品种杂乱,品种亲缘关系不清楚,在造林初期花粉配置不合理,造成产量低下、落花落果等问题[11]。而利用SSR分子标记技术可以从分子水平对薄壳山核桃进行品种分类、亲缘关系鉴定等。SSR-PCR反应体系则是进行SSR分子标记的基础和前提。本研究利用单因素试验与正交试验设计相结合的方法,确定了最佳的薄壳山核桃SSR-PCR反应体系,并根据优化后的反应体系从283对核桃及山核桃引物中筛选出12对条带清晰、多态性高的SSR引物,以期为薄壳山核桃后续的亲子关系鉴定、交配系统分析、指纹图谱构建等研究提供理论依据。
1 材料与方法
1.1 试验材料和试剂
本试验用的薄壳山核桃9个品种(YL10号、YL64号、YL35号、YL28号、YL23号、马罕、威斯顿、斯图尔特、波尼)均采自安徽佳烨农业有限公司王祠基地(116°53′E,31°46′N)。2022年4月,采摘刚长出的嫩叶在液氮速冻后于-80 ℃冰箱中保存。
植物组DNA提取试剂盒购自天根生化科技(北京)有限公司,10×PCR Buffer、Mg2+购自上海信乎生物科技有限公司,Taq酶购自爱必梦生物科技有限公司,dNTPs购自上海生工生物工程股份有限公司,从文献中收集整理出283对核桃及山核桃引物[12-18],283对引物由上海生工生物工程股份有限公司合成。
1.2 基因组DNA提取和检测
用天根试剂盒提取薄壳山核桃叶片DNA,利用1.5%琼脂糖凝胶电泳和核酸检测仪检测DNA的质量和浓度,放置于-20 ℃冰箱中保存。
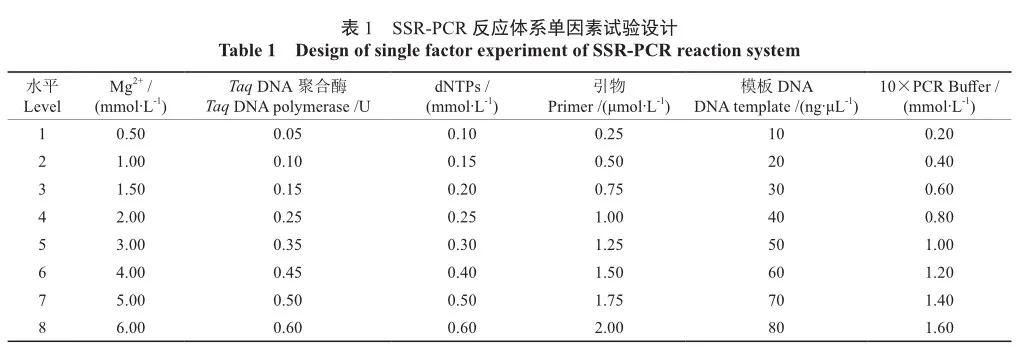
1.3 试验设计
从9种薄壳山核桃品种中随机选取YL10号作为DNA模板,采用单因素与正交试验相结合的方法,对影响SSR-PCR反应体系的6个因素(DNA、Mg2+、10×PCR Buffer、dNTPs、Taq酶、引物浓度)进行优化。从283对引物中随机选取一对引物(Cc119)作为正交试验和单因素试验的引物,另外再选取3对引物作为验证引物。单因素试验设计方案如表1所示。根据单因素试验结果确定各影响因素适宜的用量范围,以此设计正交试验表,进一步优化薄壳山核桃的PCR反应体系。总反应体系为10 μL,除上述6个组分外,不足的部分用ddH2O补齐。反应程序:94 ℃预变性 4 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,循环30次,72 ℃最后延伸7 min,4 ℃保存。在双槽梯度PCR仪中进行扩增,PCR产物用1.5%琼脂糖凝胶电泳检测,电压120 V,时间为30 min。
1.4 PCR反应体系的验证
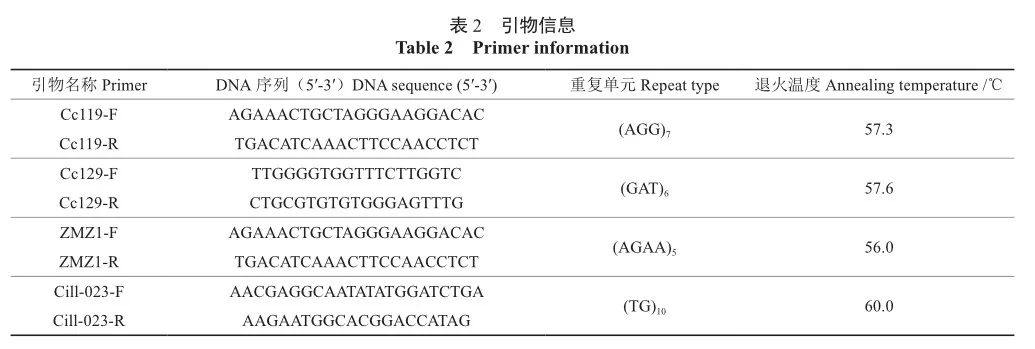
以8个品种的薄壳山核桃作为DNA模板,从283对引物中再选取3对引物,和Cc119一起作为验证引物,用优化后的体系进行PCR扩增。如果扩增后的产物在琼脂糖凝胶电泳上的条带清晰明亮,则说明优化后的体系稳定且适用于多对引物和不同品种的薄壳山核桃。选取的4对引物信息如表2所示。
1.5 多态性引物筛选及退火温度优化
从文献中收集整理出283对核桃及山核桃引物,以8个品种的薄壳山核桃(YL64号、YL35号、YL28号、YL23号、马罕、威斯顿、斯图尔特、波尼)作为DNA模板,用优化后的体系进行PCR扩增。从中筛选适合用于薄壳山核桃的多态性引物,并对文献中引物的预测退火温度进行优化。
1.6 数据处理


2 结果与分析
2.1 单因素试验结果分析
2.1.1 DNA浓度
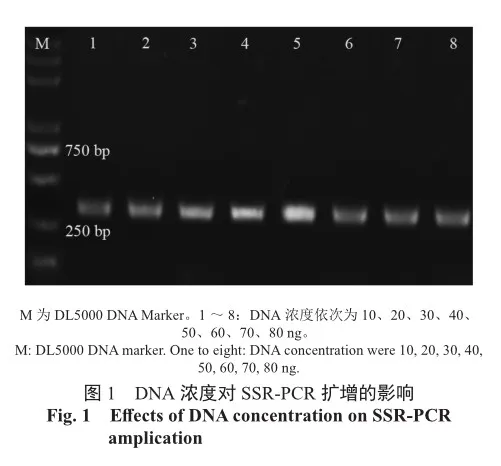
模板DNA浓度也会对PCR反应产生影响,模板DNA浓度单因素试验结果如图1所示。由图1可知,DNA浓度由10 ng增加到50 ng时,扩增出的条带越发明亮清晰;但随着DNA浓度从50 ng增加到80 ng,扩增条带亮度明显降低。因此,将正交试验中模板DNA浓度设为20~50 ng。
2.1.2 Mg2+浓度
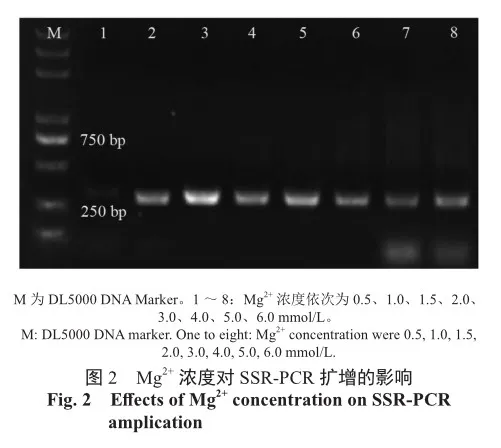
在PCR反应体系中,Mg2+浓度对Taq酶的活性具有直接影响,Mg2+浓度过低会导致PCR扩增条带减少或扩增不出条带,过高则会出现非特异性扩增。由图2可知,Mg2+浓度为0.5 mmol/L时未能成功扩增;Mg2+浓度由1.0 mmol/L增加到1.5 mmol/L时,扩增出的条带亮度明显增加;Mg2+浓度为1.5 mmol/L时,扩增出的条带最为明亮清晰;但随着Mg2+浓度从1.5 mmol/L逐步增加到4.0 mmol/L时,扩增出的条带亮度逐渐变暗;Mg2+浓度增加到5.0~6.0 mmol/L时,出现了非特异性扩增且条带亮度变弱。考虑到扩增效果,正交试验中将Mg2+浓度设为1.0~4.0 mmol/L。
2.1.3 Buffer浓度
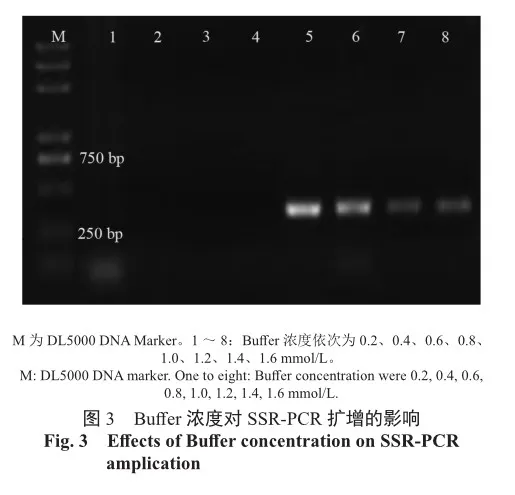
由图3可知,PCR Buffer浓度为0.2~0.8 mmol/L时,未能成功扩增出目的条带;Buffer浓度为 1.0 mmol/L时,扩增出的条带最为明亮清晰;随着Buffer浓度的增加,由1.0 mmol/L逐步增加至1.6 mmol/L时,扩增条带亮度逐步变暗。故而在正交试验中,将PCR Buffer浓度定为1.0 mmol/L。
2.1.4 dNTPs浓度
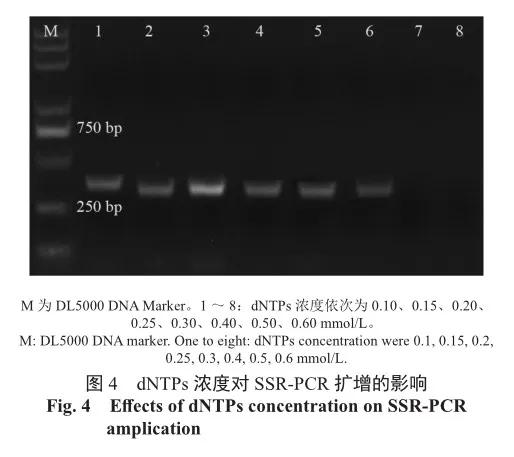
dNTPs是DNA复制的原材料,其浓度过高或过低均会影响PCR扩增的结果。由图4可知,dNTPs浓度为0.10~0.25 mmol/L时,随着浓度的增加,扩增出的条带逐渐明亮清晰;当浓度为0.25~0.40 mmol/L时,条带开始逐渐暗淡模糊;当dNTPs浓度为0.50~0.60 mmol/L时,未能成功扩增出目的条带。由此设计正交试验时,将dNTPs的浓度调整为0.10~0.40 mmol/L。
2.1.5 Taq酶浓度
Taq酶是PCR反应的催化剂,其浓度对扩增效果有重要的影响,Taq酶浓度过高会造成非特异性扩增,过低则会扩增不出条带。由图5可知,Taq酶浓度为0.05~0.25 U时,随着浓度的增加,扩增条带逐渐清晰明亮;当Taq酶浓度为0.35~0.45 U时,扩增条带明显变弱;Taq酶浓度为0.50~0.60 U时,未能成功扩增出目的条带。考虑到扩增效果,将Taq酶的浓度定为0.05~0.35 U。

2.1.6 引物浓度
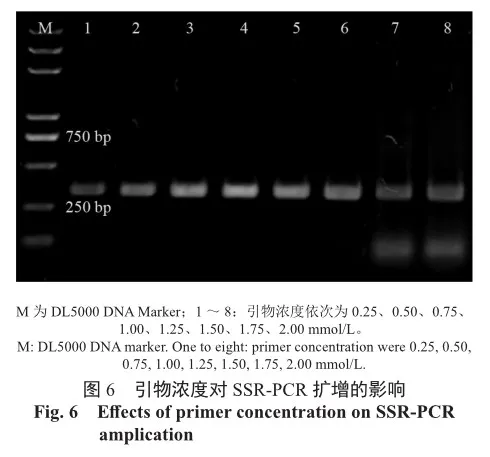
引物浓度也是影响PCR反应的重要因素,浓度过高会造成错配和非特异性扩增,引物之间形成二聚体的几率增大,浓度过低则会扩增不出条带或条带较弱。由图6可知,引物浓度为0.25~1.50 μmol/L时,随着浓度的增加,扩增的条带逐渐明亮;当浓度增加到1.75~2.00 μmol/L时,出现了二聚体,且条带变弱。考虑到扩增效果和成本,将引物浓度定为0.25~1.00 μmol/L。
2.2 正交试验结果分析
根据单因素试验结果,设定各因素用量范围,确定正交试验设计(表3)。选用引物Cc119,以 YL10号作为DNA模板进行PCR扩增,扩增结果如图7所示。由图7可知,不同浓度因素组合扩增出的结果存在较大差异。其中,处理1、5、7、9、13、14均未扩增出条带,处理2、3、8、10、11、12扩增出的条带相对较弱,其余处理均扩增出明亮清晰的条带。
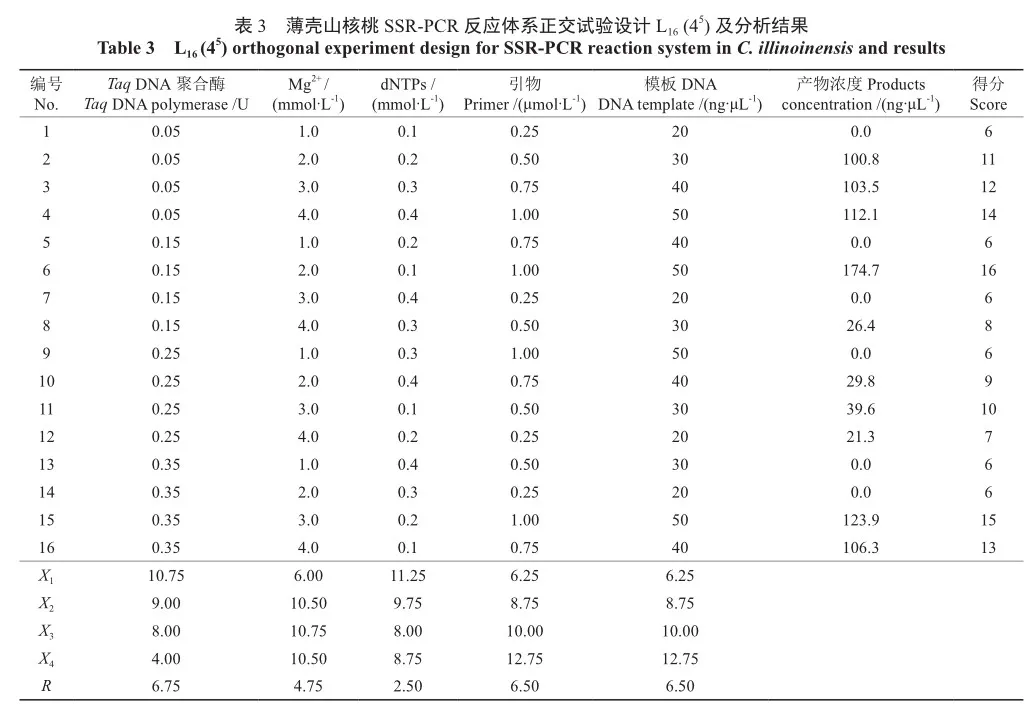
利用核酸检测仪检测正交试验16个处理的PCR产物浓度,依据浓度值进行打分,产物浓度最高得分16分,以此类推,16个处理的得分依次为6、11、12、14、6、16、6、8、6、9、10、7、6、6、15、13。计算同一因素相同水平下的分值平均值Xi(i=1,2,3,4,表示4个不同水平)和各因素的极差R,结果如表3所示。
其中,各因素对薄壳山核桃SSR-PCR反应体系的影响程度大小为:Taq酶>引物=DNA>Mg2+>dNTPs,处理6的扩增效果最好,即最佳反应体系为Taq酶0.15 U,Mg2+ 2.0 mmol/L,dNTPs 0.1 mmol/L,引物1.0 μmol/L,50 ng模板DNA 1.0 μL,10×PCR Buffer 1.0 μL,ddH2O 5.8 μL。

2.3 优化反应体系的验证
利用薄壳山核桃的8个品种(YL64号、YL35号、YL28号、YL23号、马罕、威斯顿、斯图尔特、波尼)和4对引物(Cc119、Cc129、ZMZ1、Cill-023),对优化后的体系进行验证。扩增产物进行1.5%琼脂糖凝胶电泳检测,电压120 V,时间30 min,电泳结果如图8所示。由图8可知,每对引物均扩增出明亮清晰的条带,说明优化后的反应体系稳定、可靠,适于多种薄壳山核桃品种及不同山核桃引物,可用于后续试验。

2.4 多态性引物筛选及退火温度优化
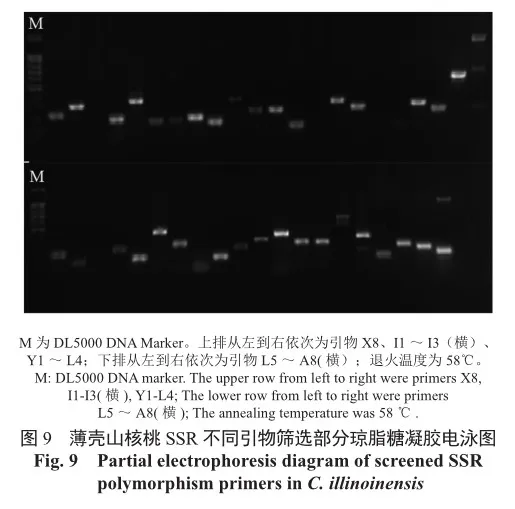
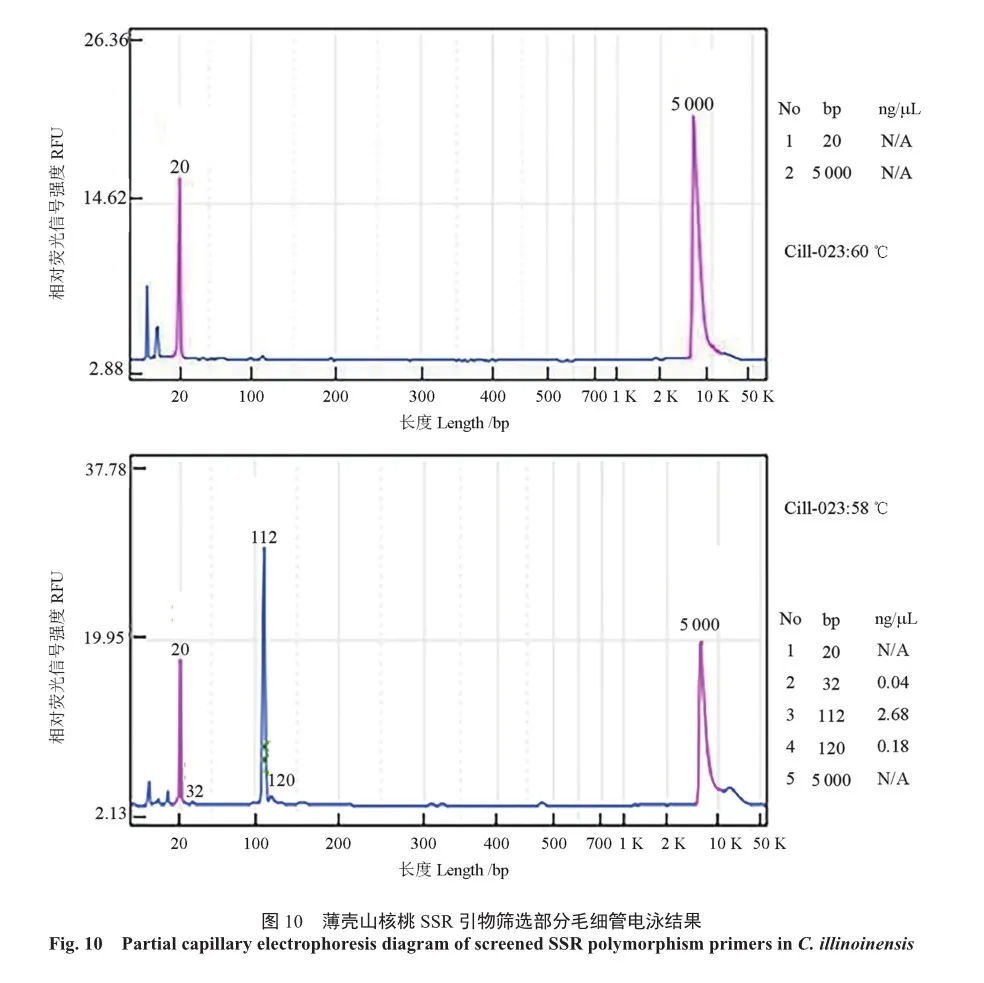
从文献中收集整理出283对核桃及山核桃引物,用优化后的反应体系,以8种薄壳山核桃DNA为模板(YL64号、YL35号、YL28号、YL23号、马罕、威斯顿、斯图尔特、波尼),在退火温度58 ℃的条件下进行PCR扩增。扩增产物进行1.5%琼脂糖凝胶电泳(电压220 V,30 min)检测,根据电泳结果初步筛选引物,部分电泳结果如图9所示。初步筛选结果表明,283对引物在58 ℃退火温度下有85对引物扩增出的条带明亮清晰,33对引物出现多条带,102对引物扩增出的条带较为暗淡,主带不清晰,剩余63对引物未能成功扩增。在SSR-PCR反应过程中,除了上述5个影响因素(Mg2+浓度、Taq酶浓度、引物浓度、dNTPs浓度、DNA模板浓度),引物的退火温度同样影响扩增效果。退火温度过低会出现杂带,即非特异性扩增。反之,退火温度过高则会降低整体扩增的效率,扩增效果差。根据初步筛选的结果,对出现多条带的33对引物进行升温处理,升高退火温度至60 ℃,最终有5对引物扩增出的条带单一明亮。对扩增条带效果不理想的102对引物进行降温处理,降低退火温度至56 ℃,最终有16对引物扩增出的条带明亮清晰,无拖带杂带。在3个退火温度(56、58、60 ℃)处理下,初步筛选出106对引物。用8个品种的薄壳山核桃为模板和优化后的反应体系,将106对引物扩增出的PCR产物送往杭州厚泽生物有限公司进行毛细管电泳检测。部分引物筛选毛细管电泳结果如图10所示。由图10可知,引物Cill-023在退火温度60 ℃时未能成功扩增,退火温度降至58 ℃时成功扩增出目的条带。根据毛细管电泳的结果,计算106对引物的等位基因频率,根据PIC值大小筛选出12对高多态性SSR引物,结果显示12对引物(表4)的多态性位点信息数(PIC)均大于0.5。
3 讨论与结论
3.1 讨 论
PCR反应体系的优良与否直接影响着PCR扩增产物的质量和数量[9],因而建立最佳的SSR-PCR反应体系,是应用SSR分子标记技术的前提[20]。通过设计单因素试验,确定各个影响因素的适宜浓度范围,以此来设计正交试验,可以更好地把控影响因素间的用量范围。对影响薄壳山核桃SSR-PCR反应的5个影响因素(Mg2+、Taq酶、引物、dNTPs、DNA)进行16个不同水平处理,能够快速筛选出最佳的反应体系。通过比较PCR扩增产物的浓度,计算同一因素相同水平下的分值平均值和各因素的极差,可以得知各因素对扩增效果的影响程度。正交试验也是目前最常用来建立SSR-PCR反应体系的方法[20-25]。本研究各因素对反应体系的影响程度大小为:Taq酶>引物=DNA>Mg2+>dNTPs。从前人对SSR-PCR反应体系优化的研究中发现,不同作物的SSRPCR反应体系的最大影响因素不同。禾本科植物冰草[10]为dNTPs浓度,元宝枫[25]为Taq酶浓度,桑树[26]、鹅掌楸[27]为Mg2+浓度。
尽管前人研究已经完成薄壳山核桃全基因组测序[28],并据此设计了大量的山核桃及核桃引物[12-18],但在薄壳山核桃分子标记试验中,稳定的高多态性引物仍是开展薄壳山核桃亲子鉴定、遗传多样性分析及交配系统分析等分子标记研究的前提。本试验以前人研究开发的核桃及山核桃引物为基础,利用优化后的薄壳山核桃SSR-PCR反应体系,从283对核桃及山核桃引物中,最终筛选出12对高多态性薄壳山核桃SSR引物。在引物筛选过程中,对前人开发出的核桃及山核桃引物退火温度进行优化,最终筛选出的薄壳山核桃SSR引物理论适宜退火温度与实际适宜退火温度存在一定差别,通过调整退火温度可确保引物及目的序列在PCR反应过程中顺利退火,保证扩增条带的清晰稳定。合适的退火温度亦是影响PCR扩增效果的关键因素,退火温度过低容易产生非特异性扩增,增大引物与模板错配的概率。而退火温度过高,则会降低整体扩增的效率,扩增条带减少,扩增效果差[30]。
在试验操作过程中,不可避免地会存在误差问题,多数反应体系优化试验利用正交实验直观分析法对结果进行分析,本试验采用核酸检测仪对PCR产物进行浓度检测,一定程度上降低了主观分析的误差性,提高了试验结果的准确性。且优化后的反应体系经过验证后稳定可靠,将优化后的反应体系应用于薄壳山核桃SSR引物筛选,从283对引物中筛选出12对SSR引物。
3.2 结 论
本研究采用单因素试验与正交试验相结合的方法,对影响薄壳山核桃SSR-PCR反应的5个影响因素进行16个处理,得出最佳的反应体系为:Taq酶0.15 U,Mg2+ 2.0 mmol/L,dNTPs 0.1 mmol/L,引物1.0 μmol/L,50 ng模板DNA 1.0 μL,10×PCR Buffer 1.0 μL,ddH2O 5.8 μL。筛选出的12对SSR引物经Cervus3.0软件测算等位基因频率,PIC值均高于0.5,为高度多态性,可直接用于后续薄壳山核桃分子标记试验,丰富了薄壳山核桃SSR分子标记的数量,为薄壳山核桃的良种选育、亲缘关系分析、父本鉴定等提供有效工具。
参考文献:
[1] 王艺颖,周文君,卢琨,等.湖南省4个薄壳山核桃品种果实形态和品质变化规律[J].经济林研究,2022,40(4):52-60. WANG Y Y, ZHOU W J, LU K, et al. Fruit morphology and quality variation patterns of four Carya illinoensis varieties in Hunan province[J]. Non-wood Forest Research,2022,40(4): 52-60.
[2] 何的明,傅松玲,毕慧慧,等.江淮地区薄壳山核桃主栽品种开花物候观测[J].经济林研究,2022,40(4):42-51. HE D M, FU S L, BI H H, et al. Observation on flowering phenology of main varieties of pecan in the area between Yangtze River and Huaihe River[J]. Non-wood Forest Research,2022,40(4): 42-51.
[3] 汤文华,窦全琴,潘平平,等.不同薄壳山核桃品种光合特性研究[J].南京林业大学学报(自然科学版),2020,44(3):81-88. TANG W H, DOU Q Q, PAN P P, et al. Photosynthetic characteristics of grafted plants of different Carya illinoinensis varieties[J]. Journal of Nanjing Forestry University (Natural Sciences Edition),2020,44(3):81-88.
[4] 常君,任华东,姚小华,等.薄壳山核桃‘Mahan’品种果实动态发育分析及营养物质积累规律研究[J].经济林研究,2019, 37(3):90-94,127. CHANG J, REN H D, YAO X H, et al. Dynamic development and nutrient accumulation regularity in Carya illinoensis ‘Mahan’ nut[J]. Non-wood Forest Research,2019,37(3):90-94,127.
[5] 刘国豪,杨倩,余速航,等.基于SSR分子标记的茶树新品种‘戚家美铭’的亲缘关系分析[J].中国野生植物资源,2023,42(3): 67-73. LIU G H, YANG Q, YU S H, et al. Genetic relationship analysis of a new tea variety ‘Qijiameiming’ based on SSR molecular markers[J]. Chinese Wild Plant Resources,2023,42(3):67-73.
[6] 罗冉,吴委林,张旸,等.SSR分子标记在作物遗传育种中的应用[J].基因组学与应用生物学,2010,29(1):137-143. LUO R, WU W L, ZHANG Y, et al. SSR marker and its application to crop genetics and breeding[J]. Genomics and Applied Biology,2010,29(1):137-143.
[7] 黄秦军,苏晓华,张香华.SSR分子标记与林木遗传育种[J].世界林业研究,2002,15(3):14-21. HUANG Q J, SU X H, ZHANG X H. Microsatellite marker and its application in tree genetics and breeding[J]. World Forestry Research, 2002,15(3):14-21.
[8] 韩洪强.利用形态学标记和SSR分子标记分析茄子种质资源遗传多样性[D].上海:上海交通大学,2009. HAN H Q. Analysis of genetic diversity of eggplants (Solanum melongena L.) by morphological markers and SSR markers[D]. Shanghai: Shanghai Jiao Tong University,2009.
[9] 黄彬,倪州献,周婷,等.观赏海棠SSR-PCR体系优化及引物筛选应用[J].南京林业大学学报(自然科学版),2018,42(3): 51-57. HUANG B, NI Z X, ZHOU T, et al. Optimization of SSR-PCR reaction system and primer screening in Malus crabapple[J]. Journal of Nanjing Forestry University (Natural Sciences Edition), 2018,42(3):51-57.
[10] 于肖夏,杨东升,于卓,等.冰草SSR-PCR反应体系优化及验证[J].草业科学,2020,37(9):1760-1769. YU X X, YANG D S, YU Z, et al. Optimization and validation of a simple sequence repeat (SSR)-PCR system in wheatgrass[J]. Pratacultural Science,2020,37(9):1760-1769.
[11] 黄国帅.薄壳山核桃品种差异性比较研究[D].杭州:浙江农林大学,2019. HUANG G S. A comparative study on variety differences of Carya illinoensis[D]. Hangzhou: Zhejiang A & F University,2019.
[12] GRAUKE L J, IQBAL M J, REDDY A S, et al. Developing microsatellite DNA markers in pecan[J]. Journal of the American Society for Horticultural Science,2003,128(3):374-380.
[13] LI J, ZENG Y R, SHEN D F, et al. Development of SSR markers in hickory (Carya cathayensis Sarg.) and their transferability to other species of Carya[J]. Current Genomics,2014,15(5):357-379.
[14] 戴晓港,房芝馨,胡雪纯,等.薄壳山核桃全基因组SSR分析及多态性引物筛选[J].分子植物育种,2021,19(24):8199-8207. DAI X G, FANG Z X, HU X C, et al. Whole genome SSR analysis and polymorphic primer screening in Carya illinoinensis[J]. Molecular Plant Breeding,2021,19(24):8199-8207.
[15] 齐建勋,王克建,吴春林,等.核桃EST-SSR标记的开发[J].农业生物技术学报,2009,17(5):872-876. QI J X, WANG K J, WU C L, et al. Development of EST-SSR markers in Juglans regia[J]. Journal of Agricultural Biotechnology, 2009,17(5):872-876.
[16] 张智勇.核桃EST-SSR引物开发及其在喙核桃保育遗传学中的应用[D].北京:北京林业大学,2013. ZHANG Z Y. Development and characterization of EST-SSRs for walnut (Juglans regia) and their utilization in conservation genetics of Annamocarya sinensis (Dode) Leroy[D]. Beijing: Beijing Forestry University,2013.
[17] 李晖,张瑞,彭方仁,等.美国山核桃种质资源遗传多样性ISSR分析[J].南京林业大学学报(自然科学版),2015,39(4): 7-12. LI H, ZHANG R, PENG F R, et al. Genetic diversity and identification of pecan by ISSR markers[J]. Journal of Nanjing Forestry University (Natural Sciences Edition),2015,39(4):7-12.
[18] 宋双,黄银芝,董雷鸣,等.山核桃SSR标记的初步开发[J].浙江农林大学学报,2012,29(4):626-633. SONG S, HUANG Y Z, DONG L M, et aNHpmuJftdaQvY9TKJmoKliyJVqtRZACPwXrpLMzv2Kw=l. Simple sequence repeat (SSR) markers in Carya cathayensis[J]. Journal of Zhejiang A & F University,2012,29(4):626-633.
[19] BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980,32(3):314-331.
[20] 李显煌,高丽云,王娟,等.云南金花茶SSR-PCR反应体系的建立与优化[J].中南林业科技大学学报,2019,39(8):101-108. LI X H, GAO L Y, WANG J, et al. Establishment and optimization of the SSR-PCR reaction system in Camellia fascicularis H. T. Chang[J]. Journal of Central South University of Forestry & Technology,2019,39(8):101-108.
[21] 黄芳芳,何长青,闫丽君,等.观光木SSR-PCR反应体系优化及引物筛选[J].中南林业科技大学学报,2015,35(10):142-146. HUANG F F, HE C Q, YAN L J, et al. Optimization of SSRPCR reaction system and prime screening for Tsoongiodendron odorum[J]. Journal of Central South University of Forestry & Technology,2015,35(10):142-146.
[22] 李守明,史荣梅,李辉玲.西北地区大蒜及其野生蒜SSRPCR体系优化及引物筛选[J].江苏农业科学,2022,50(18): 274-279. LI S M, SHI R M, LI H L. Optimization of SSR-PCR system and primers screening of Allium sativum L. and wild-garlic in Northwestern China[J]. Jiangsu Agricultural Sciences,2022,50(18): 274-279.
[23] 林萍,张含国,谢运海.正交设计优化落叶松ISSR-PCR反应体系[J].生物技术,2005,15(5):34-37. LING P, ZHANG H G, XIE Y H. Study on optimization for ISSR reaction system of Larix using orthogonal design[J]. Biotechnology, 2005,15(5):34-37.
[24] 江常胜,朱洪海,钟永丽,等.正交试验与单因子双重试验法优化玉米PCR反应体系[J].智慧农业导刊,2022,2(21):14-17. JIANG C S, ZHU H H, ZHONG Y L, et al. Optimization of PCR reaction system of corn by orthogonal test and single factor double test[J]. Journal of Smart Agriculture,2022,2(21):14-17.
[25] 燕丽萍,王因花,吴德军,等.元宝枫SSR-PCR反应体系优化及引物筛选[J].分子植物育种,2021,19(7):2279-2285. YAN L P, WANG Y H, WU D J, et al. Optimization and primers screening of SSR-PCR reaction system for Acer truncatum[J]. Molecular Plant Breeding,2021,19(7):2279-2285.
[26] 刘玲,陈祥平,范小敏,等.桑树SSR-PCR反应体系的优化[J].江苏农业科学,2019,47(14):45-49,58. LIU L, CHEN X P, FAN X M, et al. Optimization of SSR-PCR reaction system for mulberry[J]. Jiangsu Agricultural Sciences, 2019,47(14):45-49,58.
[27] 贾波,徐海滨,徐阳,等.鹅掌楸Genomic-SSR反应体系优化[J].林业科学研究,2013,26(4):506-510. JIA B, XU H B, XU Y, et al. Optimization of genomic-SSR reaction system in Liriodendron[J]. Forest Research,2013,26(4): 506-510.
[28] HUANG Y J, XIAO L H, ZHANG Z R, et al. The genomes of pecan and Chinese hickory provide insights into Carya evolution and nut nutrition[J]. Giga Science,2019,8(5):1-17.
[29] 贾展慧,宣继萍,张计育,等.薄壳山核桃转录组中的SSR位点信息分析[J].分子植物育种,2019,17(10):3305-3311. JIA Z H, XUAN J P, ZHANG J Y, et al. Analysis of SSR information in Carya illinoinensis transcriptome[J]. Molecular Plant Breeding,2019,17(10):3305-3311.
[30] 吴望蕊,王英哲,国坤,等.朝鲜淫羊藿ISSR-PCR反应体系优化及引物筛选[J].分子植物育种,2023,21(7):2318-2324. WU W R, WANG Y Z, GUO K, et al. Optimization of ISSR-PCR reaction system and primer selection of Epimedium koreanum Nakai[J]. Molecular Plant Breeding,2023,21(7):2318-2324.
[本文编校:谢荣秀]

