花生GATA 基因家族鉴定和表达分析







摘要:[目的]探究GATA 基因家族在花生生长发育和抗旱调控中的作用。[方法]通过生物信息学方法鉴定花生GATA 家族成员,分析其系统进化、基因结构、组织表达模式,最后结合转录组数据和实时荧光定量PCR 技术筛选花生GATA 家族响应干旱胁迫的关键基因。[结果]本文鉴定到47 个GATA 类转录因子编码基因,它们不均匀地分布在除2 号、4 号和14 号染色体以外的17 条染色体上。理化性质分析表明,该家族属于亲水性蛋白,大部分基因呈酸性,只有Arahy. QG9XFC. 1 具有信号肽。根据拟南芥家族同源基因聚类分析结果,可将花生GATA 家族蛋白分为4 个亚家族。其中第Ⅰ、Ⅱ、Ⅲ亚家族成员的锌指结构域特征模体为CX2CX18CX2C,而第Ⅳ亚家族为CX2CX20CX2C 类型。大部分成员在A 亚基因组和B 亚基因组中呈对称分布,组织表达模式分析显示,超过半数的GATA 家族成员在22 个组织中表现不同程度的特异性表达。其中第Ⅰ亚家族Arahy. LJYJ4M、Arahy. VS8GG1、Arahy. 3S8P0L 和Arahy. I6JZDT 在所有组织中均显示较高的表达水平。然而Arahy. QY7BN7 和Arahy. F7WS4I 在叶片,Arahy. C6NV9N 在花被中表达较高,Arahy. 4Y7J59 和Arahy. D56WMI 在种子中呈现组织特异性高表达特性。转录组数据分析发现,34 个GA ⁃TA 基因响应干旱、低温、ABA 和BR 激素处理,5 个GATA 基因在干旱和低温胁迫下表达量发生显著上调,1 个基因发生显著下调。进一步RT-qPCR 分析表明,Arahy. C6NV9N 和Arahy. 2SUK7S 特异性响应PEG 胁迫的基因,尤其前者在叶组织中胁迫早期呈现持续上调表达。[结论]花生中有47 个GATA 基因,其中Arahy. C6NV9N 和Ara⁃hy. 2SUK7S 可能是花生GATA 家族响应干旱胁迫关键基因。
关键词:花生; 转录因子; GATA; 组织表达模式; 干旱胁迫
中图分类号:S565.2 文献标识码:A 文章编号:1671-8151(2024)02-0010-13
转录因子,又称反式作用因子,能够特异性识别和结合靶基因的启动子,进而调控下游靶基因的表达[1]。研究人员根据转录因子与DNA 上特定序列的结合方式的不同,将其分为不同的家族,比如:bZIP(碱性亮氨酸拉链)、MYB、ZFP(锌指蛋白)、MADS、WRKY、NAC(NAM、ATA-F1/2 和CUC1/2)、PHD(植物保守结构域)等[2]。锌指蛋白(zinc-finger protein,ZFP)是指具备指状结构特征并且能够与锌结合的一类蛋白质的总称,是植物中重要的转录因子之一。根据锌指蛋白中氨基酸残基的数量和位置对其进一步分类,比如:C2H2型、GATA 型、C3H6 型等。已有研究报道表明,GATA 在植物种子萌发、器官发育、碳代谢、氮代谢、逆境胁迫、器官形成等生物学过程中发挥着重要的调控作用[3]。
1988 年,研究人员首次在鸡的珠蛋白基因的启动子上发现并报道了GATA 蛋白,证明了该转录因子参与鸡的造血过程[4-5]。之后,研究人员又鉴定出了GATA-1~GATA-6 等多个GATA 蛋白[6],1993 年,研究人员首次在植物烟草中鉴定出了GATA 转录因子,并且命名为NTL1,研究表明该转录因子参与烟草氮代谢生物学过程[7]。因此表明GATA 基因不仅仅存在于高等动物中,在植物中同样存在。随后研究人员在拟南芥中发现了调控拟南芥下胚轴和叶柄伸长的GATA 转录因子ZIM[8]、调控拟南芥种子萌发的GATA 转录因子BME3[9]、调控拟南芥光形态建成转录因子GATA2[10];在水稻中发现了调控水稻器官形成的GATA 家族转录因子NECKLEAF1[11]和叶绿素合成及叶绿体发育的GATA 家族转录因子Cga1[12];在小麦中发现了转录因子TaGATA1 通过直接调控TaABI5 的表达增强小麦种子休眠能力,进而提高穗发芽抗性[13];在玉米中GATA 家族转录因子GATA7 和GATA33 参与调控玉米顶尖分生组织生长和发育[14];在构树中发现GATA 家族转录因子可以参与调控冷胁迫[15];杨树(Popu⁃lus)转录因子GATA13 和GATA19 通过调控气孔的大小或关闭,提高植株的水分利用效率,进一步提高抗旱性[16]。
目前,关于GATA 基因家族方面的研究大多数都还是停留在拟南芥和水稻等模式作物中,而花生GATA 基因家族还鲜有报道,关于该家族生物信息学分析更少。因此,本研究通过对花生全基因组中的GATA 基因家族进行鉴定和组织表达模式进行分析,旨在为花生GATA 转录因子对于花生生长发育和抗逆调控分析奠定基础。
1 材料与方法
1. 1 材料
本研究所选用的种子(周花5 号、远杂9102),聚乙二醇6000(科密欧化学试剂有限公司)。
1. 2 方法
1. 2. 1 花生GATA 基因家族成员鉴定
以“GATA”为关键词在花生基因组数据库(https://peanutbase. usda. iastate. edu/home)中Geneamp;Gene Family 工具进行检索,下载检索到的基因氨基酸序列(存在多个转录本的取最长),将氨基酸序列上传至NBCI-CDD 网站((https://www. ncbi. nlm. nih. gov/cdd/))和SMART 网站(https://smart. embl. de/)进行结构域分析,去除不含“ZFP-GATA”结构域的序列最终得到47 个花生GATA 基因家族成员。将花生GATA 基因家族成员氨基酸序列上传至Expasy 网站(https://web. expasy. org/protparam/)进行氨基酸理化性质分析包括:氨基酸数量、分子量、等电点和总平均亲水性,将所有成员理化性质汇总,使用Excel2016 软件制表。
1. 2. 2 花生GATA 基因家族成员染色体定位
将GATA 家族成员在花生基因组数据库中用Geneamp;Gene Family 工具进行检索,下载染色体编号、起始位置以及终止位置,使用MapGene2Chrom(http://mg2c. iask. in/mg2c_v2. 1/)工具进行可视化。
1. 2. 3 花生GATA 基因家族系统进化关系
从拟南芥数据库TAIR(https://www. arabi⁃dopsis. org/)中获得拟南芥GATA 基因家族氨基酸序列,将花生和拟南芥GATA 基因家族氨基酸序列上传至MEGA 7. 0 软件,使用Clustal 工具进行序列比对,比对结果进行合理的删除,使用邻接法(Neighbor-Joining Method)构建系统进化树,校验参数Bootstrap 设置为重复1000 次。导出并保存. nwk 文件,使用ITOL(https://itol. embl. de/)工具对进化树进行美化,最后根据拟南芥GATA基因家族同源基因聚类分析结果,对花生GATA基因家族进行分类。
1. 2. 4 花生GATA 基因家族基因结构分析
将花生GATA 基因家族氨基酸序列上传至MEME 网站进行motif 分析,下载mast. xml 文件;花生GATA 基因家族氨基酸上传至CDD 网站进行结构域分析,下载结果;将1. 3 中MEGA 7. 0 比对之后花生的氨基酸序列单独提取出来使用邻接法(Neighbor-Joining Method)构建系统进化树,校验参数Bootstrap 设置为重复1000 次),导出并保存. nwk 文件;最后使用TBrools 中Gene StructureView 工具对上述步骤结果进行可视化。
1. 2. 5 花生GATA 基因家族序列比较
将花生GATA 基因家族氨基酸序列上传至MEGA 7. 0 软件,使用Clustal 工具进行比对,找到GATA 结构域并截图保存。
1. 2. 6 花生GATA 蛋白信号肽、亚细胞定位预测
将花生GATA 基因家族氨基酸序列上传至SignalP 6. 0 工具网站(https://services. health⁃tech. dtu. dk/services/SignalP-6. 0/)进行信号肽预测;将花生GATA 基因家族氨基酸序列上传至WoLF PSORT 工具网站(https://wolfpsort. hgc.jp/)进行亚细胞定位。
1. 2. 7 花生GATA 基因家族共线性分析
使用TBtools- Ⅱ(Toolbox for Biologist)v2. 001 软件中Text merge for MCScanX 工具提取花生物种内GATA 基因间的关系,使用CircleGene View 工具进行可视化。
1. 2. 8 花生GATA 基因家族组织表达、不同激素、干旱和低温胁迫处理响应分析
22 个花生不同发育时期、不同组织[17]和不同激素、干旱和低温胁迫处理转录组数据分别来自于花生基因组数据库(https://peanutbase. usda. ia⁃state. edu/home)和PGR 网站(http://peanutgr.fafu. edu. cn/index. php)。使用R 软件对基因FPKM 值进行log2 标准化后,使用Excel 2016 绘制热图。
1. 2. 9 花生GATA 基因家族在干旱响应分析
以耐旱品种远杂9102 和周花5 号为试验对象,使用75% 酒精消毒10 min 后,放置清水中进行催芽,待种子露白,定植于水培盒中,使用清水于人工气候箱(16 h 光照/8 h 黑暗;28 °C)中培养,待植株发育至三叶期时使用20%PEG6000 进行开始处理,于0、3、6、9、12 h 分别对叶片和根系进行取样,液氮速冻后用于总RNA 的提取。RT-qPCR分析采用诺唯赞生物科技股份有限公司荧光定量酶试剂盒(ChamQ Universal SYBR qPCR masterMix),在Bio-Rad CFX 96 平台上进行,以Actin11为内参,利用primer 5. 0 进行引物设计(表1),每个样品3 次重复。
2 结果与分析
2. 1 花生GATA 基因家族的鉴定与理化性质分析
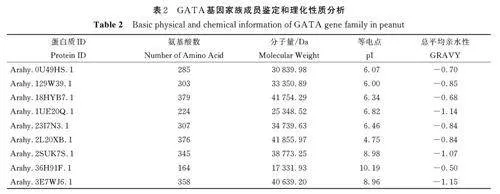
通过花生基因组数据库检索,NCBI-CDD 和SMART 数据库验证,最终鉴定得到47 个花生GATA 基因家族成员,经Expasy 数据库理化性质进行分析,结果表明(表2):GATA 基因家族成员氨基酸数量范围在108~415 个,分子量在12 119. 71~45 242. 82 Da,其中Arahy. C6NV9N分子量最小,Arahy. V4I8CJ 分子量最大;等电点在4. 5~10. 19,小于7 的有27 个,占总数目的57. 45%,而大于7 的有20 个,占总数目的42. 55%;总平均亲水性均小于零。由此说明该家族成员大部分呈酸性,且均为亲水性蛋白。
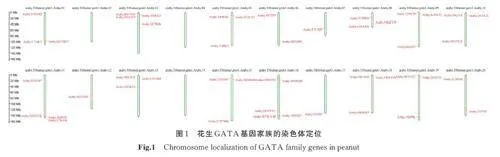
2. 2 花生GATA 基因家族成员染色体定位
根据花生基因组数据库中GATA 基因家族染色体位置信息,绘制染色体定位图(图1),结果显示:GATA 基因家族分布在17 条染色体上,第2、4和14 号染色体上未检测到GATA 基因。第16 号染色体分布最多有5 个GATA 基因;其次是第3 和11 号染色体分布4 个GATA 基因;第1、5、6、9、10、13、15、18、19 号染色体均分布3 个GATA 基因;第6 和20 号染色体上分布2 个GATA 基因,最后是第7、12、17 号染色体分布最少只有1 个GATA基因。说明GATA 家族基因不均匀的分布在花生染色体上。
2. 3 花生GATA 基因家族系统进化树
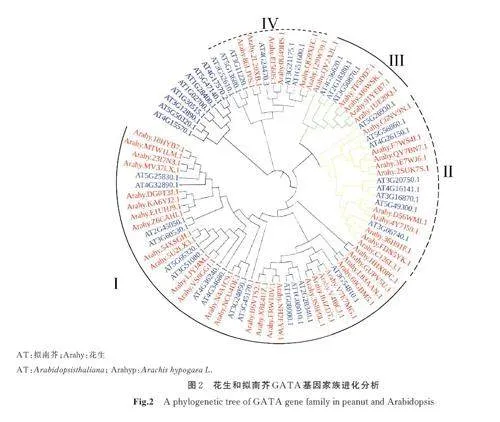
为了进一步探究花生GATA 基因家族成员之间进化关系,使用MAGA 7. 0 软件对花生GATA基因家族成员(红色)和拟南芥家族成员(蓝色)进行多重比较,构建系统进化树(图2)。结果表明:根据拟南芥家族同源基因聚类分析结果,将花生GATA 基因家族分为4 个亚家族,第Ⅰ 亚家族成员有26 个,第Ⅱ 亚家族成员数量次之有10 个,第Ⅲ 亚家族成员最少有5 个,第Ⅳ 亚家族有6 个成员。根据系统发育关系,可以推测基因亚家族成员之间具有较近进化距离,可能具有相同的生物学功能,因此可以根据家族成员进化关系推测GATA 基因家族成员的功能。
2. 4 花生GATA 基因家族结构域和比对和分类
为了进一步揭示花生GATA 基因功能特性,对该家族基因进行了基因结构分析,结果表明(图3、图4):改为“根据图 3B 可知,所有亚家族中第Ⅰ亚家族中检索到的基序最多含有8 个不同基序,第Ⅲ亚家族含有基序最少,只有1 个基序,进一步研究发现第Ⅰ 亚家族均含有Motif2,第Ⅱ 亚家族均含有Motif8,第Ⅲ亚家族均含有Motif1,第Ⅳ亚家族均含有Motif1 和Motif5,不同的亚家族之间Motif 数量和种类也不相同,这说明不同亚族中基序分布的不同导致其可能在进化过程中出现了功能多样性演变;图3C 可知,除了第Ⅱ 亚家族部分GATA 结构域在氨基酸序列的前端,其它基因GATA 结构域皆在氨基酸序列后端;除Arahy. 18HYB7、Arahy. MTW1LM、Arahy. 69GBM5、Arahy. V4I8CJ 以外所有基因均包含CDS 区和UTR 区,GATA 基因外显子呈现1 个(Arahy. 69GBM5)到11 个(Arahy. 2L20XB、Arahy. 86LJVS)的结构变化,不同亚家族成员具有不同的CDS 数量,而相同的亚家族中CDS 数量相近(图3D)。同时,在相同亚族中GATA 转录因子相似的结构特征表明其含有相似的功能。
通过MEGA 7. 0 中ClustalW 工具比对发现,第Ⅰ 、Ⅱ 、Ⅲ 亚家族成员锌指结构域类型为CX2CX18CX2C,而第Ⅳ亚家族为CX2CX20CX2C 类型(图4)。其它氨基酸同一亚家族存在相似性,不同亚家族存在特异性,推测为物种进化的结果。
2. 5 花生GATA 基因家族信号肽预测
对花生GATA 蛋白进行信号肽预测,发现除Arahy. QG9XFC. 1 具有信号肽以外其它蛋白均不具备信号肽(图5)。
2. 6 花生GATA 基因家族亚细胞定位
为了进一步探索GATA 基因家族蛋白的表达位置,我们使用WoLF PSORT 工具对该家族蛋白进行亚细胞定位预测,结果显示(图6):有39 个蛋白定位于细胞核中,占总数的83%,只有8 个蛋白(Arahy. 0U49HS. 1、Arahy. 3J6WSK. 1、Arahy. 4Y7J59. 1、Arahy. EI5605. 1、Arahy. LJYJ4M. 1、Arahy. TF5DD7. 1、Arahy. V7E7MG. 1、Arahy.F7WS4I. 1)定位于叶绿体,占总数17%。说明大多数蛋白在细胞核中表达。
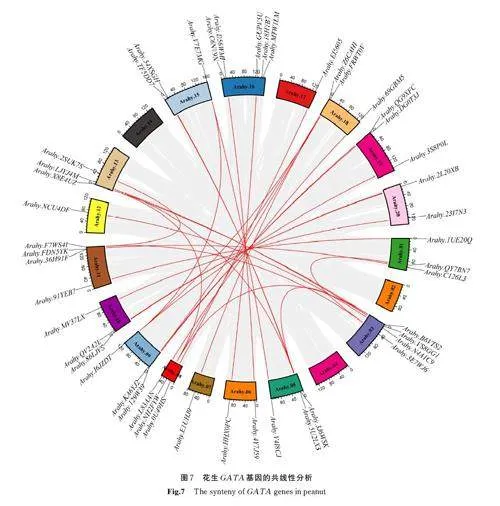
2. 7 花生GATA 基因家族共线性分析
为进一步探究花生GATA 基因家族成员间的进化关系,使用TBtools 软件对家族成员进行共线性分析。研究发现在47 个成员中,共有19 对家族成员在花生A 亚基因组和B 亚基因组相应的位置,占花生GATA 基因家族成员总数的80%(图7)。进一步研究又发现这些成对出现的家族成员,在系统进化树也有较近的距离,因此可以推测为异源四倍体栽培种花生形成时,由2 个二倍体野生种花生各自携带而来。剩下20% 没有成对出现的基因家族成员则可能是在栽培花生形成时其染色体发生了倒位、易位,反转录转座等事件。
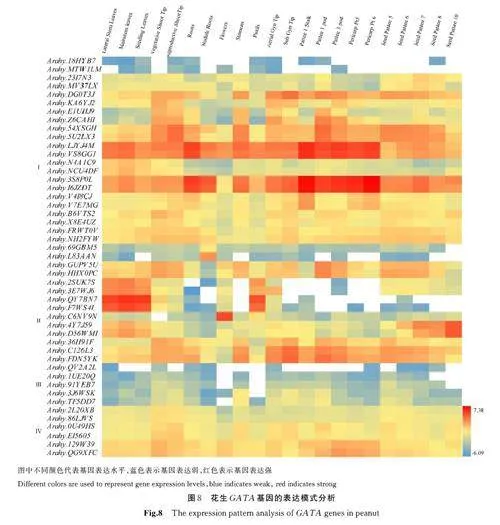
2. 8 花生GATA 基因家族组织表达模式分析
为进一步探究花生GATA 基因家族生物学功能,对基因组数据库不同组织转录组数据,绘制热图(图8),结果表明:在47 个家族成员中有34 个成员在22 个组织中均有不同程度的表达(白色表示未检测到表达),其中Arahy. LJYJ4M、Arahy.VS8GG1、Arahy. 3S8P0L、Arahy. I6JZDT 在所有组织中都具有较高的表达,而且他们均属第Ⅰ 亚家族,而Arahy. QY7BN7 和Arahy. F7WS4I 在叶片中有较高的表达,Arahy. C6NV9N 在花被中具有较高的表达,Arahy. 4Y7J59 和Arahy. D56WMI在种子中具有较高的表达,进一步研究发现,该家族成员组织表达模式相似的基因其进化关系也比较近,因此可以推测其生物学功能也可能相似,根据其组织表达情况,推测GATA 家族成员在花生生长发育过程中的功能,但是需要相关分子生物学试验加以论证。
2. 9 花生GATA 基因家族对不同激素、干旱和低温胁迫处理响应分析
为了进一步探究不同非生物胁迫和激素处理下GATA 基因的表达模式,对干旱、低温、ABA 和BR 处理之后花生幼苗叶片的转录组数据进行分析,结果显示(图9):34 个基因家族成员响应干旱、低温、ABA 和BR 处理(白色表示未检测到表达)。其中在干旱和低温处理下Arahy. C6NV9N、Arahy. 4Y7J59、Arahy. 3E7WJ6、Arahy. D56WMI和Arahy. 2SUK7S 表达量均发生显著上调,而Arahy. Z6CAHI 下调最为明显,进一步研究发现Arahy. D56WMI、Arahy. 4Y7J59、Arahy. 2SUK7S在受到干旱处理和ABA 处理后基因表达变化出现了一致性,表明它们对于干旱胁迫的调控与ABA 信号通路相关,通过对比干旱胁迫和低温胁迫基因表达变化情况可知,该家族成员对于干旱胁迫更为敏感且大多数基因表达量上调。
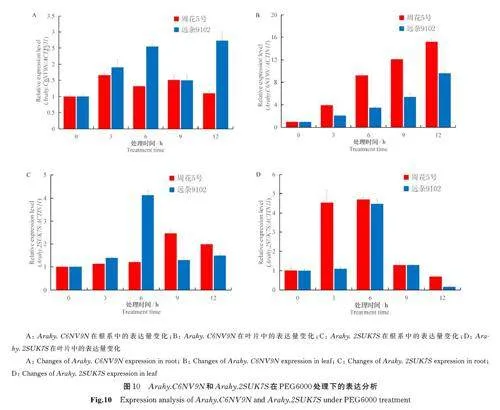
2. 10 花生GATA 基因家族PEG6000 处理下表达分析
基于干旱处理的转录组数据,对干旱处理后表达量明显上升的5 个基因(Arahy. C6NV9N、Arahy. 4Y7J59、Arahy. 3E7WJ6、Arahy.D56WMI、Arahy. 2SUK7S)进行PEG 处理。采用20% 的PEG 模拟干旱进行处理,结果显示:只有Arahy. C6NV9N 和Arahy. 2SUK7S 响应PEG 胁迫且表现出规律性变化,但响应模式不同(图10)。其中Arahy. C6NV9N 在受到PEG 胁迫后在根系中的表达量在3 h 时显著增高,但在周花5 号材料遗传背景下其表达差异仅为1. 7 倍,而在远杂9102材料遗传背景下表现为PEG 胁迫后6 和12 h 双峰表达模式,表达量为未胁迫对照的2. 6 倍;叶片中Arahy. C6NV9N 受到胁迫以后在2 个品种中皆显著上调并且均在12 h 时达到最高表达水平,最低上调幅度为4 倍,最大上调幅度达16 倍以上。Arahy. 2SUK7S 在受到PEG 胁迫后在根系中的表达量表现为先上升后下降,在周花5 号中其表达量峰值在6 h 达到显著水平,而远杂9102 中峰值在9 h 达到显著水平;在叶片中Arahy. 2SUK7S 受到胁迫后,在周花5 号中3 h 时显著升高,胁迫9 h 后降低到一般水平,而在远杂9102 中其表达量在3 h无明显变化,在6 h 时达到峰值,之后也降低到一般水平,在12 h 时降到最低。
3 讨论与结论
能够特异性识别并结合靶基因的顺式元件的蛋白被称为转录因子。锌指转录因子是目前研究较为深入的转录因子家族之一,GATA 型转录因子又是锌指蛋白转录因子中的一种重要类型。已有研究报道该家族影响着植物种子萌发、器官发育、碳代谢、氮代谢等[3]过程,本研究在栽培花生中鉴定得到47 个GATA 基因家族成员,成员数目比水稻(29 个)[18]、玉米(37 个)[19]、高粱(30 个)[20]、辣椒(24 个)[21]多,但是又比烟草(57 个)[22]少,推测可能与水稻、拟南芥和辣椒属于二倍体,而烟草和花生属于异源四倍体有关。
一级序列结构分析表明,花生GATA 蛋白均为不稳定的亲水蛋白,有27 个呈酸性,20 个呈碱性;编码108~415 个氨基酸,分子质量12 119. 71~45 242. 82 Da,其锌指结构域包括2 种类型:CX2CX18CX2C 型和CX2CX20CX2C 型,与烟草GATA 基因家族锌指结构域相似,且同一亚家族基因具有相同锌指结构域类型[22]。进一步对GATA 家族成员基因结构研究发现,与已报道基因家族相似,同一亚家族成员之间其基因结构具有相似性,而不同亚家族成员之间具有特异性。因此可见或许正是由于这些家族成员之间的理化性质和基因结构的相似性和特异性,才使得GATA 基因家族成员具有相似而又不同的生物学功能。
通过共线性分析发现在GATA 基因家族中半数以上成员在A、B 亚基因组之间对应位置存在共线性,这与花生中另外一个重要的转录因子家族bHLH 转录因子共线性结果一致[23],也从侧面印证了异源四倍体栽培花生基因组来自于二倍体野生花生A. duranensis 和A. ipaensis 祖先种。通过组织表达模式分析,发现第Ⅰ 亚家族中表达量最高组织为花生发育后期生殖生长阶段的器官中,因此推测第Ⅰ亚家族成员其生物学功能可能与后期种子发育成熟相关,而第Ⅱ 亚家族则主要在叶片中突出表达,第Ⅲ、Ⅳ亚家族表达量都比较低,因此研究家族成员时要把重点放到第Ⅰ、Ⅱ亚家族。利用非生物胁迫和激素处理的花生转录组数据对GATA 基因家族表达模式进行进一步分析,发现半数以上基因参与花生非生物胁迫应答,如在干旱胁迫和激素处理下Arahy. D56WMI、Arahy. 4Y7J59、Arahy. 2SUK7S 基因表达变化出现了一致性。因此可以推测这3 个基因对于干旱胁迫的调控可能是依赖ABA 信号途径[24]。RT-qPCR 验证结果显示Arahy. C6NV9N 和Arahy. 2SUK7S 在PEG 处理后表达均发生显著变化,然而花生GATA 基因具体抗旱调控功能有待进一步研究。
综上所述,本研究以花生GATA 家族基因为对象,主要分析了GATA 基因家族系统理化性质、进化关系、基因结构、共线性以及组织表达模式和非生物胁迫与激素响应,进一步加深了对于花生GATA 基因家族成员的了解,为花生GATA 基因家族成员进行下一步研究提供一定的基础,为抗逆、高产花生品种的选育提供新思路。
参考文献
[1]Zhang J Z. Overexpression analysis of plant transcription factors
[J]. Current Opinion in Plant Biology, 2003, 6(5): 430-440.
[2]侯思宇,孙朝霞,郭彬,等. 大豆两个C2H2 型转录因子基因序
列特征及表达分析[J].植物生理学报,2014,50( 5): 665-674.
Hou S Y, Sun C X, Guo B, et al. Cloning and expression
analysis of two C2H2 transcription factors in soybean [J]. Plant
Physiology Journal, 2014, 50(5): 665-674.
[3]王娟,兰海燕.GATA 转录因子对植物发育和胁迫响应调控的
研究进展[J]. 植物生理学报,2016,52(12):1785-1794.
Wang J, Lan H Y. Advances in regulation of GATA
transcription factor to plant development and stress responses
[J]. Plant Physiology Journal, 2016, 52(12): 1785-1794.
[4]Evans T, Reitman M, Felsenfeld G. An erythrocyte-specific
DNA-binding factor recognizes a regulatory sequence common
to all chicken globin genes [J]. Proceedings of the National
Academy of Sciences of the United States of America, 1988, 85
(16): 5976-5980.
[5]Omichinski J G, Clore G M, Schaad O, et al. NMR structure
of a specific DNA complex of Zn-containing DNA binding
domain of GATA-1[J]. Science, 1993, 261(5120): 438-446.
[6]张未丽,刘智皓,吴风瑞,等. 转录因子GATA-2 在脊椎动物发
育过程中的作用[J]. 四川动物,2009,28(6):945-948.
Zhang W L, Liu Z H, Wu F R, et al. Role of the GATA-2
transcription factor in vertebrates during development [J].
Sichuan Journal of Zoology, 2009, 28(6): 945-948
[7]Daniel-Vedele F, Caboche M. A tobacco cDNA clone encoding
a GATA-1 zinc finger protein homologous to regulators of
nitrogen metabolism in fungi [J]. Molecular Genetics and
Genomics, 1993, 240(3): 365-373.
[8]Shikata M, Matsuda Y, Ando K, et al. Characterization of
Arabidopsis ZIM, a member of a novel plant-specific GATA
factor gene family [J]. Journal of Experimental Botany, 2004,
55(397): 631-639.
[9]Liu P P, Koizuka N, Martin R C, et al. The BME3 (Blue
Micropylar End 3) GATA zinc finger transcription factor is a
positive regulator of Arabidopsis seed germination [J]. The
Plant Journal for Cell and Molecular Biology, 2005, 44(6):
960-971.
[10]Luo X M, Lin W H, Zhu S, et al. Integration of light- and
brassinosteroid-signaling pathways by a GATA transcription
factor in Arabidopsis [J]. Developmental Cell, 2010, 19(6):
872-883.
[11]Wang F, Zhu D M, Huang X, et al. Biochemical insights on
degradation of Arabidopsis DELLA proteins gained from a cellfree
assay system [J]. The Plant Cell, 2009, 21(8): 2378-
2390.
[12]Hudson D, Guevara D R, Hand A J, et al. Rice cytokinin
GATA transcription factor1 regulates chloroplast development
and plant architecture [J]. Plant Physiology, 2013, 162(1):
132-144.
[13]Wei X N, Li Y Y, Zhu X L, et al. The GATA transcription
factor TaGATA1 recruits demethylase TaELF6-A1 and
enhances seed dormancy in wheat by directly regulating
TaABI5 [J]. Journal of Integrative Plant Biology, 2023, 65
(5): 1262-1276.
[14]Zhan J, Thakare D, Ma C, et al. RNA sequencing of lasercapture
microdissected compartments of the maize kernel
identifies regulatory modules associated with endosperm cell
differentiation[ J]. The Plant Cell, 2015, 27(3): 513-531.
[15]Peng X, Wu Q Q, Teng L H, et al. Transcriptional regulation
of the paper mulberry under cold stress as revealed by a
comprehensive analysis of transcription factors [J]. BMC Plant
Biology, 2015, 15(1): 108.
[16]沈超. 杨树PdGATA19 和PdGATA13 在干旱和低氮胁迫下
的功能研究[D]. 北京:北京林业大学,2021.
Shen C. Functional analyses of poplar PdGATA19 and
PdGATA13 in response to low-nitrogen and drought stress
[D]. BeiJing: Beijing forestry university, 2021.
[17]Clevenger J, Chu Y, Scheffler B, et al. A developmental
transcriptome map for allotetraploid Arachis hypogaea [J].
Frontiers Plant Science, 2016, 7: 1446.
[18]骆鹰,王有成,王伟平,等. 水稻GATA 基因家族生物信息学
分析[J]. 分子植物育种,2018,16(17):5514-5522.
Luo Y, Wang Y C, Wang W P, et al. Bioinformatics analysis
of GATA gene family in rice [J]. Molecular Plant Breeding,
2018, 16(17): 5514-5522.
[19]王延召,周波,韩小花,等. 玉米GATA 基因家族的全基因组
鉴定及热胁迫下的表达分析[J]. 河南农业科学,2020,49
(11):19-25.
Wang Y Z, Zhou B, Han X H, et al. Genome-wide
identification of maize GATA gene family and expression
analysis under heat stress [J]. Journal of Henan Agricultural
Sciences, 2020, 49(11): 19-25.
[20]宋迎辉,朱灿灿,代书桃,等. 高粱GATA 基因家族的全基因
组鉴定及表达分析[J]. 山东农业科学,2022,54(8):14-23.
Song Y H, Zhu C C, Dai S T, et al. Genome-wide
identification and expression of GATA gene family in Sorghum
bicolor [J]. Shandong Agricultural Sciences, 2022, 54(8):
14-23.
[21]袁岐,张春利,赵婷婷,等. 辣椒GATA 转录因子的生物信息
学分析[J]. 中国农学通报,2017,33(17):24-31.
Yuan Q, Zhang C L, Zhao T T, et al. Bioinformatics analysis
of GATA transcription factor in Pepper [J]. Chinese
Agricultural Science Bulletin, 2017, 33(17): 24-31.
[22]杨玄松,谢雯榕,顾钢,等. 普通烟草GATA 转录因子家族鉴
定及基因表达分析[J]. 烟草科技,2023,56(1):11-23.
Yang X S, Xie W R, Gu G, et al. Identification and gene
expression analysis of GATA transcription factor family in
Nicotiana tabacum [J]. Tobacco Science amp; Technology,
2023, 56(1): 11-23.
[23]王勇江,陈克平,姚勤.bHLH 转录因子家族研究进展[J]. 遗
传,2008,30(7):821-830.
Wang Y J, Chen K P, Yao Q. Progress of studies on bHLH
transcription factor families [J]. Hereditas, 2008, 30(7):
821-830.
[24]Agurla S, Gahir S, Munemasa S, et al. Mechanism of
stomatal closure in plants exposed to drought and cold stress
[J]. Advances in Experimental Medicine and Biology, 2018,
1081: 215-232.
(编辑:吕俊俐)

